长江流域主要常规双季早籼稻的遗传相似性分析及指纹图谱构建
2017-02-20戴冬青张华丽张萌李西明马良勇
戴冬青张华丽张萌 李西明 马良勇
(中国水稻研究所,杭州310006;#共同第一作者;∗通讯联系人,E-mail:maliangyong@caas.cn)
长江流域主要常规双季早籼稻的遗传相似性分析及指纹图谱构建
戴冬青#张华丽#张萌 李西明 马良勇∗
(中国水稻研究所,杭州310006;#共同第一作者;∗通讯联系人,E-mail:maliangyong@caas.cn)
【目的】构建常规早稻品种的指纹图谱以及分析其遗传相似性,为早稻品种鉴定、育种者有效选配亲本提供参考依据。【方法】以2000年以来长江流域双季稻区通过省级以上审定的常规早稻品种为材料,利用50对多态性高,稳定性好,且均匀分布于水稻12条染色体的SSR引物,建立常规早稻的SSR指纹图谱。利用NTSYS PC Version 2.10e软件、UPGMA法进行聚类分析。【结果】50对引物共检测到多态性位点154个,每对引物检测到的等位基因数为2~6个,平均3.08个;引物多态信息含量(PIC)值为0.03~0.75,平均值为0.36。62份材料的遗传相似系数变化范围为0.66~1,并且在相似系数为0.73处可分为浙江省内品种及省外品种2组,各组间差异不大。【结论】结合系谱关系发现,本研究中所用早稻品种的遗传背景较为狭窄。
早稻;系谱;DNA指纹图谱;聚类分析
早籼稻是我国水稻的主要生态类型之一,通常用作双季稻栽培,主要分布于华南双季稻作区和华中双、单季稻作区。包括江西、湖南、广西、广东、湖北、安徽、福建、河南、浙江、云南、四川等省的部分地区[1]。其生产具有独特的社会和经济意义,表现在育种、粮食政策、商品加工性及粮食安全等多方面[2,3]。早中熟早稻品种的推广,给晚稻产量大幅度增长创造了条件,为我国粮食生产的发展做出了巨大贡献[4]。据2014年中国农业年鉴统计资料显示, 2013年早稻种植面积为580.44万hm2,约占整个水稻种植面积的19.14%,总产量为3413.5万t,约占当年水稻总产量的16.76%,产量为5881 kg/hm2,是中国粮食安全的重要支撑[5]。
随着水稻育种事业的发展,总体育种目标趋于一致,少数骨干亲本被反复利用,导致品种间亲缘关系越来越近,遗传差异越来越小,完全依据形态性状进行品种的辨别越来越困难[6]。此外,虽然我国在品种选育方面取得了喜人成果,但在品种鉴定和品种权保护上进展却比较缓慢,品种保护意识和手段的欠缺直接影响到新品种的保护,品种侵权事件经常发生,严重影响到育种者的积极性和育种成果的合法推广应用[7]。传统的水稻新品种特异性、一致性和稳定性(DUS)测试具有耗时久、鉴定指标有限及准确性相对不高等特点[8]。20世纪90年代之前,作物品种鉴定工作中经常会用到生化指纹图谱辅助鉴定。1985年,英国科学家Jeffreys等[9]用人类基因组高变区的高度保守序列作探针进行Southern杂交,得到了类似指纹的特异性杂交图谱,并将其称作DNA指纹图谱。1988年,水稻的DNA图谱诞生[10]。新兴的基于SSR标记的DNA指纹图谱鉴定速度快、结果准确,已迅速推广到科研、育种及生产指导工作中[11-15]。SSR技术具有操作简便、效率高和重演性强等特点,现已被广泛应用于水稻遗传图谱构建、重要性状基因定位和标记辅助育种等众多领域,也是当前主要农作物品种(如玉米、水稻、大豆)真伪性鉴定中应用最广泛的分子标记[16-20]。
近年来,基于SSR标记构建了水稻[21]、小麦[22]、玉米[23]、烟草[24]、大豆[25]、棉花[26]等主要农作物的DNA指纹图谱数据库。从夕汉等[27]构建了56份杂交水稻骨干亲本的指纹图谱;马红勃等[28]构建了福建省若干水稻品种的指纹图谱;陈英华等[29]构建了东北地区区试新品种的指纹图谱;朱良勇等[30]构建了太湖稻区及国内部分香稻的SSR指纹图谱。综合前人研究结果,可以看出,在水稻中对指纹图谱的研究大多集中于杂交水稻这一方面,对于常规稻尤其是常规早稻的指纹图谱研究相对较少。
本研究以58份常规早稻品种及4份祖先品种为研究材料,从水稻的12条染色体上筛选出50对SSR引物构建了这些材料的DNA指纹图谱,同时结合系谱关系对品种间的遗传相似性进行了分析,以期为以后的研究提供针对早稻品种准确、科学的鉴定方法,保护育种者的知识产权和权益,也为育种者选配亲本提供参考依据。
1 材料与方法
1.1 试验材料
选用2000年以后长江流域双季稻区通过省级以上审定的58份常规早稻品种及4份2000年以前通过审定的祖先品种IR8、浙辐802、嘉育293和嘉育948。材料来源于中国水稻研究所早稻育种组及中国水稻研究所国家水稻中期库,品种名及其系谱关系见图1。SSR引物由北京鼎国生物工程公司合成,2×PCR Mix及电泳试剂等购自北京擎科新业生物技术有限公司。
1.2 系谱分析
根据中国水稻品种及其系谱数据库网站(http://www.ricedata.com)上的系谱信息,追溯供试材料至配组时的亲本。审定的品种的亲本为审定的品种或中间材料时,大多数追溯到上3代,外引品种一般不再追溯其系谱而视为原始亲本[31]。
1.3 DNA提取及标记检测
用TPS法[32]提取供试的所有62份水稻材料的总DNA。本研究所用的50对SSR标记(表1)来源于两部分,其中38对是从新的“水稻品种鉴定SSR标记法”[33]中的48对微卫星引物筛选而来;其余的为本实验室从水稻12条染色体上筛选到的多态性和稳定性较好、条带清晰的12对微卫星引物。PCR扩增、电泳等严格按照“新国标”技术要求进行。
1.4 指纹的建立
根据PCR扩增产物在电泳凝胶上的位置,视每条多态性带为一个等位基因,对每对引物生成的不同基因型进行编号,构建62份常规水稻品种的DNA指纹图谱。电泳结果采用1,0系统记录每个位置等位变异的有无,即某个扩增条带有带记为1,无带记为0。并且将每对引物在品种间扩增得到的二进制(0,1)数据转化为十进制数据,以所得的十进制数据代表每对引物扩增的结果,以50对引物组合的十进制字符串作为每一个品种的数字指纹。
1.5 统计分析
根据公式PIC=1-ΣPi2计算每对引物的多态信息量(Polymorphism information content, PIC)[29],其中Pi指第i个等位变异出现的频率。通过NTSYS-pc V2.10e软件,利用UPGMA(unweighted pair-group method with arithmetic mean,非加权平均法)进行聚类分析,并绘制树状聚类图。同时,利用Qualitative data计算品种间的遗传相似系数(Jaccard系数)[26]。

图1 主要早稻品种的系谱关系Fig.1.Pedigree diagram of the main early rice varieties.
2 结果与分析
2.1 系谱图的绘制
以本研究所涉及的早稻品种为基础绘制其系谱关系图(图1)。从系谱图中可以看出,早稻品种大部分可追溯到一个祖先品种IR8及其衍生的浙辐802、嘉育293和中选181等,不同的早稻品种之间在配组时经常会相互组合,很难明确分出不同的派系,亲缘关系相互融合。而且在新品种选育的过程中,以籼-籼交为主,遗传背景越来越狭窄。
2.2 SSR标记多态性分析
利用50对多态性高,稳定性好,且均匀分布于水稻12条染色体的SSR引物(表1),共检测到等位变异位点154个,平均等位变异数为3.08个,每个标记检测到的等位变异数为2~6个;共检测到基因型总数为162个,每个标记检测到的基因型个数为2~6个,标记RM208在62份水稻品种中共检测到5种不同基因型(图2)。如表1所示,标记间的多样性指数介于0.03和0.75之间,平均多样性指数为0.36,大于平均数的标记29个,引物编号为1、3、4、6、7、8、10、12、16、17、23、24、26、27、28、29、30、31、32、36、37、38、41、42、43、46、47、48、50;等位基因数和多样性指数均大于平均值的标记有14个,引物编号为3、7、8、16、27、29、36、37、38、41、42、44、47、48,表明这些引物在区分早籼稻品种时具有较强的鉴别能力。
2.3 指纹图谱的构建
将每对引物在品种间扩增得到的二进制数据(0,1)转化为十进制数据,以该十进制结果代表每对引物扩增出的结果,将每个品种用50个标记扩增出的结果串联在一起,构成每个品种特有的指纹信息。常规SSR引物在早稻品种鉴别中多样性指数偏低,且参试早稻品种遗传背景较为狭窄,很难找出某个品种能够明显区别于其他品种的含有特有指纹信息的某一个特征引物,于是利用全部50对引物的组合信息构建2000年以后长江流域主要栽培早稻品种的指纹图谱(表2)。在引物较多的情况下,对于少数品种可以采用组合少量引物的方式区分。如对于本研究中的4份2000年以前通过审定的祖先品种,可以通过引物组合的方式找到其特征引物,如IR8可以通过RM3331和RM258的组合区别于其他品种;嘉育293可以通过RM208、RM552和RM583的组合区别于其他品种;浙辐802可以通过RM224、
RM552和RM3331的组合区别于其他品种;嘉育948可以通过RM209、RM493和RM7102的组合区别于其他品种。

表1 50对SSR标记在62份水稻品种中的引物多态性信息Table 1.Polymorphism information of 50 SSR loci in 62 rice varieties.
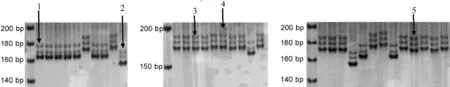
图2 标记RM208在62份材料中电泳扩增等位基因特征Fig.2.Alleles feature of marker RM208 in 62 rice varieties.

表2 利用50对引物构建的62份水稻品种的指纹信息Table 2.DNA fingerprinting of 62 rice varieties with 50 primers.

续表2
2.4 聚类分析
根据SSR分子标记所检测到的154个多态性片段,利用NTSYS-pc V2.10e软件对62份材料进行聚类分析,得到树状聚类图(图3)。利用Jaccard系数计算62个水稻品种的遗传相似系数,得到相似系数矩阵。由聚类图可看出,品种间的遗传变异较小,遗传相似系数变化范围为0.66~1.00,表明参试早稻品种之间遗传多样性较狭窄。
其中,遗传相似系数最大的是嘉育253和中冷23,二者相似系数为1,表明这50对SSR并未检测出这两个品种之间的差异。遗传相似系数最小的是湘早143和中选056,说明这两个品种之间遗传差异较大,亲缘关系较远。当相似系数为0.73时,可以大致将浙江省育成的早稻品种与其他省份育成品种分成两组。从审定年份来看,在2000年以前通过审定的4个品种IR8、浙辐802、嘉育293和嘉育948与2000年后通过审定的品种的遗传相似系数的范围分别是0.59~0.77、0.61~0.85、0.60~0.88和0.65~0.90。随着年份的推移,遗传相似系数越来越大,在一定程度上符合系谱中育种选择的进程;而2005年通过浙江省审定、2006年通过国家审定的高产早稻品种嘉育253与其衍生品种的相似系数分别为0.72(嘉育140)、0.75(早籼788)、0.79(中组7号)、0.80(早籼615)、0.87(中早39)、0.89(甬籼975)、0.88(嘉早311)、0.91(中早35)、0.94(中嘉早32)、0.94(中嘉早17)、0.96(台早518)、0.96(嘉育67)和1(中冷23),相似系数均较高,并且中早35、中早39和中嘉早17也是农业部冠名的超级稻示范推广品种,这说明嘉育253在早稻育种过程中是重要的骨干亲本。
3 讨论

图3 62个水稻品种的聚类分析Fig.3.Dendrogram of genetic similarity among 62 rice varieties.
系谱信息在育种工作中有重要的参考价值,以系谱为线索研究品种群体,对重要基因的来源有很好的追踪。目前,利用分子标记对不同作物家系有很多研究。Sun等[34]利用RAPD分子标记结合家系的系谱信息研究了45个马铃薯的遗传多样性,结果表明基于分子标记得到的遗传相似性的聚类结果,并不能完全符合家系的信息,可能是由于来自相同亲缘的家系受到严格的选择而出现分化。
从系谱关系图和聚类分析图可以看出(图1,图3),目前早稻的主栽品种亲缘关系十分相近,相互渗透,尤其是浙江省内的品种,很难明确划分为几个类群,这可能是由于不同育种单位或育种者在早稻品种的选育过程中总体目标一致,且对少数骨干亲本重复利用。并且基于遗传相似性的聚类结果并不能与系谱关系完全吻合(图1,图3),这可能是由于育种过程中的随机误差所造成的。从遗传相似系数来看,通过审定的早稻品种之间年份越接近,系谱中的亲缘关系越近,相似系数越高,一定程度上符合育种选择的进程。
目前,利用微卫星标记进行品种鉴定的研究和应用越来越多,充分体现出了该方法的优越性。本研究所用50对SSR引物在62份早稻品种中多态信息含量(PIC)普遍偏低,这可能是由于这些早稻品种本身属于籼稻亚群,且在早稻育种过程中,育种者的目标一致,选用的亲本组合血缘关系相近,导致主要栽培品种遗传基础狭窄,多样性下降[35-36],容易造成对逆境反应的遗传脆弱性[33]。并且国标“新标准”中的引物代表性不足,多态性对这些早稻品种不完全适用。总体而言,在水稻品种DNA指纹鉴别上,至今未能形成一个广适性的标准。究其原因,可能有以下两个方面:一是研究中选用的样本数少、来源地窄,导致获得的核心SSR标记的代表性不足,往往只能用于当地品种鉴别,一旦扩大到其他区域或增加品种数量时,便出现核心SSR标记并不核心的现象[16];二是我国绝大多数水稻品种间的遗传基础窄,尤其是不同省内审定的适用于特定生态区内种植的品种[19],遗传多样性更低,这导致基于不同生态区间的品种研发的多态性SSR标记在一些省内审定品种间没有多态性[33]。所以针对早稻品种的特殊分子标记如SNP等标记亟待发掘和应用。在没有适用性好的特殊引物时,我们可以采取用足够多的分子标记来构建指纹图谱的策略。
在今后的研究中应该开发更多籼稻亚属内差异的分子标记,尤其是早籼稻特有的分子标记,并将分子标记与形态特征相结合,形成信息更完善、准确的指纹图谱。
[1] 林世成,闵绍楷.中国水稻品种及系谱.上海:上海科学技术出版社,1991:10-11.Ling SC,Min S K.Rice Varieties and Their Genealogy in China.Shanghai:Shanghai Scientific and Technical Publishers, 1991:10-11.
[2] 程式华,李建.现代中国水稻.北京:金盾出版社,2007:575-577.Cheng S H,Li J.Modern Chinese Rice.Beijing:Shield Press, 2007:575-577.
[3] 方福平,王磊,廖西元.中国早稻生产波动及成因分析.中国农村经济,2006(2):11-17,26.Fang F P,Wang L,Liao X Y.The analysis about rice production fluctuation and reasion in China.Chin Rural Ecol,2006 (2):11-17,26.(in Chinese)
[4] 中国农业年鉴编辑委员会.中国农业年鉴.北京:中国农业出版社,2013:209-212. Editiorial Board of China Agriculture Yearbook.China Agriculture Yearbook:2013.Beijing:China Agriculture Press,2013.209-212
[5] 舒庆尧,夏英武.长江中下游地区优质早稻育种与生产应用.杭州:浙江大学出版社,1999:1.Shu Q Y,Xia Y W.The middle and lower reaches of the Yangtze river region of high quality rice breeding and production applications.Hangzhou:Zhejiang University Press,1999:1.(in Chinese)
[6] 张玉翠.我国棉花品种SSR指纹数据库的初步构建.北京:中国农业科学院,2012.Zhang Y C.Construction a primary SSR fingerprint database for cotton cultivars.Beijing:CAAS,2012.(in Chinese with English abstract)
[7] 李茂柏,王慧,白建江,朴钟泽.利用SSR分子标记构建水稻品种DNA指纹图谱的研究进展.中国稻米,2011(1):4-6.Li M B,Wang H,Bai J J,Piao Z Z.Using SSR molecular marker research progress of rice varieties DNA fingerprint.China Rice,2011(1):4-6.(in Chinese)
[8] 程本义,施勇烽,沈伟峰,庄杰云,杨仕华.南方稻区国家水稻区域试验品种的微卫星标记分析.中国水稻科学,2007,21 (1):7-12.Cheng B Y,Shi Y F,Shen W F,Zhuang J Y,Yang S H.Microsatellite marker-based analysis of rice varieties in national regional yield trial of Southern China.Chin J Rice Sci,2007,21 (1):7-12.(in Chinese with English abstract)
[9] Jeffreys A J,Wilson V,Kelly R,Taylor B A,Bulfield G.Mouse DNA‘fingerprints’:Analysis of chromosome localization and germ-line stability of hypervariable loci in recombinant inbred strains.Nucleic Acids Res,1987,15(7):2823-26.(in Chinese with English abstract)
[10]Dallas J F.Detection of DNA"fingerprints"of cultivated rice by hybridization with a human minisatellite DNA probe.Proc Natl Acad Sci,1988,85(18):6831-6835.
[11]朱作峰,孙传清,李白超,王象坤.用SSR标记对水稻品种的分类研究.农业生物技术学报,2001,9(1):58-61.Zhu Z F,Sun C Q,Li B C,Wang X K.Study on the classification of rice varieties with SSR molecular markers.J Agric Biotech,2001,9(1):58-61.(in Chinese with English abstract)
[12]Akagi H,Yokozeki Y,Inagaki A,Fujimura T.Highly polymorphic mi-crosatellites of rice consist of AT repeats,and a classification of closely related cultivars with these microsatellite loci.Theor Appl Genet,1997,94:61-67.
[13]汪爱顺,李进波,刘汉珍.DNA指纹用于杂交水稻种子纯度和真伪鉴定的研究进展.分子植物育种,2005,3(3):393-400.Wang A S,Li J B,Liu H Z.Research advance on application of DNA fingerprinting to identification seed purity and authenticity in hybrid rice.Mol Plant Breeding,2005,3(3):393-400.(in Chinese with English abstract)
[14]彭锁堂,庄杰云,颜启传,郑康乐.我国主要杂交水稻组合及其亲本SSR标记和纯度鉴定.中国水稻科学,2003,14(1):2-6.Peng S T,Zhuang J Y,Yan Q C,Zheng K L.SSR markers selection and purity detection of major hybrid rice combinations and their parents in China.Chin J Rice Sci,2003,14 (1):2-6.(in Chinese with English abstract)
[15]李晶,何平,李仕贵,鲁润龙,朱立煌.利用微卫星标记鉴定杂交水稻冈优22种子纯度的研究.生物工程学报,2000,16 (2):211-214.Li J,He P,Li S G,Lu R L,Zhu L H.Identification seed purity of hybrid rice with 22 microsatellite markers.Chin J Biol, 2000,16(2):211-214.(in Chinese)
[16]李汝玉,李群,张文兰,张晗,宋国安,王东建.利用SSR标记进行小麦品种鉴定和新品种保护研究.山东农业科学,2007 (6):14-17.Li R Y,Li Q,Zhang W L,Zhang H,Song G A,Wang D J.Application of SSR markers to identification and protection of wheat variety.Shandong Agric Sci,2007(6):14-17.(in Chinese with English abstract)
[17]杨旭,谭学林.滇型杂交粳稻主要亲本的SSR指纹图谱及其遗传差异分析.杂交水稻,2009,24(6):54-58.Yang X,Tan X L.SSR fingerprint and genetic diversity of major parents of Dian-type japonica hybrid rice.Hybrid Rice, 2009,24(6):54-58.(in Chinese with English abstract)
[18]戴剑,洪德林.SSR分子标记技术在杂交水稻品种鉴定中的应用.金陵科技学院学报,2012,28(1):45-51.Dai J,Hong D L.The application of SSR molecular marker in identification of hybrid rice varieties.Jinling Inst Techol, 2012,28(1):45-51.(in Chinese with English abstract)
[19]周振玲,王宝祥,樊继伟,卢百关,赵志刚,江玲,秦德荣,万建民,徐大勇.江淮稻区不同生态型粳稻品种的籼粳分化度和遗传多样性.中国水稻科学,2012,26(4):431-437.Zhou Z L,Wang B X,Fan J W,Lu B G,Zhao Z G,Jiang L, Qin D R,Wan J M,Xu D Y.Indica-japonica differentiation degree and genetic diversity of japonica cultivars belonging to different ecotypes from the Yangtze-Huaihe region.Chin J Rice Sci,2012,26(4):431-437.(in Chinese with English abstract)
[20]王凤格,赵久然,戴景瑞,易红梅,匡猛,孙艳美,于新艳,郭景伦,王璐.玉米通用SSR核心引物筛选及高通量多重PCR复合扩增体系建立.科学通报,2006,51(23):2738-2746.Wang F G,Zhao J R,Dai J R,Yi H M,Kuang M,Sun M Y, Yu X Y,Guo J L,Wang L.Screening common core SSR primers and Building high flux multiple PCR compound amplification system.Sci Bull,2006,51(23):2738-2746.(in Chinese)
[21]程保山,万志兵,洪德林.35个粳稻品种SSR指纹图谱的构建及遗传相似性分析.南京农业大学学报,2007,30(3):1-8.Cheng BS,Wan Z B,Hong D L.Establishment of SSR fingerprint map and analysis of genetic similarity among 35 varieties in japonica rice(Oryza sativa L.).J Nanjing Agric Univ, 2007,30(3):1-8.(in Chinese with English abstract)
[22]李根英,Dreisigacker S,Warburton M L,夏先春,何中虎,孙其信.小麦指纹图谱数据库的建立及SSR分子标记试剂盒的研发.作物学报,2006,32(12):1771-1778.Li G Y,Dreisigacker S,Warburton M L,Xia X C,He Z H, Sun Q X.Development of a fingerprinting database and assembling an SSR reference kit for genetic diversity analysis of wheat.Acta Agron Sin,2006,32(12):1771-1778.(in Chinese with English abstract)
[23]赵久然,王凤格,郭景伦,陈刚,廖琴,孙世贤,陈如明,刘龙洲.中国玉米新品种DNA指纹库建立系列研究:Ⅱ.适于玉米自交系和杂交种指纹图谱绘制的SSR核心引物的确定.玉米科学,2003(2):3-5,8.Zhao J R,Wang F G,Guo J L,Chen G,Liao Q,Sun S X, Chen R M,Liu L Z.Series of research on establishing DNA fingerprint pool of Chinese new maize cultivars:Ⅱ.Confirmation of a set of SSR core primer pairs.J Maize Sci,2003(2): 3-5,8.(in Chinese with English abstract)
[24]徐军,刘艳华,任民,牟建民,张兴伟,陈雅琼,王志德.普通烟草种质资源的SSR标记与指纹图谱分析.中国烟草科学, 2011(2):62-65.Xu J,Liu Y H,Ren M,Mou J M,Zhang X W,Chen Y Q, Wang Z D.SSR fingerprint map analysis of tobacco germplasms.Chin Tob Sci,2011(2):62-65.(in Chinese with English abstract)
[25]陈亮,郑宇宏,范旭红,孟凡凡,孙星邈,张云峰,王明亮,王曙明.大豆SSR指纹图谱身份证的研究进展与展望.大豆科技,2015(2):38-43.Chen L,Zheng H Y,Fan X H,Meng F F,Sun X M,Zhang Y F,Wang M L,Wang S M.Progress and perspective on SSR fingerprint identification establishment in soybean.Soy Sci Techol,2015(2):38-43.(in Chinese with English abstract)
[26]聂新辉,尤春源,李晓方,秦江鸿,黄聪,郭欢乐,王夏青,赵文霞,林忠旭.新陆早棉花品种DNA指纹图谱的构建及遗传多样性分析.作物学报,2014,12:2104-2117.Nie X H,You C Y,Li X F,Qin J H,Huang C,Guo H L, Wang X Q,Zhao W X,Lin Z X.Construction of DNA fingerprinting and analysis of genetic diversity for Xinluzao cotton varieties.Acta Agron Sin,2014,12:2104-2117.(in Chinese with English abstract)
[27]从夕汉,李莉,滕斌,刘海珍,倪大虎,陆徐忠,宋丰顺,杨剑波.56个杂交水稻骨干亲本SSR指纹图谱的构建及遗传相似性分析.生物学杂志,2010(1):87-91.Cong X H,Li L,Teng B,Liu H Z,Ni D H,Lu X Z,Song F S,Yang J B.Establishment of SSR fingerprint map and analysis of genetic similarity among 56 backbone parental lines in hybrid rice.J Biol,2010(1):87-91.(in Chinese with English abstract)
[28]马红勃,许旭明,韦新宇,杨旺兴,邹文广.基于SSR标记的福建省若干水稻品种DNA指纹图谱构建及遗传多样性分析.福建农业学报,2010,25(1):33-38.Ma H B,Xu X M,Wei X Y,Yang X W,Zou W G.DNA fingerprints and genetic diversity analysis based on SSR markers for rice cultivars in Fujian.Fujian J Agric Sci,2010,25(1): 33-38.(in Chinese with English abstract)
[29]陈英华,侯昱铭,李宏宇,李茂柏,袁媛,徐正进等.东北地区水稻区试新品种的DNA指纹图谱构建及遗传多样性分析.种子,2009,28(3):28-35.Cheng Y H,Hou Y M,Li H Y,Li M B,Yuan Y,Xu Z J.Establishment of DNA fingerprinting and analysis of genetic diversity among japonica rice cultivars attended regional trials in northeast region of China.Seed,2009,28(3):28-35.(in Chinese with English abstract)
[30]朱勇良,谢裕林,黄凌哲,吴建祥,乔中英,王建平,黄萌,陈培峰.太湖稻区及国内部分香稻SSR指纹图谱构建及遗传多样性初析.植物遗传资源学报,2012,13(4):666-671.Zhu L Y,Xie Y L,Huang L Z,Wu J X,Qiao Z Y,Wang J P,Huang M,Chen P F.Establishment of SSR fingerprint map and preliminary analysis of genetic diversity among aromatic rice from Taihu and other areas in China.J Plant Genet Resour,2012,13(4):666-671.(in Chinese with English abstract)
[31]刘化龙,王敬国,赵宏伟,邹德堂,陈温福,徐正进.黑龙江水稻育种骨干亲本及系谱分析.东北农业大学学报,2011,42 (4):18-21.Liu H L,Wang J G,Zhao H W,Zou D T,Chen W F,Xu Z J.Analysis of founder parents for cultivar varieties in Heilongjiang Province.J Northeast Agric Univ,2011,42(4): 18-21.(in Chinese with English abstract)
[32]张向前,邹金松,朱海涛,李晓燕,曾瑞珍.水稻早熟多子房突变体fon5的遗传分析和基因定位.遗传,2008,10:1349-1355.Zhang X Q,Zhou JS,Zhu H T,Li X Y,Zeng R Z.Genetic analysis and gene mapping of an early flowering and multi-ovary mutant in rice(Oryza sativa L.).Hereditas,2008,10: 1349-1355.(in Chinese with English abstract)
[33]左示敏,周娜娜,陈宗祥,张亚芳,汤义华,毛从亚,许学宏,许明,许如根,潘学彪.DNA指纹在水稻品种鉴定中的应用与展望.江苏农业科学,2014(12):4-8.Zuo S M,Zhou N N,Chen Z X,Zhang Y F,Tang Y H,Mao C Y,Xu X H,Xu M,Xu R G,Pan X B.Application and prospect of DNA fingerprint in rice cultivars.Jiangsu Agric Sci,2014(12):4-8.(in Chinese with English abstract)
[34]Sun G,Wang P G M,Jong H.RAPD and pedigree-based genetic diversity estimates in cultivated diploid potato hybrids.Theor App Genet,2003,107(1):110-115.
[35]FAO.The state of the world′s plant genetic resources for food and agriculture.Rome:FAO,1998:30-40.
[36]Vellve R.The decline of diversity in European agriculture.E-cologist,1993,23:64-69.
[37]Manifesto MM,Schlatter A R,Hopp H E,Suárez E Y,Dubcovsky J.Quantita-tive evaluation of genetic diversity in wheat germplasm using molecular markers.Crop Sci,2001,41: 682-690.
Analysis of Genetic Similarity and Construction of DNA Fingerprinting for Conventional Double Cropping Early Rice Varieties in the Yangtze River Basin
DAI Dongqing#,ZHANG Huali#,ZHANG Meng,LI Ximing,MA Liangyong∗
(China National Rice Research Institute,Hangzhou 310006,China;#These authors contributed equally to this work;∗Corresponding author,E-mail:maliangyong@caas.cn)
【Objective】To provide a reference for rice variety identification and parents selection,we constructed fingerprints of conventional early rice varieties and analyzed its genetic similarity.【Method】Conventional early rice varieties registered at or above the provincial level since 2000 in the Yangtze River Basin were analyzed using 50 pairs of SSR primers with high polymorphism,good stability,and even distribution on 12 chromosomes of rice to construct SSR fingerprints of conventional early rice.Cluster analysis was conducted with NTSYS PC Version 2.10e software and UPGMA cluster analysis method.【Results】A total of 154 polymorphic bands were detected among 62 rice lines.Two to six alleles were detected for each marker with an average of 3.08.Primers polymorphism information content(PIC) value ranged from 0.03 to 0.75,with an average of 0.36.Cluster analysis with NTSYS PC Version 2.10e software showed that the genetic similarities ranged from 0.66 to 1.00.The 62 varieties could be divided into two groups as varieties in Zhejiang Province and varieties outside the province at the similarity coefficient of 0.73 with small difference between groups.【Conclusion】Combination with the pedigree relationship,it was revealed that the genetic background of early rice varieties in this study was relatively narrow.
early rice;pedigree;DNA fingerprinting;clustering analysis
S511.032;S511.2+1
A
1001-7216(2017)01-0040-10
2016-08-01;修改稿收到日期:2016-09-24。
国家863计划资助项目(2014 AA10A604-15);国家自然科学基金资助项目(31501288);浙江省自然科学基金资助项目(LQ16C130002);浙江省高产优质多抗水稻新品种选育(9410计划)资助项目(2012C12901-2);中国水稻研究所中央级公益性科研院所基本科研业务费专项(2014RG003-3)。
