基于荧光检测技术的多花黑麦草EST-SSR指纹图谱的构建
2017-02-16刘欢张新全马啸张瑞珍何光武潘玲金梦雅
刘欢,张新全,马啸,张瑞珍,何光武,潘玲,金梦雅
(1四川农业大学草业科学系,成都 611130;2四川省草原工作总站,成都 610041)
基于荧光检测技术的多花黑麦草EST-SSR指纹图谱的构建
刘欢1,张新全1,马啸1,张瑞珍2,何光武2,潘玲1,金梦雅1
(1四川农业大学草业科学系,成都 611130;2四川省草原工作总站,成都 610041)
【目的】构建基于EST-SSR荧光标记的多花黑麦草(Lolium multiflorum Lam.)DNA指纹鉴定体系,为多花黑麦草品种鉴定提供高通量技术手段,为不同品种的合理应用提供参考依据,有效保护农民利益和育种权益。【方法】利用表型性状差异大的3个品种(特高Tetragold、长江2号ChangjiangNo.2和阿伯德Aubade),通过聚丙烯酰胺凝胶电泳从200对EST-SSR引物中筛选出扩增条带清晰、多态性好、扩增稳定的30对引物。在筛选出的每对引物5′端添加荧光标记FAM后,采用毛细管法通过DNA分析仪检测200个单株不同等位变异的扩增片段,从30对EST-SSR引物中筛选出25对扩增稳定的荧光引物,建立基于高通量荧光SSR标记的多花黑麦草品种鉴定体系。【结果】通过25对EST-SSR引物构建的DNA指纹图谱来进行10个多花黑麦草材料的品种鉴定。25对EST-SSR引物共检测到127个等位基因,等位变异扩增片段长度范围为51—249 bp,每对引物可检测到有效等位基因数为2—11个,特异等位基因最多可检测到11个(N101),平均每对引物4.00个;多态性位点的比率范围为33.33%—100.00%。平均PIC值为0.702,Shannon指数最大为3.322(N101),平均为1.929,基因多样性指数变幅为0.159—0.500,平均0.318,可鉴别的材料数为0—10个;其中14对特征引物在10个品种(系)上可检测出25个特异等位基因。综合来看,引物N101在200对引物中鉴别效率最高,可直接将10个多花黑麦草品种(系)区分开,在长江2号、赣选1号和川农2号上同时检测出特异等位基因。但由于多花黑麦草在品种间与品种内变异均较高,因此为鉴定更多材料,从25对引物中选择了6对扩增和检测效果较好的引物(N54、N101、N146、N151、N154、N156),6对引物可检测到的稳定等位基因数均不小于19个,在达伯瑞和邦德上最多可检测到22个等位基因。通过6对高效引物构建了10个多花黑麦草品种(系)的DNA指纹图谱,包括标准模式图、图谱代码和图谱QR编码。首次利用EST-SSR荧光标记毛细管电泳检测,为10个多花黑麦草材料分别构建了唯一的指纹代码和QR编码。【结论】利用6对高效引物构建了多花黑麦草SSR高通量鉴定体系,其中荧光引物N101多态性最高,可直接鉴别10个多花黑麦草品种(系)。
多花黑麦草;EST-SSR分子标记;指纹图谱;荧光检测;毛细管电泳
0 引言
【研究意义】多花黑麦草(Lolium multiflorum Lam.)作为中国南方种植面积最大的一年生牧草,受到广大农牧民的关注[1]。在长期引种、育种和选种的过程中,栽培范围不断扩大,品种不断增加,市场销售的商品种子出现了同名异物、同物异名、多种混卖、种子不纯等现象,阻碍了优良品种推广及应用,严重危害了育种者和生产者的利益,因此,能有效地鉴定区别不同品种是合理利用多花黑麦草的前提条件之一。【前人研究进展】目前,多花黑麦草品种常见的鉴定方法包括DUS鉴定法(distinctness,uniformity,stability)和DNA指纹图谱鉴定法,但由于DUS鉴定成本高,且易受环境因素和人为测量因素影响[2-3],因此,在不断完善多花黑麦草 DUS测试指南的同时,DNA指纹图谱在多花黑麦草的品种鉴定中的应用也不可忽视[4]。国际植物新品种保护联盟(International Union for the Protection of New Varieties of Plants,UPOV)已批准在已有DNA指纹图谱的基础上[5],DNA分子标记技术可以应用于近似品种的辅助筛选[6]。HIRATA等[7]研究证明SSR分子标记可以有效鉴定多花黑麦草和其相似的品种;PASAKINAKIENE等[8]利用SSR标记对多花黑麦草、多年生黑麦草、草地羊茅和高羊茅进行了鉴定;赵欣欣等[9]通过SSR分子标记鉴定了多花黑麦草杂种后代真实性和表型差异;罗永聪等[10]通过12对SSR引物构建的指纹图谱区分了21个多花黑麦草品种(系);黄婷等[11]通过 20对 SSR引物对 6个多花黑麦草品种进行了鉴定分析。PRAKASH等[12]虽然对琼脂糖凝胶电泳技术和聚丙烯酰胺凝胶电泳技术进行了优化,但仍难以满足试验中高效、准确、大样品数的需求,因此,在这样的现状中,毛细管电泳荧光检测技术逐渐受到关注。SSR荧光标记毛细管电泳法是以DNA测序技术为基础发展起来的检测方法,具有高效、自动化等优点。程本义等[13]和陈雅琼等[14]研究表明荧光标记技术检测效率显著高于聚丙烯凝胶电泳,结果更为精确灵敏,更适用于材料的高通量检测分析。易红梅等[15]通过对7个SSR位点的检测分析,通过毛细管电泳荧光标记检测
法和变性PAGE银染法分别构建了192份玉米品种的指纹图谱,结果得出荧光标记技术检测法效率更高;许鲲等[16]也通过40对SSR荧光引物构建了163份国家冬油菜的指纹图谱。SANCHEZ-PEREZ等[17]探讨了琼脂糖凝胶电泳、聚丙烯酰胺凝胶电泳和毛细管电泳3种技术在杏仁研究上的优劣,结果证明聚丙烯酰胺凝胶电泳与毛细管电泳比琼脂糖凝胶电泳效果更佳,但DNA自动测序仪成本较高,而PAGE更耗费时间。因此,目前的检测方法都各有优缺点,单一的检测技术在品种鉴定和指纹图谱构建中已经难以满足所有试验需求,因此,在不同的试验阶段按实际需求选择合适的检测技术,可显著提高鉴定效率。【本研究切入点】EST-SSR分子标记是基于植物表达序列标签(expressed sequence tags,ESTs)进行开发的SSR标记,物种间通用性高、侧翼序列保守性高,可直接反映出转录区的差异[18],被广泛应用在结缕草[19]、牛鞭草[20]和高丹草[21]等禾本科植物DNA指纹鉴定中。目前,已通过RAPD、SSR和ISSR等分子标记构建了黑麦草的DNA指纹图谱,但是EST-SSR分子标记在黑麦草指纹图谱构建上的应用未见报道,仍未见SSR荧光标记毛细管电泳检测法在多花黑麦草上的应用。【拟解决的关键问题】本研究结合聚丙烯酰胺凝胶电泳和毛细管电泳荧光检测技术,构建10个多花黑麦草品种(系)的指纹图谱,为多花黑麦草品种育成、品种鉴定提供高通量技术手段,完善多花黑麦草现有的DNA指纹数据库。
1 材料与方法
1.1 试验材料
选择10个四倍体多花黑麦草材料,包括8个国审品种,2个新品系,均由四川农业大学草业科学系2014—2015年收集(表1)。于2015年9月在光照培养箱中水培发芽育苗,待植株长至4—5叶龄取样。

表1 供试10个多花黑麦草品种(系)信息Table 1 Informations of 10 L.multiflorum varieties (strains) used in this study
1.2 DNA的提取
参考行业标准《植物品种鉴定DNA指纹方法 总则》(NY/T 2594-2014)[22],由于多花黑麦草是异花授粉植物,因此,采用单株鉴定的方式,每份材料选取20个单株,共200个单株的幼嫩植株,通过试剂盒(北京天根生化公司)提取基因组DNA,然后用超微量分光光度仪(NanoVue Plus)检测DNA纯度和浓度,选取合格的DNA样品于-20℃保存备用。
1.3 EST-SSR引物筛选
引物筛选采用3个田间表型性状差异较大的多花黑麦草品种(特高Tetragold、长江2号ChangjiangNo.2和阿伯德Aubade),每个品种选择4个单株,共12个单株提取DNA。所用引物序列由多花黑麦草转录组自行测序开发而来,引物由南京金斯瑞生物科技有限公司合成。PCR所用94 kD分子量Taq酶、PCR-Mix混合液(含有10×PCR buffer、Mg2+、dNTPs)和50 bp DNA ladder Marker均购自北京天根生化公司。
引物筛选采用聚丙烯酰胺凝胶电泳进行,EST-SSR优化反应体系为15 µL,在前人的基础上稍作修改[11],包括DNA 1.5 μL(10 ng·μL-1)、正反向引物各0.3 μL(10 pmol·μL-1)、Taq酶0.3 μL(2.5 U·μL-1)、PCR-Mix 7.5 μL(10 ng·μL-1)和ddH2O 5.1 μL。PCR反应程序为94℃ 5 min;94℃ 30 s,退火45 s,72℃ 1 min,35个循环;72℃ 10 min,4℃保存。扩增产物在 8%非变性聚丙烯酰胺凝胶(丙烯酰胺Acr﹕N'-N'甲叉双丙烯酰胺Bis =29﹕1,1×TBE缓冲液)上进行检测。每样品点样5.5 μL,50 bp DNA Ladder为分子量标准,200 V电压预电泳30 min,而后400 V电压电泳 2 h,在 0.1%的 AgNO3溶液中银染,在NaOH溶液中显色[23],凝胶用Bio-6000凝胶扫描仪扫描保存以供分析。最终从 200对自主开发的EST-SSR引物(分别编号为 N1—N200)中共筛选出30对高效引物,并根据反复试验的结果调整最终的退火温度与循环数(表2)。

表2 30对EST-SSR荧光引物信息Table 2 Informations of 30 EST-SSR fluorescence primer pairs
1.4 荧光标记毛细管电泳
将从200对引物中筛选出的30对EST-SSR引物(表 2),在每对引物 5′端添加荧光标记 FAM(6-carboxy-fluorescein)。试验中所用荧光引物由上海英骏生物技术有限公司合成。引物用灭菌ddH2O溶解并稀释到10 µmol·μL-1备用。
毛细管电泳的PCR扩增采用10 µL的反应体系,其中包括 5 ng DNA、3U Taq DNA聚合酶、2 mmol·L-1Mg2+、5 mmol·L-1dNTPs、2 μmol·L-1正向引物和反向引物、2 μmol·L-1FAX荧光标记和7 μL ddH2O。PCR反应体系为94℃ 5 min;94℃ 30 s,不同退火温度退火30 s,72℃ 1 min,35或40个循环;72℃ 10 min,4℃保存。将PCR产物稀释,在96孔板的各孔中分别加入9 μL去离子甲酰胺、0.05 μL GS3730-500分子量内标和1 μL稀释后的PCR产物,95℃变性5 min,于4℃冷却10 min,再置于ABI 3730XL DNA分析仪(Applied Biosystems,Foster City,USA)上进行自动荧光检测。毛细管电泳程序为预电泳15 kV,2 min;2 kV电压进样10 s;电泳 15 kV,20 min。最后设置分子内标(Size Standard)和参数(Parameters)用Data Collection和Gene Marker软件进行数据收集与图像分析。
1.5 数据统计与分析
原始数据采用Data Collection软件收集,并根据引物筛选时确定的分子量范围及引物颜色进行分子量的确定,用Gene Marker 2.2.0软件进行分析,最终将电泳结果转化成PDF图片形式导出。将毛细管电泳检测所得的SSR扩增片段信息输入EXCEL表格,以多花黑麦草品种编号和基因型编号为横列,以扩增核苷酸片段大小为纵列,有稳定扩增核苷酸片段记为“1”,没有或扩增不稳定的记为“0”,生成由“1”和“0”构成的原始数据矩阵。然后根据表征矩阵,数据通过DCFA 1.1[24]生成,利用POPGENE 1.31软件[25]计算I值(Shannon’s information index)和H值(Nei’s gene diversity)。多态性信息含量 PIC值(Polymorphism information content)参考 Nei[26]的方法进行计算,PIC=1-ΣPi2(Pi表示第i个等位位点出现的频率,反映每个SSR位点的多态性水平)。
指纹图谱代码采用陈昌文等[27]方法,并根据指纹图谱代码构建10个多花黑麦草材料的DNA指纹图谱;最终将每个材料的信息分别整合到QR编码中,编码中扩增信息标注方式参考宋海斌等[28]标注方法,依据扩增位点分子量从小到大的顺序记录各检测位点的分子量(单位为bp,略去单位bp,只记录数字),如果一份材料经同一引物扩增同时检测出多个位点,则用“-”连接数字,即可简单表示每个材料的扩增信息。
2 结果
2.1 EST-SSR标记多态性分析
利用初步筛选出的30对EST-SSR引物对10个多花黑麦草材料进行了荧光标记毛细管电泳检测。发现部分由聚丙烯酰胺凝胶电泳筛选出的引物,添加荧光标记后在毛细管电泳中难以检测到稳定片段或检测效果不佳,不适用于多花黑麦草高通量指纹鉴定中,因此,根据DNA分析仪检测到的片段,排除检测异常、具有较多异常峰型的、较多位点不易判别的引物,筛选出能检测到稳定变异等位基因的25对EST-SSR引物,每条扩增片段的长度范围为51—249 bp。采取每个材料20个单株进行毛细管电泳[6],删除部分不稳定扩增片段后,获得稳定性较高的扩增片段信息(表3)。
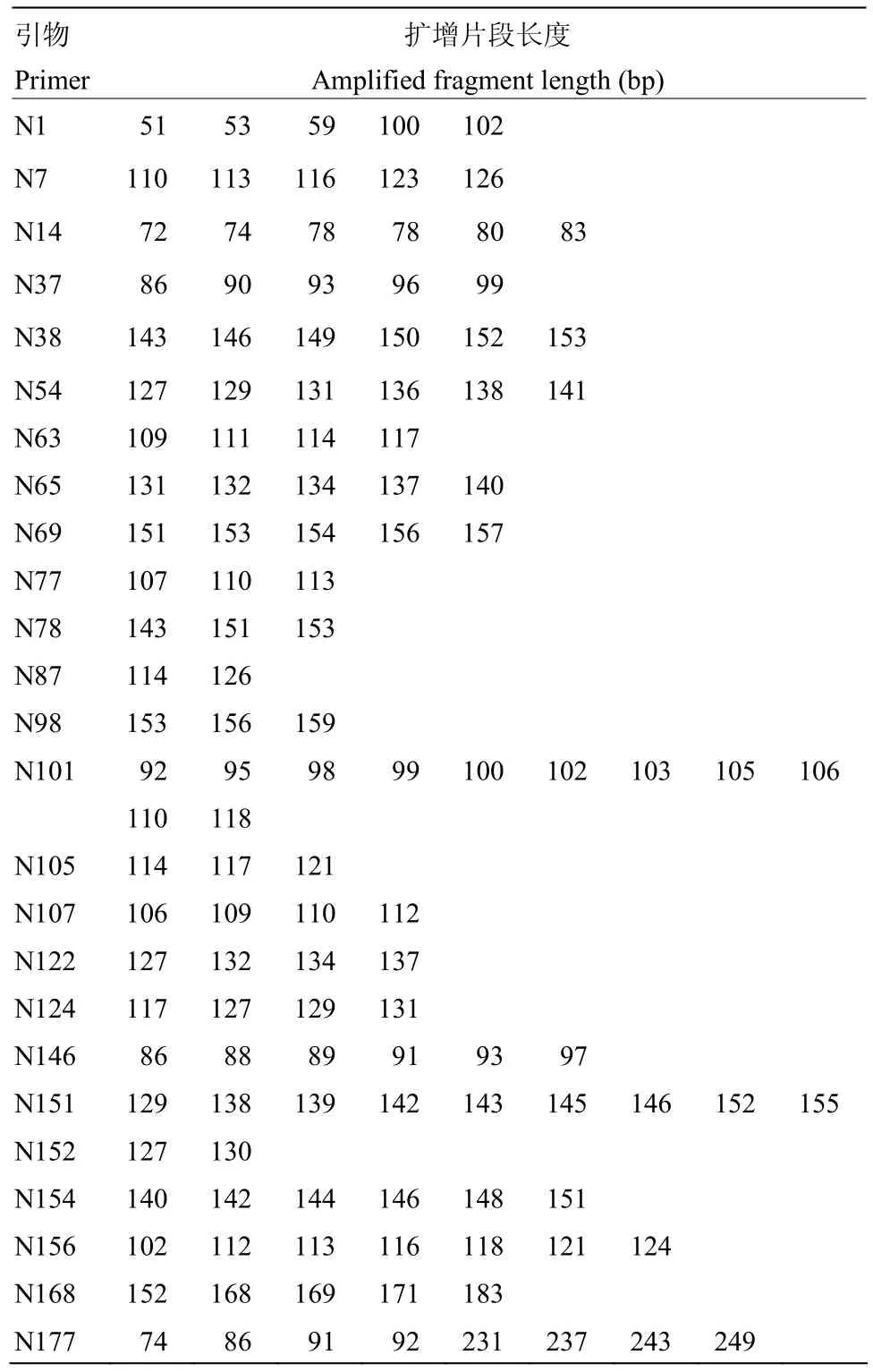
表3 25对EST-SSR引物扩增片段信息Table 3 Amplified fragment informations of 25 EST-SSR primer pairs
25对EST-SSR引物对10个多花黑麦草品种(系)进行扩增,共检测出127个等位基因。不同引物扩增的等位基因数明显不同,可检测到有效等位基因数为 2(N87)—11(N101)个,平均每个引物5.08个等位基因;特异等位基因数范围为0(N152)—11(N101)个,平均每对引物 4.00个,多态性位点比率为 33.33%—100.00%,平均73.02%(表4)。PIC变动范围为0.484(N87)—0.877(N101),平均 0.702,表明多花黑麦草材料间变异较大,具有丰富的遗传多样性。Shannon指数最大为3.322(N101),均值为1.929;基因多样性指数变动范围为0.159(N101)—0.500(N152),平均0.318;可鉴别的材料数为0—10个。综上所述,可得出引物N101是所有引物中鉴定效率最高的引物,该引物可完全区分本试验所有的多花黑麦草材料。测得的序列若按二倍性估算,最多可能鉴别121种材料,在以后多花黑麦草DNA指纹数据库的构建中应重点应用。

表4 EST-SSR引物扩增结果及多态性信息Table 4 Results and the polymorphism informations of EST-SSR primers
2.2 EST-SSR标记引物高效性分析
对25对EST-SSR荧光引物在10个多花黑麦草材料的毛细管电泳结果进行统计分析,结果表明,10个多花黑麦草品种(系)均可检测到特异等位基因(表5),即均可利用一个引物就可鉴别相应的品种。其中,特高、达伯瑞和川农2号只具有1个特征引物;长江2号和邦德具有2个特征引物;赣选1号、阿伯德、杰威、川农1号和LG1具有3个特征引物。另外,一个引物可以同时鉴定多个品种,即它能在多个品种上检测到特异等位基因。如图1,可观察到N101引物在长江2号(118 bp)、赣选1号(105 bp)和川农2号(92 bp)上同时检测到特异等位基因;N168引物在特高(152 bp)、杰威(169 bp)和LG1(168 bp)上同时检测到特异等位基因;N151引物在长江2号(143 bp)和邦德(152 bp)上同时检测到特异等位基因;N156引物在赣选1号(102 bp)和达伯瑞(112 bp)上同时检测到特异等位基因;N38引物在阿伯德(143 bp)和杰威(152 bp)上,N54在杰威(136 bp)和川农1号(138 bp)上,N146引物在赣选1号(93 bp)和LG1(89 bp)上同时检测到特异等位基因;其余引物均只在一个材料上出现特异等位基因。结果表明,25对EST-SSR引物在毛细管电泳下能检测到丰富的等位基因,可有效用于多花黑麦草指纹图谱的构建。

图1 荧光引物N101在长江2号、赣选1号和川农2号(从上至下)单株上的特异峰图Fig. 1 Specific peak figures of Changjiang No.2, Ganxuan No.1 and Chuannong No.2 (from up to down) at fluorescent primer N101
2.3 多花黑麦草品种(系)DNA指纹图谱构建
2.3.1 指纹图谱标准模式图 由于多花黑麦草是异花授粉植物,在品种间和品种内变异均较大,为避免单个引物构建的指纹图谱在更多材料鉴定时的误差,因此,采用引物组合法进行了指纹图谱构建[29]。根据毛细管检测峰型、PIC值、引物多态性、引物鉴定效率(表4和表5),从25对EST-SSR引物中选择了6对扩增和检测效果较好的引物(N54、N101、N146、N151、N154、N156)[30],6对引物可检测到的稳定等位基因数均不小于19个(表6),在达伯瑞和邦德上可检测到的等位基因数最多,均为22个,其中,基因数较少的是杰威和川农2号,但材料间可检测到的等位基因数差异不显著。

表5 10个多花黑麦草材料的14对特征引物Table 5 14 specific primer pairs for 10 L.multiflorum materials
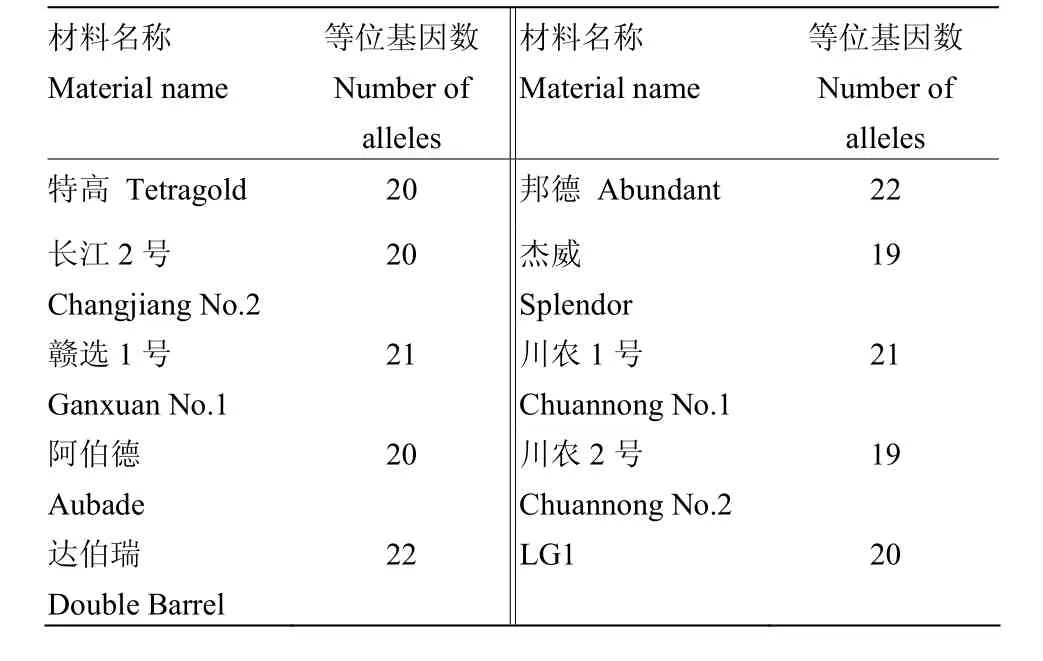
表6 6对引物在不同材料中可检测到的等位基因数Table 6 The number of alleles in different materials by 6 primer pairs
利用筛选出的6对EST-SSR荧光引物分别构建了10个多花黑麦草材料检测位点的标准模式图(图2)。以品种长江2号为例,横坐标表示不同的EST-SSR多态性引物,纵坐标表示引物扩增峰图的分子量。长江2号标准模式图表示的是,长江2号分别在引物N54的127、129和131 bp,引物N101的95、100、103、110和118 bp,N146的86和91 bp,引物N151的138、139、142、143、145和146 bp,引物N154的144、146和148 bp,N156的116 bp处检测到核苷酸片段,可鉴定区别于其他品种,这种标准模式图能将毛细管电泳峰图转变成更直观的多态性谱带图,为品种鉴定提供了更简单易操作的方式。
2.3.2 指纹图谱代码 参考陈昌文等[27]方法,对筛选出来的6个引物扩检测出的位点进行赋值(表7),采用1—9字符进行赋值,对于等位基因数超过9的引物,采用只考虑其中9个基因位点的方法来进行,优先删除鉴定性差的等位基因位点。根据这些原则,对25对引物扩增的等位基因筛选后,选择了其中42个等位基因位点进行赋值。根据引物扩增峰值的选择和赋值标准,编码时保证每对引物下的编码只有一位数字,并且按照6个引物下的扩增片段大小,先选择位点较小的原则进行赋值,编码了多花黑麦草指纹图谱代码(表8)。编码后,每个材料的DNA指纹图谱代码具有6位数字,代表6个引物上的6个电泳峰。如特高,图谱代码为 331233,表示特高在引物 N54的131 bp(3)、N101的98 bp(3)、N146的86 bp(1)、N151的138 bp(2)、N154的144 bp(3)和N156的113 bp(3)处均能检测出峰。

表7 等位基因电泳峰的选择和赋值标准Table 7 The selection and encoded standard of allelic peak

图2 10个多花黑麦草材料的标准模式图Fig. 2 The standard model pattern of 10 L.multiflorum materials
编码结果表明,6位指纹图谱代码能够完全区分10个多花黑麦草材料,每份材料均具有特异的指纹图谱代码。根据指纹图谱代码,简化多花黑麦草材料的指纹图谱,图3中谱带与表8中的指纹图谱代码完全一致,由图3可知,10个多花黑麦草材料明显具有不同位点检测到等位基因,可明显区别不同材料。
2.3.3 指纹图谱QR编码 利用QrPlant Generator植物二维码生成工具对所有供试材料进行编码,为每份材料构建了一份指纹图谱QR编码(图4),其中包含了材料名称、材料类型、材料的植物学分类、指纹图谱代码、引物扩增信息[28]。如扫描特高的QR编码,会获得如下信息:

图3 10个多花黑麦草材料指纹图谱Fig. 3 Fingerprintings of 10 L.multiflorum materials
材料名称Cultivars Name:特高Tetragold;
类型Type:引进品种The introduced varieties;
植物学分类 Botanic classification:L. multiflorum.cv. Tetragold;
EST-SSR指纹图谱代码EST-SSR fingerprinting codes:331233;
扩增信息Amplification information:
N54-131-141N101-98-102N146-86-88-91-97N151-138-145-14 6N154-144-146-148-151N156-113-116-118-121-12。

图4 多花黑麦草指纹图谱QR编码Fig. 4 Fingerprinting QR code of L.multiflorum

表8 10个多花黑麦草材料的DNA指纹图谱代码Table 8 DNA fingerprinting codes of 10 L.multiflorum materials
3 讨论
3.1 EST-SSR标记效果分析
EST-SSR分子标记不仅具有SSR标记共显性、重复性高、多态性高等优点[31-32],而且通用性也较SSR分子标记高,因此,逐渐成为植物种质遗传多样性分析、系谱分析、品种鉴定及DNA指纹图谱构建中广泛应用的分子标记[33-34]。本试验根据荧光检测毛细管电泳检测结果,选择了25对EST-SSR引物进行多花黑麦草材料的鉴定。25对引物共检测出127个等位基因,特异等位基因数在0—11个不等,平均每对引物4.00个,PIC变幅为0.484—0.877,Nei氏基因多样性指数变幅为 0.159—0.500。同时,引物 N101在 200对引物中鉴定效率最高,在多花黑麦草遗传多样性、品种鉴定和DNA指纹图谱的构建中具有重要的应用价值。
多花黑麦草品种(系)间存在相似的遗传背景和近缘关系,特别是杂交材料,一般是通过杂交后代连续混合选择或多个品种开放授粉连续混合选择育成的,因此,品系间、品系与亲本间亲缘关系相近,在品种鉴定时难以区分。本试验筛选出的6对引物成功将亲缘关系近的材料区分开,如赣选1号,同时是川农1号(牧杰×赣选1号)和LG1(F4N×赣选1号)的亲本,在6对引物下的指纹图谱代码分别为151131、231214和223224,证明了引物多态性好且鉴别效率高,在近亲品种的鉴定中也具有品种特异性片段,可形成不同的指纹代码。
3.2 DNA指纹图谱构建分析
DNA指纹图谱是快速鉴别植物品种时的有效工具,效率较 DUS鉴定法更加准确快速,错误率更低[35-36];较物理化学鉴定法和生化鉴定法更具有普遍性,不需要特殊的物理化学反应或者特殊的蛋白质,能在各类植物中普遍应用。DNA指纹图谱构建采用的方法一般有特征位点法[30]、单引物法[37]和核心引物组合法[38]。但由于利用单一引物法获取不同的品种特异性等位基因需要较多引物和更多的供试品种,导致鉴定效率较不同引物组合法更低[39-40],因此,本试验采用6对引物组合构建了10个多花黑麦草材料的指纹图谱,图谱包括指纹图谱标准模式图、指纹图谱代码和指纹图谱QR编码。虽然指纹图谱标准模式图与指纹图谱代码易于理解,但包含信息量较少;而指纹图谱QR编码信息量更大,可编码该品种的所有相关信息,检索该品种时更方便且全面,也更加有利于多花黑麦草指纹鉴定工作的快速进行。同时配合品种 DUS测试,可以更全面的解决生产实际中多花黑麦草品种的鉴定区分问题。本研究通过EST-SSR荧光标记毛细电泳检测法初步建立了多花黑麦草高通量指纹鉴定体系,为多色荧光检测法、种子纯度鉴定、指纹数据库的构建奠定了基础。
4 结论
本研究筛选应用了25对EST-SSR引物,构建了 10个多花黑麦草材料的DNA指纹图谱。通过引物多态性和高效性分析表明,荧光引物N101在多花黑麦草材料上多态性高,可直接鉴别本试验中的所有多花黑麦草材料。采用6对引物构建了多花黑麦草指纹图谱,通过标准模式图、图谱代码和图谱QR编码3种方式鉴定了10个多花黑麦草材料,分别构建了唯一的指纹代码和QR编码,建立了基于 SSR荧光标记的高通量多花黑麦草指纹鉴定体系。
[1] 董宽虎, 沈益新. 饲草生产学. 北京: 中国农业出版社, 2003: 113-117.
DONG K H, SHEN Y X. Forage Production. Beijing: China Agriculture Press, 2003: 113-117. (in Chinese)
[2] BOLARIC S, BARTH S, MELCHINGER A E, POSSELT U K. Genetic diversity in European perennial ryegrass cultivars investigated with RAPD makers. Plant Breeding, 2005, 124(2): 161-166.
[3] ROLDAN-RUIZ I, VAN EUWIJK F A, GILLILAND T J, DUBREUIL P, SILLMANN C, LALLEMAND J, LOOSE M D, BARIL C P. A comparative study of molecular and morphological methods of describing relationships between perennial ryegrass (Lolium perenne L.) varieties. Theoretical and Applied Genetics, 2001, 103(8): 1138-1150.
[4] GUSTAVO. DNA Markers. New York: Wiley-vch Press, 1998.
[5] International Union for the Protection of New Varieties of Plants. Guidelines for DNA-profiling: Molecular markers selection and database construction. Geneva: UPOV, 2006.
[6] International Union for the Protection of New Varieties of Plants. Possible use of molecular markers in the examination of distinctness, uniformity and stability. Geneva: UPOV, 2011.
[7] HIRATA M, CAI H W, INOUE M, YUYAMA N, MIURA Y, KOMATSU T, TAKAMIZO T, FUJIMORI M. Development of simple sequence repeat (SSR) markers and construction of an SSR-based linkage map in Italian ryegrass (Lolium multiflorum Lam.). Theoretical and Applied Genetics, 2006, 113(2): 42-47.
[8] PASAKINSKENE I, GRIFFITHS C M, BETTANY A J E. Simple-sequence repeats as primers to generate species-specific DNA markers in Lolium and Festuca grasses. Theoretical and Applied Genetics, 2000, 100(4): 384-390.
[9] 赵欣欣, 张新全, 苗佳敏, 黄琳凯, 季杨. 多花黑麦草杂交种 SSR分子标记鉴定及表型比较分析. 农业生物技术学报, 2013(7): 811-819.
ZHAO X X, ZHANG X Q, MIAO J M, HUANG L K, JI Y. Identification by SSR molecular markers and comparative analysis in morphological characterization in Lolium multiflorum hybrid progenies. Journal of Agricultural Biotechnology, 2013(7): 811-819. (in Chinese)
[10] 罗永聪, 马啸, 张新全. 利用SSR技术构建多花黑麦草品种指纹图谱. 农业生物技术学报, 2013, 21(7): 799-810.
LUO Y C, MA X, ZHANG X Q. DNA Fingerprinting of annual ryegrass (Lolium multiflorum Lam.) varieties using SSR makers. Journal of Agricultural Biotechnology, 2013, 21(7): 799-810. (in Chinese)
[11] 黄婷, 马啸, 张新全, 张新跃, 张瑞珍, 符开欣. 多花黑麦草 DUS测定中 SSR标记品种鉴定比较分析. 中国农业科学, 2015, 48(2): 381-389.
HUANG T, MA X, ZHANG X Q, ZHANG X Y, ZHANG R Z, FU K X. Comparation of SSR molecular markers analysis of annual ryegrass varieties in DUS testing. Scientia Agricultura Sinica, 2015, 48(2): 381-389. (in Chinese)
[12] PRAKASH P G, GOWDA M B, KUNDUR P J, VIMARSHA H S, SHASHIDHAR H E. Upgraded horizontal polyacrylamide gel units for DNA marker genotyping. Indian Journal of Science and Technology, 2015, 8(9): 822.
[13] 程本义, 夏俊辉, 龚俊义, 杨仕华. SSR荧光标记毛细管电泳检测法在水稻 DNA指纹鉴定中的应用. 中国水稻科学, 2011, 25(6): 672-676.
CHENG B Y, XIA J H, GONG J Y, YANG S H. Application of capillary electrophoresis detection with fluorescent SSR markers in rice DNA fingerprint identification. Chinese Journal of Rice Science, 2011, 25(6): 672-676. (in Chinese)
[14] 陈雅琼, 李凤霞, 李锡坤, 徐军, 张磊, 王绍美, 孙玉合. 烟草SSR荧光标记与毛细管电泳检测技术研究. 中国烟草科学, 2011, 32(2): 66-70.
CHEN Y Q, LI F X, LI X K, XU J, ZHANG L, WANG S M, SUN Y H. Fluorescent detection and capillary electrophoresis in tobacco SSR. Chinese Tobacco Science, 2011, 32(2): 66-70. (in Chinese)
[15] 易红梅. 基于毛细管电泳荧光标记的玉米品种 SSR高通量检测体系的建立研究[D]. 北京: 首都师范大学, 2006.
YI H M. Establishment a high throughput detection system of SSR markers fitting for maize variety based on capillary electrophoresis detection with fluorescent[D]. Beijing: Capital Normal University, 2006. (in Chinese)
[16] 许鲲, 李锋, 吴金锋, 谷铁城, 陈碧云, 高桂珍, 闫贵欣, 李俊,乔江伟, 汪念, 伍晓明. SSR荧光标记毛细管电泳法与国家冬油菜区试指纹鉴定平台的构建. 中国油料作物学报, 2014, 36(2): 150-159.
XU K, LI F, WU J F, GU T C, CHEN B Y, GAO G Z, YAN G X, LI J, QIAO J W, WANG N, WU X M. Fingerprint identification platform of capillary electrophoresis detection with fluorescent SSR markers on national winter rapeseed varieties (lines) field trials. Chinese Journal of Oil Crop Sciences, 2014, 36(2): 150-159. (in Chinese)
[17] SANCHEZ-PEREZ R, BALLESTER J, DICENTA F, ARUS P, MARTINEZ-GOMEZ P. Comparison of SSR polymorphisms using automated capillary sequencers, and polyacrylamide and agarose gel electrophoresis: Implications for the assessment of genetic diversity and relatedness in almond. Scientia Horticulturae, 2006, 108(3): 310-316.
[18] 张利达, 唐克轩. 植物 EST-SSR标记开发及其应用. 基因组学与应用生物学, 2010, 29(3): 534-541.
ZHANG L D, TANG K X. Development of plant EST-SSR markers and its application. Genomics and Applied Biology, 2010, 29(3): 534-541. (in Chinese)
[19] 赵岩, 孔凡美, 丁承强, 李斯深. EST-SSR标记在结缕草中的通用性. 中国草地学报, 2008, 30(3): 69-73.
ZHAO Y, KONG F M, DING C Q, LI S S. Transferability of EST-SSR markers in zoysiagrasses. Chinese Journal of Grassland, 2008, 30(3): 69-73. (in Chinese)
[20] 黄秀, 曾捷, 聂刚, 刘欢, 黄琳凯, 张新全, 蒋晓梅, 张瑜, 张博涛.牛鞭草品种EST-SSR指纹图谱构建及遗传多样性分析. 热带亚热带植物学报, 2014, 22(2): 165-171.
HUANG X, ZENG J, NIE G, LIU H, HUANG L K, ZHANG X Q, JIANG X M, ZHANG Y, ZHANG B T. Construction of fingerprinting and genetic diversity analysis of Hemarthia cultivars by EST-SSR. Journal of Tropical and Subtropical Botany, 2014, 22(2): 165-171. (in Chinese)
[21] 温莹, 逯晓萍, 任锐, 米福贵, 韩平安, 薛春雷. 高丹草 EST-SSR标记的开发及其遗传多样性. 遗传, 2013, 35(2): 225-232.
WEN Y, LU X P, REN R, MI F G, HAN P A, XUE C L. Development of EST-SSR marker and genetic diversity analysis in Sorghum bicolor×Sorghum sudanenes. Hereditas, 2013, 35(2): 225-232. (in Chinese)
[22] 王凤格, 易红梅, 赵久然, 吕波, 堵苑苑, 田红丽. 植物品种鉴定DNA指纹方法总则(NY/T2594-2014). 北京: 中国人民共和国农业部, 2014.
WANG F G, YI H M, ZHAO J R, LÜ B, DU Y Y, TIAN H L. General guideline for identification of plant varieties by DNA fingerprinting(NY/T2594-2014). Beijing: Ministry of Agriculture of the People's Republic of China, 2014. (in Chinese)
[23] 梁宏伟, 王长忠, 李忠, 罗相忠, 邹桂. 聚丙烯酰胺凝胶快速、高效银染方法的建立. 遗传, 2008, 30(10): 1379-1382.
LIANG H W, WANG C Z, LI Z, LUO X Z, ZOU G. Improvement of the silver-stained technique of polyacrylamide gel electrophoresis. Hereditas, 2008, 30(10): 1379-1382. (in Chinese)
[24] 张富民, 葛颂. 群体遗传学研究中的数据处理方法I.RAPD数据的AMOVA分析. 生物多样性, 2002, 10(4): 438-444.
ZHANG F M, GE S. Data analysis in population genetics I. Analysis of RAPD data with AMOVA. Biodiversity Science, 2002, 10(4): 438-444. (in Chinese)
[25] YEH F C, YANG R C, BOYLE T. Pop Gene, Version 1.31 Microsoft Windows-based Freeware for Population Genetic Analysis, Quick User-guide. University of Alberta: Center for the International Forestry Research, 1999.
[26] NEI M. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences of the USA, 1973, 70(12): 2223-3321.
[27] 陈昌文, 曹珂, 王力荣, 朱更瑞, 方伟超. 中国桃主要品种资源及其野生近缘种的分子身份证构建. 中国农业科学, 2011, 44(10): 2081-2093.
CHEN C W, CAO K, WANG L R, ZHU G R, FANG W C. Molecular ID establishment of main China peach varieties and peach related species. Scientia Agricultura Sinica, 2011, 44(10): 2081-2093. (in Chinese)
[28] 宋海斌, 崔喜波, 马鸿艳, 朱子成, 栾非时. 基于 SSR标记的甜瓜品种(系)DNA指纹图谱库的构建. 中国农业科学, 2012, 45(13): 2676-2689.
SONG H B, CUI X B, MA H Y, ZHU Z C, LUAN F S. Construction of DNA fingerprint database based on SSR marker for varieties (lines) of Cucumis melo L.. Scientia Agricultura Sinica, 2012, 45(13): 2676-2689. (in Chinese)
[29] 王凤格, 赵久然, 郭景伦, 佘花娣, 陈刚. 比较三种DNA指纹分析方法在玉米品种纯度及真伪鉴定中的应用. 分子植物育种, 2003, 5(1): 655-661.
WANG F G, ZHAO J R, GUO J L, SHE H D, CHEN G. Comparison of three DNA fingerprint analyzing methods for maize cultivars' identification. Molecular Plant Breeding, 2003, 5(1): 655-661. (in Chinese)
[30] 岳静, 朱志成, 申雅娟, 张卉, 王汝宝. 利用 SSR标记技术鉴定玉米品种真实性的研究. 中国农学通报, 2011, 27(12): 201-204.
YUE J, ZHU Z C, SHEN Y J, ZHANG H, WANG R B. Study on identifying the authenticity of maize varieties by SSR markers. Chinese Agricultural Science Bulletin, 2011, 27(12): 201-204. (in Chinese)
[31] MORGANTE M, OLIVIERI A M. PCR-amplified microsatellites as markers in plant genetics. The Plant Journal, 1993, 3(1): 175-182.
[32] ROJAS G, MENDEZ M A, MUNOZ C, LEMUS G, HINRICHSEN P. Identification of a minimal microsatellite marker panel for the fingerprinting of peach and nectarine cultivars. Electronic Journal of Biotechnology, 2008, 11(5): 4-5.
[33] DANIN-POLEGY, REIS N, BAUDRACCO-ARNAS S, PITRAT M, STAUB J E, OLIVER M, ARUS P, DE VICENTE C M, KATZIR N.Simple sequence repeats in Cucumis mapping and map merging. Genome, 2000, 43: 963-974.
[34] AKKAYA M S, SHOEMAKER R C, SPECHT J E, BHAGWAT A A, CREGAN P B. Integration of simple sequence repeat DNA markers into a soybean linkage map. Crop Science, 1995, 35(5): 1439-1445.
[35] 葛亚英, 张飞, 沈晓岚, 刘建新, 王炜勇, 田丹青, 张智. 丽穗凤梨ISSR遗传多样性分析与指纹图谱构建. 中国农业科学, 2012, 45(4): 726-733.
GE Y Y, ZHANG F, SHEN X L, LIU J X, WANG W Y, TIAN D Q, ZHANG Z. Analysis of genetic diversity and construction of fingerprint of Vriesea by ISSR. Scientia Agricultura Sinica, 2012, 45(4): 726-733. (in Chinese)
[36] HECKENBERGER M, BOHN M, ZIEGLE J S, JOE L K, HAUSER J D, HUTTON M, MELCHINGER A E. Variation of DNA fingerprints among accessions within maize inbred lines and implications for identification of essentially derived varieties: I. Genetic and technical sources of variation in SSR data. Molecular Breeding, 2002, 10: 181-191.
[37] 徐宗大, 赵兰勇, 张玲, 杨志莹. 玫瑰SRAP遗传多样性分析与品种指纹图谱构建. 中国农业科学, 2011, 44(8): 1662-1669.
XU Z D, ZHAO L Y, ZHANG L, YANG Z Y. Analysis of genetic diversity and construction of fingerprint of Rosa rugosa by SRAP. Scientia Agricultura Sinica, 2011, 44(8): 1662-1669. (in Chinese)
[38] 蒋林峰, 张新全, 黄琳凯, 马啸, 罗登, 张宗瑜, 蒙芬. 中国鸭茅主栽品种 DNA指纹图谱构建. 植物遗传资源学报, 2014, 15(3): 604-614.
JIANG L F, ZHANG X Q, HUANG L K, MA X, LUO D, ZHANG Z Y, MENG F. Construction of DNA fingerprinting of dominant orchardgrass (Dactylis glomerata) varieties of China. Journal of Plant Genetic Resources, 2014, 15(3): 604-614. (in Chinese)
[39] ARCHAK S, GAIKWARDA B, GAUTAM D, RAO E V V B, SWAMY K R M, KARIHALOO J L. DNA fingerprinting of Indian cashew (Anacardium occodentale L.) varieties using RAPD and ISSR techniques. Euphytica, 2003, 130(3): 397-404.
[40] FANG D Q, ROOSE M L. Identification of closely related citrus cultivars with inter-simple sequence repeat markers. Theoretical and Applied Genetics, 1997, 95(3): 408-417.
(责任编辑 李莉)
Construction of EST-SSR Fingerprinting Based on Fluorescence Detection Technology for Italian Ryegrass
LIU Huan1, ZHANG XinQuan1, MA Xiao1, ZHANG RuiZhen2, HE GuangWu2, PAN Ling1, JIN MengYa1
(1Department of Grassland Science, Sichuan Agricultural University, Chengdu 611130;2Grassland Station of Sichuan Province, Chengdu 610041)
【Objective】In this study, a Italian ryegrass (Lolium multiflorum Lam.) variety identification system based on fluorescently labeled ETS-SSR markers was developed to provide a high-throughput DNA profiling means for identification of Italian ryegrass varieties, which can provide valuable information for the use of Italian ryegrass production and an effective method of protecting farmers’ benefits and breeders’ rights.【Method】Using three Italian ryegrass varieties (Tetragold, ChangjiangNo.2 andAubade) with high difference of phenotypic traits, 30 primers were screened from the original 200 EST-SSR primers by polyacrylamide gel electrophoresis, which had clear amplification bands, rich polymorphism and stable amplification. Markers selected were labeled at the 5′ end of forward primer using fluorescent tags FAM, DNA analyzer was employed to detect different allelic variations of 200 individual PCR-amplified fragments by capillary electrophoresis. After further screening, 25 out of 30 fluorescent markers were chosen based on stable amplification to construct a high throughput identification system for Lolium multiflorum L..【Result】DNA fingerprint of ten Italian ryegrass materials were constructed using 25 EST-SSR primers for variety identification. A total of 127 alleles were amplified by 25 primer pairs, and the amplification fragment length ranged from 51 to 249. The effective number of alleles ranged from 2 to 11 on each pair of primers among them, primer N101 with 11 specific alleles was the most of all primers and each pair of primers had 4.00 specific alleles on average. The ratio of polymorphism sites ranged from 33.33% to 100.00%. The average value of polymorphic information content (PIC) was 0.702, the largest Shannon’s value (I) was 3.322 (N101) and the average value was 1.929, the Nei’s genetic diversity (H) ranged from 0.159 to 0.500 and the average value was 0.318. The amount of identified materials was from 0 to 10, and 14 of 25 primer pairs had 25 specific amplification sites among ten varieties (strains). Taken together, the results showed that the N101 fluorescence primer had the highest identification capability, which can identify these varieties (strains) directly. In addition, specific alleles were detected in primer ‘N101’ in three varieties such as Changjiang No.2, Ganxuan No.1 and Chuannong No.2. But due to high variation exists in same variety and different varieties of Lolium multiflorum L., in order to identify other materials, 6 EST-SSR primers (N54, N101, N146, N151, N154, N156) with good amplification and detection effect were chosen from 25 primers, the numbers of stable alleles were no less than 19, and the varieties “Double Barrel” and “Abundant” with 22 alleles were the most of all materials. Six high-efficiency primers were used for construction of DNA fingerprint spectrum including the standard model, fingerprint code and QR encodes of fingerprint spectrum. In this study, the fingerprint code and the unique QR encode for ten varieties of Italian ryegrass were firstly obtained using EST-SSR molecular marker with the aid of capillary electrophoresis and fluorescence labeling technology.【Conclusion】In this study, a SSR high-throughput identification system was constructed according to six pairs high efficiency primers, in which the ‘N101’ fluorescence primer having the most polymorphism can directly apply to identification of 10 Italian ryegrass varieties (strains).
Italian ryegrass (Lolium multiflorum Lam.); EST-SSR marker; fingerprinting; fluorescence detection; capillary electrophoresis
2016-08-04;接受日期:2016-11-04
国家现代牧草产业技术体系(CARS-35-05)、农业部草品种DUS测定项目(20130106)、四川省饲草育种攻关(2011NZ0098-11)
联系方式:刘欢,Tel:18728157834;E-mail:snlh9231@163.com。通信作者张新全,E-mail:zhangxq@sicau.edu.cn
