TNFα诱导NF-κB转录调控SLC2A3基因
2017-02-05彭仲特孙延杰王茂先
周 飞,彭仲特,王 微,孙延杰,王茂先
(韩山师范学院食品工程与生物科技学院,广东潮州 521041)
葡萄糖转运蛋白3编码基因SLC2A3是SLC2A基因家族的一员,定位于染色体12p13.31,起初被认为神经元特异表达[1-2].后来证明SLC2A3在CD45-阳性的白细胞中也表达[3].除此之外,SLC2A3在其他很多正常器官和组织中低表达或不表达[1].但是SLC2A3在多种类型癌症细胞中均高表达,如间充质状态的非小细胞肺癌和胶质细胞瘤[1]、喉鳞状癌[4]、低分化的子宫内膜癌和乳腺癌中[5]等.高表达的SLC2A3可能促进癌细胞的生长增殖等过程[1].但是至今癌细胞中SLC2A3高表达的分子机制不清楚.
NF-κB是1986年发现的DNA结合型转录因子,其家族包含NF-κB1(p50)、NF-κB2(p52)、p65/RelA、c-Rel和RelB共5个成员[6].通常该转录因子与IκBα等抑制蛋白结合以“静息状态”存在于细胞质中.但是TNFα等细胞因子可以诱导激活NF-κB进入细胞核,并结合靶基因的特异DNA序列(κB位点)转录调控它们的表达[6].在乳腺癌和肺癌等许多肿瘤组织中NF-κB被组成型激活[7].NF-κB可能通过转录调控其靶基因在肿瘤进展中发挥关键作用.以往发现,p53缺失的小鼠胚胎成纤维细胞(Mouse embryonic fibroblast,MEF)中NF-κB转录上调SLC2A3基因[8].此结果提示,人类癌症中SLC2A3高表达原因可能是被NF-κB转录上调的.据此,本研究先后分析了NF-κB对SLC2A3基因的结合情况和调控情况.结果首次在宫颈癌和肝癌细胞中发现SLC2A3基因是NF-κB的靶基因,此结果揭示了癌细胞中SLC2A3上调表达的分子机制,为揭示NF-κB在癌症中的分子功能提供了新视角.
1 材料与方法
1.1 主要试剂材料
人宫颈癌细胞株HeLa和人肝癌细胞株HepG2均购自中国科学院细胞库,胎牛血清购自Hy⁃Clone,胰酶、青霉素和链霉素购自碧云天,TNFα购自Sigma公司,脂质体2000和Trizol试剂购自Invitrogen公司,RelA/p65 siRNA和control siRNA均购自Cell signaling Technology公司,兔抗人-NF-κB/p65抗体购自Abcam 7970公司,正常兔IgG购自Santa Cruz公司,逆转录试剂盒购自Takara公司,荧光实时定量PCR试剂盒购自Roche公司,本研究所用引物均为南京金斯瑞公司合成.
1.2 细胞培养及处理
人宫颈癌细胞HeLa和人肝癌细胞HepG2均用含10%的胎牛血清的DMEM培养基,置5%CO2和37°C培养箱培养.首先接种3组HeLa细胞,次日采用脂质体2000向其中1组HeLa细胞转染30 nM RelA/p65 siRNA,48 h后再用30 ng/mL TNFα处理1 h,作为siRNA干扰组.与此同时,向余下2组细胞转染control siRNA,48 h后,将其中1组细胞用同量TNFα诱导1 h,作为TNFα诱导组;另一转染control siRNA且未经TNFα处理的细胞作为阴性对照组.本文使用的HepG2细胞也按类似的方法诱导处理.
1.3 ChIP-Seq数据及NF-κB结合峰分析
为了揭示NF-κB的分子功能,用染色质免疫共沉淀-测序(Chromatin Immunoprecipitation Se⁃quencing,ChIP-Seq)技术研究了NF-κB全基因组结合图谱[9].简言之,首先用30 ng/mL TNFα诱导HeLa细胞1 h.用兔抗人-NF-κB/p65抗体富集NF-κB结合的DNA,正常兔IgG作阴性对照.随后,用Illumina二代高通量测序仪检测富集的NF-κB结合的DNA片段.测序获得的reads数据用ELAND软件mapping到人参考基因组(hg19),再经ChIP-Peak在线软件(http://ccg.vital-it.ch/chipseq)识别ChIP-Seq结合峰(即NF-κB结合峰).ChIP-Peak软件运行参数设置如下,窗口宽度:200;邻近范围:200;峰值阈值:1;计数阈值:10.之后采用基因组浏览器UCSC可视化NF-κB结合峰.由于NF-κB可以通过结合远离靶基因的DNA元件并转录调控靶基因的表达[6,9].因此根据以往的研究结果[6,9],本文将SLC2A3基因的基因区设定为chr12:7 971 824~8 188 892(GRCh37/hg19),并用UCSC可视化该基因区内测序获得的NF-κB结合峰,统计分析NF-κB结合峰特征.
1.4 κB位点分析
本研究采用TRANSFAC在线软件分析SLC2A3基因的NF-κB结合峰内的κB位点分布情况.一般认为TRANSFAC计算的核心匹配分值(core match score,CMS)和矩阵匹配分值(matrix match score,MMS)均超过0.75的κB位点为经典的NF-κB结合位点[10].为了提高分析结果的可靠性,将CMS和MMS的阈值均设置为0.80.分析κB位点分布情况所采用的NF-κB结合矩阵为M00208、M00194、M00054、M00051、M00052和M00053.
1.5 RNA抽提和qPCR
首先采用Trizol试剂抽提HeLa和HepG2细胞内总RNA.然后于10 μL反应体系中采用逆转录试剂盒将总RNA逆转录为cDNA.最后使用1μL逆转录产物进行SYBR荧光实时定量PCR检测(qPCR检测),GAPDH为检测内参.所检测基因的引物见表1.
1.6 统计学分析
本研究荧光定量计算结果采用平均数±标准差表示,并采用SPSS13.0软件包进行统计T-检验.P<0.05为差异有统计学意义,用一个星号表示.P<0.01为差异有显著统计学意义,用两个星号表示.P<0.001为差异极显著,用三个星号表示.

表1 荧光定量PCR的引物
2 结 果
2.1 SLC2A3基因区内NF-κB结合峰的特征
以往采用ChIP-Seq技术在TNFα诱导的HeLa细胞中鉴定出NF-κB全基因组结合图谱[9].此图谱为揭示NF-κB新的靶基因提供了理论依据和线索.为了探索癌细胞中SLC2A3高表达的机制,进一步分析了NF-κB全基因组结合图谱.结果见表2,SLC2A3基因区内富含大量NF-κB结合峰,其中富集倍数(Fold Enrichment,FE)大于1的结合峰158个,FE大于10的结合峰57个,FE大于20的结合峰5个.

表2 SLC2A3基因区内NF-κB结合峰分布情况
自SLC2A3基因区的3′端起依次将以上FE大于20的NF-κB结合峰命名为结合峰1~结合峰5.分析发现,结合峰1~3位于SLC2A3基因的3′端,结合峰4和5位于SLC2A3基因的5′端.结合峰1的FE最高,为29.34,结合峰3的FE次高,为26.413.最短和最长的结合峰分别为结合峰1和结合峰3(表3).而且,结合峰1、3和4与DNase I超敏感位点(DNase I hypersensitive sites,DHSs)重合,结合峰1、2、3和4与H3K27Ac富集区重合(图1).以上结果表明,TNFα诱导NF-κB结合SLC2A3基因.

表3 SLC2A3基因区内富集倍数大于20的NF-κB结合峰特征

图1 SLC2A3基因的NF-κB结合峰(FE>20)
2.2 NF-κB结合峰内的κB位点特征
为了鉴定NF-κB是否直接结合SLC2A3基因,我们分析了NF-κB结合峰(FE大于20)内的κB位点分布情况.结果发现,NF-κB结合峰1~5分别含有3、4、6、8和4个κB位点(表4).这些κB位点的CMS和MMS均超过0.8,被认为是经典κB位点.并且结合峰2~5各含1个平均CMS为1的经典κB位点,分别为“GGAAAACACT”(6~15)、“GGAAACTGCA”(1603~1612)、“GTGTATTTCC”(175~184)和“GGAAATTCGC”(589~598).此结果表明这些κB位点与NF-κB结合矩阵的核心矩阵完全匹配(表4).而且有些经典κB位点同时被多个NF-κB结合矩阵匹配,比如结合峰3中的κB位点“GGGGGAAATCCCCTC”(700~713),被6个矩阵同时匹配(表4).综上所述,TNFα诱导NF-κB直接结合SLC2A3基因.此结果提示,TNFα可能诱导NF-κB调控SLC2A3基因表达.

表4 富集倍数大于20的NF-κB结合峰内κB位点特征

(续表4)
2.3 NF-κB转录活性的调节
利用GCBI数据库(https://www.gcbi.com.cn/gclib/html/index)分析发现,与正常组织相比,TNFα在很多类型的癌症中高表达,比如TNFα在宫颈癌、食管癌和子宫内膜癌等癌症中明显高表达(图2).结合上述结果,推测TNFα诱导NF-κB转录调控SLC2A3可能是其在癌症中高表达的分子机制.
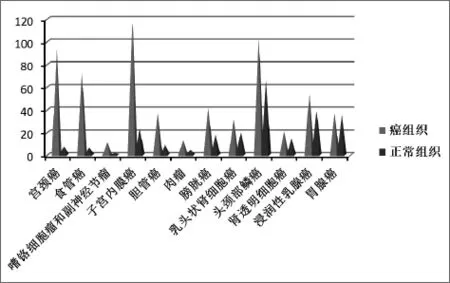
图2 TNFα的表达丰度
为了证明此推论,本研究用NF-κB活 性 诱 导 剂 (TNFα) 和 抑 制 剂(NF-κB/p65 siRNA)构建NF-κB转录活性差异的细胞模型.结果见图3A,TNFα上调 HeLa细胞的 NF-κB,同时NF-κB/p65 siRNA 干 扰 TNFα 上 调 的NF-κB.
为了进一步验证此细胞模型,我们检测IL6、IL8和IL7R共3个已知NF-κB靶基因的表达情况.结果发现,TNFα诱导NF-κB显著上调IL6、IL8和IL7R的表达,且此表达上调又能被NF-κB/p65 siRNA显著干扰(图4A).这些检测结果表明,我们已建立了NF-κB转录活性差异的HeLa细胞模型.采用类似的方法,我们还建立了NF-κB转录活性差异的HepG2细胞模型(图3B和图4B).

图3 qPCR检测NF-κB的转录水平

图4 qPCR检测TNFα诱导NF-κB调控IL6,IL8和IL7R的情况
2.4 NF-κB转录调控SLC2A3表达
利用上述细胞模型检测NF-κB对SLC2A3基因表达的调控情况,结果发现,HeLa细胞内TNFα诱导NF-κB上调SLC2A3的表达,NF-κB/p65 siRNA抑制TNFα诱导上调的SLC2A3(图5A).为了验证此结果,我们又在NF-κB转录活性差异的HepG2细胞模型中进行类似的检测并得出了一致的结果(图5B).以上检测结果表明,TNFα诱导NF-κB上调SLC2A3基因的转录表达.换句话说,SLC2A3是NF-κB的上调靶基因.

图5 qPCR检测TNFα诱导NF-κB调控SLC2A3的情况
3 讨 论
研究发现人类癌症中高表达的SLC2A3基因起重要作用[11].比如,高表达的SLC2A3与口腔癌的侵袭、大小、病理分期以及复发呈显著正相关[12],抑制SLC2A3的高表达能废止mir-106a介导的胶质瘤细胞异常增殖和葡萄糖吸收[11].因此,SLC2A3被认为是一个新的癌基因[11].但是,至今SLC2A3高表达的机制不清.
分析NF-κB全基因组结合谱数据[9],发现许多TNFα诱导的NF-κB结合峰分布于SLC2A3基因区.生物信息学方法分析表明,这些结合峰是由NF-κB结合产生的.而且这些NF-κB结合峰与DHSs等染色质上转录调控活跃区[13]重合或相邻,提示NF-κB的结合可能对SLC2A3的转录表达有调控作用.而且我们也发现,TNFα在宫颈癌等多种癌症中明显高表达,因此推测TNFα诱导NF-κB转录调控SLC2A3可能是其在癌症中高表达的分子机制.此后,我们构建并利用NF-κB转录活性差异的细胞模型研究发现,TNFα诱导NF-κB转录上调SLC2A3基因.此发现与在p53缺失的小鼠MEF细胞中检测得到的结果相一致[8].至此我们总结提出,NF-κB结合并转录调控SLC2A3基因.即,SLC2A3为NF-κB的靶基因.
众所周知,代谢重编程是肿瘤细胞标志之一.肿瘤细胞改变并增加了对葡萄糖吸收和消耗的方式来满足其快速生长增殖所需的生物量[14].最近研究发现,肿瘤细胞上调SLC2A家族基因可能是其高效利用葡萄糖的关键机制之一[5,15].已经发现,p53缺失的小鼠MEF细胞中NF-κB通过其靶基因SLC2A3介导的糖酵解相关的信号调控回路可能促进H-Ras诱导MEF细胞恶性转化[8].因此,抑制NF-κB活性可降低癌细胞的代谢活性[16].本研究同时在两种人类癌细胞中证明SLC2A3为NF-κB的上调靶基因,提示NF-κB可能通过转录上调其靶基因SLC2A3参与人类癌症的代谢过程,从而影响癌症进展.因此,本研究为深入洞察NF-κB在人类癌症发展中的分子功能提供了新的视角.
[1]Masin M,Vazquez J,Rossi S,et al.GLUT3 is induced during epithelial-mesenchymal transition and promotes tumor cell pro⁃liferation in non-small cell lung cancer[J].Cancer Metab,2014,2:11.
[2]Simpson I A,Dwyer D,Malide D,et al.The facilitative glucose transporter GLUT3:20 years of distinction[J].Am J Physiol Endocrinol Metab,2008,295(2):E242-253.
[3]McKinnon B,Bertschi D,Wotzkow C,et al.Glucose transporter expression in eutopic endometrial tissue and ectopic endome⁃triotic lesions[J].J Mol Endocrinol,2014,52(2):169-179.
[4]Starska K,Forma E,Jozwiak P,et al.Gene and protein expression of glucose transporter 1 and glucose transporter 3 in human laryngeal cancer-the relationship with regulatory hypoxia-inducible factor-1alpha expression,tumor invasiveness,and pa⁃tient prognosis[J].Tumour Biol,2015,36(4):2309-2321.
[5]Krzeslak A,Wojcik K K,Forma E,et al.Expression of GLUT1 and GLUT3 glucose transporters in endometrial and breast can⁃cers[J].Pathol Oncol Res,2012,18(3):721-728.
[6]Zhou F,Xu X,Wang D,et al.Identification of novel NF-kappaB transcriptional targets in TNFalpha-treated HeLa and HepG2 cells[J].Cell Biol Int,2017,41(5):555-569.
[7]Gupta S C,Sundaram C,Reuter S,et al.Inhibiting NF-kappaB activation by small molecules as a therapeutic strategy[J].Bio⁃chim Biophys Acta,2010,1799(10-12):775-787.
[8]Kawauchi K,Araki K,Tobiume K,et al.p53 regulates glucose metabolism through an IKK-NF-kappaB pathway and inhibits cell transformation[J].Nat Cell Biol,2008,10(5):611-618.
[9]Xing Y,Yang Y,Zhou F,et al.Characterization of genome-wide binding of NF-kappaB in TNFalpha-stimulated HeLa cells[J].Gene,2013,526(2):142-149.
[10]Wong D,Teixeira A,Oikonomopoulos S,et al.Extensive characterization of NF-kappaB binding uncovers non-canonical mo⁃tifs and advances the interpretation of genetic functional traits[J].Genome Biol,2011,12(7):R70.
[11]Dai D W,Lu Q,Wang L X,et al.Decreased miR-106a inhibits glioma cell glucose uptake and proliferation by targeting SLC2A3 in GBM[J].BMC Cancer,2013,13:478.
[12]Estilo C L,O-charoenrat P,Talbot S,et al.Oral tongue cancer gene expression profiling:Identification of novel potential prognosticators by oligonucleotide microarray analysis[J].BMC Cancer,2009,9:11.
[13]Deng T,Zhu Z I,Zhang S,et al.Functional compensation among HMGN variants modulates the DNase I hypersensitive sites at enhancers[J].Genome Res,2015,25(9):1295-1308.
[14]Renner K,Singer K,Koehl G E,et al.Metabolic Hallmarks of Tumor and Immune Cells in the Tumor Microenvironment[J].Front Immunol,2017,8:248.
[15]Ha T K,Chi S G.CAV1/caveolin 1 enhances aerobic glycolysis in colon cancer cells via activation of SLC2A3/GLUT3 tran⁃scription[J].Autophagy,2012,8(11):1684-1685.
[16]Zhou F,Xu X,Wu J,et al.NF-kappaB controls four genes encoding core enzymes of tricarboxylic acid cycle[J].Gene,2017,621:12-20.
