奶牛SREBP1蛋白在乳腺上皮细胞的表达定位及对SCD1基因启动子的转录调控
2017-01-13韩立强王月影王林枫朱河水褚贝贝杨国宇
韩立强,王月影,王林枫,朱河水,钟 凯,褚贝贝,杨国宇
(河南农业大学牧医工程学院/农业部动物生化与营养重点实验室,郑州 450002)
奶牛SREBP1蛋白在乳腺上皮细胞的表达定位及对SCD1基因启动子的转录调控
韩立强,王月影,王林枫,朱河水,钟 凯,褚贝贝,杨国宇
(河南农业大学牧医工程学院/农业部动物生化与营养重点实验室,郑州 450002)
【目的】固醇调节元件结合蛋白 1( SREBP1)作为核转录因子,对于细胞脂肪合成酶基因的表达发挥着重要的调控作用。论文旨在奶牛乳腺上皮细胞中研究SREBP1对于SCD1基因启动子的转录调控作用,为进一步明确SREBP1对于靶基因的转录调控机制提供理论基础。【方法】以荷斯坦奶牛乳腺组织的cDNA为模板,采用分段克隆的方法获得SREBP1基因的编码序列,通过重组酶与pcDNA3.1载体进行重组环化构建pcDNA3.1-SREBP1表达载体,将构建的载体测序验证后提取质粒,转染奶牛乳腺上皮细胞。以EIF3K基因为内参基因,采用荧光定量PCR检测SREBP1基因mRNA的表达差异;采用免疫荧光的方法对SREBP1进行标记,以DAPI复染细胞核,激光共聚焦观察SREBP1蛋白的亚细胞定位;转染含有不同调控元件的SCD基因启动子,同时转染1.0 µg pcDNA3.1-SREBP1作为处理,荧光素酶报告基因系统分析启动子活性;分别转染0.25、0.5和1µg的pcDNA3.1-SREBP1载体,分析pGL3-SCD 2 和pGL3-SCD3启动子活性与SREBP1之间的量效关系。【结果】分段克隆得到的PCR产物分别为1 170、1 116、363和900 bp的片段,经过与pcDNA3.1载体重组后获得pcDNA3.1-SREBP1表达载体,经酶切和测序验证,发现除1个无义突变外,与标准序列完全相同,整个序列长度达到3 510bp;将pcDNA3.1-SREBP1载体转染乳腺上皮细胞后,Real-time PCR检测发现与转染空载体的对照组相比,SREBP1基因的mRNA表达倍数增强130.4倍(P<0.001);激光共聚焦观察发现,DAPI染色的细胞核呈蓝色,免疫荧光标记的SREBP1呈绿色,二者融合后呈现青色,共定位在乳腺上皮细胞核中;启动子活性检测发现,与pGL3-SCD1、pGL3SCD 2相比,SREBP1处理能够极显著增加pGL3-SCD3、pGL3-CD4启动子的活性(P<0.001),分别比对照组提高了1.0倍和0.7倍,进一步分析发现,在用0.25—1 µg的pcDNA3.1-SREBP1处理后,与pGL3-SCD2的启动子活性持续下降相比,pGL3-SCD3的启动子活性从59.81上升到108.43(P<0.001),二者存在剂量效应关系,结合SCD2和SCD 3启动子上主要的结构差异SRE元件(5′-AGCAGATTGCG-3′),推测此序列可能是SREBP1调控SCD基因启动子转录的结合序列。【结论】克隆构建奶牛SREBP1基因表达载体,亚细胞定位SREBP1蛋白主要在乳腺上皮细胞核中,SREBP1可以与SRE调控元件结合促进SCD1基因启动子的转录。
奶牛;固醇调节元件结合蛋白 1;乳腺上皮细胞;亚细胞定位;转录调控
Abstract:【Objective】As a member of nuclear transcription factor, sterol regulatory element binding protein1(SREBP1)plays a significant role in the expression of the lipogenic gene . The objective of this study is to investigate the effect of SREBP1 on the regulation of transcription of SCD1 gene in the mammary epithelial cells, which will provide a fundamental basis for the transcription regulation mechanism of SREBP1 to target gene. 【Method】 The coding sequence (CDS) of SREBP1 gene was cloned using the method of subsection cloning with the cDNA of Holstein mammary tissues as the template. The expression vector pcDNA3.1-SREBP1 was recombined and constructed by recombining vector pcDNA3.1. Plasmids were extracted from the constructed vector after being verified by sequencing, and transfected to mammary epithelial cells. With gene EIF3K as the reference gene, the expressional difference of mRNA in gene SREBP1 was detected with Real-time PCR. SREBP1 was marked with the immunofluorescence method. The cell nucleus was restained with DAPI, and the subcellular localization of protein SREBP1 was observed with laser con-focal microscopy. The SCD1 gene promoters with different regulatory elements and 1.0 μg pcDNA3.1-SREBP1 were restained as treatments, and the promoter activity was analyzed systematically with luciferase reporter gene. Vectors pcDNA3.1-SREBP1 of 0.25 μg, 0.5 μg and 1 μg were transfected respectively. The dose-effect relationship between the promoter activity of pGL3-SCD2 , pGL3-SCD3 and protein SREBP1 was analyzed. 【Result】 The PCR products cloned in subsection were fragments 1170bp, 1116bp, 363bp and 900 bp respectively. The expression vector pcDNA3.1-SREBP1 was obtained by reconstructing the fragments and vector pcDNA3.1. Confirmed by digestion and sequence, the expression vector, except one nonsense mutation, was exactly the same with the standard sequence. The whole sequence length reached 3510 bp. After vector pcDNA3.1-SREBP1 was transfected into mammary epithelial cells, compared with the control group of the empty transfected vector, the expression mRNA of gene SREBP1 increased by 130.4 times (P<0.001)by the real-time PCR detection. As shown in the observation of the laser con-focal microscopy, the cell nucleus dyed with DAPI was blue, and SREBP1 with immunofluorescent labeling was green, both of which were cyan after being merged and were localized in the mammary epithelial cell nucleus. The results of the detection of the promoter activity showed that, compared with those of pGL3-SCD1 and pGL3SCD 2, the treatment with SREBP1 increased the promoter activity of pGL3-SCD3 and pGL3-CD4 significantly (P<0.001), and their promoter activity increased by 1.0 and 0.7 times. The further study showed that, after treatment with 0.25-1µg pcDNA3.1-SREBP1, compared with the continuous decrease of the promoter activity of pGL3-SCD2, the promoter activity of pGL3-SCD3 increased from 59.81 to 108.43(P<0.001). There is a dose-effect relationship between them. Combined with the element SRE(5′-AGCAGATTGCG-3′), the architectural difference between promoter SCD2 and promoter SCD 3, it could be speculated that the sequence was the incorporating sequence in which SREBP1 regulated the transcription of gene promoter SCD1.【Conclusion】 It was verified that the gene expression vector SREBP1 was constructed by cloning, the subcellular localization of protein SREBP1 was in the mammary epithelial cell nucleus, and SREBP1 promoted the regulation of the transcription of SCD1 promoter together with regulatory element SRE.
0 引言
【研究意义】乳腺做为牛、羊等哺乳动物合成分泌乳汁的主要器官,其基因表达调控机制一直是畜牧工作者进行科学研究的重要内容。BIONAZ等采集奶牛乳腺组织分析基因表达谱[1],发现固醇调节元件结合蛋白1(Sterol-regulatory element binding protein1,SREBP1)作为一种转录因子,是乳腺中脂肪合成代谢基因表达调控网络的中心,其他研究发现 一些营养因素可以影响 SREBP1蛋白的活性和脂肪合成酶基因的表达[2-3]。因此研究SREBP1及脂肪合成酶基因在乳腺上皮细胞中的表达调控机制,可以为阐明营养元素影响乳腺脂肪合成的机制提供理论基础。【前人研究进展】早期在生产中发现对奶牛饲喂油脂会造成奶牛的乳脂率下降,形成生产上低脂乳症(milk fat depression, MFD)的现象[4],后来发现奶牛在发生MFD时,不仅乳脂肪含量与组成发生变化,而且乳腺组织的脂肪合成关键酶的基因表达也受到抑制[4-6],进一步研究发现在降低脂肪合成酶基因表达的同时也能够影响SREBP1基因mRNA的表达[3,7]。SREBP1作为重要的核转录因子,主要调控细胞内的胆固醇浓度和脂类的稳态[8-10]。对于SREBP1调控靶基因的促转录功能,通过采用CHIP-chip和CHIP-seq的研究,在人和小鼠全基因组上已发现上百个可能受SREBP1调控的靶基因,其中主要包括硬脂酰辅酶 A去饱和酶(stearoyl-coenzyme A desaturase,SCD)等脂肪合成酶基因[11-13]。SCD是催化饱和脂肪酸脱氢形成不饱和脂肪酸的关键酶,在奶牛中主要存在SCD1和SCD5两种亚型[14],并且SCD1在奶牛乳腺基因表达谱中所占的比例远远超过其他脂肪合成酶基因[1]。已有多个研究表明SCD1和SREBP1基因的核酸多态性均与乳脂肪性状相关[15-16]。【本研究切入点】目前在细胞上研究SREBP1与靶基因的转录调控机制多是采用间接研究模式,在培养细胞中加入诱导SREBP1的刺激因素(如胰岛素/葡萄糖),通过引起SREBP1及其靶基因mRNA的表达变化来进行推测[17],而这些基因表达变化是SREBP1直接调控还是由于代谢通路的其他因子引起的,目前还不十分清楚[18]。还有研究采用表达SREBP1成熟核蛋白来研究其转录调控作用[19],但对于SREBP1全长蛋白在乳腺上皮细胞的转录调控机制还缺乏了解,特别是关于奶牛SREBP1在乳腺上皮细胞中调控 SCD1基因转录的研究还未见报道。【拟解决的关键问题】因此本研究结合前期试验建立的含不同调控元件奶牛 SCD1基因启动子[20],通过克隆表达奶牛SREBP1蛋白,在奶牛乳腺上皮细胞中研究SREBP1对于SCD1基因启动子的转录调控作用,有助于阐明SREBP1对于靶基因的转录调控机制。
1 材料与方法
相关试验在2014年1月至2015年4月在河南农业大学农业部动物生化与营养重点实验室完成。
1.1 材料与仪器
奶牛乳腺组织于2014年1月采自郑州市屠宰场,乳腺上皮细胞为实验室保存,4个不同长度 pGL3-SCD1/SCD2/SCD3/SCD4荧光素酶报告基因表达载体为实验室构建,所用试剂AxyPrep DNA Gel Extraction Kit 试剂盒购自Axygen 公司,ClonExpress Multis kit试剂盒购自Vazyme公司,Lipofectamine2000购自Invitrogen公司,Star XL扩增酶购自Takala公司,双荧光素酶检测试剂盒购自Promega公司,DMEM培养基购自Hyclone公司,无血清培养基Opti-mem购自 Gibco公司,标签蛋白抗体 c-myc Antibody(9E10)购自Santa Cruz公司,荧光二抗Alexa Fluor® 488 Donkey Anti-Mouse IgG (H+L)购自Invitrogen公司,细胞核染料DAPI、抗荧光封片淬灭液购自碧云天,细胞培养板购自 Corning公司,引物合成及测序由上海生工完成。二氧化碳培养箱和 Fluroskan Ascent FL荧光和化学发光检测仪购自 Thermo公司,激光共聚焦显微镜LSM 5 PASCAL购自Carl Zeiss公司。
1.2 方法
1.2.1 奶牛SREBP1基因的克隆 采用TRIZOL提取乳腺组织的RNA,反转录成cDNA,根据NCBI公布的奶牛SREBP1基因序列(NM_001113302.1),设计引物时在 SREBP1序列 ATG后加入 c-myc序列(GAACAAAAACTCATCTCAGAAGAGGATCTG)作为标签蛋白,采用分段克隆 PCR方法对奶牛SREBP1基因进行克隆,具体引物序列见表 1。将SREBP1总共分为四段分别进行扩增,同时PCR扩增pcDNA3.1+序列作为载体序列,分别对目的基因1、2、3、4各片段纯化、电泳后定量。
1.2.2 pcDNA3.1-SREBP1载体的构建及鉴定 采用重组酶 Exnase Multis对各片段与载体进行重组环化,重组反应体系:5×CE Multis Buffer 4 μL,vector 108 ng,4个回收片段,Exnase Multis 2 μL,同时做无酶的空白对照,在37℃水浴30 min,置于冰水浴中反应5 min。取反应产物10 μL,加入到感受态细胞中进行转化,获得克隆后挑取单菌落扩增,提取质粒进行分别采用HindⅢ、XhoI双酶切和HindⅢ、XbaI双酶切鉴定,鉴定正确的质粒送测序公司进行测序。

表 1 引物序列Table 1 The sequences of primer
1.2.3 SREBP1基因mRNA在乳腺上皮细胞中的超表达将培养好的奶牛乳腺上皮细胞经胰酶消化后,按比例分散培养在12孔细胞培养板中,放入37℃、5% CO2培养箱培养,到细胞达到80%的融合度时,弃去培养基,采用转染试剂(Lipofectamine 2000)转染pcDNA3.1-SREBP1质粒6 h后,更换新鲜培养基,同时以pcDNA3.1空载体作为对照组,转染48 h后提取细胞RNA,反转录成cDNA,采用荧光定量PCR引物(F-CCAGCTGACAGCTCCATTGA,R-TGCGCGC CACAAGGA)检测SREBP1基因mRNA的表达,同时检测奶牛EIF3K基因(F-CCAGGCCCACCAAGA AGAA,R- TTATACCTTCCAGGAGGTCCATGT)作为内参基因[21],计算SREBP1基因的mRNA表达差异倍数。
1.2.4 SREBP1蛋白在乳腺上皮细胞的定位 在24孔板中加入细胞爬片,接种乳腺上皮细胞,细胞培养过夜后,转染pcDNA3.1-SREBP1质粒,培养24 h后用预冷的PBS洗3遍,4%多聚甲醛固定10 min,PBS洗3遍,透化剂(0.2%Triton100)作用15 min,封闭液(90%PBS+10%胎牛血清)封闭1 h;弃封闭液,用一抗c-MYC Antibody(9E10)(1∶500)4 ℃过夜孵育;PBS洗3遍,加入荧光标记二抗Alexa Fluor® 488 Donkey Anti-Mouse IgG(H+L)(1∶1 000)室温避光孵育1 h;然后采用DAPI染色液(500µg·mL-1)对于细胞核染色8 min,PBS洗3遍,滴加抗荧光封片淬灭液,封片,晾干放 4℃,在激光共聚焦显微镜下观察SREBP1蛋白亚细胞定位。
1.2.5 SREBP1蛋白对含不同调控元件SCD1启动子的转录调控作用 培养乳腺上皮细胞,以 pGL3-Basic荧光素酶空载体作为对照,转染实验室前期构建的4个含有不同调控元件的奶牛 SCD1基因启动子载体[20],分别是pGL3-SCD1(212bp,序列中有TATA-box元件)、pGL3-SCD2(380bp,有CAAT-box元件)、pGL3-SCD3(416 bp,有SRE元件)和pGL3-SCD4(760 bp,有SRE、Sp1元件)(图1),同时分别转染1.0 µg pcDNA3.1-SREBP1质粒作为处理因素,作用24 h后收集细胞加入裂解液,采用荧光和化学发光检测仪进行荧光素酶活性检测,采用萤火虫荧光素酶活性/海肾荧光素酶活性计算启动子活性,结合调控元件分析SREBP1处理对于启动子活性的影响。
1.2.6 SREBP1调控SCD1基因启动子转录的剂量关系 培养乳腺上皮细胞,接种12孔细胞培养板后,转染1.0 µg pGL3-SCD2、pGL3-SCD3启动子载体,同时分别转染0.25、0.5、1.0µg的pcDNA3.1-SREBP1质粒作为处理因素,作用24 h后收集细胞,加入裂解液,进行荧光素酶活性检测并计算启动子相对活性,分析不同剂量SREBP1的处理对于启动子活性的影响。

图1 含不同调控元件的奶牛SCD1启动子结构Fig. 1 The structure of bovine SCD1 promoter with different regulatory elements
1.2.7 数据统计 试验中每个处理中重复4次,试验数据采用SPSS10.0软件进行统计学分析,**P<0.01,***P<0.001。
2 结果
2.1 SREBP1基因的克隆
对 SREBP1基因采用分段克隆的方法,分别获得大小约5 400 bp 的pcDNA3.1载体片段和4个不同长度的目的基因片段(图2),将各个片段切胶回收后进行基因重组获得连接产物,将连接产物转化感受态细胞进行阳性克隆筛选,提取阳性细菌质粒后进行酶切鉴定。分别采用HindIII、XhoI双酶切,获得3个片段,片段长度分别为5 356、3 057和390 bp,采用HindIII、Xbal双酶切,获得2个片段,分别为5 350和3 453 bp(图3),最后经测序验证后发现,克隆的序列与 NCBI公布的标准序列相比,除在1 509位有一个无义突变外,其他序列均与标准序列相一致,加上标签蛋白myc序列,整个表达序列长度达到3 510 bp。
2.2 SREBP1在乳腺上皮细胞中的超表达
将pcDNA3.1-SREBP1质粒转染乳腺上皮细胞后48h收集细胞,采用荧光定量PCR检测SREBP1基因的表达丰度,结果发现,与转染空载体的对照相比,SREBP1基因的mRNA表达显著升高(图4),比对照组增加了 130.4倍(P<0.001),表明载体转染后在奶牛乳腺上皮细胞中能够高效表达SREBP1基因的mRNA。
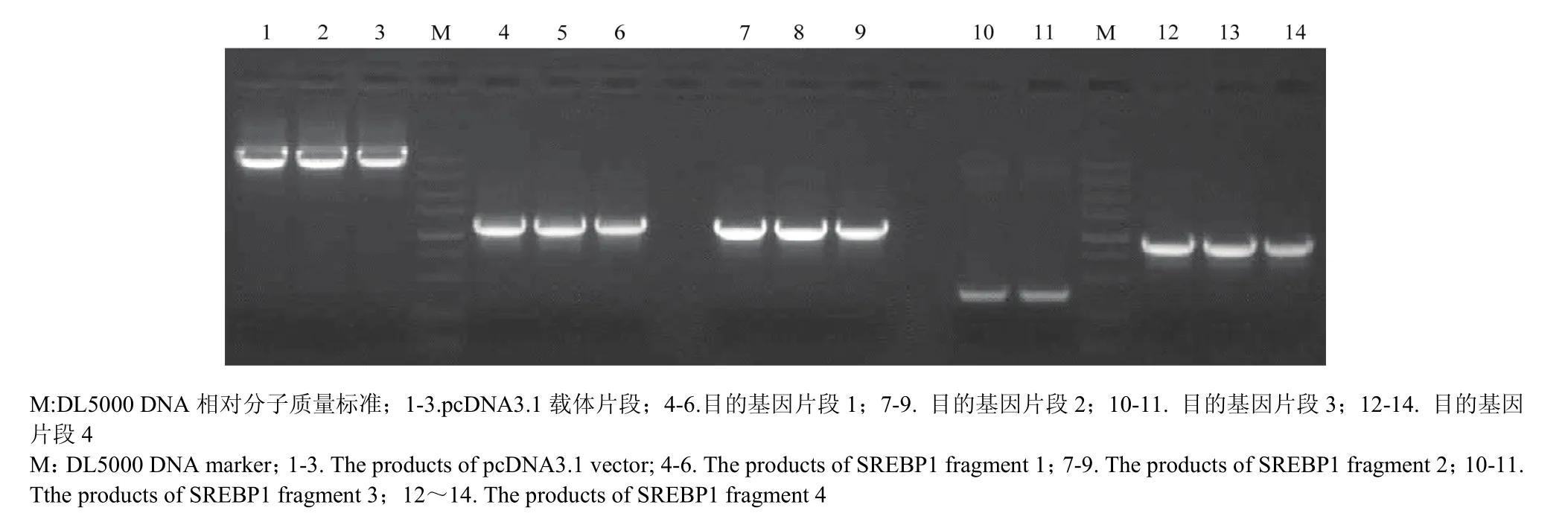
图2 奶牛SREBP1基因的克隆Fig. 2 The clone of bovine SREBP1
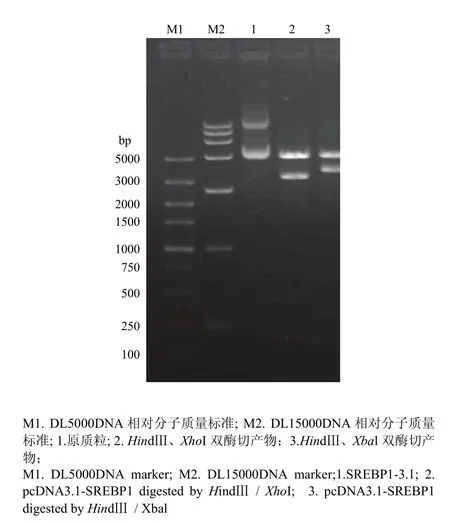
图3 pcDNA3.1-SREBP1质粒酶切鉴定图Fig. 3 The restriction enzyme digestion of pcDNA3.1-SREBP1
2.3 SREBP1蛋白在乳腺上皮细胞中的定位
将 pcDNA3.1-SREBP1载体转染乳腺上皮细胞后,采用激光共聚焦显微镜观察发现(图 5),进行免疫荧光标记的SREBP1发出绿色荧光,细胞核经 DAPI染色后发出蓝色荧光,共定位发现绿色和蓝色融合形成一种青色荧光定位在细胞核位置,表明SREBP1作为转录因子能够转运到乳腺上皮细胞核中进行表达。
2.4 SREBP1蛋白对于不同SCD1启动子的调控作用
将含不同调控元件的 SCD1基因启动子 pGL3-SCD1、SCD2、SCD3和SCD4转染细胞后,同时转染SREBP1质粒进行处理,检测启动子活性发现(图6),对照组basic启动子活性为2.32,在SREBP1刺激后启动子活性降低到1.29(P<0.01),与此类似,SCD1和SCD2启动子进行SREBP1处理后,荧光值均显著降低(P<0.01),而对 SCD3、SCD4启动子进行SREBP1处理后,启动子活性均有显著增加。其中SCD3启动子活性由59.92增加到124.37,增加了1.0倍(P<0.001),SCD4进行SREBP1处理后,启动子活性由 83.55增加到 145.48,增加了 0.7倍(P< 0.001),表明SREBP1蛋白表达后,显著增加了SCD3、SCD4启动子的活性,其中对于SCD3启动子的活性促进最为明显。

图4 SREBP1基因mRNA在乳腺上皮细胞中的表达丰度Fig. 4 The mRNA expression of SREBP1 in mammary epithelial cell (n=4)
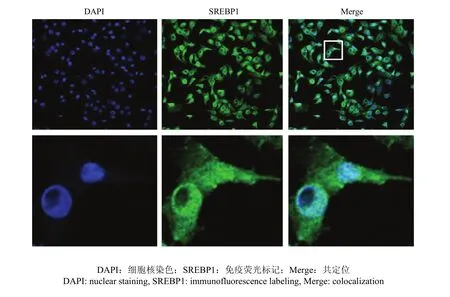
图5 SREBP1蛋白在乳腺上皮细胞的表达定位Fig. 5 The localization of SREBP1 in mammary epithelial cell

图6 SREBP1对不同SCD1启动子活性的调控Fig. 6 The regulation of different SCD1 promoter activities by SREBP1
2.5 SREBP1调控SCD基因启动子转录的量效关系

图7 不同含量SREBP1对pGL3-SCD2、3启动子活性的调控Fig. 7 The regulation of pGL3-SCD2 and SCD3 promoter activities by different doses of SREBP1
在转染0.25、0.5、1.0 μg的 SREBP1质粒后,pGL3-SCD2启动子的活性呈现持续下降的趋势(图7),从19.90降低到10.31,而pGL3- SCD3启动子的活性随着质粒含量的增加则呈现上升趋势,其活性值从59.81分别增加到70.76、101.48和108.43(P<0.001),表明SREBP1能够剂量依赖性的增强pGL3- SCD3启动子的活性。
3 讨论
固醇调节元件结合蛋白(SREBP)属于“碱性螺旋-环-螺旋亮氨酸拉链”转录因子家族,其中SREBP1蛋白结构域一般分为3部分,包括480个氨基酸的N-端,80个氨基酸的跨膜结构域和590个氨基酸的C-端[22-23]。本研究中荷斯坦奶牛SREBP1基因的编码区全长3 440 bp,加上标签蛋白序列后整个基因长度在3 510 bp左右。由于SREBP1基因序列中含有复杂的二级结构,为提高克隆的准确性和效率,本研究采用分段克隆、基因重组的方法,得到了完整的SREBP1基因编码区序列,表明此分段克隆重组的方法对于长片段的基因克隆具有较好的效果。经过细胞转染和荧光定量PCR检测发现,SREBP1基因的mRNA有显著表达(图4),表明载体构建成功。
SREBP1作为在内质网合成的蛋白质,首先合成无活性的前体(大约1 150个AA),然后通过与SCAP的相互作用后进入到高尔基体进行水解活化,暴露出NH2-末端活性结构域形成入核蛋白(大约500个AA),进入到细胞核内与靶基因启动子结合调控转录[24]。因此本研究采用激光共聚焦对表达的SREBP1蛋白进行亚细胞定位后,发现SREBP1能够在细胞核中表达(图5),这说明构建的 SREBP1质粒在细胞内表达全长蛋白后能够水解活化形成入核蛋白,转运到细胞核内发挥功能。
对于在乳腺上皮细胞中 SREBP1与靶基因的研究发现,SREBP1通过激活PⅢ启动子来调节乙酰辅酶A羧化酶基因的转录[25]。MA等在乳腺上皮细胞中通过构建SREBP1的siRNA抑制载体,能够降低SCD、ACC和FAS等多种脂肪酸合成酶基因mRNA的表达[26]。本研究采用不同调控元件的 SCD1启动子,通过在乳腺上皮细胞表达SREBP1后发现pGL3-SCD1-2启动子的活性显著降低,而 pGL3-SCD3-4启动子的活性显著增加(图6)。研究表明SREBPs 自身是非常低效的转录因子, 只有接近结合位点时才能刺激靶基因的表达[27],而 pGL3-SCD3、4比pGL3-SCD1、2在结构上主要多出了一个SRE调控元件(图1),表明SRE调控元件可能对于SREBP1调控 SCD1基因转录具有重要作用。ZULKIFLI在HEK细胞中采用表达人 SREBP1-c成熟核蛋白来调控羊SCD基因启动子的转录,发现去除羊SCD基因启动子的SRE元件显著降低了启动子活性,其结果与本研究一致[19]。为了进一步验证SRE调控元件的作用,试验又采用不同浓度的 SREBP1刺激 pGL3-SCD2、pGL3-SCD3启动子,结果发现 pGL3-SCD3启动子的活性增加与 SREBP1的浓度呈现剂量依赖性,结合pGL3-SCD3与pGL3-SCD2启动子结构上相比只有36bp的差异(其中含有SRE调控元件),说明SREBP1可以通过与SRE元件结合促进SCD1基因转录。
启动子上与转录因子结合的位点具有特定模式,称为模体,一般长度在5—20 bp范围内。转录因子在细胞内可以同时调控多个基因,而在不同基因上的结合位点并不完全相同。研究认为SREBP1结合的经典模体为(5′-TCACNCCAC-3′)/E-和(5′-CANNTG-3′)两种[13,18]。本研究中 SRE调控元件含有的模体为5′-AGCAGATTGCG-3′(图1),与经典的SREBP1模体序列并不相符。TABOR 等在HepG2细胞上研究小鼠SREBP1a与鼠SCD基因启动子的关系,发现了与本研究一致的SRE新模体(5′-AGCAGATTG CG-3′)对于启动子活性至关重要,而经典模体反而对启动子活性没有显著影响[28],ZULKIFLI比对人、猪、鼠、羊的SCD1基因启动子序列后发现此模体在不同物种间是高度保守的[19]。这些结果说明SREBP1可能通过与SCD启动子上(5′-AGCAGAT TGCG-3′)模体的结合,发挥了转录因子的促转录作用。SEO等在小鼠组织中曾经发现了 1 个新的SREBP1模体,并认为是经典模体的一个功能变体[12],因此本研究中发现的 SREBP1模体序列可能是SCD1基因所特有的一个功能变体,这还需要进一步的研究证实。
4 结论
本研究通过克隆构建奶牛固醇调节元件结合蛋白1真核表达载体,发现固醇调节元件结合蛋白1能够定位在乳腺上皮细胞核中,并且固醇调节元件结合蛋白1可以与SRE调控元件结合促进SCD1基因启动子的转录。
[1] BIONAZ M, LOOR J J. Gene Networks driving bovine milk fatsynthesis during the lactation cycle. BMC Genomics, 2008, 9: 366.
[2] BERNARD L, LEROUX C, CHILLIARD Y. Expression and nutritional regulation of lipogenic genes in the ruminant lactating mammary gland. Advances in Experimental Medicine and Biology, 2008, 606(1): 67-108.
[3] HARVATINE K J, BAUMAN D E. SREBP1 and thyroid hormone responsive spot 14 (S14) are involved in the regulation of bovine mammary lipid synthesis during diet-induced milk fat depression and treatment with CLA. Journal of Nutrition, 2006, 136(10): 2468-2474.
[4] BAUMAN D E, GRINARI J M. Regulation and nutritional manipulation of milk fat:low-fat milk syndrome. Advanced Experimental Medicine and Biology, 2000, 480:209-216.
[5] PETERSON D G, MATITASHVILI E A, BAUMAN D E. Diet-induced milk fat depression in dairy cows results in increased trans-10, cis-12 CLA in milk fat and coordinate suppression of mRNA abundance for mammary enzymes involved in milk fat synthesis. Journal of Nutrition, 2003, 133(10): 3098-3102.
[6] AHNADI C E, BESWICK N, DELBECCHI L, KENNELLY J J, LACASSE P. Addition of fish oil to diets for dairy cows. II. Effects on milk fat and gene expression of mammary lipogenic enzymes. Journal of Dairy Research, 2002, 69(4): 521-531.
[7] HAN LQ, PANG K, LI H J, ZHU S B, WANG L F. Conjugated linoleic acid-induced milk fat reduction associated with depressed expression of lipogenic genes in lactating Holstein mammary glands. Genetics and Molecular Research, 2012, 11(4):4754-4764.
[8] DAEMEN S, KUTMON M, EVELO C T. A pathway approach to investigate the function and regulationof SREBPs. Genes and Nutrition, 2013, 8(3):289-300.
[9] WEBER L W, BOLL M, STAMPFL A. Maintaining cholesterol homeostasis: sterol regulatory element-binding proteins. World journal of gastroenterology, 2004, 10(21): 3081-3087.
[10] JEON T I, OSBORNE T F. SREBPs: metabolic integrators in physiology and metabolism. Trends in Endocrinology and Metabolism, 2012, 23(2):65-72.
[11] ROME S, LECOMTE V, MEUGNIER E, RIEUSSET J, DEBARD C,EUTHINE V, VIDAL H, LEFAI E. Microarray analyses of SREBP-1a and SREBP-1c target genes identify new regulatory pathways in muscle. Physiological Genomics, 2008, 34(3): 327-337.
[12] SEO Y K, CHONG H K, INFANTE A M, IM S S, XIE X H, OSBORNE T F. Genome-wide analysis of SREBP-1 binding in mouse liver chromatin reveals a preference for promoter proximal binding to a new motif. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(33): 13765-13769.
[13] REED B D, CHAROS A E, SZEKELY A M, WEISSMAN S M, SNYDER M. Genome-wide occupancy of SREBP1 and its partners NFY and SP1 reveals novel functional roles and combinatorial regulation of distinct classes of genes. PLoS Genetics, 2008, 4(7): e1000133.
[14] 张蕊, 张宜辉, 邵丹, 王来娣, 龚道清. 硬脂酰辅酶 A去饱和酶基因的功能与调控. 生命科学, 2013, 25(4):378-382.
ZHANG R, ZHANG Y H, SHAO D, WANG L D, GONG D Q. The function and regulation of stearoyl-CoA desaturase gene. Chinese Bulletin of Life Sciences, 2013, 25(4):378-382. (in Chinese)
[15] CECCHINATO A, RIBECA C, MAURMAYR A, PENASA M, DE MARCHI M, MACCIOTTA N P, MELE M, SECCHIARI P, PAGNACCO G, BITTANTE G. Short communication: Effects of β-lactoglobulin, stearoyl-coenzyme A desaturase 1, and sterol regulatory element binding protein gene allelic variants on milk production, composition, acidity, and coagulation properties of Brown Swiss cows. Journal of Dairy Science, 2012, 95(1): 450-454.
[16] RINCON G, ISLASTREJO A, CASTILLO A , BAUMAN D E, GERMAN B J, MEDRANO J F. Polymorphisms in genes in the SREBP1 signalling pathway and SCD are associated with milk fatty acid composition in Holstein cattle. The Journal of Dairy Reasearch, 2012, 79(1):66-75.
[17] ZHANG Y, CHEN W, LI R, LI Y, GE Y B, CHEN G X. Insu-lin-regulated Srebp-1c and Pck1 mRNA expression in primary hepatocytes from zucker fatty but not lean rats is affected by feeding conditions. PLoS One, 2011, 6(6): e21342.
[18] 汤晓丽, 邓立彬, 林加日, 张伟龙, 刘双梅, 魏懿, 梅普明, 汪雁,梁尚. 固醇调节元件结合蛋白 1及其靶基因网络. 遗传, 2013, 35(5): 607-615.
TANG X L, DENG L B, LIN J R, ZHANG W L, LIU S M, WEI Y, MEI P M, WANG Y, LIANG S D. Sterol regulatory element binding protein 1 and its target gene networks. Yi Chuan, 2013, 35(5):607-615. (in Chinese)
[19] ZULKIFLI R M, PARR T, SALTER A M, BRAMELD J M. Regulation of ovine and porcine stearoyl coenzyme A desaturase gene promoters by fatty acids and sterols. Journal of Animal Science, 2010, 88(8): 2565-2575.
[20] 韩立强, 曹菁菁, 付彤, 魏占勇, 王亚宾, 杨国宇. 奶牛硬脂酰辅酶 A去饱和酶基因(SCD)启动子的克隆及活性分析. 农业生物技术学报, 2013, 21(4): 435-440.
HAN L Q, CAO J J, FU T, WEI Z Y, WANG Y B, Yang G Y.Cloning and activity analysis of promoter of bovine stearoyl-CoA desaturase gene(SCD). Journal of Agricultural Biotechnology, 2013, 21(4): 435-440. (in Chinese)
[21] Kadegowda A K G, Bionaz M, Thering B, Piperova L S, Erdman R A. Identification of internal control genes for quantitative polymerase chain reaction in mammary tissue of lactating cows receiving lipid supplements. Journal of Dairy Science, 2009, 92 (5): 2007-2019.
[22] BROWN M S, GOLDSTEIN J L. The SREBP pathway: regulation of cholesterol metabolism by proteolysis of a membrane-bound transcription factor. Cell, 1997, 89(3): 331-340.
[23] 付常振,昝林森,王虹,成功,王洪宝,李耀坤,姜碧杰,高建斌,杨宁. 牛SREBP1基因shRNA序列的筛选及其腺病毒载体的构建与鉴定. 中国农业科学, 2013, 46(23): 5026-5036.
FU C Z, ZAN L S, WANG H, CHENG G, WANG H B, LI Y K, JIANG B J, GAO J B, YANG N. Selection of effective SREBP1 shRNA in cattle and the construction of recombinant adenovirus vector. Scientia Agricultura Sinica, 2013, 46(23): 5026-5036. (in Chinese)
[24] YANG T, ESPENSHADE P J, WRIGHT M E, YABE D, GONG Y, AEBERSOLD R, GOLDSTEIN J L, BROWN M S. Crucial step in cholesterol homeostasis: Sterols promote binding of SCAP to INSIG-1, a membrane protein that facilitates retention of SREBPs in ER. Cell, 2002, 110(4): 489-500.
[25] BARBER M C, VALLANCE A J, KENNEDY H T, TRAVERS M T. Induction of transcripts derived from promoter III of the acetyl-CoA carboxylase-alpha gene in mammary gland is associated with recruitment of SREBP-1 to a region of the proximal promoter defined by a DNase I hypersensitive site. The Biochemical Journal, 2003, 375(2): 489-501.
[26] MA L, CORL B A. Transcriptional regulation of lipid synthesis in bovine mammary epithelial cells by sterol regulatory element binding protein-1. Journal of Dairy Science, 2012, 5(7): 3743-3755.
[27] Seo Y K, Jeon T I, Chong H K, Biesinger J, Xie X H, Osborne T F. Genome-wide localization of SREBP-2 in hepatic chromatin predicts a role in autophagy. Cell Metabolism, 2011, 13(4): 367-375.
[28] Tabor D E, Kim J B, Spiegelman B M, Edwards P A. Identification of conserved cis-Elements and transcription factors required for sterol-regulated transcription of Stearoyl-CoA desaturase 1 and 2. The Journal of Biology Chemistry, 1999, 274(29): 20603-20610.
(责任编辑 林鉴非)
Expression and Localization of Bovine SREBP1 Protein and Regulation of the Transcription of SCD1 Promoter in Bovine Mammary Epithelial Cell
HAN Li-qiang, WANG Yue-ying, WANG Lin-feng, ZHU He-shui, ZHONG Kai, CHU Bei-bei, YANG Guo-yu
(College of Animal Science and Veterinary Medicine, Henan Agricultural University,Key Laboratory of Animal Biochemistry and Nutrition of Ministry of Agriculture, Zhengzhou 450002)
bovine; SREBP1; mammary epithelial cell; subcellular localization; gene regulation
ntia Agricultura Sinica
10.3864/j.issn.0578-1752.2016.24.011
2015-07-29;接受日期:2016-10-17
国家“973”项目(2011CB100802)、国家自然科学基金(31072009)、国家转基因重大专项(2014ZX0801015B)、河南省高等学校重点科研项目(15A230020)、河南省基础与前沿项目(162300410258)
联系方式:韩立强,E-mail:qlhan2001@126.com。通信作者杨国宇,E-mail:haubiochem@163.com
