山西野生黄芩种质资源的RAPD分析
2017-01-06刘晓伶郝建平付琳
刘晓伶,郝建平,付琳
(山西大学生命科学学院,山西太原030006)
山西野生黄芩种质资源的RAPD分析
刘晓伶,郝建平,付琳
(山西大学生命科学学院,山西太原030006)
应用RAPD分子标记技术对7个山西不同产地的野生黄芩试管植株进行了分析,应用SPSS 19.0软件对结果进行了聚类分析。结果表明,5条引物扩增出20条片段,其中,多态性条带17条,多态率为85%;7个野生种质的遗传距离在0~1,可聚为两类,说明7个不同产地的山西野生黄芩之间存在明显差异,具有丰富的遗传多样性。研究结果为山西野生黄芩种质资源的保护和开发、利用以及黄芩优良种质的选育提供了试验依据。
黄芩;试管植株;RAPD;遗传多样性
黄芩(Scutellaria baicalensis Georgi)为唇形科黄芩属多年生草本植物,是我国的传统中药材,也是最常用和用量最大的中药材之一,具有清热燥湿、凉血安胎、解毒等功效[1],其所含的黄芩苷、黄芩素、汉黄芩苷、汉黄芩素等黄酮类衍生物具有抗病原体、抗炎、抗自由基损伤、抗肿瘤、抗过敏、抗氧化、镇痛、保肝、增强免疫功能以及调节中枢神经系统等作用[2-4]。黄芩是山西省的道地药材,全省共有野生黄芩13.52万hm2,家种黄芩2.91万hm2,野生资源分布区域和人工种植面积均位于全国前列[5]。RAPD是常用的遗传标记方法[6-7],通过PCR来反映特异性扩增的DNA片段的多态性,可以快速有效地进行遗传多样性评价[8-12]。冯学锋等[13]和邵爱娟等[14]利用RAPD方法分别对黄芩和并头黄芩样本以及34个不同种源的黄芩进行了种源间亲缘关系的分析。RAPD分子标记技术在北柴胡[15]、拟南芥[16]、太行菊[17]、蓖麻[18-19]、珍珠黄杨[20]、紫花苜蓿[21]等许多植物中也得到了普遍的应用[12-13]。
本研究以山西7个产地的野生黄芩试管植株为材料,应用RAPD技术分析不同种源试管植株的遗传多样性。黄芩种质间的遗传背景及亲缘关系的确定,可为不同种质的鉴定提供了新的试验技术平台,为山西黄芩种质资源的保护和开发、利用以及优良种质资源的选育提供科学依据。而以试管植株为试验材料,可以弥补由于个别野生种质资源分布稀少所引起的数量限制,克服由于野生植株生长年限难以确定所带来的原始误差,以保证试验数据的可比性和可信度。
1 材料和方法
1.1 材料
分别在山西省7个不同产地采集野生黄芩植株(种质来源如表1所示),并应用快速繁殖技术获得试管植株。选取生长30 d长势良好的试管植株的幼嫩叶片作为试验材料。
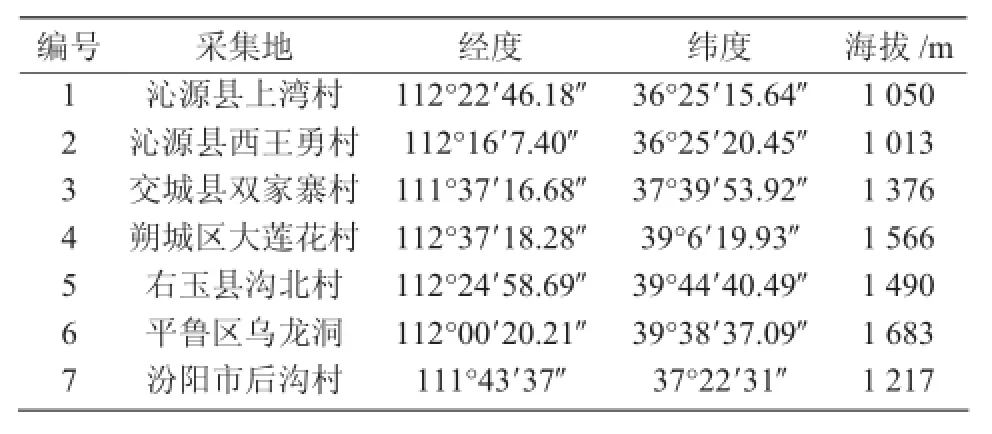
表1 山西野生黄芩试管植株种质来源
1.2 试验仪器
Blue pard人工气候箱(上海一恒科学仪器有限公司),YXQ-LS立式压力蒸汽灭菌锅(上海博迅实业有限公司医疗设备厂),HH-2数显恒温水浴锅(国华电器有限公司),TGL-16G台式离心机(上海安亭科学仪器厂),Eppendorf移液枪,BIOER LifeECO基因扩增仪(杭州博日科技有限公司),DYY-8C型电泳仪(北京市六一仪器厂),ZF-2紫外分析仪(上海嘉鹏科技有限公司)。
1.3 试验试剂
CTAB,β-巯基乙醇,Tris,EDTA,无水乙醇,异戊醇,氯仿和异丙醇等,均为分析纯试剂。
1.4 试验方法
1.4.1 总DNA的提取采用CTAB法提取DNA。取150mg黄芩试管植株叶片置于研钵中,加入1 mL CTAB提取液置冰上研磨,研磨均匀后倒入2 mL EP管中,65℃水浴2 h,10 000 r/min离心10 min,取上清液加氯仿-异戊醇(24∶1)抽提3次,取上清液加入1/10体积NaAc冰浴10 min后再加入等体积异丙醇,置于4℃过夜沉淀DNA,次日取出后12 000 r/min离心10 min,弃上清液,用70%乙醇洗涤DNA沉淀3次,留沉淀自然风干,加20 μL灭菌ddH2O溶解,并根据OD260值将各基因组DNA稀释为40 ng/μL浓度,置于-20℃保存备用。
1.4.2 RCR反应体系及扩增程序PCR扩增反应在20 μL体系中进行,其中含有10×Buffer(含Mg2+)缓冲液2 μL,dNTP 0.63 μL(其中含dATP,dGTP,dTTP,dCTP各10 mmol/L),扩增引物(浓度为10 μmol/L)1 μL,模板DNA稀释液2.5 μL,Taq聚合酶0.3 μL,ddH2O13.57 μL。
PCR反应程序:94℃预变性5 min;94℃变性1 min,34℃复性50 s,72℃延伸2 min,37个循环;72℃8 min补齐。4℃保存备用。
1.4.3 RAPD扩增产物检测取5 μLLoadingBuffer与PCR扩增产物混匀,于含4%的GoldenView的质量分数为1%的琼脂糖凝胶上电泳检测,DNA Marker DL5000作为参照。
1.5 数据统计与分析
将PCR产物在琼脂糖凝胶上同一位置的条带按有带(包括显带和弱带)、无带分别记为“1”和“0”,将试验结果转化为数字形式,用SPSS 19.0软件分析计算7个处理种之间的遗传相似系数及遗传距离,并绘制聚类图。
2 结果与分析
2.1 7种晋产野生黄芩试管植株PCR扩增结果
在查询相关文献的基础上,设计20条引物,经反复筛选,其中,5条引物扩增的条带清晰、多态性良好,可用于RAPD检测。图1、图2为部分引物的扩增结果。
RAPD统计结果表明,5条引物共扩增出20条片段,其中,平行性条带3条,多态性条带17条,多态率为85%(表2)。

表2 山西野生黄芩试管植株的PCR扩增结果
2.2 7种山西野生黄芩种质聚类分析
由表3,4可知,7个不同产地的山西野生黄芩间的遗传距离在0~1。其中,产自汾阳市后沟村的种质与产自朔城区大莲花村、右玉县沟北村、平鲁区乌龙洞的种质之间遗传距离最小,为0;产自沁源县上湾村的种质与产自朔城区大莲花村、右玉县沟北村、平鲁区乌龙洞及汾阳市后沟村的种质之间的遗传距离最大,为1。
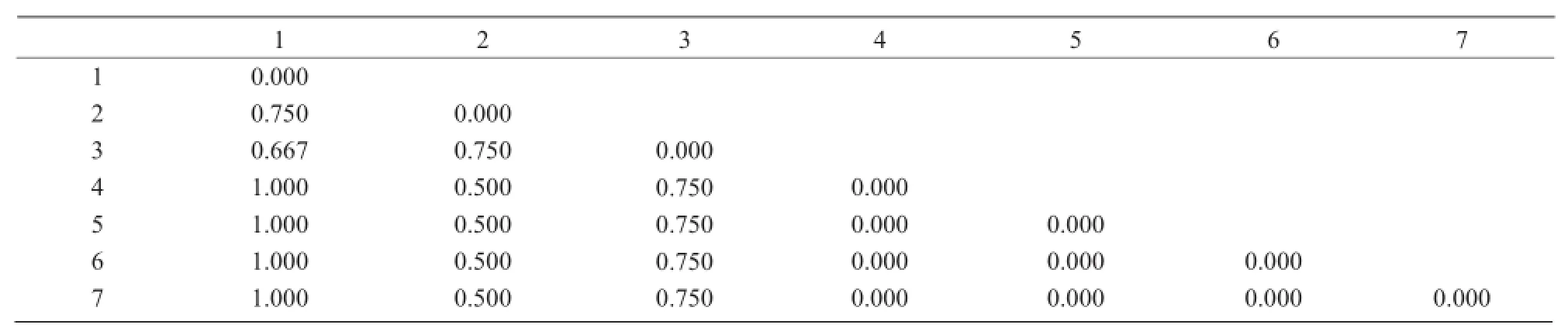
表3 7种山西野生黄芩试管苗植株的遗传距离
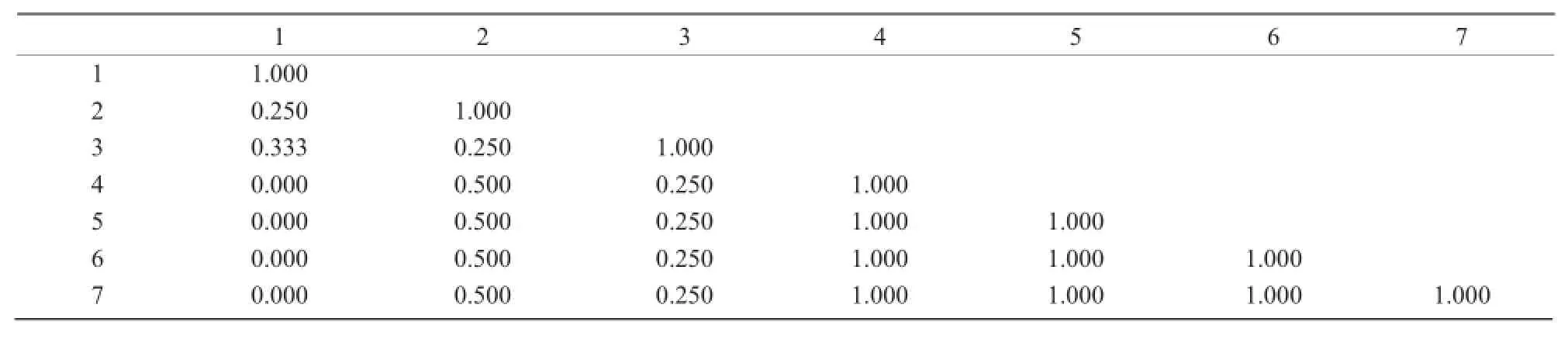
表4 7种山西野生黄芩试管植株的遗传相似系数
由图3可知,7个产地的山西野生黄芩种质可划分为2个类群:产自朔城区大莲花村、右玉县沟北村、平鲁区乌龙洞及汾阳市后沟村的种质首先聚在一起,然后与沁源县西王勇村的种质聚在一起形成类群Ⅰ;产自交城县双家寨村的种质与沁源县上湾村的种质遗传距离较近,首先聚到一起,然后与类群Ⅰ聚到一起形成类群Ⅱ。

3 讨论与结论
7个不同产地的山西野生黄芩试管植株的PCR扩增多态率达到了85%,说明山西野生黄芩具有丰富的遗传多样性,也有着一定程度的相似性。本研究以不同产地的野生黄芩试管植株为试验材料,不受生长季节、生长年限以及气候条件的限制;试管植株生长环境一致,遗传稳定性相对更高,使试验结果更加可靠。
从生长地理位置及环境来看,产自山西省朔州市大莲花村、沟北村、乌龙洞以及产自山西省汾阳市后沟村的4种黄芩种质的遗传距离为0;产自山西省长治市沁源县上湾村和西王勇村的2种黄芩种质的遗传距离为0.750,差异较大;产自山西省吕梁市交城县双家寨村的黄芩种质与产自山西省吕梁市汾阳市后沟村的黄芩种质产地相距较近,但遗传距离为0.750,差异较大。本研究结果表明,山西野生黄芩分布的地理位置与其遗传多样性没有直接的相关性。
[1]国家药典委员会.中华人民共和国药典(一部)[M].北京:中国医药科技出版社,2015:301.
[2]杨全,白音,陈千良.黄芩资源现状及可持续利用的研究[J].时珍国医国药,2006,17(7):1159-1160.
[3]刘金花,张永清,王修奇.不同方法繁殖黄芩药材的产量与质量比较[J].中国中医药科技,2009,16(5):394-395.
[4]李化.黄芩采收规律及其数学模型的研究[D].北京:中国中医科学院,2008.
[5]李树军,陈廷珠,徐国平,等.山西省黄芩蜜源植物分布特点及开花流蜜规律[J].中国蜂业,2014,65(10):30-32.
[6]Williams J G K,Kubelik A R,Livak K L,et al.DNA polymorphisms amplified byarbitraryprimers are useful as genetic markers[J].Nucleic Acids Research,1990(18):6531-6535.
[7]Welsh J,Mclelland M.Finger printing genomes using PCR with arbitraryprimers[J].Nucleic Acids Research,1990(18):7213-7218.
[8]荆玉祥.植物分子生物学:成就与前景[M].北京:科学出版社,1995:95-302.
[9]王华,李渊,郭尚,等.5种食用菌遗传多样性的RAPD分析[J].山西农业科学,2015,43(5):516-517,547.
[10]武青山,马建华,武峻新,等.芹菜种质资源遗传多样性RAPD分析[J].山西农业科学,2013,41(8):774-777.
[11]杨慧珍,牛西午,任志强,等.RAPD标记在林木育种中的应用[J].山西农业科学,2007,35(1):73-76.
[12]车代弟,秦智伟,王金刚.仙客来的种质资源RAPD分析[J].植物研究,2002,22(3):314-317.
[13]冯学锋,胡世林,郭宝林,等.黄芩种群遗传多样性初步研究[J].世界科学技术:中药现代化,2002,4(4):38-43.
[14]邵爱娟,李欣,黄璐琦,等.不同种源黄芩的RAPD分析[J].中国中药杂志,2006,31(6):452-455.
[15]赵瑒,高可青,郝建平,等.晋产北柴胡试管植株的RAPD分析[J].山西大学学报:自然科学版,2013,36(2):267-270.
[16]郭美丽,郝建平,裴雁曦.拟南芥抗盐突变体的RAPD分析[J].西北植物学报,2004,24(8):1514-1516.
[17]赵利新,张安世,刘莹.珍稀濒危植物太行菊遗传多样性的RAPD分析[J].河南农业科学,2014,43(4):106-109.
[18]黄凤兰,赵永,彭木,等.与蓖麻果刺性状连锁的RAPD标记[J].华北农学报,2014,29(1):83-88.
[19]包春光,黄凤兰,朱国立,等.与蓖麻种子大小性状连锁的RAPD分析[J].华北农学报,2015,30(5):108-114.
[20]姜自红.珍珠黄杨RAPD最佳反应体系研究[J].天津农业科学,2015,21(4):15-18.
[21]刘青松,陈立波,李志勇.基因组DNA分子标记在紫花苜蓿遗传多样性研究中的应用[J].华北农学报,2013,28(增刊):36-40.
RAPD Analysis of Germplasm Resources of WildScutellaria baicalensisfrom Shanxi
LIUXiao-ling,HAOJian-ping,FULin
(College ofLife Sciences,Shanxi University,Taiyuan 030006,China)
Toknowthe genetic distance and phylogenetic relationsships amongthe 7 varies oftest-tube plantlets ofwild Scutellaria baicalensis from Shanxi,the paper used the RAPD technique to analyze and make a cluster analysis to the result with the software SPSS 19.0.The results showed that 5 primers amplificated 20 pieces,including17 polymorphic bands,and the polymorphic rate was 85%.The genetic distance among 7 varies of test-tube plantlets of wild Scitellarie baicalensis was from 0 to 1,and it could be divided into two classes.This revealed that there was a obvious difference and the abundant genetic diversity among the test-tube plantlets of wild Scitellarie baicalensis in 7 different regions of Shanxi,which provided experimental basis for genetic resources breeding,artificial cultivation and resources protection.
Scutellaria baicalensis;test-tube plantlet;RAPD;genetic diversity
R282.71
A
1002-2481(2016)03-0288-04
10.3969/j.issn.1002-2481.2016.03.03
2015-08-14
山西省科技攻关计划项目(振东专项)(2014ZD0503)
刘晓伶(1991-),女,山西朔州人,在读硕士,研究方向:药用植物细胞工程。郝建平为通信作者。
