猪流行性腹泻病毒云南流行毒株N基因序列分析
2017-01-06李美荃石於友李华春杨荣丽
宋 聪,刘 宁 ,李美荃,石於友,李华春,高 林 ,杨荣丽 ,姚 俊*
(1.云南农业大学动物科技学院,云南昆明 650201 ;2.云南省巍山县动物疫病预防与控制中心,云南巍山 672400; 3.云南省畜牧兽医科学院云南省热带亚热带动物病毒病重点实验室,云南昆明 650224)
猪流行性腹泻病毒云南流行毒株N基因序列分析
宋 聪1,刘 宁1,李美荃1,石於友2,李华春3,高 林3,杨荣丽3,姚 俊3*
(1.云南农业大学动物科技学院,云南昆明 650201 ;2.云南省巍山县动物疫病预防与控制中心,云南巍山 672400; 3.云南省畜牧兽医科学院云南省热带亚热带动物病毒病重点实验室,云南昆明 650224)
为了解近年来云南省PEDV流行毒株的变异情况,选取了采集自昆明西翥、富源、厂口、 玉溪红塔等地方猪场的不同时间的6份PEDV阳性水样腹泻粪便样品,进行N基因的RT-PCR扩增、测序及序列分析。结果表明,来自同一猪场不同流行时间的TY1、TY2、TY3毒株之间,TY1与TY2毒株N基因核苷酸序列完全相同,仅TY3毒株在第80和第1 238位置处的碱基发生了变异(A→C、C→T),TY3与TY1、TY2的毒株N基因核苷酸序列的同源性达99.8%。云南6个流行毒株TY1、TY2、TY3、YXHT、FYDH及Changkou的核苷酸序列的同源性为99.2%~100%。云南6个流行毒株与2014年德国毒株GER-L00721-2014(Accession: LM645057.1)、法国毒株KR011756-FR001-2014(Accession: KR011756.1)及我国2010年河北毒株JF700126.1-HB-HS-2010(Accession: LM645057.1)的核苷酸序列同源性为98.6%~100%,并同属于一个进化分支。云南6个流行毒株与CV777经典疫苗毒株之间的同源性仅为95.2%~95.5%,存在一定的变异,这或许是云南省猪群经PEDV疫苗免疫后保护率较低的原因之一。
猪流行性腹泻病毒;N基因;序列分析;云南株
猪流行性腹泻病毒(Porcine epidemic diarrhea virus, PEDV)属于冠状病毒科(Coronaviridae)冠状病毒属成员(Coronavirus)之一,在粪便病料中所见的病毒粒子是多形态的,大致呈球形,其平均直径(包括纤突)约130 nm,范围在95 nm~190 nm之间[1]。PEDV基因组全长约为28 kb,为线性单链正股RNA[1-2]。猪流行性腹泻病毒与猪传染性胃肠炎病毒(Porcine transmissible gastroenteritis virus, TGEV)有相似的形态结构,位于病毒内部的基因组转录成亚基因组基因后可以编码几种结构性和非结构性蛋白,其中主要的蛋白包括小膜蛋白(E蛋白,大小为7 ku)、膜蛋白(M蛋白,大小约20 ku~30 ku)、纤突糖蛋白(S蛋白,大小约150 ku~220 ku)、核衣壳蛋白(N蛋白,大小为58 ku),其中,PEDV的结构蛋白中所占比例最大的为N基因所编码的N蛋白,保守性很强且能在宿主细胞中大量表达[3]。另外,除以上主要的蛋白外,猪流行性腹泻病毒内部的基因组5′端还具有1a和1b两大开放阅读框(ORFs),分别编码pp1a和pp1b这两种非结构性蛋白,且可以直接进行基因组复制和转录[2]。其3′端则是非翻译区,末端链接Poly(A)序列[4]。其中间则是包括6个开放阅读框,自5′-3′端依次是编码酶多聚蛋白、S蛋白、ORF3蛋白、E蛋白、M蛋白和N蛋白的基因[4-5]。
由PEDV所引发的疾病称为猪流行性腹泻(Porcine epidemic diarrhea,PED),病毒主要是通过破坏各年龄段猪肠道上皮绒毛细胞而使猪出现呕吐、脱水、腹泻等病理症状,其中尤以哺乳仔猪最易感染[6]。相较于夏季而言,每年的10月底至第2年的2月是流行性腹泻病的传染高发期,此病传染率高、发病力强、流行性广,可以给养猪业造成巨大的经济损失并制约养猪业的发展,是流行于世界范围内的传染性猪病之一[7]。
早在1971年,英国首次发现并报道了猪流行性腹泻[8]。1978年,比利时和英国首次分离到此种病毒并将其命名为猪流行性腹泻病毒(CV777),属冠状病毒科[1]。自那时起,猪流性腹泻多暴发于欧洲。20世纪80年代至90年代,猪流行性腹泻病相继在中国、韩国、日本等亚洲国家发现存在并开始大范围暴发,其导致的仔猪(小于1周龄)死亡率高达30%~80%[9-11]。直到现在,猪流性腹泻仍在亚洲地区横行,时有暴发,对亚洲养猪业造成了巨大的经济损失。一直以来,美洲都未见有关于猪流行性腹泻病的报告,直到2013年5月,美国首次发现并确认猪流行性腹泻病毒的感染,并在2013年-2014年间迅速蔓延至美国的33个州,甚至古巴、加拿大、墨西哥、哥伦比亚、秘鲁等其他美洲国家也相继报道了猪流行性腹泻病疫情[12-12]。截止到2014年底,全球已经有超过30个国家和地区发现并确认有猪流行性腹泻病毒的存在[9-14]。特别是自2010年以来,猪流行性腹泻在我国全国范围再次肆掠,导致大批哺乳仔猪的死亡,有的猪场死亡率甚至高达100%,给我国生猪养殖业造成毁灭性的打击,即使通过给妊娠母猪接种灭活疫苗或者弱毒活疫苗,预防效果均不理想,大部分猪场通过给妊娠母猪返饲本场发病哺乳仔猪粪便或肠内容物后起到较好的预防效果。2010年底以前,PEDV的流行相对弱一些,且仅仅为散发,但2011年以来,包括台湾省在内的很多国家和地区出现了PEDV的变异毒株,新生的哺乳仔猪呈现出严重的水样腹泻及更高的死亡率[15-16]。
近年来云南省生猪养殖业不断遭受到猪流行性腹泻暴发流行的沉重打击,大多数规模化猪场哺乳仔猪死亡率高达100%,给生猪养殖企业及养殖散户造成巨大的经济损失。猪群妊娠母猪即使经过国产或进口灭活疫苗、弱毒活疫苗产品免疫预防,但均以失败告终,反而大大提高了猪场的疫苗成本。部分猪场通过给妊娠母猪返饲本场早期发病哺乳仔猪小肠内容物及粪便病料后,均取得立竿见影的预防效果,虽然预防效果较好,但此种原始的免疫方法存在散毒,以及传播伪狂犬等其他疫病的风险。使用本场PEDV病料返饲妊娠母猪预防PEDV效果理想的事实,足以证明当前使用的基于CV777来源的疫苗毒株诱导产生的中和抗体不能完全中和近年来的PEDV流行毒株,许多国内外研究学者的研究结果也提示当前的流行毒株与历史毒株及CV777疫苗毒株比较均出现了较大的变异,因此,非常有必要弄清云南省当前PEDV流行毒株的变异情况,明确其近年来的PEDV流行毒株与国内外历史毒株及最近几年引起全球暴发流行的变异毒株之间的遗传学关系,为云南省猪流行性腹泻的防控及将来新型疫苗、特异性治疗及预防用生物制品的研发,以及抗PEDV转基因动植物等研究提供基础理论依据。为此,本文对近年来暴发流行于云南省的猪流行性腹泻进行了检测,并选取了相对保守的N基因进行了克隆、测序及序列分析,弄清了云南省PEDV流行毒株与历史毒株及国内外变异毒株的遗传学关系。
1 材料与方法
1.1 材料
1.1.1 样品 2013年7月至2015年4月采集自不同地理位置的云南省6家大规模化养猪场出现水样腹泻症状的哺乳仔猪肠系膜淋巴结或粪便样品9份,经RT-PCR方法检测鉴定,选出6份猪流行性腹泻病毒呈阳性的粪便样品作为N基因测序样品(表1)。
1.1.2 主要试剂和仪器 病毒RNA提取试剂盒(Viral RNA Kit),美国OMEGA生物技术有限公司产品;DNA Marker 2000,天根生物技术(北京)有限公司产品;一步法RT-PCR试剂盒(Prime ScriptTMOne Step RT-PCR Kit),宝生物工程(大连)有限公司产品;PCR 仪 Gene Amp PCR system 9700、梯度PCR仪, ABI(Applied Biosystems Inc.)公司产品。
1.1.3 引物 根据经典毒株CV777基因序列设计一对引物,该引物能够扩增N基因全长序列,扩增全长为1 697 bp,由昆明硕擎生物有限公司合成后,加ddH2O稀释至10 μmol/L(表2)。
1.2 方法
1.2.1 病毒RNA提取 将6份样品按照质量体积比1∶5的比例加入1×MEM后匀浆后,4℃条件下3 000 r/min离心5 min,取上清,按照病毒RNA提取试剂盒说明书进行病毒RNA提取,将提取的RNA存储于-80℃冰箱,备用。
1.2.2 猪流行性腹泻病毒N基因的PCR扩增 RT-PCR反应体系(25 μL)如下:25×Prime script one step enzyme 1 μL,2×One step buffer 12.5 μL,10 μmol/L上、下游引物各1 μL,阳性对照及样品RNA 3 μL,ddH2O 6.5 μL。RT-PCR反应条件为:50℃ 30 min;95℃ 2 min;95℃ 1 min;52℃ 1 min,72℃ 2 min 10 s,35个循环;72℃延伸5 min。RT-PCR反应结束后进行琼脂糖凝胶电泳鉴定。
1.2.3 目的基因片段的切胶回收、纯化、测序及序列分析 RT-PCR产物经切胶回收、纯化后,送昆明硕擎生物有限公司进行测序,所得序列登录NCBI GenBank进行Blast比对分析。
2 结果
2.1 PEDV N基因的PCR扩增结果
利用上、下游引物和RT-PCR技术扩增出6株PEDV云南流行毒株的N基因片段,RT-PCR产物经琼脂糖凝胶电泳后,目的片段大小与预期结果一致,大小约为1 700 bp左右(图1)。
表1 样品来源信息
Table 1 The information of sample sources

样品采集地点Samplelocation样品数量Samplenumbers毒株Strains测序基因Sequencinggene送检时间Time西翥Xizhu3TY1/TY2/TY3N2014.1/2014.4/2015.3富源Fuyuan1FYDHN2014.4厂口Changkou1ChangkouN2013.7玉溪红塔Yuxihongta1YXHTN2015.4
表2 PCR 引物
Table 2 PCR primers
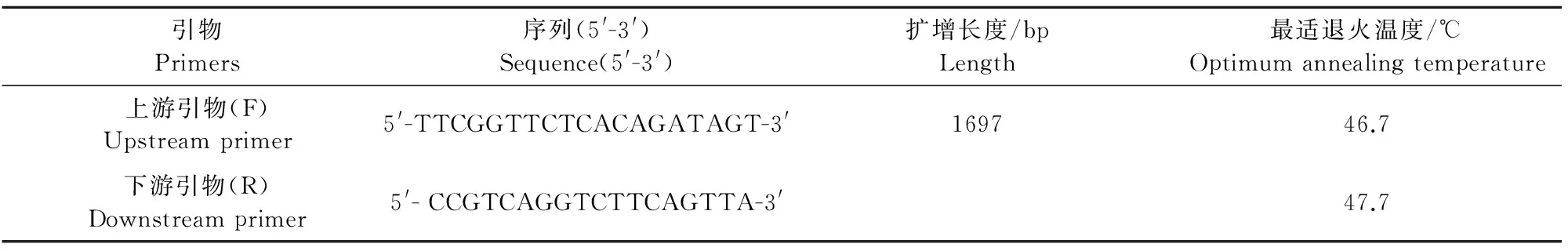
引物Primers序列(5'-3')Sequence(5'-3')扩增长度/bpLength最适退火温度/℃Optimumannealingtemperature上游引物(F)Upstreamprimer5'-TTCGGTTCTCACAGATAGT-3'169746.7下游引物(R)Downstreamprimer5'-CCGTCAGGTCTTCAGTTA-3'47.7

1.空白;2.阴性对照;3.阳性对照;4.样品;M. DNA 标准 DL 2 000
1.Black;2. Negative control; 3. Positive control; 4. Sample;M. DNA Marker DL 2 000
图1 RT-PCR扩增结果
Fig.1 The results of RT-PCR
2.2 PEDV N基因 序列分析
利用DNA Star分析软件对测定出的核苷酸序列进行拼接、校对,获得6株PEDV云南流行毒株完整的N基因片段(大小为1 326 bp),并与NCBI GenBank中国内外一些经典参考毒株进行比对分析。结果显示,来自于同一地方不同时间的TY1、TY2、TY3毒株之间,TY1与TY2毒株核苷酸序列完全相同,仅TY3毒株在第80和第1 238位置处的碱基发生了变异(A→C、C→T),TY3与TY1、TY2的同源性达99.8%。云南6个流行毒株TY1、TY2、TY3、YXHT、FYDH及Changkou的同源性介于99.2%~100%, 云南6个流行毒株与2014年德国毒株(GER-L00721-2014)、法国毒株(KR011756-FR001-2014)及我国2010河北毒株(JF700126.1-HB-HS-2010)介于98.6%~100%。云南6个流行毒株与CV777经典疫苗毒株之间的同源性仅仅介于95.2%~95.5%,存在一定的变异。云南6个流行毒株与2014年德国毒株GER-L00721-2014(GenBank: LM645057.1)、法国毒株KR011756-FR001-2014(GenBank: KR011756.1)及国内河北毒株JF700126.1-HB-HS-2010(GenBank: LM645057.1)同属于一个进化分支(图2)。
3 讨论
猪流行性腹泻病毒属于冠状病毒科冠状病毒属,该病毒形态结构与猪传染性胃肠炎病毒(TGEV)相似,基因组全长约为28kb,为线性单链正股RNA[5-6],该病毒的传播和流行使国内外养猪业蒙受了巨大的损失。2013年,美洲各大猪场流行性腹泻的发现与感染,使得PED的传播范围扩大,变异毒株增多[7-8]。针对目前关于云南PEDV地方流行毒株的变异性研究几乎为空白,所以本研究采集不同地理位置不同流行时期的云南规模化猪场PEDV流行毒株,进行N基因全基因片段的测序和序列分析,以期找出与国内外流行毒株及经典疫苗毒株CV777的异同,为进一步研究PEDV云南地方流行毒株的病原性及变异性提供参考依据。PEDV的N蛋白在病毒感染宿主后进行自我复制时发挥重要作用,编码病毒的核衣壳蛋白[9]。由于N蛋白具有抗原性,所以在PEDV感染早期就能在猪体内产生较高水平的抗体并诱导病猪体内进行细胞免疫反应[10]。因此N蛋白可以作为一个重要指标来快速有效的进行检测PEDV的感染状况。本试验采用RT-PCR 技术扩增了6份PED云南流行毒株的N基因,测序分析结果表明,云南6个流行毒株TY1、TY2、TY3、YXHT、FYDH及Changkou的同源性介于99.2%~100%,变异较小。与经典毒株CV777相比,云南6个流行毒株与CV777经典疫苗毒株之间的同源性仅仅95.2%~95.5%,存在一定的变异,这或许是疫苗免疫后猪群无法获得较为理想保护率的原因。云南6个流行毒株与2014年德国毒株(GER-L00721-2014)、2014法国毒株(KR011756-FR001-2014)及我国2010河北毒株(JF700126.1-HB-HS-2010)介于98.6%~100%,同源性较高,且同处于一个进化分支。欧洲与中国在地里位置上相距遥远,然而德国及法国2014毒株N基因与我国云南流行毒株及我国河北毒株(2010)同源性较其他毒株要高,其中的原因有待进一步研究。综上所述,本研究中的6份PEDV云南流行毒株N基因序列与CV777经典毒株之间存在一定的差异,这可能是云南地区PEDV致病力增强且猪群经PEDV疫苗免疫后保护率较低的重要原因之一。PEDV在云南省的猪群感染和发病必须引起业内人士的高度重视,其内在原因和机理需进一步的研究和探索。

图2 基于PEDV N基因核苷酸序列构建的进化树
Fig.2 Phylogenetic tree based on N gene nucleotide sequence of PEDV
[1] Pensaert M B, de Bouck P. A new coronavirus-like particle associated with diarrhea in swine[J]. Arch Virol,1978,58(3):243-247.
[2] Anastasia N, Douglas M. Distinct characteristcs and complex evolution of PEDV strains, North America, May 2013-February 2014[J].Emerg Infect Dis,2014,20(10):1620-1628
[3] Zhao P D, Bai J, Jiang P,et al.Development of a multiplexTaqMan probe-based real-time PCR for discrimination of variant and classical porcine epidemic diarrhea virus[J].J Virol Meth,2014,206: 150-155.
[4] 孙 振.猪流行性腹泻病毒荧光定量PCR方法和间接ELISA方法的建立与初步应用[D].山东泰安:山东农业大学硕士学位论文,2014:1-59.
[5] Brian D A, Baric R S. Coronavirus genome structure and replication [J].Cur Topi Microbiol Immunol,2005,287:1-30.
[6] Wang X P,Niu B B,Yan H,et al.Genetic properties of endemic Chinese porcine epidemic diarrhea virus strains isolated since 2010[J].Arch Virol,2013,158(12):2487-2494.
[7] 何启盖. 猪腹泻病的鉴别诊断与防控措施[J].兽医导刊,2011(12) : 22-24.
[8] Wood E N. An apparently new syndrome of porcine epidemic diarrhoea[J]. Arch Virol,1977,100:243-244.
[9] Takahashi K,Okada K,Ohshima K. An outbreak of swine diarrhoea of a newtype associated with coronavirus-like particles in Japan[J]. Jap J Vet Sci,1983,45: 829-832.
[10] Kweon C H, Kwon B J, Jung T S, et al.Isolation of porcine epidemic diarrhea virus infection in Korea[J]. J Vet Res,1993,33:249-254.
[11] Sozzi E, Luppi A, Lelli D,et al.Comparison of enzyme-linked immunosorbent assay and RT-PCR for the detection of porcine epidemic diarrhoea virus[J].Res Vet Sci,2010,88(1):166-168.
[12] Stevenson G W, Hoang H, Schwartz K J, et al. Emergence of porcine epidemic diarrhea virus in the United States: clinical signs, lesions, and viral genomic sequences[J]. J Vet Diagn Invest,2013,25(5): 649-654.
[13] 翁善钢.2014年国际猪病疫情回顾-猪流行性腹泻在全球的流行与传播[J].肉类工业,2015(2):49-50.
[14] 田建兴.猪流行性腹泻病毒实时荧光定量PCR检测方法的建立及应用[D].江苏南京:南京农业大学硕士学位论文,2009:1-59.
[15] Tian Y. Molecular characterization and phylogenetic analysis of new variants of the porcine epidemic diarrhea virus in Gansu, China in 2012[J]. Viruses, 2013,5(8):1991-2004.
[16] Huang Y W, Dickerman A W. Origin, evolution and genotyping of emergent porcine epidemic diarrhea virus strains in the United States[J]. Mbio, 2013,4(5):e00737-13.
Sequence Analysis of N Gene of Porcine Epidemic Diarrhea Virus Epidemic Strains in Yunnan Province
SONG Cong1, LIU Ning1, LI Mei-quan1, SHI Yu-you2, LI Hua-chun3, GAO Lin3, YANG Rong-li3, YAO Jun3
(1.CollegeofAnimalScienceandTechnology,YunnanAgriculturalUniversity,Kunming,Yunnan,650201,China;2.WeishanCenterforPreventionandControlofAnimalDisease,Weishan,Yunnan,672400,China;3.YunnanTropicalandSubtropicalAnimalVirusDiseasesLaboratory,YunnanAnimalScienceandVeterinaryInstitute,Kunming,Yunnan,650224,China)
In order to understand the variation of PEDV epidemic strains in Yunnan Province in recent years, six PEDV positive stool samples, which were collected from four large scale pig farms of different geographical locations and different time, were selected to do RT-PCR, sequencing and sequence analysis. The results showed that nucleotide sequence identity of N gene of TY1, TY2 and TY3 strains was 99.8%, and all this three PEDV strains were collected from the same pig farm in different time. Among them, nucleotide sequence identity of TY1 and TY2 stains was identical, only two mutations had occurr at the 80th and 1238th base pair of N gene sequence of TY3 strain (A→C,C→T).The TY1, TY2, TY3, YXHT, FYDH, Changkou stains of PEDV collected from Yunnan shared 99.2%-100% nucleotide identity with each other. The six Yunnan epidemic strains and three reference strains of German strain GER-L00721-2014(Accession: LM645057.1), French strain KR011756-FR001-2014(Accession: KR011756.1)and Chinese strain JF700126.1-HB-HS-2010(Accession: LM645057.1)shared 98.6%-100% identity, and they were belong to the same evolutionary branch. The nucleotide identity of N gene of the six PEDV Yunnan strains and traditional PEDV live vaccine CV777 strain was 95.2% to 95.5%. There were some variation between the six PEDV Yunnan epidemic strains and CV777 vaccine strain. This might be one of the reasons why the efficiency of immune protection with CV777 vaccine in pig herds in Yunnan is low.
PEDV; N gene; sequence analysis;Yunnan strain
2015-12-08
云南省科技厅重大科技专项(2012ZA017)
宋 聪(1989-),女,山东济宁人,硕士研究生,主要从事猪的营养与免疫基础研究。 *通讯作者
S852.659.6
A
1007-5038(2016)05-0015-05
