高粱苯丙氨酸解氨酶(SbPAL)编码蛋白的生物信息学分析
2017-01-05张晓瑛刘宝玲郝青婷薛金爱
张晓瑛,刘宝玲,郝青婷,薛金爱
(1.山西省农机局,山西太原030002;2.山西农业大学农学院,山西太谷030801)
高粱苯丙氨酸解氨酶(SbPAL)编码蛋白的生物信息学分析
张晓瑛1,刘宝玲2,郝青婷2,薛金爱2
(1.山西省农机局,山西太原030002;2.山西农业大学农学院,山西太谷030801)
高粱是世界上一种重要的粮食作物,具有耐寒耐高温等优良性质,挖掘高粱抗病基因(如PAL),在基因水平上进行遗传改良,提高高粱对贫瘠土地的耐受性仍是研究热点之一。以高粱的苯丙氨酸解氨酶基因(SbPAL)为研究对象,运用生物信息学方法对其编码蛋白的结构、理化性质及功能结构域进行分析,以期揭示苯丙解氨酶的基本信息。结果表明,高粱SbPAL基因的编码序列(Codingsequences,CDS)全长为2 145 bp,编码714个氨基酸;高粱SbPAL蛋白主要位于细胞基质中,二级结构以无规则卷曲和α-螺旋为主,三级结构预测其为同源四聚体结构,由4个相同亚基对称性组成,各亚基之间由配体DTT共价连接。多序列比对结果表明,高粱SbPAL蛋白与拟南芥、水稻、葡萄、烟草等蛋白的同源性较高,为83.31%,SbPAL基因与玉米、水稻和大麦有着较近的亲缘关系,PAL在这些作物中主要参与木质素和植保素的合成。此分析结果将为后续进一步研究SbPAL对木质素、植保素等合成的分子机制与抗病性关系奠定一定的理论基础。
高粱;苯丙氨酸解氨酶;木质素;抗病性;生物信息学
植物的抗病性在一定程度上是植物与使其致病的病原微生物在长期的协同进化环境中相互适应和选择所形成的一种可遗传的特性[1]。植物受到病原物的侵染后,会产生一定的自身防御反应和生理变化,包括合成并积累木质素等抗病物质,而木质素是苯丙烷类代谢途径中的产物之一,与植物的抗病性密切相关。苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL,EC 4.3.1.24)是一种主要存在于细胞质能够催化L-苯丙氨酸非氧化脱氨生成反式肉桂酸(E-cinnamic acid,CA)的限速酶,也是苯丙烷类物质合成途径中的第1个关键酶[2-4]。反式肉桂酸是植保素、木质素、类黄酮、花青素及某些酚类等生物合成所通用的前体[5-6]。有研究发现,由苯丙氨酸解氨酶调控生成的次生代谢产物还影响着植物的生长发育、色素合成等代谢过程[7]。此外,苯丙氨酸途径还被用来减少细胞壁木质素的合成,以此将大量生物质转化为更多的生物燃料[8]。因此,苯丙氨酸解氨酶在植物病理学及其抗逆方面都有着非常重要的研究地位[9]。目前,在很多植物中发现,并克隆研究了保守性较强的PAL基因,例如毛果杨(Populus trichocarpa)[10]、水稻(Oryza sativa)[1]、黄瓜(Cucum issativus)[11]、甜樱桃(Prunus avium)[12]、葡萄(Vitis vinifera)[13]、烟草(Nicotiana tabacum)[14]和大豆(Glycine max)[15]。
高粱(Sorghum bicolor L.)是世界上一种重要的耐旱性粮食作物,有着优良的耐旱耐高温性[16]。由于不同环境中存在的病虫害会对高粱的产量和品质造成巨大威胁,因此,研究高粱抗病害的分子机制和遗传改良迫在眉睫,而作为控制与抗病性相关的次生代谢产物合成的PAL防御基因则可被优选为良好的调控靶点。近几年来,随着高粱的基因组[17]和转录组[18]测序的完成,利用生物信息学方法从序列出发,经过先预测后验证的方法来摸索具体分子机制的突破口已经成为研究热点。
本研究利用生物信息学的方法,对高粱SbPAL基因的核苷酸以及相应氨基酸序列的理化性质、活性位点、结构特征和系统发育等方面进行了预测性分析,旨在为今后深入研究高粱SbPAL蛋白的具体催化特性及其植物病害胁迫的分子机制提供一定的理论依据。
1 材料和方法
1.1 数据来源
高粱SbPAL基因cDNA序列及其蛋白序列(Genbank号:NC_012873/XP_002454200)及另外13种植物的PAL蛋白序列均来源于NCBI(http: //www.ncbi.nlm.nih.gov/)中的Genbank数据库,这13种植物的蛋白序列分别为:水稻(Oryza sativa)(AAO72666)、陆地棉(Gossypiumhirsutum)(AER12109)、玉米(Zea mays)(NP_001105334)、槿麻(Hibiscus cannabinus)(AFN85669)、紫苏(Perilla frutescens)(AEZ67457)、黄瓜(Cucumis sativus)(AER58180)、马铃薯(Solanumtuberosum)(AGT63063)、葡萄(Vitis vinifera)(AEX32790)、野生大豆(Glycine soja)(ACT32033)、拟南芥(Arabidopsis thaliana)(AAC18870)、蓖麻(Ricinus communis)(AGY49231)、烟草(Nicotiana tabacum)(AAA34122)、大麦(Hordeum vulgare)(BAJ97194)。
1.2 数据分析方法
用ExPasy网站提供的ProtParam(http://web.expasy.org/protparam/)工具分析蛋白理化性质。通过在线工具Target P 1.1 Server(http://www.cbs.dtu.dk/services/TargetP-1.1/)来查看该蛋白的亚细胞定位;对该酶蛋白的跨膜结构和疏水性分析分别使用在线网站TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)和ProtScale(http://web.expasy.org/protscale/);通过使用Conserved Domains网站(http://www.ncbi. nlm.nih.gov/Structure/cdd/wrpsb.cgi)来分析该蛋白是否具有典型的苯丙氨酸解氨酶特征结构;使用SWISS-MODEL网站(http://swissmodel.expasy.org/)来预测SbPAL的三级结构进行同源建模,3D模型在Rasmol软件查看。利用Genedoc软件对高粱SbPAL蛋白和另外13种蛋白序列进行同源性比对;利用MEGA7.0软件将高粱SbPAL蛋白与13条来自不同植物的PAL蛋白进行Clustal W多序列比对,并生成.meg格式的比对文件。然后,采用邻接法(Neighbor-Joining,N-J)在.meg比对文件基础上构建无根系统发育树,自举检验值设为1 000个循环,采用p-distance模式。
2 结果与分析
2.1 高粱SbPAL蛋白序列的理化性质分析
高粱SbPAL基因的编码序列全长为2 145 bp,利用在线开放阅读框ORF Finder工具,其开放阅读框(ORF)长2 144 bp,得到的氨基酸序列全长为714 aa,起始密码子和终止密码子分别为ATG和TGA。高粱SbPAL基因编码的蛋白含有C,H,N,O和S共5种原子,且分子式为C3375H5436N952O1030S31,相对分子质量为76.82 kD,理论等电点为6.05,摩尔消光系数为37 590,肽链长为671 aa,酸性氨基酸有81个,碱性氨基酸为73个,总之,带电荷的氨基酸在总氨基酸占比为21.57%,含量较高的氨基酸有5种,即Ala,Leu,Val,Cly,Glu,其所占比例分别为12.5%,10.2%,7.4%,8.3%,8.0%。该蛋白不稳定指数为33.52,在生理条件为稳定蛋白(一般小于40视为稳定)。
2.2 高粱SbPAL蛋白的基本结构分析
经TargetP 1.1分析,高粱SbPAL蛋白位于细胞质基质中,没有任何转运肽(cTP:0.087<0.73,mTP:0.163<0.86,SP:0.045<0.43。前者数值为预测值,后者数值为阈值)。通过TMHMM工具进行跨膜分析,该蛋白不存在跨膜现象,也没有信号肽剪切位点(图1),说明该蛋白在细胞质核糖体上合成后直接留到了细胞质中,并发挥其催化功能;用Protscale网站分析其疏水性(图2),此蛋白主要有2个较长的疏水区,氨基酸位置分别为150~214 aa和256~291 aa,此外还有一些较短、数目较多的疏水区,总体疏水区域不是很明显,与无跨膜区相一致,其位于SbPAL蛋白核心处,共同组成酶催化中心,进行催化反应。此肽链在280位置亮氨酸(Leu)处有最高得分值(2.711),即疏水性最强;在483位置谷氨酰胺(Gln)处有最低得分值(-2.578),亲水性较强。
2.3 高粱SbPAL蛋白序列的高级结构分析
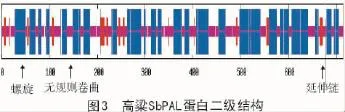
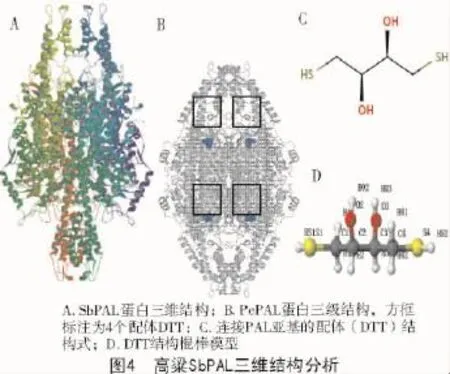
通过对高粱SbPAL氨基酸序列的二级和三级结构进行预测性分析发现,其二级结构(图3)中主要有α-螺旋和无规则卷曲,分别占50.56%和42.58%,而延伸片段占比很少,仅为6.86%。通过在线网站Swiss-model工具对高粱SbPAL三级结构进行同源建模,预测到的三级结构如图4所示,使用欧芹(Petroselinum Crispu)苯丙解氨酶(PcPAL)蛋白作为模板[19],该蛋白与高粱SbPAL蛋白序列相似性为75.99%,且保守序列包括范围为23~714 aa,覆盖度为99%。利用X-射线晶体衍射发现该蛋白有同源四聚体,由4个相同的亚基组成,这些亚基通过与4个配体(2,3-二羟基-1,4-二硫代丁烷,简称DTT)形成共价键而连接到一起。这些配体有着小分子结构(图4-C,4-D),两端各含一个巯基(-SH),中间含2个羟基(-OH),而巯基能与亚基上的巯基形成二硫键,维系着亚基之间的稳定结构,而羟基的亲水性,则可以使各亚基之间保持着一定的空隙,方便底物分子结合进入催化中心,从而更好地发生化学反应。这些连接到一起的亚基上的部分氨基酸共同组成酶与底物结合中心和催化中心,催化着L-苯丙氨酸断裂C-N键,从而非氧化脱氨基形成E-肉桂酸。对比图4-A和图4-B三级结构,高粱的SbPAL蛋白也为同源四聚体,相同的亚基对称性排列,其结构外形(呈蝶形)与欧芹PcPAL蛋白(呈椭圆形)略有差别,但其催化中心和与配体结合部位大致相似,配体与4个亚基结合部位均位于同源四聚体的中心部位(图4-B)。
2.4 高粱SbPAL基因的氨基酸多序列比对分析
使用Genedoc软件对高粱SbPAL与另外13种PAL蛋白进行多序列比对分析可知,这些植物的PAL蛋白与高粱SbPAL蛋白均具有较高的同源性,序列一致性可达到83.31%。可见,PAL蛋白的功能区极其保守,但是肽链的N端(图5)在序列长度和氨基酸组成上有着很大的差异,且因物种不同而序列差异大小都不相同。推测可能由于苯丙烷类合成途径是植物从初生代谢转向次生代谢过程中重要通路,PAL作为苯丙烷类次生代谢产物合成过程中的首要关键酶,其序列的稳定遗传性和物种间进化趋同性明显表现出其结构与功能之重要性[20]。


另外,多序列比对中发现,高粱SbPAL蛋白也和其他物种蛋白一样,在215 aa处有连续的Ala-Ser-Gly三肽序列,此序列能够通过自身脱水环化形成3'5-二氢-5-亚甲基-4H-咪唑-4-酮(MIO)结构,此结构在水稻OsPAL蛋白中紧邻底物结合位点,负责催化E-肉桂酸酯/氨的生成[21-22],且此结构与PAL和HAL(组氨酸解氨酶)中的典型MIO结构相同。
2.5 高粱SbPAL蛋白系统进化分析
进化树用MEGA 7.0软件在Clustal W多序列比对基础上,运用邻接法(Neighbor-Joining,即N-J法)设置,重复循环数为1 000,构建系统发育树(图6)。依据系统发育关系,可大体划分为A,B,C共3类。在C组,高粱SbPAL蛋白与同属禾本科的玉米、水稻和大麦的PAL蛋白聚在一起,且有的聚类值达到100,说明这些PAL蛋白更多参与着木质素及植保素等的合成,在生物胁迫和非生物胁迫下对其抗逆性(包括抗病)起着至关重要的作用。而A,B组PAL蛋白可能在类黄酮、花青素(如紫苏)和多酚类物质(如马铃薯、烟草)等代谢过程中起着重要调控作用。植物间这种序列、结构和功能分化如此明显的现象与各物种在漫长进化过程中适应不同环境的能力有着密切的联系。
3 讨论与结论


苯丙氨酸解氨酶主要存在于植物和真菌中,是苯丙烷类代谢途径中的第1个限速酶,催化L-苯丙氨酸转化为E-肉桂酸,从而形成木质素、植保素和酚类等一系列抗病性次生代谢产物。有研究表明,当植物受到病原体侵染时,PAL基因的表达量会显著增加,抗病代谢产物也会相应积累。分析表明,高粱SbPAL蛋白相对分子量为76.82 kD,分子量明显小于正常的PAL蛋白(一般为220~330 kD)[23],其活性最适pH值为6.05,且含有大量的中性小分子氨基酸,如Ala,Gly和Leu,使得SbPAL蛋白分子量很小。苯丙氨酸解氨酶(PAL)属于芳香环解氨酶类I_like超家族的成员之一,与组氨酸解氨酶(PAL)催化特点极其相似,都催化β-消除反应及非氧化脱氨,且其只有在同源四聚体形式时才具有催化活性。该酶的4个活性位点由3个不同亚基上的氨基酸共同组成,且其所在部位形状不断发生适应性变化,使得各亚基能够相互配合,有条不紊地与底物结合,催化发生化学反应,然后再释放,重新接收新的底物。这些关键氨基酸在锦紫苏中为处于不同位置的酪氨酸(Y103)和甲硫氨酸(M335和M545),说明SsPAL和PcPAL类似,均为功能蛋白[24],而高粱SbPAL蛋白的三级结构和欧芹PcPAL结构极为相似,很有可能也有类似的流动型酪氨酸和甲硫氨酸,并且参与着重要的催化功能。本研究对SbPAL单个蛋白所进行的一系列生物信息学分析,将为后期继续研究高粱苯丙氨酸解氨酶家族基因通过调控植保素和木质素等的含量,以间接参与高粱抗病性等研究提供一系列理论基础。从序列角度进一步解析基因及其蛋白结构特征,以期找到重要的靶位点,从而在分子水平上对高粱的抗病性进行遗传改良,获得抗病虫害植株,增加其耐逆性,最终提高作物产量。因此,本研究所做的一系列上游分析对后期实验理论指导有着重要的参考价值。
[1]康晓慧,雷桅,张梅.水稻苯丙氨酸解氨酶的生物信息学分析[J].湖南师范大学自然科学学报,2010,33(4):89-94.
[2]吕晓玲,孙雪梅,王芳,等.紫苏苯丙氨酸解氨酶基因片段克隆及序列分析[J].中国农学通报,2011(9):347-351.
[3]Shi R,Shuford C M,Wang J P,et al.Regulation of phenylalanine ammonia-lyase(PAL)gene family in wood forming tissue of Populus trichocarpa[J].Planta,2013,238:487-497.
[4]Vogt T.Phenylpropanoid biosynthesis[J].Mol Plant,2010,3(1):2-20.
[5]Babst B A,Harding S A,Tsai C J.Biosynthesis of phenolic glycosides from phenylpropanoid and benzenoid precursors in Populus [J].Chem Ecol,2010,36:286-297.
[6]杜何为,蔡荣,徐树德,等.克隆植物苯丙氨酸解氨酶基因通用引物的发掘与应用[J].河北农业科学,2010(7):45-47,76.
[7]侯鹏,粱冬,张卫国,等.苯丙氨酸解氨酶基因家族在大豆中的时空表达研究[J].作物杂志,2016(2):57-62.
[8]Cynthia L C,Antoine P,Patrick F D,et al.Effects of phenylalanine ammonia lyase(PAL)knockdown on cell wall composition,biomass digestibility,and biotic and abiotic stress responses in Brachy podium[J].Journal of Experimental Botany,2015,66(14):4317-4335.
[9]Wang J P,Naik P P,Chen H C,et al.Complete proteomic-based enzyme reaction and inhibition kinetics reveal how monolignol biosynthetic enzyme families affect metabolic flux and lignin in Populus trichocarpa[J].Plant Cell,2014,26(3):894-914.
[10]JongF D,HanleyS J,Beale MH,et al.Characterisation of the willow phenylalanine ammonia-lyase(PAL)gene family reveals expression differences compared with poplar[J].Phytochemistry,2015,117:90-97.
[11]董春娟,李亮,曹宁,等.苯丙氨酸解氨酶在诱导黄瓜幼苗抗寒性中的作用[J].应用生态学报,2015,26(7):2041-2049.
[12]Wiersma P A,Wu Z A.Full Length cDNA for phenylalanine ammonia-lyase cloned from ripe sweet cherry fruit[J].Plant Physiol,1998,118(3):1102.
[13]孙润泽,张雪,成果,等.葡萄苯丙氨酸解氨酶基因家族的全基因组鉴定及表达分析[J].植物生理学报,2016(2):195-208.
[14]Reichert A I,He X Z,Dixon R A.Phenylalanine ammonia-lyase(PAL)from tobacco(Nicotiana tabacum):characterization of the fourtobacco PAL genes and active heterotetrameric enzymes[J]. BiochemJ,2009,424(2):233-242.
[15]Frank R L,Vodkin L O.Sequence and structure of a phenylalanine ammonia-lyase gene from Glycine max[J].DNA Seq,1991,1(5):335-46.
[16]邢慧清,黄赛楠,张云鹤,等.分子标记在高粱抗病虫基因定位上的研究进展[J].黑龙江农业科学,2012(9):139-142.
[17]Paterson A H,Bowers J E,Bruggmann R,et al.The Sorghum bicolor genome and the diversification of grasses[J].Nature,2009,457:551-556.
[18]Li J,Wang L,Zhan Q,et al.Sorghum bmr6 mutant analysis demonstrates that a shared MYB1 transcription factor binding site in the promoter links the expression of genes in related pathways [J].Funct Integr Genomics,2013,13(4):445-453.
[19]Ritter H,Schulz G E.Structural basis for the entrance into the phenylpropanoid metabolism catalyzed by phenylalanine ammonia-lyase[J].Plant Cell,2004,16(12):3426-3436.
[20]关巍.彩色马铃薯花色苷的积累与合成酶的关系研究[D].哈尔滨:东北农业大学,2013.
[21]Langer B,Langer M,Rétey J.Methylidene-imidazolone(MIO)from histidine and phenylalanine ammonia-lyase[J].Advances in Protein Chemistry,2001,58:175-214.
[22]Mac Donald M J,D'Cunha G B.A modern view of phenylalanine ammonia lyase[J].Biochemistry and Cell Biology,2007,85(3):273-282.
[23]欧阳光察,薛应龙.植物苯丙烷类代谢的生理意义及其调控[J].植物生理学通讯,1988(3):9-26.
[24]Zhu Q L,Xie X R,Lin H X.Isolation and functional characterization ofa phenylalanine ammonia-lyase gene(Ss PAL1)from coleus(Solenoste mon scutellarioides(L.)Codd)[J].Molecules,2015,20(9):16833-16851.
Bioinformatics Analysis of Phenylalanine Ammonia-lyase(SbPAL)Protein-coding in Sorghum
ZHANGXiaoying1,LIUBaoling2,HAOQingting2,XUE Jin'ai2
(1.Agricultural Bureau of Shanxi Province,Taiyuan 030002,China;2.College of Agronomy,Shanxi Agricultural University,Taigu 030801,China)
Sorghum is an important food crop in the world,it can resist cold as well as hot temperature and has other excellent properties.Mining resistance genes(such as PAL)and geneticly modifying it at the gene level in sorghum to improve its tolerance of sorghum on marginal land is still one of the hot topics in research field.In this paper,sorghum phenylalanine ammonia-lyase gene(SbPAL)was seen as the subject and was studied by using the method of bioinformatics to analyze its structure of the encoded protein, physicochemical properties and functional domains,to reveal the basic information of phenylalanyl ammonia-lyase.The results showed that coding sequence of sorghum SbPAL gene(CDS)had a total length of2 145 bp,encoding 714 amino acids.Sorghum SbPAL protein was mainly located in the extracellular matrix.Its secondary structure concluded random coil and α-helix and the three-dimensional structure which had a homologous tetramer.Four identical subunits made up the symmetry structure which was connected by covalent ligands DTT between two subunits.The multiple sequence alignment results showed that they had a closer relationship between sorghum SbPAL and other protein of various species,such as Arabidopsis,rice,grapes,tobacco,and high homology whose value was 83.31%.The SbPAL gene shared closer relationship between maize,rice and barley in which PAL was mainly involved in the synthesis of lignin and phytoalexin.The result of this analysis will lay a theoretical foundation for further study of the molecular mechanisms of SbPAL involving in synthese of lignin and phytoalexins related to disease resistance.
sorghum(Sorghum bicolor L.);phenylalanine ammonia-lyase(SbPAL);lignin;disease resistance;bioinformatics
S514
A
1002-2481(2016)11-1584-05
10.3969/j.issn.1002-2481.2016.11.02
2016-06-14
山西农业大学引进人才科研启动项目(2014ZZ06)
张晓瑛(1981-),女,山西五寨人,农艺师,硕士,主要从事昆虫天敌与害虫综合防治研究工作。薛金爱为通信作者。
