小麦UROS基因的可变剪接
2016-12-30郎丹莹吕庚寅梁广旺李瑞航郭晓光郭蔼光
郎丹莹,吕庚寅,梁广旺,李瑞航,郭晓光,徐 虹,2,郭蔼光,2
(1.西北农林科技大学生命科学学院,陕西杨凌 712100; 2.旱区作物逆境生物学研究国家重点实验室,陕西杨凌 712100)
小麦UROS基因的可变剪接
郎丹莹1,吕庚寅1,梁广旺1,李瑞航1,郭晓光1,徐 虹1,2,郭蔼光1,2
(1.西北农林科技大学生命科学学院,陕西杨凌 712100; 2.旱区作物逆境生物学研究国家重点实验室,陕西杨凌 712100)
可变剪接是一种重要的基因表达调控机制,对植物的发育、生理、代谢、信号转导以及外界逆境的响应等多个过程都有调控。为进一步探讨小麦基因的可变剪接机制以及深入研究小麦UROS基因的功能,以返白系、矮变1号和中国春三个普通六倍体小麦品种为材料,克隆了尿卟啉原Ⅲ合成酶基因(UROS)的gDNA和cDNA序列,并利用生物信息学技术研究了小麦叶片中UROS基因的可变剪接方式。结果表明,根据在线小麦基因组数据库,将克隆到的UROS基因组DNA序列分别定位在4BL和4DL两个基因位点,命名为 UROS-4B和 UROS-4D。从三个品种中克隆得到大约50个不同的UROS基因cDNA序列,通过在线预测和与基因组序列比对,发现UROS基因存在可变剪接,并鉴定出两个UROS基因位点各有一个标准剪接转录本和两个非标准剪接转录本。可变剪接方式以内含子保留为主,还有可变的供体剪接位点和可变的受体剪接位点,品种间的剪接方式不完全相同。由不同转录本预测编码的多肽氨基酸序列也存在差异,两个基因位点选取的六个转录本所编码的多肽有12个氨基酸位点的变异。
小麦;返白系;尿卟啉原Ⅲ合成酶;可变剪接;标准剪接与非标准剪接
可变剪接也被称为选择性剪接,是高等真核生物细胞中广泛存在的一种基因表达调控机制。在mRNA的转录后加工过程中,通过剪接位点的可变选择,一个基因可以产生多个mRNA的转录本,翻译为多种多肽序列,增加了蛋白质的多样性,并影响mRNA的稳定性[1-4]。一些剪接方式导致编码区的提前终止,引起无义mRNA降解[5]。可变剪接的过程是由剪接体来完成,剪接体是一个大的RNA-蛋白复合体,由5个小RNA分子和大约180个具有不同功能的蛋白亚基组成[6]。1977年,Berget等[7]和Chow等[8]在对腺病毒2型mRNA的研究过程中发现,基因内含子序列可以在转录后加工过程中被移除,当时称之为特定重排。1981年,Rosenfeld 等[9]首次报道在高等哺乳动物小鼠降血钙素基因中发现了多种RNA分子。现有研究表明,在人体中大约有95%以上的基因都存在可变剪接[10]。1989年,在拟南芥和菠菜中发现1,5-二磷酸核酮糖羧化加氧酶活化酶(Rubisco activase)基因存在可变剪接[11]。二十几年的研究表明,可变剪接也是植物基因表达的重要调控方式[4],涉及到植物的很多生理代谢过程、信号转导以及对外界生物和非生物胁迫的响应[12-17],大约60%的植物基因都存在可变剪接[12]。对拟南芥[18-24]、玉米[25-28]、水稻[29]、二穗短柄草[30]、马铃薯[31]、山葡萄[32-33]等的研究表明,植物中主要有5种前体mRNA的剪接方式,其中最多的是内含子保留(intron rention,IR)[5,15-16,29,34]。
I型淀粉分支酶基因 Sbe1是最早报道的小麦可变剪接基因[35],基因全长5.4 kb,大约有14个外显子,有三个不同的转录本,在胚乳的不同阶段表达。此后,分别有光周期响应基因 Ppd-B1[36]、干旱胁迫响应转录因子编码基因 DREB2[37-38]、多酚氧化酶基因 Ppo-A1[39]、ε-环化酶基因ε-LCY和八氢血红素合成酶基因 Psy-A1的相关研究报道[40],这些基因涉及到了小麦的发育、生理生化代谢、逆境胁迫响应等多个过程,也揭示出小麦中基因的可变剪接具有普遍性。
尿卟啉原Ⅲ合成酶(Uroporphyrinogen Ⅲ synthase,UROS)是天然四吡咯化合物生成过程中的关键酶之一,它催化线性的四吡咯底物羟甲基胆素经过一次重要的分子内重排形成天然四吡咯化合物的共同前体——尿卟啉原Ⅲ[41-42]。1975年Higuchi等[43]开展了小麦UROS的分离鉴定工作。笔者所在实验室在研究小麦低温诱导阶段性白化突变材料返白系的白化机理过程中发现,返白系叶绿素合成途径中从胆色素原至尿卟啉原Ⅲ位点受阻[44-47],进而对小麦UROS分离纯化[48],并用同源比对和电子克隆的方法获得UROS基因的cDNA序列[49]。本研究克隆了返白系、矮变1号和中国春三个普通六倍体小麦UROS基因的gDNA与cDNA序列,利用生物信息学软件探索不同染色体位点UROS基因对应的可变剪接方式,以期为进一步研究小麦基因的可变剪接机制以及深入研究小麦UROS基因的功能提供参考依据。
1 材料与方法
1.1 供试材料及其处理
以普通六倍体小麦品种中国春、返白系及其对照亲本矮变1号为材料,种子最初由郭蔼光教授惠赠,由本实验室长期保存。
小麦种子先用70%乙醇溶液消毒1 min,再用10% NaClO溶液消毒10 min,无菌水冲洗3次,浸泡24 h出芽后,移植在营养钵中,在25 ℃下培养10 d,至三叶期,剪取叶片用于提取基因组DNA与总RNA。
1.2 基因组DNA的分离与UROS基因gDNA序列的扩增
采用CTAB法提取基因组DNA,利用NanoDrop 1000分光光度计检测总DNA浓度和纯度,并用1%琼脂糖凝胶检测。参考本实验室前期克隆且已发表的UROS基因cDNA序列(GenBank No. EF160077.1)[49]设计基因组DNA扩增引物UROS-F/UROS-R(UROS-F:5′-CTCCGCAAC GGAAGCCGCGGGATCG-3′; UROS-R: 5′-GG GAACACTTGTGCATATGCCGTGCCCG-3′)。扩增体系(20 μL):10×LA Buffer 2.0 μL、上下游引物各0.5 μL、DMSO 0.2 μL、LATaq(TaKaRa)0.2 μL、DNA 1 μL、dNTPs 1 μL、RNase Free ddH2O 14.6 μL。扩增条件:94 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 4 min,30个循环;72 ℃ 7 min。用1%琼脂糖电泳检测等体积的PCR产物,Bio-Rad凝胶成像系统照相,图片采用Gel-pro analyzer软件进行分析。
1.3 总RNA的分离、cDNA的合成及UROS基因cDNA序列的扩增
用Trizol试剂(TaKaRa)提取总RNA。用NanoDrop 1000分光光度计检测不同样品的总RNA纯度。配制1%的甲醛变性胶,通过电泳检测RNA质量。以1 μg总RNA为模板进行RT-PCR。RT-PCR体系为20 μL,包括50 mmol·L-1Tris-HCl(pH 8.3)、75 mmol·L-1KCl、3 mmol·L-1MgCl2、10 mmol·L-1DTT、1 μL oligo dT primer、50 μmol·L-1dNTPs、200 U M-MLV(Invitrogen),最终用RNase Free ddH2O补充足总体积。RT-PCR条件:37 ℃ 60 min,80 ℃ 5 min。
将获得的cDNA第一链用RNase Free ddH2O稀释5倍,用于扩增UROS基因cDNA。cDNA扩增所用引物同1.2中基因组扩增所用引物。扩增体系20 μL:10×Ex Buffer 2.5 μL、上下游引物各0.5 μL、DMSO 0.2 μL、ExTaq(Takara) 0.2 μL、cDNA 1 μL、dNTPs 1 μL、RNase Free ddH2O 14.1 μL。扩增条件:94 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 5 min。用1%琼脂糖电泳检测等体积的PCR产物,Bio-Rad凝胶成像系统照相,图片采用Gel-pro analyzer软件进行分析。
1.4 gDNA与cDNA的克隆测序
从凝胶上切割目标条带,用凝胶回收试剂盒(TaKaRa)回收PCR产物,得到的回收产物用凝胶检测,并连接在pMD18-T Vector(TaKaRa)上, 热激转化E.coilDH5α感受态,挑取阳性克隆送Invitrogen测序。凝胶回收和克隆测序严格按照操作手册进行。
1.5 序列比对与在线分析
在GrainGenes(http://wheat.pw.usda.gov/GG3/)和IWGSC(http://www.wheatgenome.org/)中进行EST序列和基因组序列比对及染色体电子定位;在Ensembl plants(http://plants.ensembl.org/index.html)和PGSB(Plant Genome and Systems Biology, http://pgsb.helmholtz-muenchen.de/plant/ plantsdb.jsp)中进行基因转录本预测等分析;用DNAMAN 6.0对已克隆测序的基因序列进行比对分析。
2 结果与分析
2.1 返白系和矮变1号中UROS基因gDNA序列的扩增及测序结果
2007年曹 凤等[49]从返白系中克隆到UROS基因cDNA序列(GenBank No. EF160077.1),全长1 077 bp,编码308个氨基酸,并预测1~63位氨基酸是叶绿体信号导肽,这是关于小麦UROS基因的最早报道。根据这个cDNA序列,本研究设计了引物UROS-F/UROS-R,包括部分5′-UTR和3′-UTR序列,从小麦品种矮变1号与返白系中扩增UROS基因的gDNA,结果如图1所示,扩增条带大约3.3 kb。
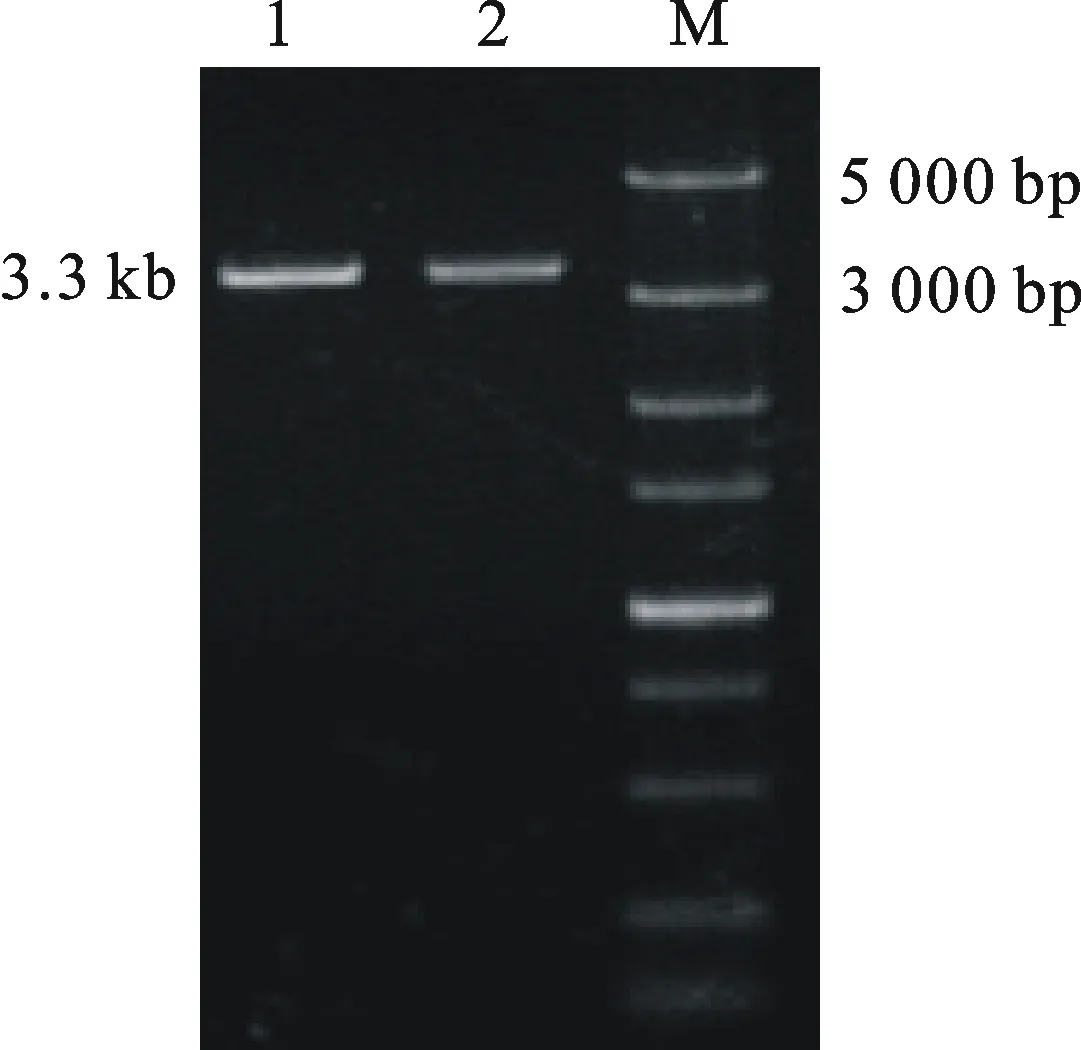
1:返白系;2:矮变1号;M:DL5000。
1:Albinism line; 2:Aibian No.1; M:DL5000.
图1 返白系和矮变1号中UROS基因gDNA的扩增结果
Fig.1 PCR fragments ofUROSgene amplified from genomic DNA of Albinism line and Aibian 1
对PCR扩增产物进行回收和克隆,每个品种送10个克隆去测序,分别在返白系中测通8个克隆的全长(两个克隆未测通),得到6个不同长度的序列;在矮变1号中测通10个克隆,得到9个不同长度的序列(表1)。用DNAMAN 6.0软件进行序列比对分析表明,序列间的相似性是99%。经在线软件和数据库GrainGenes(http://wheat.pw.usda.gov/GG3/)、PGSB(Plant Genome and Systems Biology)比对分析,分别将这些序列定位在小麦的4BL和4DL染色体上,并将两个基因位点分别命名为 UROS-4B 和 UROS-4D。
返白系4D基因位点gDNA序列平均长度为3 307 bp,矮变1号4D位点gDNA序列平均长度为3 305 bp;返白系4B位点仅得到一个gDNA序列,长度为3 325 bp,矮变1号4B位点gDNA平均长度为3 332 bp。除了PCR过程或测序造成的碱基替换或插入、缺失外,可以看出同一位点的同源基因存在品种间差异。
表1 在返白系与矮变1号中扩增得到的UROS基因的gDNA序列
Table 1UROSclones from gDNA of Albinism line and Aibian 1

品种CultivargDNA长度LengthofgDNA/bp克隆数No.ofclones染色体定位Chromosomelocation返白系332514BLAlbinismline330524DL330824DL330414DL331014DL331114DL矮变1号333014BLAibian1333114BL333214BL333714BL330024DL330214DL330614DL330914DL331414DL
长度相同的序列并不完全一致。下同。
Sequences with equal length are not absolutely the same.The same as below.
2.2 返白系和矮变1号中UROS基因cDNA序列的扩增及测序结果
利用与扩增UROS基因gDNA序列一致的引物,在返白系和矮变1号的第一链反转录产物中扩增得到UROS基因cDNA条带,结果如图2所示,扩增条带约在1.2 kb。
对PCR产物进行回收和克隆,每个品种送20个克隆去测序,返白系成功得到20个克隆的全长,矮变1号得到19个克隆的全长(表2)。在返白系中, UROS-4B基因位点可以转录出两个不同的转录本,本研究将它们命名 UROS-4B-a与 UROS-4B-b,后者较前者有一段54 bp序列的插入。由 UROS-4B-a 克隆测序得到1 231 bp与1 233 bp两种不同长度的序列,序列间有99.5%的同源性,存在个别碱基的替换与缺失。该位点可编码3种多肽,最长有299个氨基酸,其余两种多肽序列较短,应属于编码区终止密码子提前。 UROS-4D基因位点也转录出2个不同的转录本,分别命名为 UROS-4D-a与 UROS-4D-b,分别主要编码含有308和299个氨基酸的多肽, UROS-4D-b位点还可编码一个含有199个氨基酸的多肽,也是因终止密码子提前造成。上述结果表明,返白系两个UROS等位基因位点,可转录4个转录本,预测编码6种多肽。矮变1号与返白系略有差异, UROS-4B位点仅转录出一个转录本 UROS-4B-a,可编码含有299和314个氨基酸的两种多肽序列,后者是由于碱基序列突变,终止密码子后移。 UROS-4D位点与返白系类似,也有两个转录本,主要编码两种多肽, UROS-4D-b还可编码含有65、183及314个氨基酸的多肽。所以,矮变1号的两个UROS基因位点,可转录3个转录本,预测编码7种多肽。

1:返白系;2:矮变1号;M:DL2000。
1:Albinism line;2:Aibian 1;M:DL2000.
图2 返白系和矮变1号中UROS基因cDNA的扩增结果
Fig.2 PCR fragments ofUROSgene amplified from cDNA of Albinism line and Aibian 1
根据不同转录本的克隆所占比例及其编码的多肽序列,推测各转录本中, UROS-4B-a与 UROS-4D-a是标准剪接(canonical splicing)方式,而 UROS-4B-b与 UROS-4D-b是非标准剪接(non-canonical splicing)。在预测编码的多肽中,本研究推测含有299、308、314个氨基酸的多肽具有UROS活性,其余多肽由于序列缺失,可能会丧失UROS活性或活性降低。
表2 在返白系与矮变1号中扩增得到的UROS基因的cDNA序列
Table 2 URPOS clones amplified from cDNA of Abian 1 and Albinism line
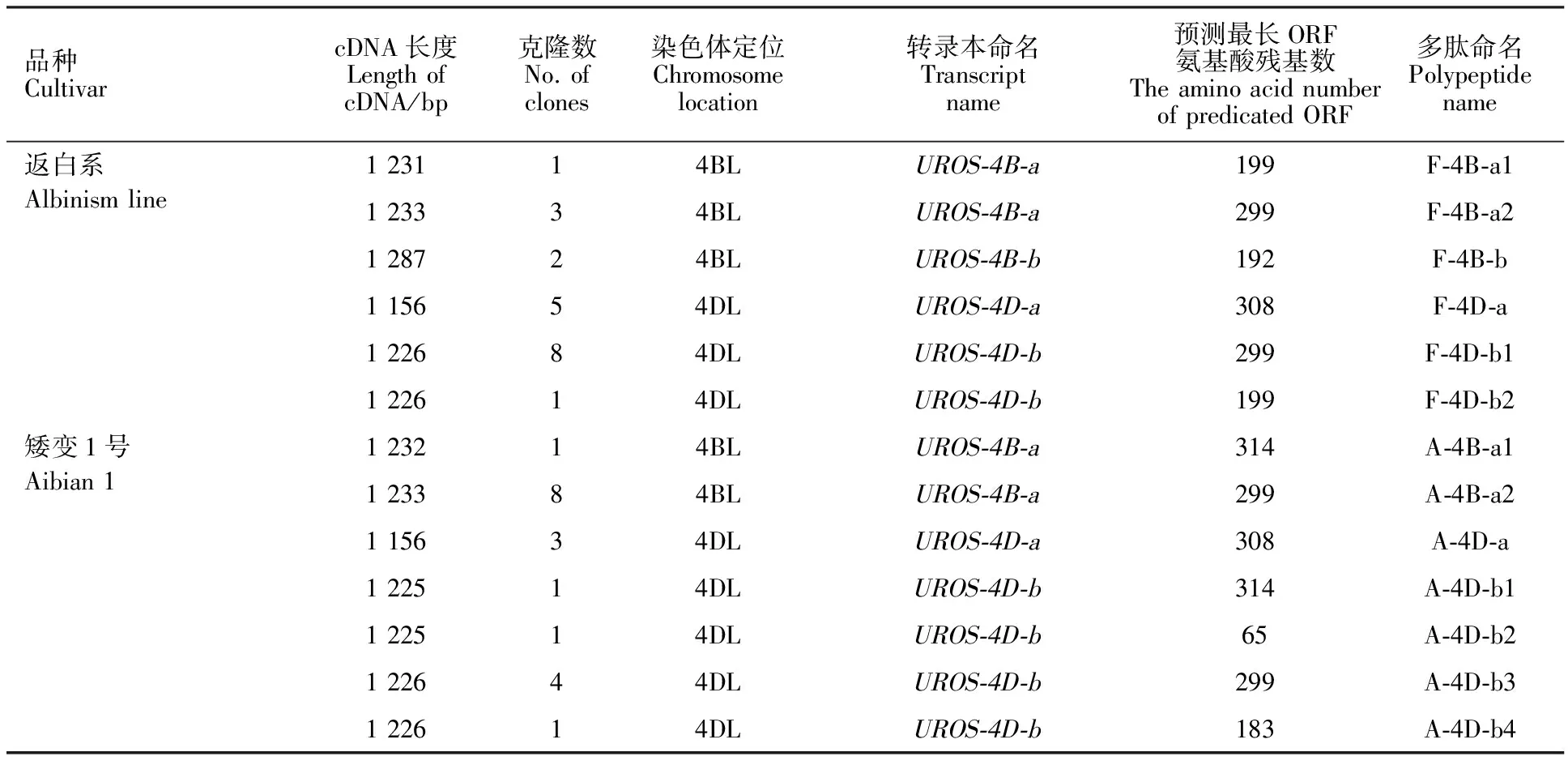
品种CultivarcDNA长度LengthofcDNA/bp克隆数No.ofclones染色体定位Chromosomelocation转录本命名Transcriptname预测最长ORF氨基酸残基数TheaminoacidnumberofpredicatedORF多肽命名Polypeptidename返白系123114BLUROS⁃4B⁃a199F⁃4B⁃a1Albinismline123334BLUROS⁃4B⁃a299F⁃4B⁃a2128724BLUROS⁃4B⁃b192F⁃4B⁃b115654DLUROS⁃4D⁃a308F⁃4D⁃a122684DLUROS⁃4D⁃b299F⁃4D⁃b1122614DLUROS⁃4D⁃b199F⁃4D⁃b2矮变1号123214BLUROS⁃4B⁃a314A⁃4B⁃a1Aibian1123384BLUROS⁃4B⁃a299A⁃4B⁃a2115634DLUROS⁃4D⁃a308A⁃4D⁃a122514DLUROS⁃4D⁃b314A⁃4D⁃b1122514DLUROS⁃4D⁃b65A⁃4D⁃b2122644DLUROS⁃4D⁃b299A⁃4D⁃b3122614DLUROS⁃4D⁃b183A⁃4D⁃b4
2.3 中国春中UROS基因gDNA与cDNA的扩增及测序结果
在线数据库GrainGenes、IWGSC、PGSB中的小麦基因组序列主要源自中国春,为了鉴定UROS基因在品种间是否有差异,以及上述试验结果的可靠性,对中国春的gDNA与cDNA也进行了扩增、克隆和测序,方法同前。结果表明,中国春中 UROS-4B和 UROS-4D两个基因位点基因组序列分别为3 339 bp和3 310 bp,与前两个品种类似,但有差异。
在IWGSC中序列比对结果显示,中国春4AS染色体上也有UROS基因同源序列,即4AS存在UROS基因位点,将其命名为 UROS-4A。序列分析结果表明,该基因位点的UROS基因编码区上下游的序列与 UROS-4B、 UROS-4D差异较大,所以用前文设计的引物UROS-F/UROS-R是不能扩增出其gDNA和cDNA的,故重新设计引物UROS-4A-F/UROS-4A-R(UROS-4A-F:5′-GTTGCTCTGTGGTTCATCTAGTG-3′,UR OS-4A-R:5′-AGCCAAGAGGAAGAAGTTGT AGT-3′)。用引物UROS-4A-F/UROS-4A-R从中国春中克隆、测序得到 UROS-4A基因gDNA序列,全长3 379 bp,与网上的中国春序列比对有99.5%的同源性。本研究对中国春 UROS-4B和 UROS-4D位点的cDNA序列也进行了PCR扩增、克隆和测序,得到12个克隆的全长,结果如表3所示。
经过序列分析表明,中国春的 UROS-4B位点有两个转录本,其中一个与在返白系和矮变1号中的不同,将其命名为 UROS-4B-c; UROS-4D位点有三个转录本,一个在前两个品种中没有出现,将其命名为 UROS-4D-c。所以,在中国春的两个UROS基因位点上共鉴定出5个转录本,预测编码5种多肽序列。
2.4 小麦中UROS基因的可变剪接
对前文得到的 UROS-4B与 UROS-4D两个基因位点的几个转录本进行分析,初步认为在这两个位点上的mRNA前体加工过程中,分别有1个标准剪接和2个非标准剪接方式,在不同品种间存在差异(表4、图3)。
UROS-4B的3个转录本中, UROS-4B-a是标准剪接方式,共有9个外显子(长度分别是250、88、68、119、96、56、86、135、541 bp)和8个内含子区,5′-UTR是52 bp,3′-UTR是486 bp。 UROS-4B-b与 UROS-4B-a相比,在第6个外显子区5′端有54 bp的内含子区转录,可变剪接方式属于可变的受体剪接位点(alternative acceptor introns,AA),也称为可变的3′端(alternative 3′ splicing)。 UROS-4B-c转录本中,第1和第2外显子区中间的第一内含子区(208 bp)转录,可变剪接方式属于IR。
表3 中国春中扩增得到的UROS基因的cDNA
Table 3UROSclones amplified from cDNA of Chinese Spring

cDNA长度LengthofcDNA/bp克隆数No.ofclones染色体定位Chromosomelocation转录本命名Transcriptname预测最长ORF氨基酸残基数TheaminoacidnumberofpredicatedORF多肽命名Polypeptidename123314BLUROS⁃4B⁃a282CS⁃4B⁃a1143414BLUROS⁃4B⁃c88CS⁃4B⁃c115654DLUROS⁃4D⁃a308CS⁃4D⁃a122644DLUROS⁃4D⁃b299CS⁃4D⁃b1133414DLUROS⁃4D⁃c118CS⁃4D⁃c
表4 本研究鉴定的小麦 UROS-4B与 UROS-4D基因位点的可变剪接
Table 4 Alternative splicings of UROS-4B and UROS-4D identified in this research

基因位点Genelocation转录本名称Transcriptname剪接方式Splicingwayofpre⁃mRNAcDNA长度LengthofcDNA/bp出现的品种Cultivar预测ORF氨基酸残基数PredictedaminoacidnumberUROS⁃4BUROS⁃4B⁃a标准剪接Canonicalsplicing1233A、F、CS299UROS⁃4B⁃b非标准剪接Non⁃canonicalsplicing1287F192UROS⁃4B⁃c非标准剪接Non⁃canonicalsplicing1434CS88UROS⁃4DUROS⁃4D⁃a标准剪接Canonicalsplicing1156A、F、CS308UROS⁃4D⁃b非标准剪接Non⁃canonicalsplicing1226A、F、CS299UROS⁃4D⁃c非标准剪接Non⁃canonicalsplicing1334CS138
A代表矮变1号;F代表返白系;CS代表中国春。
A indicated Aibian 1; F indicated Albinism line; CS indicated Chinese Spring.

虚线框表示内含子区;实线框表示外显子区;黑色实线框表示蛋白编码区。
Dashed frames indicate introns; Solid frames indicate exons; Solid and dark frames indicate protein coding regions.
图3 小麦 UROS-4B (A)与 UROS-4D (B)的可变剪接
Fig.3 Alternative splicings of UROS-4B (A) and UROS-4D (B) in wheat
UROS-4D基因位点的3个转录本中, UROS-4D-a是标准剪接方式,共有10个外显子(长度分别是245、88、68、118、96、56、86、135、51、315 bp)和9个内含子区,5′-UTR是47 bp,3′-UTR 是284 bp。与 UROS-4D-a相比, UROS-4D-b的第9个内含子区70 bp序列转录,可变剪接方式属于IR,而 UROS-4D-c除了具有IR,在第3外显子3′端有一段108 bp的内含子区转录,剪接方式还有可变的供体剪接位点(alternative donor introns,AD),也称为可变的5′端(alternative 5′ splicing)。
选取返白系和中国春不同转录本中的一个克隆,对其预测编码的多肽序列进行比对,如图4所示。标准剪接的两个转录本 UROS-4B-a和 UROS-4D-a分别编码299和308个氨基酸残基的多肽。可变剪接的转录本中,只有 UROS-4D-b可以编码较完整的多肽序列,其余剪接方式均造成肽段序列的缺失。 UROS-4B-a、 UROS-4D-a与 UROS-4D-b编码的3个多肽有95.8%的氨基酸残基序列一致,共有12个位点发生替换。在预测的信号肽区(前63位氨基酸),序列相对保守,仅第7位返白系中是丙氨酸(A),中国春中是缬氨酸(V),虽然表现出品种间的差异,但都是疏水氨基酸。这些变异位点是否会造成UROS空间结构和活性的差异,值得进一步研究。
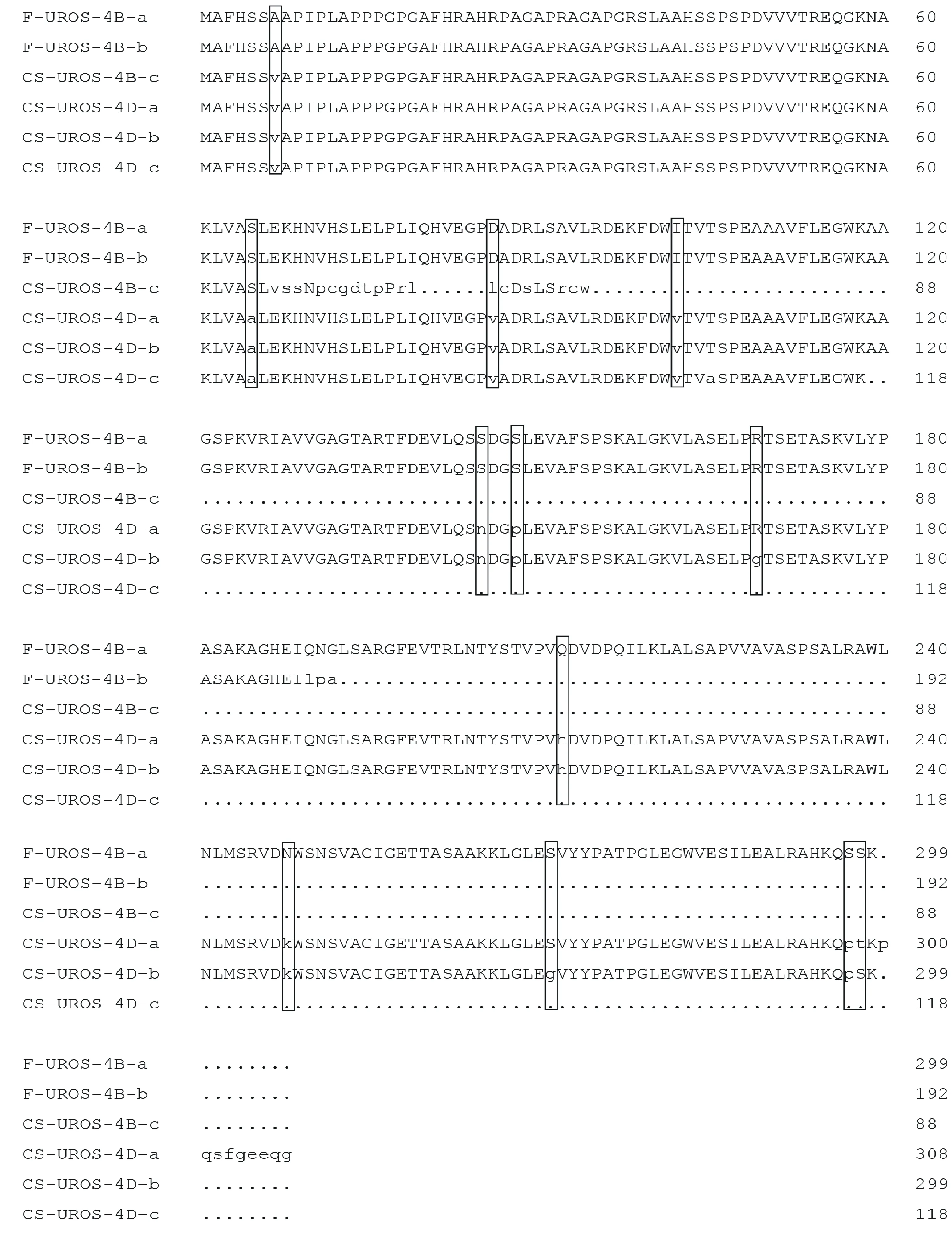
方框表示 UROS-4B-a、 UROS-4D-a与 UROS-4D-b三个转录本预测编码多肽的12个变异位点。
Frames indicated twelve variant sites of polypeptides encoded by UROS-4B-a, UROS-4D-a and UROS-4D-b.
图4 由UROS基因不同转录本预测的6种多肽氨基酸序列(用DNAMAN软件预测)
Fig.4 Six predicted amino acid sequences ofUROSgene
3 讨 论
PGSB网站可以在线预测基因的多种可变剪接方式。输入本研究得到的基因组序列,PGSB预测出 UROS-4B位点有一种标准剪接、12种非标准剪接转录本,标准剪接与本研究测序得到的 UROS-4B-a一致,非标准剪接包括本研究鉴定的 UROS-4B-b和 UROS-4B-c两种可变剪接方式。不同的是,预测 UROS-4B-c可以编码含有335个氨基酸的多肽,而本研究测序得到的序列预测仅编码含有88个氨基酸的多肽,推测是PCR扩增或测序引起序列变异造成的。PGSB网站预测 UROS-4D有一种标准剪接、5种非标准剪接转录本,标准剪接方式与 UROS-4D-a一致,非标准剪接包括 UROS-4D-b,但没有预测出本研究测序得到的 UROS-4D-c。网站也预测了UROS-4A具有一种标准剪接和3种非标准剪接方式。本研究没有检测 UROS-4A位点的可变剪接,有待后续工作继续完成。
在线预测UROS基因有多种可变剪接方式,而本研究只鉴定到了4个非标准剪接转录本,可能是有些转录本的表达量比较低,没有被克隆到,也有可能在线预测的结果并非十分准确。在本研究所鉴定到的3种可变剪接方式,主要是以IR方式进行的,24个可变剪接克隆中,有22个属于或包含IR剪接方式,这与已有的文献报道是一致的[5,15-16,29,34]。我们发现 UROS-4D-c可以同时有两种可变剪接方式,如果这也是普遍存在的,就会显著增加转录后加工的复杂性。
本文研究表明,在矮变1号、返白系与中国春三个小麦品种中UROS基因可变剪接的方式存在差异,矮变1号中的可变剪接最少,返白系在 UROS-4B和 UROS-4D位点两个基因位点各有一个可变剪接转录本,中国春有3个可变剪接转录本。这种品种间差异的分子机理及其可能具有的功能值得进一步探讨。
UROS在植物叶绿素合成代谢中具有重要的功能,已有的研究也表明在返白系与矮变1号中的UROS活性存在差异[46-47],特别是在持续低温条件下,返白系中的UROS受到抑制,是一个叶绿素合成受阻的位点,对于UROS与白化之间的关系也一直是我们所关注的。从基因组序列和启动子序列(已经克隆测序)的比对来看,两个材料间差异不大,99%的序列一致。本文的研究结果启示我们,是否由于UROS基因可变剪接方式的差异,引起UROS活性在矮变1号和返白系中不同,这些不同的可变剪接方式,是否受到发育阶段、环境条件的调控,是否与返白系受到低温诱导白化的现象有关,尚需要研究。
关于小麦基因的可变剪接已有较多报道[35-40],本研究结果再次证实了小麦基因的可变剪接很可能是广泛存在的,同一基因位点可以转录出多个不同的转录本,而且可以编码出多个不同的多肽序列。加之,小麦是异源六倍体,存在多个等位基因位点,这些更增加了小麦基因表达调控的复杂性。我们有理由相信对小麦基因可变剪接机理的研究,及其对小麦发育和逆境响应的调控研究将会成为小麦分子遗传学及小麦分子育种研究的新热点[4]。
致谢:本试验主要在西北农林科技大学生命科学学院生物化学与分子生物学科研教学平台完成,对段 敏研究员、范宁娟老师和魏兰兰老师提供的帮助和支持表示衷心的感谢!
[1] HODGES D,BERNSTEIN S I.Genetic and biochemical analysis of alternative RNA splicing [J].AdvancesinGenetics,1994,31:270.
[2] LAREAU L F,GREEN R E,BHATNAGAR R S,etal.The evolving roles of alternative splicing [J].CurrentOpinioninStructuralBiology,2004,14:273.
[3] STAMM S,BEN-ARI S,RAFALSKA I,etal.Function of alternative splicing [J].Gene,2005,344:1.
[4] SYED N H,KALYNA M,MARQUEZ Y,etal.Alternative splicing in plants- coming of age [J].TrendsinPlantScience,2012,17:616.
[5] FILICHKIN S A,PRIEST H D,GIVAN S A,etal.Genome-wide mapping of alternative splicing inArabidopsisthaliana[J].GenomeResearch,2010,20:45.
[6] WAHL M C,WILL C L,LUHRMANN R.The spliceosome:Design principles of a dynamic RNP machine [J].Cell,2009,136:701.
[7] BERGET S M,MOORE C,SHARP P A.Spliced segments at the 5′ terminus of adenovirus 2 late mRNA [J].ProceedingsoftheNationalAcademyofSciences,1977,74:3171.
[8] CHOW L T,GELINAS R E,BROKER T R,etal.An amazing sequence arrangement at the 5′ends of adenovirus 2 messenger RNA [J].Cell,1977,12:1.
[9] ROSENFELD M G,AMARA S G,ROOS B A,etal.Altered expression of the calcitonin gene associated with RNA polymorphism [J].Nature,1981,290:63.
[10] PAN Q,SHAI O,LEE L J,etal.Deep surveying of alternative splicing complexity in the human transcriptome by high throughout sequencing [J].NatureGenetics,2008,40:1413.
[11] WERNEKE J M,CHATFIELD J M,OGREN W L.Alternative mRNA splicing generates the two ribulose bisphosphate carboxylase/oxygenase activase polypeptides in spinach andArabidopsis[J].ThePlantCell,1989,1:815.
[12] BARBAZUK W B,FU Y,MCGINNIS K M.Genome-wide analyses of alternative splicing in plants:Opportunities and challenges [J].GenomeResearch,2008,18(9):1381.
[13] XU S,ZHANG Z,JING B,etal.Transportin-SR is required for proper splicing of resistance genes and plant immunity [J].PLoSGenetics,2011,7(6):e1002159.
[14] MASTRANGELO A M,MARONE D,LAIDO G,etal.Alternative splicing:Enhancing ability to cope with stress via transcriptome plasticity [J].PlantScience,2012,185-186:40.
[15] DUBROVINA A S,KISELEV K V,ZHURAVLEV Y N.The role of canonical and non-canonical pre-mRNA splicing in plant stress responses [J].BioMedResearchInternational,2013,2013:1.
[16] CARVALHO R F,FEIJAO C V,DUQUE P.On the physiological significance of alternative splicing events in higher plants [J].Protoplasma,2013,250:639.
[17] SEO P J,PARK M J,PARK C M.Alternative splicing of transcription factors in plant responses to low temperature stress:Mechanisms and functions [J].Planta,2013,237:1415.
[18] ALEXANDROV N N,TROUKHAN M E,BROVER V V,etal.Features ofArabidopsisgenes and genome discovered using full-length cDNAs [J].PlantMolecularBiology,2006,60:69.
[19] LI J,LI X,GUO L,etal.A subgroup of MYB transcription factor genes undergoes highly conserved alternative splicing inArabidopsisand rice [J].JournalofExperimentalBotany,2006,57(6):1263.
[20] CAMPBELL M A,HAAS B J,HAMILTON J P,etal.Comprehensive analysis of alternative splicing in rice and comparative analyses withArabidopsis[J].BMCGenomics,2006,7:327.
[21] MARQUEZ Y,BROWN J,SIMPSON C,etal.Transcriptome survey reveals increased complexity of the alternative splicing landscape inArabidopsis[J].GenomeResearch,2012,22:1184.
[22] KISSEN R,HYLDBAKK E,WANG C W V,etal.Ecotype dependent expression and alternative splicing of epithiospecifier protein (ESP) inArabidopsisthaliana[J].PlantMolecularBiology,2012,78:361.
[23] PETRILLO E,MICAELA A G H,FUCHS A,etal.A chloroplast retrograde signal regulates nuclear alternative splicing [J].Science,2014,344:427.
[24] SHIN K H,YANG S H,YONG L J,etal.Alternative splicing of mini-exons in theArabidopsisleaf rust receptor-like kinase LRK10 genes affects subcellular localization [J].PlantCellReport,2015,34:495.
[25] GROTEWOLD E,ATHMA P,PETERSON T.Alternatively spliced products of the maizePgene encode proteins with homology to the DNA-binding domain of myb-like transcription factors [J].ProceedingsoftheNationalAcademyofSciences,1991,88:4587.
[26] MONTAG K,SALAMINI F,THOMPSON R D.ZEMa,a member of a novel group of MADS box genes,is alternatively spliced in maize endosperm [J].NucleicAcidsResearch,1995,23:2168.
[27] CARLE-URIOSTE J C,BRENDEL V,WALBOT V.A combinatorial role for exon,intron and splice site sequences in splicing in maize [J].PlantJournal,1997,11:1253.
[28] RAUCH H B,PATRICK T L,KLUSMAN K M,etal.Discovery and expression analysis of alternative splicing events conserved among plant SR proteins [J].MolecularBiologyandEvolution,2013,31:605.
[29] E Z G,WANG L,ZHOU J H.Splicing and alternative splicing in rice and humans [J].BMBReport,2013,46(9):439.
[30] SABLOK G,GUPTA P K,BAEK J M,etal.Genome-wide survey of alternative splicing in the grassBrachypodiumdistachyon:a emerging model biosystem for plant functional genomics [J].BiotechnologyLetters,2011,33:629.
[31] BOURNAY A S,HEDLEY P E,MADDISON A,etal.Exon skipping induced by cold stress in a potato invertase gene transcript [J].NucleicAcidsResearch,1996,24:2347.
[32] DUBROVINA A S,ALEYNOVA O A,KISELEV K V,etal.True and false alternative transcripts of calcium-dependent protein kinase CPK9 and CPK3a genes inVitisamurensis[J].ActaPhysiologyPlantrum,2014,36:1727.
[33] DUBROVINA A S,ALEYNOVA O A,KISELEV K V.Influence of overexpression of the true and false alternative transcripts of calcium-dependent protein kinase CPK9 and CPK3a genes on the growth,stress tolerance,and resveratrol content inVitisamurensiscell cultures [J].ActaPhysiologyPlantrum,2016,38:78.
[34] RICHARDSON D N,ROGERS M F,LABADORF A,etal.Comparative analysis of serine/arginine-rich proteins across 27 eukaryotes:Insights into sub-family classification and extent of alternative splicing [J].PLoSOne,2011,6(9):e24542.
[35] BÅGA M,GLAZE S,MALLARD C S,etal.A starch-branching enzyme gene in wheat produces alternatively spliced transcripts [J].PlantMolecularBiology,1999,40:1019.
[36] 郭志爱,赵光耀,任正隆,等.小麦光周期基因 Ppd-B1的选择性剪接分析[J].作物学报,2009,35(10):1764.
GUO Z A,ZHAO G Y,REN Z L,etal.Alternative splicing of photoperiod response gene Ppd-B1 in wheat [J].ActaAgronomicaSinica,2009,35(10):1764.
[37] EGAWA C,KOBAYASHI F,ISHIBASHI M,etal.Differential regulation of transcript accumulation and alternative splicing of a DREB2 homolog under abiotic stress conditions in common wheat [J].Genes&GeneticsSystem,2006,81:77.
[38] TAVAKOL E,SARDARO M L S,AHID SHARIATI J,etal.Isolation,promoter analysis and expression profile of Dreb2 in response to drought stress in wheat ancestors [J].Gene,2014,549:24.
[39] SUN Y W,HE Z H,MA W J,etal.Alternative splicing in the coding region of Ppo-A1 directly influences the polyphenol oxidase activity in common wheat(TriticumaestivumL.) [J].Functional&IntegrativeGenomics,2011,11:85.
[40] HOWITT C A,CAVANAGH C R,BOWERMAN A F.Alternative splicing,activation of cryptic exons and amino acid substitutions in carotenoid biosynthetic genes are associated with lutein accumulation in wheat endosperm [J].Functional&IntegrativeGenomics,2009,9:363.
[41] BATTERSBY A R,LEEPER F J.Biosynthesis of the pigments of life:mechanistic studies on the conversion of porphobilinogen to uroporphyrinogen Ⅲ [J].ChemicalReviews,1990,90:1261.
[42] SHOOLINGIN-JORDAN P M.Porphobilinogen deaminase and uroporphyrinogen Ⅲ synthase:structure,molecular biology,and mechanism [J].JournalofBioenergeticsandBiomembranes,1995,27(2):181.
[43] HIGUCHI M,BOGORAD L.The purification and properties of uroporphyrinogen Ⅰ synthase and uroporphyrinogen Ⅲ cosynthase.Interactions between the enzymes [J].AnnalsoftheNewYorkAcademyofSciences,1975,244:401.
[44] 苏小静,汪沛洪.小麦突变体返白系返白机理的研究Ⅰ返白阶段叶绿体超微结构观察[J].西北农业大学学报,1990,18(2):73.
SU X J,WANG P H.Studies on the mechanism of albino characteristic of winter wheat mutant "the albinism line"-Ⅰ,the ultrastructure of chloroplast [J].ActaUniversitatisAgriculturalisBoreali-Occidentalis,1990,18(2):73.
[45] 郭蔼光,王振镒.小麦返白系的白化特征及其与环境因素的关系[J].西北植物学报,1991,11(5):122.
GUO A G,WANG Z Y.The relation between the albino character of wheat albinism line and environments [J].ActaBotanicaBoreali-OccidentaliaSinica,1991,11(5):122.
[46] 王保莉,郭蔼光,汪沛洪.小麦突变体返白系返白阶段叶绿素代谢的变化[J].植物学报,1996,38(7):557.
WANG B L,GUO A G,WANG P H.Changes of chlorophyll metabolism during the albinic stage of a wheat mutant [J].ActaBotanicaSinica,1996,38(7):557.
[47] LIU X G,XU H,ZHANG J Y,etal.Effect of low temperature on chlorophyll biosynthesis in albinism line of wheat (TriticumaestivumL.) FA85 [J].PhysiologiaPlantarum,2012,145(3):38490.
[48] 范 军,郭蔼光.小麦尿卟啉原Ⅲ合酶的分离和纯化[J].西北农业大学学报,2000,28 (1):11.
FAN J,GUO A G.Isolation and purification of uroporphyrinogen Ⅲ synthase from wheat leaf [J].ActaUniversitatisAgriculturalisBoreali-Occidentalis,2000,28 (1):11.
[49] 曹 凤,王亚红,金伟波,等.小麦尿卟啉原Ⅲ合成酶基因克隆及序列分析[J].西北植物学报,2007,27(7):1299 .
CAO F,WANG Y H,JIN W B,etal.Cloning and sequence analysis of uroporphyrinogen Ⅲ synthase gene in wheat [J].ActaBotanicaBoreali-OccidentaliaSinica,2007,27(7):1299.
Alternative Splicing of Uroporphyrinogen Ⅲ Synthase Genes in Wheat
LANG Danying1,LÜ Gengyin1,LIANG Guangwang1,LI Ruihang1,GUO Xiaoguang1,XU Hong1,2,GUO Aiguang1,2
(1.College of Life Sciences,Northwest A&F University,Yangling,Shaanxi 712100,China; 2.State Key Laboratory of Crop Stress Biology for Arid Areas,Yangling,Shaanxi 712100,China)
Alternative splicing (AS) is a key regulatory mechanism of gene expression,which is related to the development,physiology,metabolism,signal transduction and stress-response of plant. In this experiment,we cloned the Uroporphyrinogen Ⅲ synthase(UROS) genes from gDNA and cDNA of wheat and investigated the AS ofUROSgenes in wheat with bioinformatics techniques,using Albinism line,Aibian 1 and Chinese Spring as materials. TheUROSgDNA sequences are located on 4BL and 4DL chromosomes based on the wheat genome sequences online database,which are designated as UROS-4B and UROS-4D,respectively. About 50 differentUROScDNA sequences were cloned and sequenced from three cultivars. We found the AS inUROSpre-mRNA processing through the online predication and the comparison between cDNA and gDNA sequences,and there are one canonical and two non-canonical transcripts of each gene location. Intron retention is the primary alternative splicing type,as well as alternative donor introns and alternative receptor introns were found,and different varieties showed diverse splicing types. In addition,the predicated amino acid sequence of these transcripts were dissimilar,twelve variant amino acid sites were identified from six polypeptides encoded by six clones from different transcripts,respectively.
Wheat;Albinism line;Uroporphyrinogen Ⅲ synthase(UROS);Alternative splicing; Canonical and non-canonical splicing
徐 虹(E-mail:xuh73@163.com)
2016-05-21
2016-10-20
国家自然科学基金项目 (30971769,31371541)
E-mail:278436477@qq.com(郎丹莹);411427580@qq.com(吕庚寅,与第一作者同等贡献);545601366@qq.com(梁广旺,与第一作者同等贡献)
时间:2016-12-07
S512.1;S330
A
1009-1041(2016)12-1553-10
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20161207.1750.020.html
