喀斯特山区大眼鳜线粒体COⅠ基因序列分析
2016-12-20郭健康周其椿曹恒源陈薛伟杰
郭健康,安 苗,周其椿,曹恒源,李 珊,陈薛伟杰
( 1.贵州大学 动物科学学院,贵州 贵阳 550025; 2.贵州水产科学研究所,贵州 贵阳 550025 )
喀斯特山区大眼鳜线粒体COⅠ基因序列分析
郭健康1,安 苗1,周其椿2,曹恒源1,李 珊1,陈薛伟杰1
( 1.贵州大学 动物科学学院,贵州 贵阳 550025; 2.贵州水产科学研究所,贵州 贵阳 550025 )
为了解喀斯特山区大眼鳜3个地理群体内和群体间的遗传变异特点,采用PCR技术对169尾个体的线粒体DNA COⅠ基因进行扩增、测序,获得的序列长度为1512 bp。分析显示,碱基A、T、C、G的平均含量分别为24.9%、28.6%、28.7%、17.8%,其中A+T(53.5%)含量高于G+C(46.5%)含量。169个样本共检测到5个多态位点,并定义了5种单倍型。对喀斯特山区大眼鳜群体COⅠ基因与洞庭湖大眼鳜COⅠ基因序列进行序列差异分析,结果显示,两者之间存在10个变异位点,集中在1268~1456 nt。喀斯特山区3个群体间4种碱基在密码子3个位点上的使用频率无差别,与洞庭湖大眼鳜相比,密码子第1位上的4种碱基含量差别不大,密码子第2位上T含量(40.9%)高于后者(39.9%),而A和C含量则低于后者;与洞庭湖翘嘴鳜比较差别很小,但与斑鳜、暗鳜、长身鳜和波纹鳜4种鳜鱼比较,在密码子第1位和第3位存在不同差异,特别是暗鳜和波纹鳜密码子第3位中A含量(30.0%)明显低于喀斯特山区大眼鳜(31.8%),而波纹鳜G(8.9%)明显高于后者(7.3%)。喀斯特山区大眼鳜平均单倍型多样性为0.395,平均核苷酸多样性为0.00029,遗传多样性贫乏。
喀斯特山区;大眼鳜;COⅠ基因;密码子;遗传多样性
大眼鳜(Sinipercakneri)属硬骨鱼纲、鲈形目、科、鳜亚科、鳜属,俗称桂花鱼、白桂等,分布于中国长江及其以南水系,为中国南方特有种[1-2],是一种名贵的经济鱼类。贵州位于滇东高原到湘西丘陵之间的过渡地带,地处长江和珠江上游,属典型的喀斯特山地,自西向东的苗岭山脉将其境内的水系分隔成珠江和长江两大流域,区内河流众多,生境多样,鳜亚科鱼的种类及分布也丰富,近年来由于截流筑坝、过度捕捞、环境污染,资源锐减,有的种类已很难采到。目前,本区大眼鳜的基础生物学及群体遗传学研究仍十分滞后,仅禹真等[3-4]对锦江河特有鱼类国家级水产种质资源保护区内大眼鳜的形态特征、肌肉营养、渔获物组成和资源状况有过报道。
细胞色素c氧化酶亚基Ⅰ(Cytochrome c oxidase subunit Ⅰ, COⅠ)是线粒体中13个蛋白编码基因之一,COⅠ不仅拥有足够变异而易于被通用引物扩增, 而且其DNA序列本身很少存在插入和缺失。目前mtDNA COⅠ基因既作为DNA条码识别生物物种的有效性[5],也广泛用于水生生物群体遗传多样性及其遗传结构的研究[6-11]。本文运用PCR扩增技术对分属长江流域和珠江流域的大眼鳜3个地理群体的169尾样本mtDNA COⅠ基因进行测序分析,探讨喀斯特山区大眼鳜COⅠ基因序列特点和群体遗传多样性,为国家级水产种质资源保护区经济鱼类的种质资源评价奠定基础。
1 材料与方法
1.1 材料
野生大眼鳜采自3个水产种质资源保护区,共计169尾。其中乌江黄颡鱼国家级水产种质资源保护区沿河县城河段45尾,锦江河特有鱼类国家级水产种质资源保护区铜仁段30尾,北盘江九盘段特有鱼类省级水产种质资源保护区关岭段96尾。对活鱼样本进行常规生物学测定并解剖,取背部肌肉3~5 g浸泡于无水乙醇中,-20 ℃保存备用。利用血液/细胞/组织基因组DNA提取试剂盒(北京天根)提取基因组DNA,1.5% 琼脂糖凝胶电泳检验其完整性,用紫外分光光度计(Thermo NanoDrop 2000C)测定其吸光值。
1.2 引物设计与PCR 扩增
参照翘嘴鳜(S.chuatsi)(登录号为:JF972568.1)、斑鳜(S.scherzeri)(登录号为:AP014527.1)和中国少鳞鳜(Coreopercawhiteheadi)(登录号为:NC023957.1)等鳜亚科鱼类COⅠ基因序列设计引物,COF:5′-GGGGTTAGCCTACTTCTTT-3′;COR:5′-TGGTACTGACCTTTCTCGT-3′,引物由北京诺赛基因组研究中心有限公司合成。
PCR反应体系为20 μL:模板DNA 2 μL;2×Taq PCR Master Mix(北京天根)10 μL;上下游引物分别1 μL;剩余体积用双蒸水补齐。PCR扩增反应在AB公司梯度PCR仪(型号:Veriti)上完成;扩增条件:94 ℃预变性3 min; 94 ℃变性40 s,55.1 ℃退火40 s,72 ℃延伸1 min, 35个循坏;最后再72 ℃延伸10 min,4 ℃保存。
1.3 PCR产物的测定
根据目的片段扩增结果,切胶回收纯化,将所有PCR产物送往北京诺赛基因组研究中心有限公司双向测序。
1.4 数据的处理
利用DNAstar 5.0软件包中的Seqman做正反向链的拼接,手动校正并去掉两端引物及部分序列;运用Mega 5.0统计碱基组成、变异位点、氨基酸含量,计算遗传距离、建立系统进化树;采用DNASP 5.0软件统计单倍型多样度,计算单倍型多样度、核苷酸多样度、群体间遗传分化指数(Fst),基因流(Nm)由Nm≈(1-Fst)/(4Fst)计算。
2 结果与分析
2.1 喀斯特山区大眼鳜mtDNA COⅠ基因扩增及序列差异
用CO5F/CO5R 引物对169尾大眼鳜DNA样品进行PCR 扩增, 所有样品均扩增出约1700 bp条带(图1)。
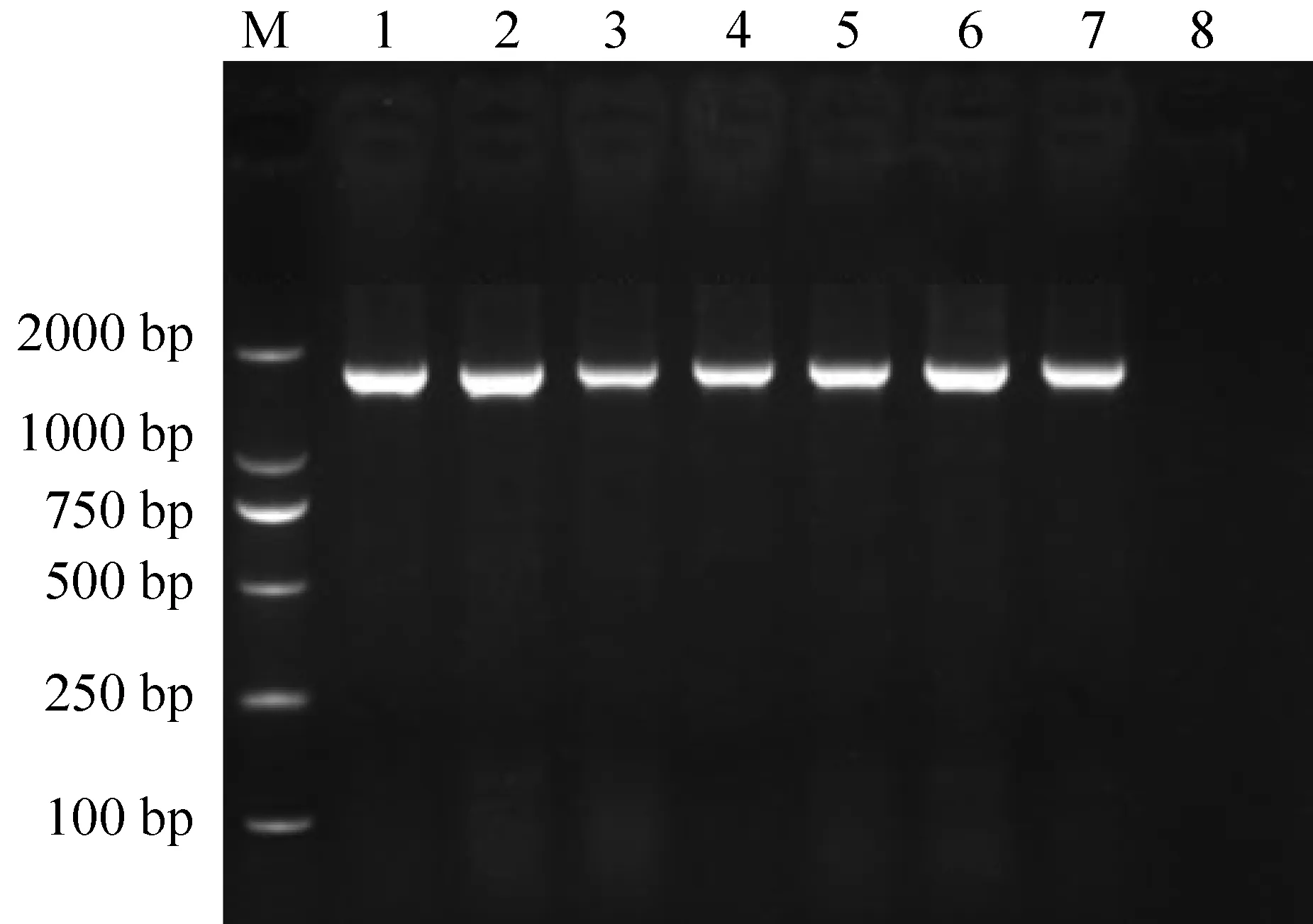
图1 喀斯特山区3个大眼鳜群体COⅠ基因PCR扩增结果
注:M.2000 bp分子标记;1、2.沿河群体;3、4.铜仁群体;5~7.关岭群体;8.空白对照.
经拼接、校对和比对后,获得169条自5′端开始的1512 bp序列,分属5种单倍型,并提交GenBank,其登录号为KU049654~KU049658,同时自GenBank中下载鳜属6种鳜鱼的10条同源序列作对比分析(表1)。

表1 本研究所用样本信息
注:Hap1~Hap5,喀斯特山区大眼鳜群体单倍型;DTD,洞庭湖大眼鳜单倍型;QZG,翘嘴鳜单倍型;BG1~BG5,斑鳜单倍型;CTG,长体鳜单倍型;ANG,暗鳜单倍型;BWG,波纹鳜单倍型. 下同.
Mega 5.0软件分析显示:喀斯特山区3个大眼鳜群体中,沿河群体存在4个变异位点(3个转换位点和1个颠换位点),而其他2个种群未检测到变异位点。169个样本共检测到5个变异位点(图2),未检测到插入或缺失位点。这些变异除在513 nt处发生了1个碱基替换外,其余变异位点均发生在880 nt以后(变异比率为0.3%)。与洞庭湖大眼鳜比较有10个变异位点,主要集中在1268~1456 nt,变异比率为0.6%。与洞庭湖翘嘴鳜相比差异小,仅有2个变异位点。与斑鳜、暗鳜、长体鳜和波纹鳜4个种间相比变异则较大,共有153个变异位点,含101个信息位点和52个单一变异位点,主要发生在213~1509 nt,变异比率为9.7%。可见,喀斯特山区大眼鳜的COⅠ基因在种内和与翘嘴鳜间,在近5′端654 bp左右变异较小,而与同属中其他鱼类间的变异较大。
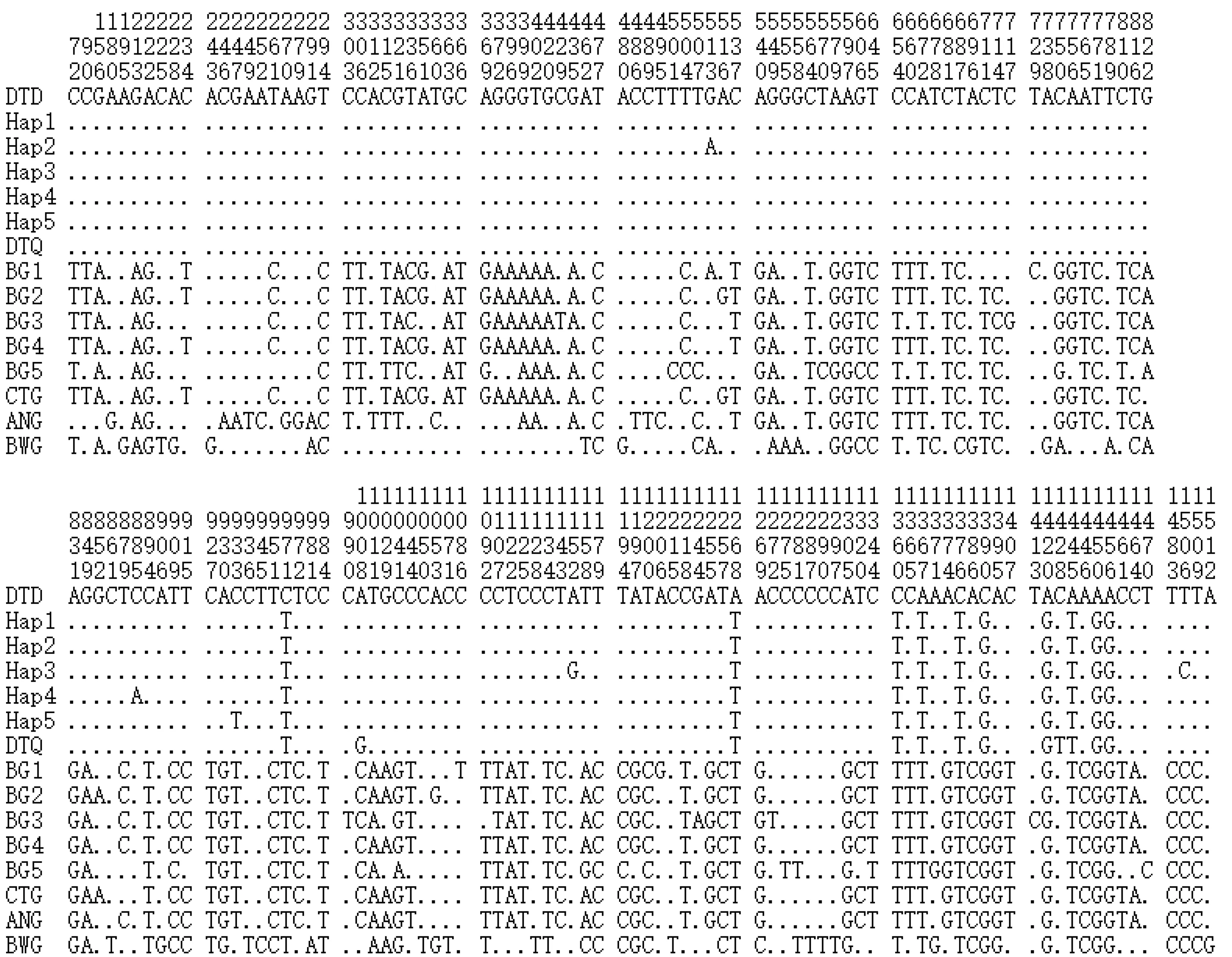
图2 鳜属鱼类线粒体COⅠ基因序列变异位点
碱基组成上,喀斯特山区3个大眼鳜群体间的A、T、C和G碱基平均含量相差不大,碱基C的含量最高,G含量最低,A+T含量都高于C+G含量且G的含量显著低于其他碱基的含量,表现出明显的反G 偏向(表2);与洞庭湖大眼鳜及其他4种鳜鱼差别较小。可以看出,喀斯特山区大眼鳜的COⅠ基因与鳜属鱼类在碱基组成上相似。
密码子的使用上,喀斯特山区3个大眼鳜群体间在第1位、第2位和第3位点上无差别。相比较于洞庭湖群体,其密码子第1位的T(20.6%)和G(30.7%)含量略高于后者(T为20.2%,G为30.0%),而A(25.5%)和C(32.2%)的含量则略低于后者(A为26.2%,C为32.6%);在密码子第2位上,喀斯特山区群体的T(40.9%)含量高于洞庭湖群体的T(39.9%)含量,而A(17.5%)和C(26.4%)含量低于后者(A为18.1%,C为26.8%)。与翘嘴鳜比较差别较小,但与斑鳜、暗鳜、长体鳜和波纹鳜4种鳜鱼比较,除密码子第2位上各碱基的使用率无差异外,密码子第1位和第3位上存在不同差异,特别是暗鳜和波纹鳜密码子第3位上A(30.0%)含量明显低于喀斯特山区大眼鳜A(31.8%),而波纹鳜G(8.9%)含量明显高于喀斯特山区大眼鳜G(7.3%)的含量。可见在密码子的使用率上,喀斯特山区大眼鳜群体的COⅠ基因在种群内差别较小;而不同种群间,在密码子第1位点和第2位点上差别较大。
1512 bp的核苷酸序列共编码504个氨基酸,喀斯特山区大眼鳜群体的5个变异位点均为同义突变且都发生在密码子第3位上,3个群体的氨基酸含量一致,其中亮氨酸含量最高(12.30%),半胱氨酸最低(0.20%)。相比于洞庭湖群体,密码子第1位(转换/颠换比为4)和密码子第2位(转换/颠换比为2/3)存在碱基变异,使10个氨基酸发生了变异,其亮氨酸和半胱氨酸含量略低(亮氨酸11.71%,半胱氨酸0.20%)。与翘嘴鳜、斑鳜、暗鳜、长体鳜、波纹鳜相比较,检测到3处氨基酸被替代。结果表明,喀斯特山区大眼鳜群体的COⅠ基因的碱基变异特点,与同属中其他鳜类的碱基变异主要表现为同义突变特点类似。
注:WYH:沿河群体;JTD:铜仁群体;BGD:关岭群体;GZD:喀斯特山区3个大眼鳜群体;DTD:洞庭湖大眼鳜;QZG:洞庭湖翘嘴鳜;BG:斑鳜;ANG:暗鳜;CTG:长体鳜;BWG:波纹鳜.
2.2 喀斯特山区大眼鳜群体的遗传距离及系统关系
喀斯特山区3个大眼鳜群体间,沿河与关岭群体间遗传距离为0.000,它们与铜仁群体间的遗传距离均为0.001;喀斯特山区3个大眼鳜群体内,沿河群体内遗传距离为0.000~0.002,关岭群体内和铜仁群体内遗传距离均为0.000。喀斯特山区大眼鳜5个单倍型间的遗传距离为0.001~0.002;与洞庭湖大眼鳜群体之间的遗传为0.007~0.008;与翘嘴鳜的遗传距离为0.002~0.003;与斑鳜之间的遗传距离为0.056~0.067;与长体鳜、暗鳜和波纹鳜之间的遗传距离为0.053~0.067;与中国少鳞鳜遗传距较远,为0.089~0.091;与日本少鳞鳜(C.kawamebari)和朝鲜少鳞鳜(C.herzi)之间的遗传距离最远,为0.184~0.190。由此表明,喀斯特山区大眼鳜的COⅠ基因在种群内遗传距离小于1%,而与其他种群间较远且在10倍以上。

表3 鳜亚科鱼类单倍型之间的遗传距离
注:Hap1~Hap5:喀斯特山区大眼鳜群体单倍型;DTD:洞庭湖大眼鳜单倍型;QZG:翘嘴鳜单倍型;BG1~GB5:斑鳜单倍型;CTG:长体鳜单倍型;BWG:波纹鳜单倍型;ANG:暗鳜单倍型;ZGS:中国少鳞鳜单倍型;RBS:日本少鳞鳜单倍型;CXS:朝鲜少鳞鳜单倍型.
将自GenBank中下载9种鳜鱼COⅠ同源序列(序列号见图3),与本文5种大眼鳜单倍型一起,以花鲈(Lateolabraxjaponicus)(GenBank:AP006789)为外群,构建邻接法分子系统发育树(图3)和最大似然法分子系统发育树(图4),邻接法树和最大似然法树拓扑结构图基本一致。由系统进化树可以看出,鳜亚科鱼类分为两大分支,中国少鳞鳜、日本少鳞鳜和朝鲜少鳞鳜聚为一支,长体鳜和另5种鳜鱼单倍型与本文大眼鳜5种单倍型簇聚成一支。

图3 基于COⅠ基因片段鳜亚科鱼类的邻接法系统发生树树枝处的数字为Bootstrap值,重复次数为1000次;Hap1~Hap5,喀斯特山区大眼鳜群体单倍型.下同.
2.3 喀斯特山区大眼鳜群体的遗传多样性和遗传分化
通过DNA SP 计算得到喀斯特山区3个群体的遗传多样性参数(表4),结果显示,沿河群体的多样性参数多态位点数、单倍型数、单倍型多样性指数、核苷酸多样性指数和平均核苷酸差异数均远远高于其他2个群体,其遗传变异最大,遗传多样性最丰富。

表4 喀斯特山区3个大眼鳜群体的遗传多样性参数
喀斯特山区大眼鳜3个群体的遗传分化指数和基因流见表5。由表5可知,铜仁群体与关岭群体之间的遗传分化指数最大为1,沿河群体与关岭群体之间的遗传分化指数最小,为0.13398;铜仁群体与沿河群体、关岭群体之间的基因流小于1,沿河群体与关岭群体之间的基因流大于1。

图4 基于COⅠ基因片段鳜亚科鱼类的最大似然法系统发生树

种群1种群2群体间遗传距离遗传分化指数基因分化系数基因流铜仁群体沿河群体0.0010.793370.608300.10588铜仁群体关岭群体0.0011.000001.000000.00000沿河群体关岭群体0.0000.133980.109893.94393
3 讨 论
3.1 喀斯特山区大眼鳜COⅠ基因片段变异及特征分析
COⅠ基因在同一种群内COⅠ基因5′端654 bp左右的片段相对保守,在3′端变异较大[12-13]。本研究初步分析并比较了喀斯特山区3个大眼鳜群体的COⅠ基因片段的差异,结果显示,169尾大眼鳜COⅠ基因片段中,除在513 nt处发生了碱基替换外,其他的变异位点均发生在880 nt以后。Hap5为30尾铜仁群体独有单倍型,其在933 nt处存在C→T替换,通过检索GenBank发现,该位点的碱基种类与同属中斑鳜和暗鳜一致,而这三种鳜类形态差异很大,因此该位点可以作为铜仁大眼鳜的分子标记。
本研究中,大眼鳜COⅠ基因的碱基C含量最高,通过检索GenBank中鳜亚科鱼类的同源序列发现,与其同属的翘嘴鳜、斑鳜、波纹鳜和暗鳜碱基含量特点一致,而少鳞鳜属的中国少鳞鳜、朝鲜少鳞鳜、日本少鳞鳜T的含量最高。从原核生物到真核生物,其基因组中广泛存在碱基使用偏向性的现象。研究发现喀斯特山区大眼鳜G的含量最低(17.8%),AT 含量明显高于GC 含量,与洞庭湖大眼鳜碱基组成一致,也与其他鱼类COⅠ基因同源序列的碱基特点一致[14-16],可见COⅠ基因对碱基的使用有明显的偏向。Fedorov等认为[17],这一现象的产生可能与基因的表达水平、翻译起始效应、碱基组分、基因长度以及密码子反密码子间结合能力的大小有关。蛋白编码基因在第3位密码子碱基上受到的功能限制较小,所以密码子第3位碱基能够更清晰地表明COⅠ基因核苷酸组成的不均一性。本研究中,喀斯特山区大眼鳜3个群体在密码子第3位各碱基的含量无差别,与翘嘴鳜比较,3个密码子位点上对各碱基的使用率差别很小,但与斑鳜、暗鳜和长身鳜比较,除密码子第2位上各碱基的使用率无差异外,密码子第1位和第3位上存在不同差异。可见,亲缘关系越近,各密码子上的碱基组成越相似。
COⅠ 基因是蛋白质编码基因之一,对于蛋白质编码区域而言,因蛋白质功能上的需要和三联体密码子结构的限制,缺失/插入很少发生或发生后很容易被选择淘汰[18]。本研究没有发现碱基的插入/缺失,变异为碱基替换,喀斯特山区大眼鳜群体转换/颠换比为4,碱基变化均发生在第3位密码子上的同义突变,与COⅠ基因遗传密码的特性保持一致。洞庭湖大眼鳜COⅠ基因与喀斯特山区大眼鳜群体差异较大,洞庭湖大眼鳜的碱基变异来源于第1位(转换/颠换比为4)和第2位密码子(转换/颠换比为2/3),使10个氨基酸发生了变化,这种差异可能与生存环境有关。
3.2 喀斯特山区大眼鳜遗传距离及鳜亚科鱼类的系统关系
Hebert等[13]经过对13 320个动物物种的分析认为,物种内COⅠ序列的遗传距离一般小于0.01,最大不超过0.02,而遗传距离大于0.01,就认为群体的变异大[19]。喀斯特山区3个大眼鳜群体COⅠ基因在群体内的遗传距离为0.000~0.002,群体间的遗传距离为0.000~0.001,小于0.01;5个单倍型与洞庭湖大眼间的遗传距离为0.007~0.008,也小于0.01。说明这些大眼鳜群体的遗传变异小。
鳜亚科鱼类的系统关系一直存在争议,本研究所建的系统进化树与刘焕章等[20]研究的鳜亚科分成鳜属和少鳞鳜属的聚类一致,长体鳜没有单独生成一枝,而是与鳜属鱼类聚在一起;本文的5种单倍型与大眼鳜和翘嘴鳜以99%的置信度聚在一起,表明大眼鳜与翘嘴鳜亲缘关系密切,而且遗传距离(0.001~0.008)并未到达种的分化标准,这与赵金良等[21-23]认为翘嘴鳜和大眼鳜是近缘姐妹种的结果一致。
3.3 喀斯特山区大眼鳜群体的遗传多样性及遗传分化
单倍型多样性指数和核苷酸多样性指数是评价物种遗传多样性的2个重要指标,值越大遗传多样性越丰富。喀斯特山区3个大眼鳜群体中,沿河群体遗传多样性较其他2个群体高,其原因可能是由于乌江流域面积广阔且又与长江连通,大眼鳜能进行广泛的基因交流,虽然沿河段下游彭水电站现今阻碍了沿河群体与下游的其他大眼鳜群体的基因交流,但彭水电站投入使用时间不足10年,所以该大眼鳜群体保存了较高的遗传多样性水平;铜仁群体单倍型单一,其可能是通过少数个体近亲繁殖而来;北盘江关岭段位于光照大坝上游,大眼鳜资源丰富但遗传多样性水平低,这可能是由于大坝的建成,为其创造一个适宜的生活环境使其大量繁殖。3个群体总体上单倍型多样性指数(0.395)低于其他鱼类[如太湖新银鱼(Neosalanxtaihuensis)为0.576、异育银鲫(Carassiusauratusgibelio)为0.690、棘头梅童鱼(Collichthyslucidus)群体为0.785][15,24-25],表现为低单倍型多样性(0.395),低核苷酸多样(0.00029),这种状况表明其可能最近出现过种群瓶颈效应或奠基者效应[26]。
基因流和遗传分化系数通常用来判断群体间遗传分化程度。若基因流大于1,表明种群间存在一定的基因交流;若基因流大于4,表明种群之间是一随机交配的群体;若基因流小于1,预示着有隔离的产生[27]。本研究中,沿河群体和关岭群体之间的基因流大于1,表明两者之间存在一定的基因交流,而北盘江与乌江是相互隔离的水体,理论上不应该有基因交流,造成这种基因交流原因,可能是2个群体曾经可能生活在一个水体中,后来由于地质变迁等因素使其彼此隔离。铜仁群体与其他两个群体的基因流均小于1,说明铜仁群体与其他两个群体有隔离,缺乏基因交流,造成这种隔离的原因,可能与生存环境有关。按照群体遗传学的标准[28],沿河群体和关岭群体间低度分化(0.15>遗传分化系数>0.05) ,而铜仁群体与其他两个群体间的遗传分化程度高 (遗传分化系数>0.25) 。
3.4 喀斯特山区大眼鳜资源的保护
遗传多样性丰富度直接反映出了物种的进化潜力,遗传多样性越丰富,物种对环境的适应能力也就越大,生存和进化的潜力也就越大[29]。本研究中,沿河群体的遗传多样性明显高于铜仁群体和关岭群体,因此应优先对沿河群体进行保护,防止稀有等位基因丢失。对于遗传多样性贫乏的铜仁群体和关岭群体,应尽快采取有效措施(如人工引种、放流等)来扩大其遗传变异和数量。
[1] 周才武, 杨青, 蔡德霖. 鳜亚科SINIPERCINAE鱼类的分类整理和地理分布[J]. 动物学研究, 1988, 9(2):113-125.
[2] 李思忠. 鳜亚科鱼类地理分布的研究[J]. 动物学杂志, 1991, 26(4):40-44.
[3] 禹真, 冉辉, 樊均德, 等. 锦江河国家级水产种质资源保护区鳜类资源调查[J]. 贵州农业科学, 2013, 41(11):146-148.
[4] 冉辉, 姚俊杰, 杨兴, 等. 锦江河大眼鳜和斑鳜肌肉中脂肪酸组成的比较[J]. 淡水渔业, 2012, 42(4):91-93.
[5] Ward R D, Hanner R, Hebert P D N. The campaign to DNA barcode all fishes, FISH-BOL[J]. Journal of Fish Biology, 2009, 74(2):329-356.
[6] 彭士明, 施兆鸿, 侯俊利. 基于线粒体 D-loop 区与 COⅠ 基因序列比较分析养殖与野生银鲳群体遗传多样性[J]. 水产学报, 2010,34 (1):19-25.
[7] 冯建彬, 孙悦娜, 程熙,等. 我国五大淡水湖日本沼虾线粒体 COⅠ 基因部分片段序列比较[J]. 水产学报, 2008, 32(4):517-525.
[8] 孟学平,高如承,申欣,等.基于COⅠ的中国西施舌DNA条形码[J].水产科学,2011,30(10):626-630.
[9] 陈淑吟,吉红九,张雪娜,等. 西施舌线粒体COⅠ与16S rRNA基因片段序列测定及其分析[J].水产科学,2010,29(8):485-488.
[10] 齐兴柱,尹绍武,张本,等. 基于mtDNA-COⅡ基因序列对中国南海裸胸鳝属鱼类分子系统进化关系的研究[J].水产科学,2010,29(10):605-609.
[11] 毕相东,杨雷,侯俊利,等.COⅠ基因在海洋动物分子系统学研究中的应用[J].水产科学,2008,27(2):105-108.
[12] Hebert P D N, Cywinska A, Ball S L. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London B:Biological Sciences, 2003, 270(1512):313-321.
[13] Hebert P D N, Ratnasingham S, de Waard J R. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society of London B:Biological Sciences, 2003, 270(Suppl 1):S96-S99.
[14] 柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体 COⅠ基因的 DNA 条形码在石首鱼科 (Sciaenidae)鱼类系统分类中的应用[J]. 海洋与湖沼, 2010, 41(2):223-232.
[15] 张迪, 雷光春, 龚成, 等. 基于 COⅠ 基因序列的太湖新银鱼遗传多样性[J]. 湖泊科学, 2012, 24(2):299-306.
[15] 于亚男, 宋超, 侯俊利, 等. 基于线粒体 COⅠ 基因部分序列的长江口虾虎鱼科鱼类系统分类[J]. 淡水渔业, 2014, 44(5):3-8.
[17] Fedorov A, Saxonov S, Gilbert W. Regularities of context-dependent codon bias in eukaryotic genes[J]. Nucleic Acids Research, 2002, 30(5):1192-1197.
[18] 单云晶, 鲁翠云, 李超, 等. 基于线粒体 COⅠ 基因序列的 5 种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 20(5):931-938.
[19] Lan H, Shi L. The origin and genetic differentiation of native breeds of pigs in southwest China:an approach from mitochondrial DNA polymorphism[J]. Biochemical Genetics, 1993, 31(1/2):51-60.
[20] 刘焕章, 陈宜瑜. 鱖类系统发育的研究及若干种类的有效性探讨[J]. 动物学研究, 1994, 15(增刊):1-12.
[21] 赵金良, 王伟伟, 李思发, 等. 鳜类鱼类的线粒体 DNA 控制区结构及其系统发育分析[J]. 遗传学报, 2006, 33(9):793-799.
[22] 余帆洋. 长江鳜和大眼鳜复合种的遗传多样性研究 [D]. 广州:暨南大学, 2011.
[23] Chen D, Guo X, Nie P. Phylogenetic studies of sinipercid fish (Perciformes:Sinipercidae) based on multiple genes, with first application of an immune-related gene, the virus-induced protein (viperin) gene[J]. Molecular Phylogenetics and Evolution, 2010, 55(3):1167-1176.
[24] 孟玮, 郭焱, 海萨, 等. 额尔齐斯河银鲫形态学及 COⅠ 基因序列分析[J]. 淡水渔业, 2010, 40(5):22-26.
[25] 赵明, 宋炜, 马春艳, 等. 基于线粒体 COⅠ 基因序列的棘头梅童鱼 7 个野生群体遗传结构分析[J]. 中国水产科学, 2015, 22(2):233-242.
[26] Grant W A S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 1998, 89(5):415-426.
[27] (美)Nei M, Kumar S著.吕宝忠等译.分子进化与系统发育[M]. 北京:高等教育出版社, 2002.
[28] Foster C B, Aswath K, Chanock S J, et al. Polymorphism analysis of six selenoprotein genes:support for a selective sweep at the glutathione peroxidase 1 locus (3p21) in Asian populations[J]. BMC Genetics, 2006, 7(1):56.
[29] 张峻德, 赵良杰, 刘其根. 千岛湖细鳞鲴和黄尾鲴 COⅠ 种群遗传结构比较的初步研究[J]. 淡水渔业, 2013, 43(5):3-7.
SequenceAnalysisofCytochromeCOxidaseSubunitⅠ (COⅠ)ofMitochondrialDNAGeneofBigeyeMandarinfishSinipercakneriinKarstMountain
GUO Jiankang1, AN Miao1, ZHOU Qichun2, CAO Hengyuan1, LI Shan1, CHEN Xueweijie1
( 1.College of Animal Science, Guizhou University, Guiyang 550025, China;2.Gui Zhou Fisheries Research Institute, Guiyang 550025, China )
The partial mitochondrial COⅠ sequences of 169 individuals of bigeye mandarinfishSinipercaknerifrom three populations in Karst Mountain were amplified by PCR technique to analyze their sequence variation and the 1512 bp nucleotide sequences were obtained.The amplification showed that the average contents of A, T, G and C were 24.9%,28.6%,28.7% and 17.8%, respectively, the average content of AT(53.5%) being higher than that of GC(46.5%). Five variable sites were detected and five different haplotypes were defined in 169 samples of the populations, and the 10 variable loci were detected between the populations ofS.kneriin Karst Mountain andS.kneriin Dongting lake, most of which were occurred from 1268 nt to 1456 nt. There were no difference about codon usage among the three populations in Karst Mountain. Comparing toS.kneriin Dongting lake, there were no significant differences in the first positions of codon usage, higher content of T(40.9%) than the latter (39.9%) in the second codon positions, whereas the content of A and C was lower than the latter in the third codon positions. The codon usage was small discriminatory between bigeye mandarinfish in Karst Mountain and mandarinfishS.chuatsiin Dongting lake. Comparison ofS.scherzeri,S.obscura,S.rouleiandS.undulatarevealed different discriminatory at the first codon positions and third codon positions, especially significantly lower content of A(30.0%) inS.obscuraandS.undulataat the codon first positions than that(31.8%) inS.akneriin Karst mountain, whereas G content (8.9%) ofS.undulatawas significantly higher than the latter (7.3%). The total haplotype diversity (Hd) was 0.395 and nucleotide diversity(Pi) was 0.00029, therefore, indicating a low genetic diversity level in the populations ofS.kneriin Karst Mountain.
Karst Mountain;Sinipercakneri; COⅠ gene; codon; genetic diversity
10.16378/j.cnki.1003-1111.2016.06.008
Q959.4
A
1003-1111(2016)06-0649-09
2015-11-25;
2016-03-14.
郭健康(1987—),男,硕士研究生;研究方向:水产动物增养殖学.E-mail:gzguojk@126.com.通讯作者:安苗(1970—),男,副教授,硕士;研究方向:水生动物增养殖与鱼类种质资源.E-mail:gzuam@163.com.
