安徽桑树品种全基因组DNA提取方法研究
2016-12-20王钰婷李瑞雪王伟汪泰初
王钰婷,李瑞雪,王伟,汪泰初
(安徽省农业科学院蚕桑研究所,合肥230061)
安徽桑树品种全基因组DNA提取方法研究
王钰婷,李瑞雪,王伟,汪泰初
(安徽省农业科学院蚕桑研究所,合肥230061)
旨在获得一种高效、稳定、廉价的DNA提取方法。以安徽桑树品种嫩叶为材料,以CTAB法提取其基因组DNA,并用紫外分光光度计、凝胶电泳、PCR扩增等方法进行鉴定。研究结果表明:提取的DNAA260/A280比值在1.80~1.90之间,DNA得率约为0.187~0.275 μg/mg,以提取的DNA为模版,PCR能够获得清晰的目的条带。证明CTAB法提取的DNA质量较好,能满足后续分子生物学操作的需要。
桑树;DNA提取;PCR扩增
0 引言
中国是蚕桑生产的发源地,拥有极其丰富的桑树种质资源。随着分子生物学技术的快速发展,从分子水平上对桑树种质资源的遗传变异进行研究已成为桑树分子生物学研究热点之一。向仲怀等[1]运用RAPD技术构建了桑属9个种的DNA指纹图谱,对桑属的植物分类进行了开创性的研究,开辟了中国分子标记技术研究桑树种质的先例。基因组DNA的提取是进行分子标记技术研究的关键步骤之一,高质量基因组DNA的获取是进行桑树生物学方面研究的基础。由于桑树是多年生木本植物,组织细胞含有大量的多糖、多酚、酯类等复杂次生代谢产物,因而要从中提取高质量的DNA有较大困难[2-5]。
本实验采用十六烷基三甲基溴化铵(CTAB)法对安徽特色桑树品种的嫩叶进行DNA的提取,在实验过程中,优化了DNA提取程序,获得了高得率、高纯度的DNA,能成功地应用于后续的分子生物学反应。旨在为桑树基因组DNA的研究提供理论与技术参考,也为进一步深入开展其亲缘关系分析、基因组比较、遗传多样性分析等分子生物学研究提供一定的基础资料。
1 材料与方法
1.1 实验材料
选取5种安徽桑树地方品种(样本采自安徽省农业科学院蚕桑研究所桑树品种选育中心)。以桑嫩叶为实验材料,品种分别为:‘金寨九龙桑’、‘皖桑1号’、‘红星5号’、‘皖花桑’、‘7707’。
1.2 DNA提取方法
桑树基因组DNA提取采用CTAB法,参照赵卫国[2]的方法,稍加改进。
①取0.1~0.2 g嫩叶剪碎,在液氮条件下磨碎成粉末,转入经55℃预热的600 μL的CTAB提取缓冲液(0.1 mol/LTris·HCl,pH 8.0,0.02 mol/LEDTA,1.4 mol/L NaCl,2%CTAB,1%PVP,0.04 mol/Lβ-巯基乙醇)中;②混匀后在65℃水浴裂解30min,期间轻摇数次以混匀,室温冷却,12000 r/min离心15 min;③加入500 μL氯仿:异戊醇(24:1),抽提细胞残渣和叶绿体,轻轻颠倒混匀至乳白色;常温13000 r/min离心10 min;④转移上清液到新离心管中,加入等体积的预冷的异丙醇混匀,室温静置3~5 min;⑤常温下,10000 r/min高速离心5 min,弃上清后倒置干燥,加500 μL ddH2O溶解;加入2 μL RNase(10mg/mL),37℃水浴1 h;⑥加等体积酚抽提1次,酚:氯仿:异戊醇(25:24:1)抽提1次,氯仿:异戊醇(24:1)抽提1次;⑦加2倍体积无水乙醇沉淀DNA,轻轻混匀,-20℃过夜;⑧13000 r/min离心10 min,弃上清,用70%无水乙醇洗涤DNA2次,每次8000r/min离心10min;倒置干燥后加入200 μL的TE缓冲液(10 mmol/L Tris-HCl pH 8.0,1 mmol/L EDTApH 8.0)溶解DNA,样品保存于4℃或-20℃。
1.3 DNA质量检测方法
1.3.1 D(λ)法DNA完全溶解后,吸取5 μL DNA溶液,加95 μL TE,稀释100倍后,在DU-70紫外分光光度计上测定OD260和OD280值。
1.3.2 电泳分析取DNA样品3 μL与1 μL 6×Loading buffer混匀,在1%琼脂糖凝胶(含EB)上电泳检测DNA质量,反应混合物在1×TAE缓冲液中120V电泳30min。电泳结果在Tanon(Gis-2008)凝胶成像系统拍照。
1.3.3 PCR分析参照文献[6]设计扩增引物ITS5(5'-GGAAGTAAAAGTCGTAACAAGG-3')和ITS4(5'-TCCTCCGCTTATTGATATGC-3')。PCR反应体系总体积25 μL:2*Master Mix 12.5 μL;模板DNA 1.0 μL;引物各1.0 μL(浓度为10 ng/μL);ddH2O 11.5 μL。PCR扩增程序如下:95℃预变性5 min;95℃变性30 s,60℃退火1 min,72℃延伸1 min,35个循环;72℃延伸5 min;PCR产物于4℃保存。
2 结果与分析
2.1 DNA样品纯度及得率
5种样品A260/A280比值均在1.80~1.90之间。说明以桑嫩叶为材料所提取的5种DNA样品中蛋白质、植物色素、多糖和酚类物含量较低,DNA的得率和纯度均较高,能够满足进一步分子生物学操作的需要。
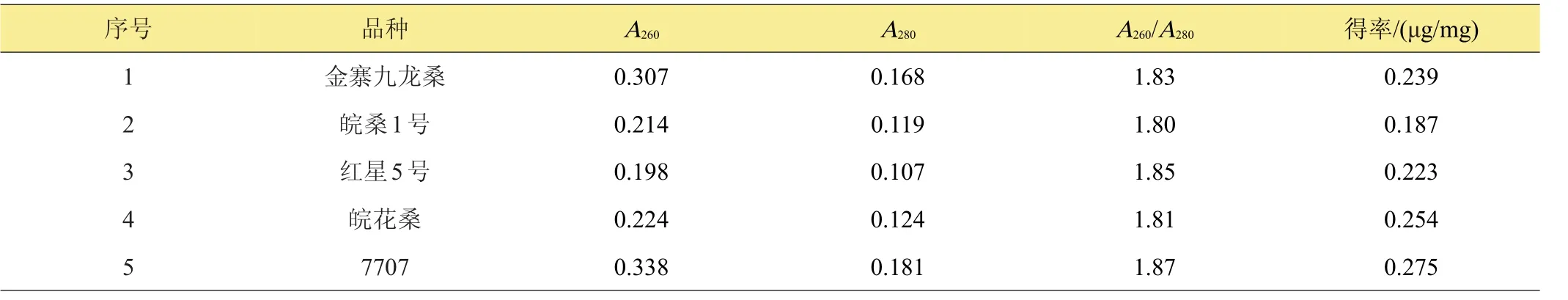
表1 5种DNA样品的测定结果
2.2 DNA样品的电泳
琼脂糖凝胶电泳结果显示,提取到的DNA总带清晰整齐,无杂带、无拖尾现象(图1),表明DNA提取纯度较高。
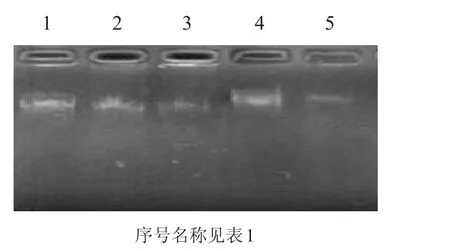
图1 桑树DNA凝胶电泳图谱

图2 提取DNA的PCR扩增结果
2.3 DNA的PCR分析
提取DNA进行PCR分析,结果显示能够获得清晰稳定的产物条带(图2),说明提取的DNA完全可用于PCR分析,可为桑树的后续分子生物学实验操作奠定基础。
3 讨论与结论
制备较完整、纯度较高的基因组DNA是进行植物分子生物学研究的基础和前提,是进一步试验的最关键因素[7-10]。植物组织与动物细胞不同,含有较高的多酚、多糖、色素和脂类物质等干扰物质,在多年生木本植物中含量更高,高质量DNA的提取更为困难[11-13]。
桑树是多年生木本植物,桑叶中含大量的多糖及多酚类物质,蛋白质含量也高于其他植物[14-15]。有研究者用氯化铯密度梯度离心法分离桑叶总DNA[16],费时且价格昂贵。本实验对传统的CTAB法进行改进和完善,尽量减少多酚、多糖、色素、果胶和脂类物质等的干扰。结果表明,采取CTAB法提取供试树种基因组DNA,结果均获得了较高质量的DNA(图1,图2)。在实验过程中,对CTAB法进行了如下改进:①在条件允许的情况下,优先选取新鲜采摘的幼嫩植物组织作为DNA提取的材料;②采集的样品及时放入液氮中冻存,减少DNA的降解;③如植物组织较老,为减少组织的褐变情况,可适当加大β-巯基乙醇的用量(可达2.5%~3%);④在组织裂解水浴加热期间添加RNaseA,有效去除了其中的RNA,而且所添加的RNase A在纯化的过程可以得到进一步的去除;⑤加入异丙醇后离心过程中,降低离心速度、缩短离心时间,能防止多酚、多糖、单宁、果胶等次生代谢物质与DNA一起被沉淀。紫外线检测、凝胶电泳及PCR鉴定分析,说明本实验采用CTAB法提取的桑树DNA可以用于后续的分子生物学研究。
[1]向仲怀,张孝勇,余茂德,等.采用随机扩增多态性DNA技术(RAPD)在桑属植物系统学研究的应用初报[J].蚕业科学,1995,21 (4):203-208.
[2]林强,邱长玉,赵卫国,等.32份广东桑类型桑树种质资源亲缘关系的SRAP标记分析[J].蚕业科学,2011,37(3):373-379.
[3]赵卫国,潘一乐.从桑树营养组织中提取高纯度DNA的方法[J].江苏蚕业,2001,28(1):19-20.
[4]林强,朱方容,邱长玉,等.广西桑树种质资源亲缘关系的SRAP分析[J].南方农业学报,2011,42(4):358-363.
[5]田智得,赵卫国,甘丽红,等.广西桑树品种全基因组DNA提取方法研究[J].广西蚕业,2006,43(2):7-10.
[6]Zhao W G,Fang R J,Pan Y L,et al.Analysis of genetic relationships of mulberry(L.)germplasm using sequence-related amplified polymorphism(SRAP)markers[J].African Journal of Biotechnology,2009,8(11):2604-2610.
[7]李军集,廖和发,刘海龙.药用植物六角莲基因组DNA提取方法[J].广西林业科学,2007,36(2):89-92.
[8]戴瑞强.华桑遗传多样性分析及核心种质构建[D].镇江:江苏科技大学,2010.
[9]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenctics[M]. California:Academic Press,1990:315-322.
[10]李金璐,王硕,于婧,等.一种改良的植物DNA提取方法[J].植物学报,2013,48(1):72-78.
[11]丁浩,郑唐春,梁德洋,等.一种简易高效提取多种植物纯净DNA的方法[J].植物研究,2015,35(3):457-461.
[12]赵卫国.桑种质资源的遗传多样性及分子系统学研究[D].北京:中国农业科学院,2005.
[13]赵卫国,潘一乐.从桑树营养组织中提取高纯度DNA的方法[J].江苏蚕业,2001,28(1):19-20.
[14]林强,邱长玉,朱方容,等.桑树二倍体及其人工诱导同源四倍体遗传差异的RAPD分析[J].南方农业学报,2011,42(1):11-15.
[15]孙璐宏,鲁周民,张丽.植物基因组DNA提取与纯化研究进展[J].西北林学院学报,2010,25(6):102-106.
[16]町井博明.桑全DNA的分离[J].国外农学-蚕业,1990(2):60-61.
Genomic DNA Extraction Method of Mulberry Varieties in Anhui
Wang Yuting,Li Ruixue,Wang Wei,Wang Taichu
(The Sericultural Research Institute,Anhui Academy of Agricultural Science,Hefei 230061,Anhui,China)
The aim is to explore an efficient,stable,low-cost DNA extraction method.The total genomic DNA was extracted from mulberry leaves with CTAB in Anhui.Ultraviolet spectrophotometer,agarose gel electrophoresis and PCR methods were used to test the DNA quality.The results showed that the value of A260/ A280was 1.80-1.90,DNA yield was 0.187-0.275 μg/mg.The extracted DNA was used as templates,the clear target bands were obtained by PCR amplification.It indicated that CTAB method was suitable to extract the total genomic DNA from leaves of mulberry,which could meet the need of subsequent analysis of molecular biology.
Mulberry;DNA Extraction;PCR Amplification
S-186
A论文编号:cjas16050018
安徽省农科院院长青年创新基金“安徽省地方桑树种质资源ITS序列及进化分析研究”(15B0634);国家蚕桑产业技术体系合肥综合试验站“现代农业产业技术体系建设专项”(CARS-22-SYZ09);安徽省农科院创新团队项目“多功能生态桑树品种的选育与利用”(11C0610)。
王钰婷,女,1985年出生,安徽岳西人,助理研究员,硕士,研究方向:分子遗传学。通信地址:230061安徽省合肥市蜀山区霍山路15号安徽省农业科学院蚕桑研究所,Tel:0551-62842195,E-mail:wyt@ahaas.com。
汪泰初,男,1970年出生,安徽池州人,研究员,硕士,研究方向:桑树栽培育种。通信地址:230061安徽省合肥市蜀山区霍山路15号安徽省农业科学院蚕桑研究所,Tel:0551-62842195,E-mail:18949853828@126.cn。
2016-05-30,
2016-08-11。
