菊粉酶的研究进展
2016-12-14张天祥丁宁杨春光孙慎侠王克鑫马君燕
张天祥,丁宁,杨春光,孙慎侠,王克鑫,马君燕*
(大连大学医学院,辽宁大连116622)
菊粉酶的研究进展
张天祥,丁宁,杨春光,孙慎侠,王克鑫,马君燕*
(大连大学医学院,辽宁大连116622)
菊粉酶是一类能水解β-2,1-D-果聚糖果糖苷键的水解酶,属于糖苷水解酶32家族。菊粉酶是菊芋生物炼制中的关键酶,它能将菊芋一步水解,获得高纯度的果糖浆,生产燃料乙醇、燃料丁醇、单细胞油脂、生物柴油、甘露醇和乳酸等其它工业产品,在食品、医药及生物能源等领域有着巨大的应用价值。该文介绍了菊粉的分子结构及来源,概述了菊粉酶的分类、来源、克隆表达、三维结构及应用,并对菊粉酶的未来发展方向进行了展望,旨在提高人们对菊粉酶的认识,推动菊芋生物炼制的研究。
菊粉酶;糖苷水解酶32家族;三维结构;生物炼制
随着不可再生能源危机的加剧,寻找新的可再生资源以摆脱对化石资源的依赖,是21世纪最为重要而又艰巨的任务之一。生物质是自然界最丰富的含碳有机大分子功能体,它可以通过生物炼制生产燃料乙醇、燃料丁醇等新的可再生生物质能源。利用来源于粮食作物的淀粉作为原料生产生物质能源已不符合社会发展的需要,所以利用非粮作物生产生物质能源产品成为中国乃至全世界关注的焦点。
菊芋俗名洋姜、鬼子姜,多年生草本植物,具有种植简单、产量高、生命力顽强、耐贫瘠、耐寒、耐旱、耐盐碱等特性,所以被广泛种植于不适宜于粮食作物和经济作物生长的盐碱、滩涂和荒漠地。每年我国菊芋的产量约为5.4 t/hm2[1]。其低廉的价格、较高的产量,更为重要的是属于非粮作物,从而使菊芋成为生物炼制关注的焦点。近年来,菊芋作为果糖浆、低聚果糖、生物乙醇、2,3-丁二醇及其他化学品的生物质原料受到广泛关注[2]。开发利用菊芋,成为生物炼制研究领域中一个重要的研究方向,在食品、医药及生物能源等行业有着巨大的开发价值和商业潜力。菊粉(inulin)又称菊糖,是一种广泛存在于自然界中的天然果聚糖,主要由果糖和葡萄糖组成,其中果糖分子通过β-2,1糖苷键相连。
菊粉酶(inulinase)EC3.2.1是一类能水解β-2,1-D-果聚糖的水解酶,广泛存在于微生物和植物中,尤其是丝状真菌和酵母菌[3-4]。菊粉酶可以在一定的温度条件下水解菊粉成果糖,广泛用于食品、医药和生物能源等行业中。因此,开发利用菊粉酶,成为科研人员关注的焦点。早在1900年,LINDER P发现马克斯克鲁维酵母(Saccharomyces marxianus)以及其他一些酵母菌能够利用菊粉。随后,其他研究者发现脆壁克鲁维酵母(Kluyveromyuces fragilis)能够在菊粉上生长。1924年,PRINGSHEIMH等首次报道了黑曲霉(Aspergillus niger)能产菊粉酶[5]。1991年,LALOUX O等[6]利用分子生物学方法,首次克隆了外切菊粉酶基因INU1。近年来,科研人员从微生物菊粉酶的菌株筛选、诱变改造、酶学性质、克隆表达及同步糖化发酵等方面进行了较多的研究,并初步开始解析其结构和功能基团,然而,我国关于菊粉酶的研究比较晚,从20世纪90年代初开始,滞后于欧美等国家,现有菊粉酶在活力和稳定性方面仍较难达到工业化生产的要求。
本文介绍了菊粉的分子结构及来源,概述了菊粉酶的分类、来源、克隆表达、三维结构及应用,并展望了菊粉酶的未来发展方向,以期提高人们对菊粉酶的认识,为进一步推动菊粉酶在菊芋生物炼制中的应用奠定基础。
1 菊粉
1.1 菊粉的分子结构
菊粉是由D-呋喃果糖分子通过β-2,1-糖苷键相连而成的链状多糖,其末端与一分子葡萄糖残基相连[3],分子式为GFn,其中G为末端葡萄糖残基,F代表果糖分子,n为聚合的果糖个数,菊粉酶结构如图1。菊粉的聚合度一般为10~30,与植物来源、生长环境、收获期和存储时间等因素相关[7],聚合度的差异对其溶解度影响较大,通常短链菊粉比长链菊粉易溶于水。
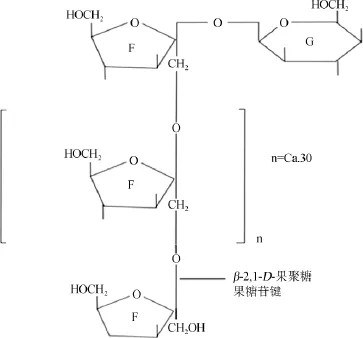
图1 菊粉的分子结构[1]Fig.1 Molecule structure of inulin
1.2 菊粉的来源
菊粉是继淀粉之后,植物中第二大储藏性多糖,目前,已经在36 000多种植物组织中发现了菊粉的存在,数量约为植物种类总数的1/3,包括双子叶植物中的菊科、橘梗科、龙胆科等11个科以及单子叶植物中的百合科、禾本科,都含有丰富的菊粉。然而,菊粉主要存在于菊科植物中,如广泛存在于菊芋(Jerusalem artichoke)、菊苣(Chicorium intybus)、大丽花(Dahlia)和雪莲果(Smallanthus sonchifolius)等植物的根和块茎中[2]。其中菊粉在菊芋块茎中的含量,可达其干质量的70%以上[8]。
菊粉可通过酸法(pH=1.0~2.0,80~100℃)或酶法直接转化为易于利用的果糖浆。然而,酸法处理时,菊粉易形成没有甜味带颜色的副产物,如5-羟基康甲醛,而且产率低(<45%),而酶法水解菊粉,具有工艺简单、转化率高、产物纯、含量高等优点,尤其是菊粉酶通过一步水解,高达95%的果糖产率[5]。因而,菊粉酶作为菊芋生物炼制中的关键酶越来越受到重视[5]。
2 菊粉酶
2.1 菊粉酶的分类
根据对底物作用方式的不同,菊粉酶可以分成外切菊粉酶和内切菊粉酶。外切菊粉酶从底物分子的非还原末端催化水解下果糖残基产生高果糖浆,其底物可以是菊粉、蔗糖或果聚糖[9];内切菊粉酶催化水解菊粉内部的β-2,1-糖苷键,使其变成三糖、四糖或五糖为主的低聚果糖,但其缺少蔗糖酶活力[1,10]。通常用I/S的大小来区分内切菊粉酶和外切菊粉酶,I是以菊粉作底物时的酶活,S是以蔗糖作底物时的酶活,I/S大于10-2定义为菊粉酶,而I/S小于10-4定义为蔗糖酶。根据菊粉酶在微生物体内的主要分布可将其分为胞内酶、胞壁结合酶和胞外酶,它们的比例主要受菌种、碳源、温度和pH的影响。根据来源,菊粉酶可以分为微生物菊粉酶和植物菊粉酶。
2.2 菊粉酶的来源
菊粉酶的来源很广,自然界中的植物以及土壤、水和动物消化道中的多种微生物都可以分泌菊粉酶。植物来源的菊粉酶主要见于菊科植物,如菊芋的块茎、天竺牡丹(大理菊)的块根、蓟的根等。植物来源的菊粉酶底物专一性强,只对菊粉具有催化活性,然而含量甚微;微生物来源的菊粉酶底物范围广,大多都能水解含有β-2,1呋喃果糖苷键的碳水化合物,如菊粉、蔗糖和棉子糖。但微生物来源的菊粉酶种类多,热稳定性好,而且微生物生长迅速,培养简单,且有些菌株可产生大量的酶,所以对菊粉酶的研究和利用主要针对各种微生物来源的酶。
根据报道,酵母菌约有10个属20余种[2]、丝状真菌约有17个属40余种[2]、细菌约有12个属10余种都能够产生菊粉酶[2],尤其是丝状真菌和酵母菌[11]。其中,酵母菌产菊粉酶的能力比真菌和细菌产菊粉酶的能力强[12]。酵母菌中的马克斯克鲁维酵母(Kluyveromyces marxianus)、脆壁克鲁维酵母(Kluyveromyces fragilis)、毕赤酵母(Pichia pastoris)和金黄色隐球酵母(Cryptococcus aureus)具有较高而且稳定的产酶活性,存在着潜在的工业应用价值[5]。尤其是马克斯克鲁维酵母。由于它和酿酒酵母(Saccharomyces cerevisiae)进化关系最近,有共同的进化起源,与乳酸克鲁维酵母(Klyuveromyces lactis)是姐妹菌株,可以分解乳糖和菊粉,并且具有生长快速、繁殖时间短、耐热性好、能大量分泌外源蛋白、食品安性高等优点,使其在生物技术应用领域面临的限制小,更加具有商业价值。K.marxianus CDBB-L-278菌株在以甘油和菊粉为碳源的培养基中生长,分泌的外切菊粉酶活力达34.78 U/mL[13]。SINGHR S等[14]报道以质量浓度2 g/100 mL的大丽花块茎提取物为碳源,培养K.marxianusYS-1菌株,在通气振荡条件下,60 h后,获得55.4 U/mL的外切菊粉酶。SHENG J等[15]发现在含有4%菊粉和0.5%酵母粉pH 5.0的人工海水中,28℃培养42 h后,CryptococcusaureusG7a酵母菌产外切菊粉酶的活力达85 U/mL。GONG F等[16]发现48 h摇瓶发酵后,季也蒙毕赤酵母(Pichia guilliermondii)产菊粉酶的活力为60 U/mL。
丝状真菌中的曲霉菌属(Aspergillussp.)和青霉菌属(Penicilliumsp.)也是很好的生产菊粉酶的来源[2]。其中,曲霉菌属是研究最多的真菌微生物。从土壤分离的无花果曲霉(Aspergillusficuum)JNSP5-06,在含菊粉的培养基中,30℃培养5 d后,产菊粉酶的活力为25 U/mL[17]。
虽然,细菌来源的菊粉酶产量远不及酵母菌和丝状真菌,但某些菌株能在较高的温度生长。为了寻找适合工业生产的高温酶,研究者对某些产菊粉酶的细菌菌株也展开了一定的研究,主要是芽孢杆菌属(Bacillussp.)[18]、假单胞菌属(Pseudomonassp.)和链霉菌属(Streptomycessp.)[19-20]。嗜热芽抱杆菌(G.stearothermophilus)KP1289菌株在41~69℃范围内分泌外切菊粉酶,酶的最适作用温度为60℃[21]。
2.3 菊粉酶的克隆表达
随着分子生物学的发展,许多不同来源的微生物菊粉酶被克隆,并在大肠杆菌、克鲁维酵母、毕赤酵母等宿主中表达[18,22-30]。外切菊粉酶中第一个被克隆和表达的基因INUI来自马克斯克鲁维酵母(Klyuveromyces marxiarzus var.marxianus)ATCC 12424菌株[6]。INU1的开放阅读框(open reading frame,ORF)为1 668 bp,编码555个氨基酸,其中前16个氨基酸是信号肽序列,共有13个可能的糖基化位点。该序列与S.cerevisiae来源的蔗糖酶有67%的同源性,它们来自共同的祖先。
WEN T Q等[31]对高产菊粉酶的菌株K.cicerisporus CBS4857的菊粉酶基因进行了克隆和分析,此基因ORF为1 665 bp,编码555个氨基酸,前23个氨基酸为该菊粉酶基因的信号肽部分,与K.marxianusCBS6556和K.marxianus var.marxianusATCC12424菊粉酶基因的同源性分别为99%和98%。
ZHANG S F等[32]将K.marxianusCBS6556菌株的菊粉酶基因克隆并在P.pastorisX-33表达,获得具有较高菊粉酶活力的重组菌株。重组菌分泌的上清中外切菊粉酶活力对伊利菊粉和Sigma菊粉分别为6 667 U/mL和1 212 U/mL。该基因的ORF编码556个氨基酸,其氨基酸序列与S.cerevisia蔗糖酶有59%的同源性,基因序列与K.marxianusvar. marxianusATCC12424来源的菊粉酶基因有95%的同源性。基因序列的5′端含有Migl结合位点(TAAATCCGGGG),说明该菊粉酶基因的表达受葡萄糖的抑制作用。
近年来,池振明课题组从中国南海海泥中分离得到可产菊粉酶的海洋酵母C.aureusG7a和C.aureusHYA,两菌株分泌的外切菊粉酶基因的ORF分别是1557bp和1653bp,基因都不含内含子,编码518和550个氨基酸,分子质量分别为60.0 ku和59.5 ku[12,15]。
2.4 菊粉酶的三维结构
菊粉酶属于糖苷水解酶32家族(goycoside hydrolase 32,GH32),此家族有共同的3D结构特征:N-端5-折叠β-螺旋浆形成的催化区域和C-端的β-折叠三明治区[33-35],如图2。N-端的每个螺旋浆包括4个反向平行的β-折叠片,形成经典的‘W'拓扑结构,辐射状围绕着中心轴,封闭带负电荷的催化活性口袋[36]。每个β-折叠片之间由发卡环连接,其中,β-折叠片1离中心轴最近,并与中心轴平行。整个N-端结构域的形状类似圆柱形,每个螺旋浆中β-折叠片4位于圆柱筒的最外面[36]。C-端结构域由2个反向平行的β-折叠片组成,每个β-折叠片包括6个β-折叠股,组装成类似三明治的结构,此区域具有协调二聚体相互作用、识别底物与底物结合的作用[36-39]。

图2 菊粉酶的三维结构[37]Fig.2 Three-dimensional structure of inulinase
有关GH32家族结构的研究,可以追溯到20世纪90年代,早期的研究主要是通过定点突变来确定酶的催化活性中心,随后开展了保守区域氨基酸所起作用以及活性位点周围氨基酸在酶催化、空间结构稳定、底物特异性方面的研究。上述研究主要以植物果聚糖1-外源水解酶、6-外源水解酶、蔗糖酶为研究对象[37,40-41],针对微生物菊粉酶结构与功能关系的研究较少,曾有报道内外切菊粉酶底物选择性不同,是因为内切菊粉酶在催化活性中心有两个loop环形成的额外的口袋[42]。目前,GH32家族已有12种酶的晶体结构得到解析。菊粉酶有两个晶体结构得到解析,一个是泡盛曲霉(Aspergillus awamori)来源的外切菊粉酶[36],另一个是无花果曲霉来源的内切菊粉酶(晶体结构数据库(protein data bank,PDB)3RWK)。天然外切菊粉酶在相同的结晶条件下呈现出两种晶型。一种晶型是P212121斜方晶系空间群(PDB:1Y9M),另一种晶型是P21单斜晶系空间群(1Y4W),这两种晶型除了糖基化程度不一致外,其它结构基本一致[36]。晶体结构的解析可以确定酶的催化活性中心、酶与底物的结合位点及最优构象、阐明催化机制,认识结构与功能之间的关系,从而帮助研究者设计动力学性质更优越的酶,适合工业化生产的需求。
2.5 菊粉酶的应用
2.5.1 生产高果糖浆
果糖是理想的甜味剂,其甜度是蔗糖的1.8倍。其代谢不受胰岛素制约,可以供糖尿病患者食用,而且不易被口腔微生物利用,对牙齿的不利影响小于蔗糖,可以预防糖尿病、肥胖症,被认为是21世纪全球代替蔗糖、葡萄糖的新型功能性糖。果糖广泛用于食品、医药和代替蔗糖生产饮料等行业中。果糖浆可由外切菊粉酶以菊粉为原料一步催化完成,果糖含量高达95%。相对于以淀粉为原料经多步酶法生产果糖的工艺及酸法水解菊粉获得果糖的方法,具有工艺简单、转化率高、产物纯等优点。因此,菊粉酶在果糖及果葡糖浆的生产上具有巨大的开发价值和应用潜力。
2.5.2 生产低聚果糖
低聚果糖的聚合度为2~7,是纯天然、高效能、无公害的绿色食品和饲料添加剂,是一种功能性多糖,有多种生理功能,如改善肠道内微生物区系,使肠道菌群中双歧杆菌等有益菌数量增加、产气荚膜梭菌等有害菌群数量降低;改善脂质代谢,降低血脂和胆固醇;促进矿物质的吸收等。低聚果糖以其优越的生理功能,日益得到人们的广泛重视。内切菊粉酶水解菊粉可以得到高纯度的低聚果糖。不同来源的内切菊粉酶可得到不同聚合度的低聚果糖,如青霉菌属(Penicilliumsp.)TN-88内切菊粉酶的产物以二聚果糖(F2)、三聚果糖(F3)和四聚果糖(F4)为主;黄单胞菌属(Xanthomonassp.)所产内切菊粉酶的水解产物主要为聚合度为大于等于5的低聚糖。
2.5.3 生产生物乙醇
生物乙醇是一种新的可再生资源,可作为石油、煤炭等能源的替代燃料和燃料添加剂,已在中国、美国、印度等国家广泛应用。以菊芋为原料,利用外切菊粉酶生产果糖,进而通过能利用果糖的微生物(如S.cerevisiae)发酵产生生物乙醇,为解决能源枯竭问题开辟了一条新途径。相较于木质纤维素的利用过程,菊粉酶预处理简单,反应条件温和,且仅需要菊粉外切酶一步就可以把菊粉转化为果糖。国内外对此方面的研究较多[2],如ZHANG T等[23]克隆季也蒙毕赤酵母(P.guilliermondii)strain l中的外切菊粉酶基因INUI,在Saccharomycessp.WO尿嘧啶突变菌株中表达,72 h发酵培养后,外切菊粉酶活力为34.2 U/mL,加入菊粉后,重组菌株可以直接利用菊粉的水解产物进行发酵生产乙醇,从100 mL发酵液中得到13.7 mL的乙醇,果糖的转化率为99.1%。
2.5.4 其他产品
以菊芋为原料,利用外切菊粉酶的水解产物果糖和葡萄糖,制备生物丁醇、生物油脂、2,3-丁二醇、乳酸、甘露醇、单细胞蛋白等产品,广泛应用在食品、医药和化工等领域。
3 展望
从自然界中寻找生产菊粉酶的微生物是科研工作者努力的方向,但大量的菌株筛选既耗时又费力,且存在菊粉酶在天然材料中表达水平低、分离纯化困难、活性低等难题,难以达到工业化生产的要求,限制了菊芋生物炼制的工业化发展。伴随分子生物学和糖生物学的发展,20世纪80年代兴起的对现有的天然蛋白质进行改造的蛋白分子改造工程和对蛋白质进行糖基化修饰的蛋白质糖基化工程,是解决这一系列问题的有效措施,可以获得适合工业生产的高活性、表达量高及热稳定性好等理化性质优越的“量身定制”的工业菊粉酶。
[1]LIU G L,CHI Z,CHI Z M.Molecular characterization and expression of microbial inulinase genes[J].Crit Rev Microbiol,2013,39(2):152-165.
[2]HE M,WU D,WU J,et al.Enhanced expression of endoinulinase from Aspergillus nigerby codon optimization inPichia pastorisand its application in inulooligosaccharide production[J].J Ind Microbiol Biotechnol,2014,41:105-114.
[3]ARTYKHOV V G,HOLYAVKA M G,KOVALEVA T A.Structural and functional properties of inulinases.Ways to regulate their activity[J].Biophysics,2013,58(4):493-501.
[4]LIU G L,FU G Y,CHI Z,et al.Enhanced expression of the codon-optimized exo-inulinase gene from the yeastMeyerozyma guilliermondiiin Saccharomycessp.W0 and bioethanol production from inulin[J].Appl Microbiol Biotechnol,2014,98(21):9129-9138.
[5]王静,金征宇.微生物菊粉酶的研究进展[J].生物技术,2002,12(2):42-45.
[6]LALOUX O,CASSART J P,DELCO J,et al.Cloning and sequencing of the inulinase gene ofKluyveromyces marxianusvar.marxianusATCC 12424[J].FEBS Lett,1999,289(1):64-68.
[7]CHI Z M,ZHANG T,CAO T S,et al.Biotechnological potential of inulin for bioprocesses[J].Bioresource Technol,2011,102(6):4295-4303.
[8]ASHOK P,SOCCOL C R,SELVAKUMAR P,et al.Recent developments in microbial inulinases its production,properties,and industrial applications[J].Appl Biochem Biotechnol,1999,81(11):3337-3345.
[9]KANGO N,JAIN S C.Production and properties of microbial inulinases: recent advances[J].Food Biotechnol,2011,25(3):165-212.
[10]MUTANDA T,MOKOENA M P,OLANIRAN A O,et al.Microbial enzymatic production and applications of short chain fructooligosaccharides and inulooligosaccharides:recent advances and current perspectives[J].J Ind Microbiol Biotechnol,2014,41:893-906.
[11]JAIN S C,JAIN P C,KANGO N.Production of inulinase from Kluyveromyces marxianususing dahlia tuber extract[J].Braz J Microbiol,2012,43(1):62-69.
[12]CHI Z M,CHI Z,ZHANG T,et al.Inulinase-expressing microorganisms and applications of inulinases[J].Appl Microbiol Biotechnol,2009,82: 211-220.
[13]CRUZ-GUERRER A,GARCIA-PERA I,BARZANA E,et al.Kluyveromyces marxianusCDBB-L-278:a wild inulinase hyper producing strain[J].J Ferment Bioeng,1995,80(2):159-163.
[14]SINGHR S,SOOCH B S,PURI M.Optimization of medium and process parameters for theproduction of inulinase from a newly isolated KluyveromycesmarxianusYS-1[J].Bioresource Technol,2007,98(13): 2518-2525.
[15]SHENG J,CHI Z M,LI J,et al.Inulinase production by the marine yeastCryptococcus aureusG7a and inulin hydrolysis by the crude inulinase[J].Process Biochem,2007,42(5):805-811.
[16]GONG F,SHENG J,CHI Z,et al.Inulinase production by a marine yeastPichia guilliermondiiand inulin hydrolysis by the crude inulinase [J].J Ind Microbiol Biot,2007,34(3):179-185.
[17]JING W,CHEN X M,LI Y,et al.Production and separation of exo-and endoinulinase fromAspergillus ficuum[J].Process Biochem,2003,39 (1):5-11.
[18]KWON H J,JEON S J,YOU D J,et al.Cloning and characterization of an exoinulinase fromBacillus polymyxa[J].Biotechnol Lett,2003,25: 155-159.
[19]LAOWKLOM N,CHANTANAPHAN R,PINPHANICHAKARN P,et al.Production,purification and characterization of inulinase from a newlyisolatedStreptomycessp.CP01[J].Nat Resour,2012,3:137-144. [20]SHARMA A D,GILL P K.Purification and characterization of heat-stable exo-inulinase fromStreptomycessp.[J].J Food Eng,2007,79(4): 1172-1178.
[21]TSUJIMOTO Y,WATANABE A,NAKANO K,et al.Purification and properties of a thermostable inulinase fromBacillus stearothermophilus KP1289[J].Appl Microbiol Biotechnol,2003,62:180-185.
[22]SOKOLENKO G G,KARPECHENKO N A.Expression of inulinase genes in the yeastsSaccharomyces cerevisiaeandKluyveromyces marxianus[J].Microbiology,2015,84(1):23-27.
[23]ZHANG T,CHI Z,CHI Z M,et al.Expression of the inulinase gene from the marine-derivedPichia guilliermondiiinSaccharomycessp. W0 and ethanol production from inulin[J].Microb Biotechnol,2010,3 (5):576-582.
[24]ZHIOU J P,GAO Y J,ZHANG R,et al.A novel low-temperature-active exo-inulinase identified based on molecular-activity strategy fromSphingobacteriumsp.GN25 isolated from feces ofGrus nigricollis[J].Process Biochem,2014,49(10):1656-1663.
[25]MORIYAMA S,TANAKA H,UWATAKI M,et al.Molecular cloning and characterization of an exoinulinase gene fromAspergilhs niger strain 12 and its expression inPichia pastoris[J].J Biosci Bioeng,2003, 96(4):324-331.
[26]GAO J,XU Y Y,YANG H M,et al.Gene cloning,expression,and characterization of an exo-inulinase fromPaenibacillus polymyxaZJ-9 [J].Appl Biochem Biotechnol,2014,173(6):1419-1430.
[27]VOLKOV P V,SINITSYNA O A,FEDOROVA E A,et al.Isolation and properties of recombinant inulinases fromAspergillussp.[J].Biochemistry,2012,77(5):492-501.
[28]ZHOU H X,XIN F H,CHI Z,et al.Enhanced expression of heterologous inulinase inKluyveromyces lactisby disruption of hap1 gene[J]. Biotechnol Lett,2010,32(4):507-512.
[29]YUAN B,HU N,SUN J,et al.Purification and characterization of a novel extracellular inulinase from a new yeast speciesCandida kutaonensissp.nov.KRF1(T)[J].Appl Microbiol Biotechnol,2012,96(6): 1517-1526.
[30]ZHANG L H,WANG J,OHTA Y,et al.Expression of the inulinase gene fromAspergillus nigerinPichia pastoris[J].Process Biochem, 2003,38(8):1209-1212.
[31]WEN T Q,LIU F,HUO K K,et al.Cloning and analysis of the inulinase gene fromKluyveromyces cicerisporus[J].World J Microb Biot,2003, 19(19):423-426.
[32]ZHANG S F,YANG F,WANG Q,et al.High-level secretory expression and characterization of the recombinantKluyveromyces marxianusinulinase[J].Process Biochem,2012,47(1):151-155.
[33]BUJACZ A,JEDRZEJCZAK-KRZEPKOWSKA M,BIELECKI S,et al. Crystal structures of the apo form of beta-fructofuranosidase fromBifidobacterium longumand its complex with fructose[J].FEBS Journal, 2011,278(10):1728-1744.
[34]VERHAEST M,VAN DEN ENDE W,ROY K L,et al.X-ray diffraction structure of a plant glycosyl hydrolase family 32 protein:fructan 1-exohydrolase IIa ofCichorium intybus[J].Plant J,2005,41(3):400-411.
[35]ALBERTO F,JORDI E,HENRISSAT B,et al.Crystal structure of inactivatedThermotoga maritimainvertase in complex with the trisaccharide substrate raffinose[J].Biochem J,2006,395(3):457-462.
[36]NAGEM R A,ROJAS A L,GOLUBEV A M,et al.Crystal structure of exo-inulinase fromAspergillus awamori:the enzyme fold and structural determinants of substrate recognition[J].J Mol Biol,2004,344(2): 471-480.
[37]SAINZ-POLO M A,RAMIREZ-ESCUDERO M,LAFRAYA A,et al. Three-dimensional structure ofSaccharomyces invertase:role of a noncatalytic domain in oligomerization and substrate specificity[J].J Biol Chem,2013,288(14):9755-9766.
[38]ALVARO-BENITO M,SAINZ-POLO M A,GONZALEZ-PEREZ D,et al.Structural and kinetic insights reveal that the amino acid pair Gln-228/Asn-254 modulates the transfructosylating specificity ofSchwanniomyces occidentalisbeta-fructofuranosidase,an enzyme that produces prebiotics[J].J Biol Chem,2012,287(23):19674-19786.
[39]PARK J,KIM M I,PARK Y D,et al.Structural and functional basis for substrate specificity and catalysis of levan fructotransferase[J].J Biol Chem,2012,287(37):31233-31241.
[40]LE ROY K,LAMMEMS W,VERHAEST M,et al.Unraveling the difference between invertases and fructan exohydrolases:a single amino acid(Asp-239)substitution transformsArabidopsiscell wall invertase1 into a fructan 1-exohydrolase[J].Plant Physiol,2007,145(3):616-625.
[41]LE ROY K,LAMMENS W,VAN LAERE A,et al.Influencing the binding configuration of sucrose in the active sites of chicory fructan 1-exohydrolase and sugar beet fructan 6-exohydrolase[J].New Phytol, 2008,178(3):572-580.
Research progress of inulinase
ZHANG Tianxiang,DING Ning,YANG Chunguang,SUN Shenxia,WANG Kexin,MA Junyan*
(Medical College,Dalian University,Dalian 116622,China)
Inulinase is a hydrolase that targets on the β-2,1-D-fructosyl-fructose bonds,belonging to glycoside hydrolase family 32.Inulinase is a key enzyme inJerusalem artichokebiorefinery.It can hydrolyzeJ.artichokeinto high purity fructose syrup by one step.The fructose can be further transformed into fuel ethanol,biobutanol,single cell oil,biodiesel,mannitol,lactic acid and other important industrial products which can be widely used in food,pharmaceutical and biofuels fields.The paper introduced the molecular structure and source of inulin,summarized the classification,source, cloning expression,three dimensional structure and application of inulinase,meanwhile,the future development of inulinase was discussed as well, which aimed to improve people's awareness of inulinase and promote the research development ofJ.artichokebiorefinery.
inulinase;glycoside hydrolase family 32;three dimensional structure;biorefinery
Q55
0254-5071(2016)11-0021-05
10.11882/j.issn.0254-5071.2016.11.004
2016-05-30
国家自然科学基金资助项目(31370937);辽宁省(国家)大学生创新创业训练计划项目(201511258026)
张天祥(1993-),男,本科生,研究方向为酶工程与糖生物学。
*通讯作者:马君燕(1981-),女,讲师,博士,研究方向为酶工程与糖生物学。
