版纳微型猪近交系CATSPER3基因克隆、序列及发育进化分析
2016-12-12肖晶查星琴成文敏霍金龙潘伟荣王淑燕王配刘海京张秀琼曾养志
肖晶查星琴,成文敏,霍金龙,潘伟荣,王淑燕,王配,刘海京张秀琼曾养志
(1. 云南农业大学动物科学技术学院,昆明 650201;2. 云南农业大学 云南省版纳微型猪近交系重点实验室,昆明 650201)
版纳微型猪近交系CATSPER3基因克隆、序列及发育进化分析
肖晶1查星琴1,2成文敏1,2霍金龙1,2潘伟荣1,2王淑燕1,2王配1,2刘海京2张秀琼2曾养志2
(1. 云南农业大学动物科学技术学院,昆明 650201;2. 云南农业大学 云南省版纳微型猪近交系重点实验室,昆明 650201)
获得版纳微型猪近交系(BMI)CATSPER3基因部分编码区序列,通过生物信息学分析其氨基酸序列和进行不同物种间的同源性比较。以版纳微型猪近交系的公猪睾丸为材料提取RNA,RT-PCR方法扩增CATSPER3基因部分编码区序列,利用在线分析软件进行生物信息学分析。结果显示,扩增出CATSPER3基因部分编码区序列。生物信息学分析表明,推导氨基酸序列,编码蛋白分子量为19.397 3 kD,理论等电点为5.05;在氨基酸组成上,酸性氨基酸(Asp+Glu)有24个、碱性氨基酸(Lys+Arg)有16个,其中以亮氨酸(Leu)占13.3%、缬氨酸(Val)占9.6%、苏氨酸(Thr)占9.0%、苯丙氨酸(Phe)占8.4%、谷氨酸(Glu)占7.2%,天冬氨酸(Asp)占7.2%等含量较高。在核苷酸相似度上与普通猪相似度最高,与山羊核苷酸的相似度较低;分子系统进化树表明与非近交系猪同处于一个分支中,亲缘关系较近,与山羊和绵羊亲缘关系较远。
版纳微型猪近交系;CATSPER3基因;系统进化分析
近年来许多相关研究发现,精子在获能和超活化过程中会出现大量钙离子内流,说明它是精子质膜上控制钙离子内流的通道,精子头部和尾部存在一种离子蛋白,对精子的运动能力、穿卵能力以及受精能力都有一定的影响。CATSPER通道是已知的仅存于生精细胞和成熟精子中的Ca2+离子通道,通过基因剔除发现这类通道直接与精子的运动和受精能力有关。早期研究表明Ca2+离子通道蛋白家族(Canon channel of sperm ptoreins Catsper)由4个成员(CATSPER1、CATSPER2、CATSPER3和CATSPER4)组成[1,2],而后又发现Ca2+离子通道蛋白的新成员Catsper β和CatsperG[3,4]。
Ca2+离子通道蛋白的表达是睾丸特异的,在睾丸以外的其他器官及免疫系统如大脑、心脏、脾、肾、胃、骨骼中皆不表达。其中CATSPER3主要定位于精子头部[1,3],而AMP和Ca2+都能诱导顶体反应的发生,因此推测CATSPER3可能参与顶体反应中胞内Ca2+浓度的调节。版纳微型猪近交系是世界上第一个培育成功的大型哺乳类实验动物近交系,CATSPER3基因是Ca2+离子通道蛋白家族的重要成员之一,本研究拟采用反转录PCR的方法扩增得到版纳微型猪近交系猪CATSPER3基因的序列,结合多种生物信息学分析软件分析,以期能够解释版纳微型猪近交系猪弱精症产生的原因。
1 材料与方法
1.1 材料
DNA纯化试剂盒,购自全式金生物技术有限公司;病毒总RNA/DNA提取试剂盒(TaKaRa MiniBEST Viral DNA/RNA Extraction Kit Ver.3.0)、RT-PCR一步法试剂盒、Taq酶、DL2000 DNA Marker、Competent Cell Preparation Kit,逆转录酶XL(AMV)、Ta-KaRa Ex TaqTM均购自宝生物工程(大连)有限公司。
1.2 方法
1.2.1 引物设计 参考GenBank上猪CATSPER3的全基因序列设计的引物,由上海生工生物工程技术服务有限公司合成,扩增的特异性片段长度约为498 bp。引物序列为:5'-GCCTTCTTTACCCTCTTCAGC-3';5'-AAGTATCAGGCTCGGCACAT-3'。
1.2.2 RNA的提取、cDNA合成 参照 RNA Plus的使用方法,从版纳微型猪近交系睾丸组织中提取总RNA,经过核酸蛋白测定仪检测出总RNA的浓度和纯度,根据测定的浓度将RNA稀释成200 ng/μL,取5 μL进行1%琼脂糖凝胶电泳检测,然后将检测合格的RNA样品反转录为cDNA,反转录体系(20 μL):50 μmol/L oligo(dT)181 μL,总RNA 1 ng-5 μg,10 mmol/L dNTP 1 μL,加入灭菌蒸馏水至12 μL,混合后65℃加热5 min,迅速冰浴冷却;短暂离心后加入:5×first buffer 4 μL,0.1 mol/L的DTT 2 μL,recombiant RNase inhibitor 1 μL,轻轻将各组分混合,37℃孵育 2 min,室温下加入1 μL reverse transcriptase M-ML,轻轻吹打混匀,37℃孵育5 min,70℃加热15 min以终止反应,产物置-20℃储存备用。
1.2.3 CATSPER3基因PCR扩增 PCR反应体系25 μL:Premix Taq 13.0 μL,引物(10 pmol/μL)各1.5 μL,DNA模 板(25 ng/μL)1.5 μL, 加 ddH2O(高压灭菌双蒸水)补足至25 μL。PCR反应程序为:95℃5 min;94℃ 30 s,56℃ 30 s,72℃ 30 s,35个循环;72℃ 7 min,4℃保存。PCR扩增产物用2%的琼脂糖凝胶电泳检测,紫外凝胶成像系统(SYGENE,GeneGenius)中观察分析并照像。
1.2.4 CATSPER3基因克隆 应用全式金生物技术有限公司DNA纯化回收试剂盒(离心柱型)进行PCR产物纯化(步骤参照说明书),将纯化后检测正确的PCR产物连接入pMD18-T克隆载体,转化DH5α感受态细胞,涂布琼脂糖平皿上,恒温培养箱37℃过夜培养,进行蓝白斑筛选,用白色吸头挑取白色单克隆菌落,接种入LB液体培养基,37℃,200 r/min震荡过夜培养,吸取微量过夜菌液进行菌液PCR鉴定,检测后送往上海生物工程技术有限公司测序。
1.2.5 CATSPER3基因序列的生物信息学分析 测序得到的结果用NCBI(National Center for Biotechnology Information)的Nucleotide Blast软件检验核苷酸的序列;然后用Generunr软件预测出开放阅读框,并得到相应的氨基酸序列;用在线分析软件ExPASy-PrtPram tool分析CATSPER3基因编码蛋白质的结构;通过NetNGlyc 1.0 Server预测蛋白质N-糖基化位点;通过NetOGlyc 4.0 Server预测蛋白质O-糖基化位点;通过NetPhos 2.0 Server预测蛋白质
磷酸化位点;通过SignalP 4.1 Server预测信号肽序列;通过TMpred server预测蛋白质中是否有跨膜结构域;用SOPMA方法预测蛋白质的二级结构;用Lasergene软件分析核苷酸的同源性。
2 结果
2.1 CATSPER3基因扩增结果
CATSPER3引物扩增的特异性片段为498 bp,与预期片段大小一致(图1)。测序结果与GenBank上猪CATSPER3基因序列比对,确定扩增序列为BMI的CATSPER3基因序列。

图1 CATSPER3引物扩增的PCR产物电泳结果
2.2 CATSPER3基因序列生物信息学分析
用ExPASy ProtParam程序预测CATSPER3基因编码蛋白分子量为19.397 3 kD,理论等电点为5.05;在氨基酸组成上,在氨基酸组成上,酸性氨基酸(Asp+Glu)有24个;碱性氨基酸(Lys+Arg)有16个;其中,亮氨酸(Leu)占13.3%、缬氨酸(Val)占9.6%、苏氨酸(Thr)占9.0%、苯丙氨酸(Phe)占8.4%、谷氨酸(Glu)占7.2%、天冬氨酸(Asp)占7.2%等含量较高。CATSPER3蛋白质不稳定指数为39.46,属于稳定蛋白;脂溶指数为103.80,总的亲水性平均系数(Grand average of hydropathicity,GRAVY)为0.020,具有弱亲水性。采用SignalP 4.1对CATSPER3蛋白进行预测(图2)表明,CATSPER3蛋白没有信号肽位点;用NetOGly 4.0和NetNGly 1.0进行预测的结果表明,CATSPER3蛋白中不含有N-糖基化位点和O-糖基化位点。
NetPhos2.0预测(图3)表明,CATSPER3蛋白存在7个磷酸化位点,其中3个丝氨酸(Ser)磷酸化位点(A29、A59、A139)、3个苏氨酸(Thr)磷酸化位点(A74、A96、A115)、1个酪氨酸(Tyr)活性位点(A156)。CATSPER3的蛋白存在着较多的磷酸化位点,说明磷酸化位点修饰对CATSPER3的蛋白活化起着重要作用。
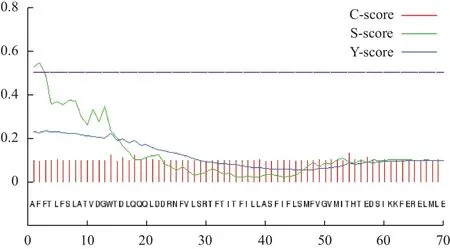
图2 CATSPER3蛋白跨膜信号肽位点预测
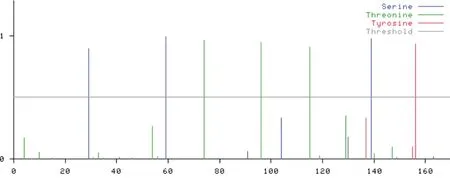
图3 CATSPER3蛋白磷酸化位点推测图
用SOPMA方法预测的CATSPER3蛋白的二级结构结果(图4)显示,参与α-螺旋的氨基酸有94个,含量达56.63%;有35个氨基酸参与延伸链,占21.08%;10个氨基酸可能参与β-转角,占6.02%;27个氨基酸可能参与无规则卷曲,占16.27%。以上分析可推测,α-螺旋是CATSPER3蛋白的主要结构。
测序结果经GenBank上Nucleotide Blast,结果(图5)显示,在第970位核苷酸位置有一个碱基由G变成C,在第1 243位核苷酸位置有一个碱基由C变成T。测序获得序列与参考序列推导氨基酸序列,结果(图6)显示,在核苷酸突变的位置氨基酸未改变,属于无意义突变。
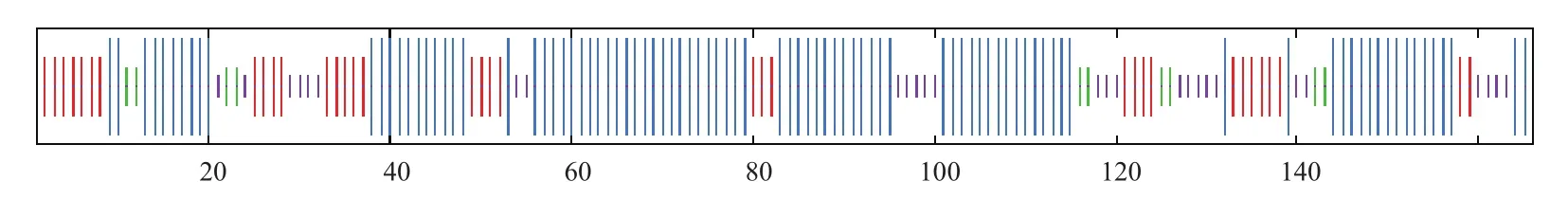
图4 SOPMA程序预测BMI CATSPER3蛋白二级结构

图5 CATSPER3基因核苷酸序列比对

图6 CATSPER3基因推导氨基酸序列比对
在NCBI中下载CATSPER3基因各物种的相关序列,通过lasergene软件编辑格式,比对得出图7,图8。图7显示,版纳微型猪近交系猪与普通猪核
苷酸相似度较近为99.6%,与山羊相似度较低为88.6%,从进化树图8中也可以得出相似的结论,版纳微型猪近交系猪与普通猪同处于一个分支中,而与山羊和绵羊在进化树中距离最远。

图7 CATSPER3基因核苷酸相似度分析

图8 CATSPER3基因系统进化树分析
3 讨论
CatSper是睾丸内生殖细胞中特异的离子型通道,定位于精子尾部鞭毛的主段,由于主段没有细胞器,因此推测 CATSPER1和 CATSPER2主要定位于主段的质膜上,对鞭毛鞭打运动起调节作用;而 CATSPER3表达于精子顶体,可能参于精子顶体反应和生殖调节[5]。Qi等[5]研究表明,CATSPER3也参与精子超活化的调控,且CATSPER1、CATSPER2、CATSPER3和CATSPER4四个蛋白都对受精有影响[6]。
本实验扩增的序列长度为498 bp,包含部分编码区和部分非编码区,对测序结果分析,在第970位和第1 243位核苷酸位置分别有一个碱基由G变成C和C变成T,但在该位置氨基酸却没有改变,属于无意义突变。
在推导的氨基酸序列中无跨膜结构和信号肽,含有7个磷酸化位点,其中3个丝氨酸(Ser)磷酸化位点(A29、A59、A139)、3个苏氨酸(Thr)磷酸化位点(A74、A96、A115)、1个酪氨酸(Tyr)活性位点(A156),推测可能在这一段序列推导的二级结构中,磷酸化位点对其的修饰和调控作用占主要方面;参与α-螺旋的氨基酸有94个,含量达到56.63%;有35个氨基酸参与延伸链,占21.08%;10个氨基酸可能参与β-转角,占6.02%;27个氨基酸可能参与无规则卷曲,占16.27%。推测α-螺旋是该段推导CATSPER3蛋白的主要结构。核苷酸相似度的比较中与普通猪相似度最高,与山羊相似度较低,这与杨媛媛[7]的研究结果一致,CATSPER3在进化上比较保守,核苷酸物种间相似性较高;在亲缘关系上与普通猪同处于一个分支,亲缘关系较近,与山羊和绵羊亲缘关系较远,与预期结果一致。
4 结论
成功扩增了CATSPER3基因部分编码区序列,特异性片段为498 bp。版纳微型猪近交系猪与非近交系猪核苷酸相似度为99.6%,与山羊相似度为88.6%。版纳微型猪近交系猪与非近交系猪同处一个分支,亲缘关系较近,与山羊和绵羊亲缘关系较远。
[1]Darszon A, Acevedo JJ, Galindo BE, et al. Sperm channel diversity and functional multiplicity[J]. Reproduction, 2006, 131(6):77-88.
[2]Inaba K. Molecular architecture of the sperm flagella:molecules for motility and signaling[J]. Zoolog Sci, 2003, 20(9):1043-1056.
[3]Liu J, Xia J, Cho KH, et al. CatSperbeta, a novel transmembrane protein in the CatSper channel complex[J]. J Biol Chem, 2007, 282(26):18945-18952.
[4]Wang H, Liu J, Cho KH, Ren D. A novel, single, transmembrane protein CATSPERG is associated with CATSPERI channel protein[J]. Biol Reprod, 2009, 81(3):539-544.
[5]Jin JL, O’Doherty AM, Wang S, et al. Catsper3 and catsper4 encode two canon channel-like proteins exclusively expressed in the testis[J]. Biol Reprod, 2005, 73(6):1235-1242.
[6] Qi H, Moran MM, Navarro B, et al. All four CatSper ion channel proteins are required for male fertility and sperm cell hyperactivated motility[J]. Proc Natl Acad Sci USA, 2007, 104(4):1219-1223.
[7]杨媛媛, 王根林. 精子超活化结构蛋白-Catsper表达特性的研究[D]. 南京:南京农业大学, 2006.
(责任编辑 李楠)
Cloning,Sequence and Phylogenetic Analysis of CATSPER3 Gene in Banna Mini-pig Inbred Line
XIAO Jing1ZHA Xing-qin1,2CHENG Wen-min1,2HUO Jin-long1,2PAN Wei-rong1,2WANG Shu-yan1,2WANG Pei1,2LIU Hai-jing2ZHANG Xiu-qiong2ZENG Yang-zhi2
(1. Faculty of Animal Science and Technology,Yunnan Agricultural University,Kunming 650201;2. Key Laboratory of Banna Mini-pig Inbred Line of Yunnan Province,Yunnan Agricultural University,Kunming 650201)
Our purpose of this work is to obtain the partial coding region sequences of CATSPER3 gene from Banna Mini-pig Inbred Line(BMIL),and to analyze the amino acid sequence and homology comparison among different species by bioinformatics. We used boar testis material of BMIL to extract RNA,RT-PCR to amplify the coding sequence of CATSPER3 gene,and online analysis software to analyze the bioinformatics. Bioinformatics analysis showed that the protein molecular weight of deduced amino acid sequence was 19.397 3 kD and theoretical isoelectric point was 5.05. Regarding the composition of amino acid,there were 24 acidic amino acids(Asp and Glu);and there were 16 alkaline amino acid(Lys+Arg);among them the leucine(Leu)accounted for 13.3%,valine(Val)9.6%,threonine(Thr)9.0%,phenylalanine(Phe)8.4%,glutamate(Glu)7.2%,aspartic acid(Asp)7.2%,and their contents were high. Compared with ordinary pig,the nucleotide similarity was the highest,however,lower with that of goat. Molecular phylogenetic tree showed that inbred pig and noninbred pig clustered in the same branch,i.e.,their genetic relationship was close,while far from goat and sheep.
Banna Mini-pig Inbred Line;CATSPER3 gene;phylogenetic analysis
10.13560/j.cnki.biotech.bull.1985.2016.12.017
2016-01-11
云南省教育厅科学研究基金项目(2014Y212)
肖晶,男,硕士,研究方向:分子生物学研究,E-mail:xiaojingjing525@163.com;查星琴为本文并列第一作者
潘伟荣,男,硕士,研究方向:胚胎生物技术;E-mail: pwr2000@sina.com
