苦马豆根瘤中内生细菌遗传多样性分析
2016-12-09邓振山
邓振山
(延安大学生命科学学院,陕西 延安 716000)
苦马豆根瘤中内生细菌遗传多样性分析
邓振山
(延安大学生命科学学院,陕西 延安 716000)
苦马豆(Sphaerophysasalsula)是西北荒漠区重要的豆科植物。本研究采用16S rDNA PCR-RFLP和16S rDNA全序列分析方法,对西北部分地区苦马豆根瘤中内生细菌的遗传多样性及系统发育进行分析。结果显示,115株供试菌株产生了28种遗传图谱类型,对每种图谱类型的代表性菌株进行了16S rDNA全序列分析,其中的根瘤菌分别归属于中华根瘤菌属(Sinorhizobium)、根瘤菌属(Rhizobium)、中慢生根瘤菌属(Mesorhizobium);其余的内生菌分别归属于副球菌属(Paracoccus)、鞘脂单胞菌属(Sphingomonas)、固氮螺菌属(Inquilinus)、假单胞菌属(Pseudomonas)、沙雷氏菌属(Serratia)、分枝杆菌属(Mycobacterium)、诺卡氏菌属(Nocardia)、链霉菌属(Streptomyces)、类芽孢杆菌属(Paenibacillus)、短短芽孢杆菌属(Brevibacillus)、Lysinibacillus、葡萄球菌属(Staphylococcus)和芽孢杆菌属(Bacillus),这表明苦马豆根瘤中内生细菌具有丰富的多样性。
苦马豆; 16S rDNA PCR-RFLP; 遗传多样性; 系统发育
豆科植物广泛分布于世界各地,它们通过根瘤菌的侵染和诱导,形成了称之为根瘤或茎瘤的固氮共生体。根瘤菌在植物根瘤中受到保护并从植物体内获得碳源,同时把空气中的氮气转化为氨,并供给植物生长。与根瘤菌相同,其它的非共生细菌也已大范围地从多种豆科植物根瘤中分离出来[1-15]。这些内生菌在根瘤中只能与共生菌共存,而且不会引起植物结瘤,也不固氮。但是,至少有一部分生命周期是存在于根瘤中的,并且不会对植物产生明显的损害作用,所以,这些非共生细菌是生活在根瘤中的内生菌[15]。研究者从根瘤中分离出不同种类的内生细菌,包括芽孢杆菌(Bacillus)、假单胞菌(Pseudomonas)和肠杆菌(Enterobacter)[9,14]。根瘤中的内生菌,分布在不同的纲和门,有变形菌门(革兰氏阴性),放线菌门、厚壁菌门[15]。目前,属于α-变形菌纲的这些根瘤菌被鉴定为Rhizobium,Bradyrhizobium,Sinorhizobium,Mesorhizobium,Azorhizobium和Allorhizobium[15]。还有些属于α-和β-变形菌纲以及一些未被分类的变形菌纲的细菌也从多种豆科植物的根瘤中被分离得到。
苦马豆(Sphaerophysasalsula)属于豆科,蝶形花亚科,苦马豆属,为多年生草本植物,主要分布于中亚和东亚,仅有2个种,其中1个种产自我国,为非引进种。苦马豆多生于海拔960-3 180 m的山坡、草原、荒地、沙滩、地埂、沟渠旁及盐池周围,耐盐耐碱、耐瘠耐涝,是改良盐碱地的先锋植物。
与植物其它组织中的内生菌相比,根瘤中的内生菌研究还比较少,对它们所发挥的生态作用的了解还远远不够。豆科作为地球上植物界最大的科之一,包括的植物种类大约有19 000种(http://www.ildis.org/ leguminosae/)[15-16],但仅有少数种类(大多为农业主栽品种)[17]被研究过,根瘤中内生菌的相关研究就更少。研究者对西北部分地区的苦马豆根瘤菌进行了遗传多样性及系统发育分析, 但主要对苦马豆根瘤菌多样性进行了描述[16]。目前相关研究主要集中在其它豆科植物根瘤内生菌的研究上[1-14],而针对苦马豆根瘤内生菌的研究尚未见报道,其内生菌的多样性与系统发育地位还不明确。鉴于此,本研究采用16S rDNA PCR-RFLP和16S rDNA 全序列分析方法,对西北部分地区苦马豆根瘤中内生细菌的遗传多样性及系统发育进行研究,旨在分析苦马豆根瘤中内生细菌的多样性,确定其系统发育地位,加深对苦马豆根瘤中内生细菌资源分布的认识,为进一步发掘和在农业生产和生态恢复中充分利用性状优良的内生菌种质资源提供依据。
1 材料与方法
1.1 试验地概况
经过系统研究和综合考虑,于2008年7月(苦马豆的花期,有利于植物体的鉴定和有效瘤的采集)选择了分布广泛和最适合苦马豆生长的两个不同的生态区(我国西北地区的甘肃省和宁夏回族自治区)作为采样地。生态区Ⅰ(甘肃省)为典型的温带季风性气候,具有砂质土和盐/碱土壤,年降水量45~132 mm,年平均温度7~9 ℃;生态区Ⅱ(宁夏回族自治区)为大陆季风性气候,位于半干旱温带地区,黏质土,年降水量179~260 mm,年平均温度8~9 ℃。分别记录各采样地的土壤类型,海拔、地理坐标(使用GPS记录)(表1),并对所采集的植物样本进行照相和标本压制,以备鉴定所用。
1.2 试验材料
依据苦马豆生长所在的不同生态型和地理条件,于2008年7月从6个采样点采集了苦马豆根瘤、植株、果实、种子及原始土样,这6个样点的根瘤采集数分别为:甘肃省张掖市,45个根瘤;甘肃省高台县,48个根瘤;甘肃省瓜洲县,63个根瘤;甘肃省民勤县,61个根瘤,甘肃省采样覆盖面积为305 km×665 km(南北×东西)。宁夏中卫市,51个根瘤;宁夏青铜峡市,55个根瘤,宁夏回族自治区采样覆盖面积为90 km×68 km。
1.3 试验方法
1.3.1 植物根瘤的采集方法 每个采样点之间的距离大于50 km,并在每个采样点随机选择地理坐标不同的3个小采样点,每个小采样点至少采集30~50个不同的植株,每个植株间距离大于30 m,并在须根上随机选择3~4个没有损伤、健康、大小相近的有效根瘤,存放于灭过菌、干燥的、装有变色硅胶和棉塞的1.5 mL离心管或马铃薯葡萄糖琼脂斜面中,置于4 ℃冰箱中保存备用[18]。
1.3.2 土壤样品的采集方法 每个采样点取样量大体一致,均匀混合后,取约2 kg装入灭菌的封口聚乙烯袋,带回实验室于-70 ℃保存备用,待测定土壤理化性质。pH用酸度计法,总碳用重铬酸钾氧化法,总氮用半微量开氏法,总磷用盐酸氟化铵-钼锑抗比色法,总钾用原子吸收分光光度法[15]。
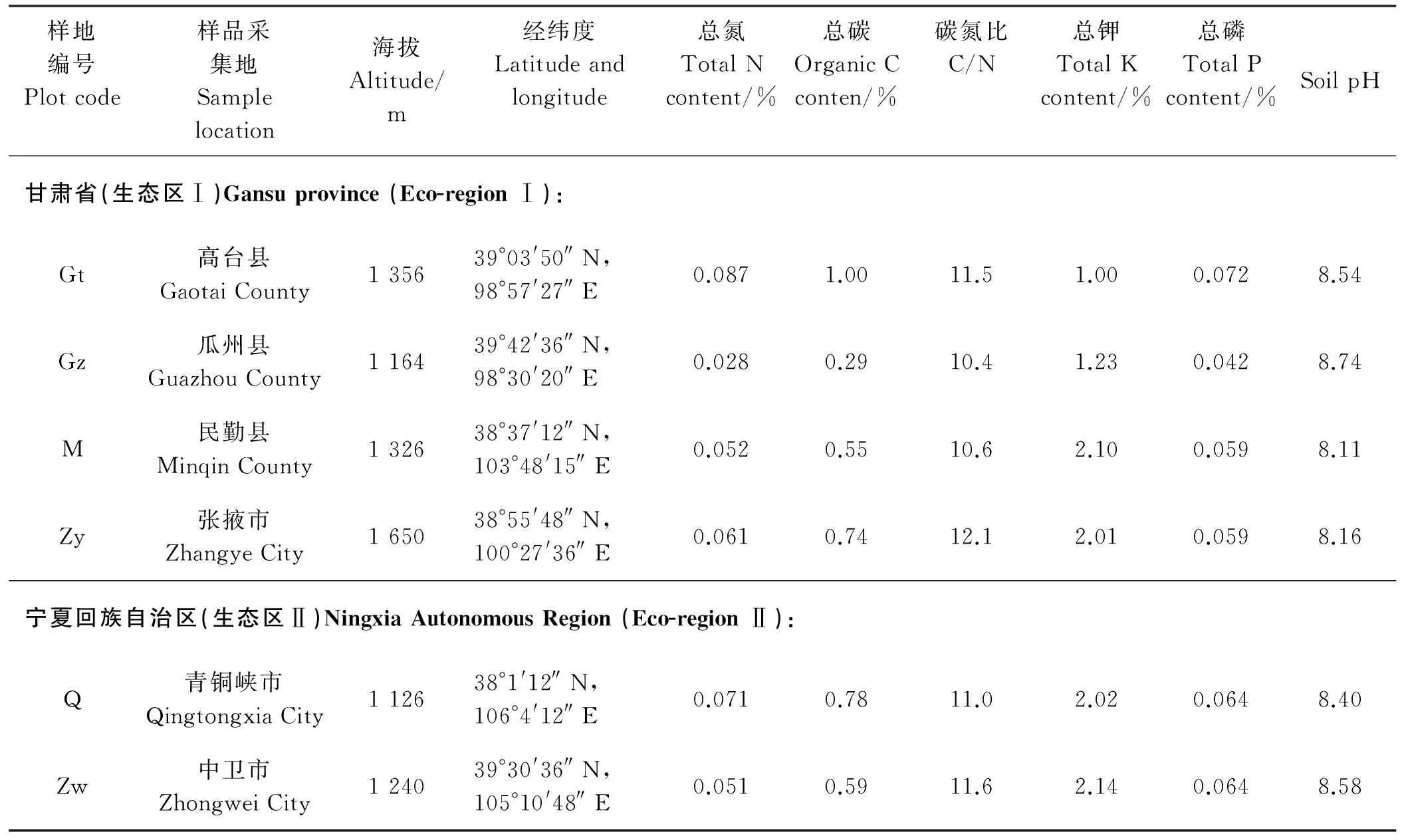
表1 供试土壤样品的地理来源和理化性质Table 1 The geographical origin and physicochemical characteristics of the studied soilsa
1.3.3 菌株的培养及分离纯化 将根瘤放在无菌水中充分浸泡湿胀后,用吸水纸吸干根瘤表面的残留水分,95%的乙醇中浸泡30 s,随即浸入5%的NaClO 3 min,之后灭菌水洗10次,最后用经火焰灭菌后的无菌镊子夹破根瘤,随即将菌悬液分别用划线法接种于yeast-mannitol agar(YMA)、Potato Dextrose Agar(PDA)、King’s B和Nutrient Agar(NA)4种不同的培养基平板上[8],于28 ℃培养两周,获得其纯培养单菌落后,将其分别转接于相应的试管斜面培养基中,部分置于4 ℃冰箱中暂时保存,部分在30%(v/v)甘油中于-80 ℃长期保存备用。为了确定根瘤表面消毒是否彻底,各处理均分别以最后一次冲洗液涂抹平板设置为对照(CK),培养4 d后平板上无菌体生长,表明表面消毒彻底,相应平板的根瘤可用于内生菌的分离[8]。
1.3.4 16S rDNA全序列分析 总DNA的提取方法参见文献[18],fD1和rD1(Escherichia coli 16S rRNA基因序列的8-27 bp和1 524-1 540 bp)用作PCR扩增的引物[19],PCR扩增的方法见文献[20]。
1.3.5 16S rDNA PCR-RFLP分析 选用扩增较好的PCR产物进行酶切。本试验选用了4种限制性内切酶,分别为HaeⅢ、HinfⅠ、MspⅠ和HhaⅠ。酶切反应体系均为20 μL,在37 ℃水浴中酶切4 h。全部酶切产物与2.5 μL上样缓冲液溴酚兰(10×Loading buffer)混匀,用2%的琼脂糖凝胶分离,70 V水平电泳4 h,电泳结束后UV扫描,文件以JPG格式保存,并对4种酶的酶切结果进行分析、记录,4种酶的酶切结果一样的菌株被看作同一种酶切类型,统计各菌株的酶切组合类型。
1.3.6 16S rDNA序列测定及其系统发育分析 根据16S rDNA PCR-RFLP遗传图谱组合类型,选取代表菌株(共25株)的16S rDNA扩增产物送上海生物工程技术服务有限公司进行测序。把得到的序列提交到Genbank(NCBI)申请并获得各菌株序列号为:GU129566到GU129569和GU201840到GU201863。根据测序结果,用BLAST软件与从Genbank(NCBI)中获得的已知种及相关种16S rDNA的全序列进行多重序列比对,应用Clustal-X 1.81和TREECONW version 1.3b软件包分析,构建以16S rDNA全序列为基础的系统发育树状图,并使用DNAMAN 6.0.40计算各测试菌株间16S rDNA序列的相似值。根据代表性菌株的16S rDNA的全序列与已知种的参比菌株序列比较,计算各菌株之间的遗传距离,采用Neighbor-joining方法和Jukes-Cantor模型构建系统发育树状图。自展数 (bootstrap)为100。
1.3.7 各采样点可操作分类单位(OTUs) 的分离频率和丰富度 可操作分类单位(operational taxonomic units,OTUs) 相似标准定义为16S rDNA基因序列差异小于3%[21]。每一种OTU的分离频率用公式F=n/N计算,其中n代表采样点的同一种OTU分离数;N代表所有采样点总数。在一个采样点,每一种OTUs的丰富度用公式R=s/S计算,其中,s表示同一种OTUs的菌株数;S表示在同一个采样点的所有菌株数。
2 结果与分析
2.1 根瘤中内生菌的分离与纯化
从4种不同配方的培养基中分离得到的根瘤内生细菌经过纯化和镜检,根据菌落和菌体的差异,最终共获得内生细菌115株,其中66株分离自生态区Ⅰ, 49株分离自生态区Ⅱ(表2)。
2.2 16S rDNA PCR-RFLP分析
用引物P1和P6对115株供试菌株的16S rDNA进行PCR扩增后,其扩增产物用1%琼脂糖凝胶电泳检测,均产生了约1.5 kb的DNA条带(以100 bp DNA Ladder Marker 作标记)。该结果表明,供试菌株与其它细菌16S rDNA的片段大小基本一致。
扩增产物分别用4种限制性内切酶酶切后,经2%的琼脂糖凝胶电泳成像。对应每一个酶切电泳图谱照片,凡是电泳图谱上不同菌株间迁移率相同的带被认为是同一个性状。4种限制性内切酶酶切图谱代表类型及遗传图谱类型结果如表3所示,西北地区苦马豆根瘤中菌体的16S rDNA扩增片段的RFLP遗传图谱比较丰富。供试菌株的16S rDNA PCR产物经HinfⅠ、HhaⅠ、HaeⅢ、MspⅠ这4种限性内切酶组合在一起所得到的16S rDNA遗传图谱类型共有25种(表3),每一种定义为一个16S rDNA遗传图谱类型。苦马豆根瘤中内生细菌16S rDNA扩增片段的RFLP电泳图谱存在较大差异,说明16S rDNA基因具有丰富的遗传多样性。综合分析可知,供试的115株苦马豆根瘤中的内生细菌,类型4(dcaa)占主要地位,由23 株供试菌株组成,并且在6个采样点均有分布(9株来自宁夏中卫市,4株来自宁夏青铜峡市,4株来自甘肃省高台县,3株来自甘肃省民勤县,2株来自甘肃省瓜洲县,1株来自甘肃省张掖市);其次是类型20(jbfe),共由21株菌组成,并且在5个采样点均有分布(9株来自甘肃省高台县,4株来自甘肃省瓜洲县,2株来自宁夏中卫市,2株来自宁夏青铜峡市,4株来自甘肃省民勤县);类型1(jhac),共由10株菌组成,分布在2个采样点(8株来自宁夏青铜峡市,2株来自甘肃省民勤县);类型2(bakn),共由6株菌组成,分布在2个采样点(3株来自宁夏青铜峡市,3株来自甘肃省张掖市);类型5(ecab),共由7株菌组成,分布在3个采样点(3株来自宁夏青铜峡市,2株来自甘肃省瓜洲县,2株来自宁夏中卫市);类型9(cakn),共由2株菌组成,分布在2个采样点(1株来自甘肃省张掖市,1株来自宁夏中卫市);类型12(ijek),共由5株菌组成,分布在3个采样点(1株来自宁夏青铜峡市,3株来自甘肃省张掖市,1株来自甘肃省瓜洲县);类型13(miha),由6 株供试菌株组成,分布在3个采样点(3株来自宁夏中卫市,1株来自宁夏青铜峡市,1株来自甘肃省高台县, 1株来自甘肃省瓜洲县);类型14(afgg),由3株供试菌株组成,分布在2个采样点(2株来自宁夏青铜峡市,1株来自甘肃省民勤县);类型17(cbej),由3株供试菌株组成,分布在2个采样点(2株来自甘肃省高台县,1株来自甘肃省民勤县);类型21(nmfd),由8株供试菌株组成,分布在3个采样点(2株来自甘肃省高台县,4株来自甘肃省瓜洲县,2株来自宁夏中卫市);类型23(bebe),由7株供试菌株组成,全部来自甘肃省高台县;类型24(ecfb),由2株供试菌株组成,全部来自宁夏青铜峡市;类型3(ecam)、类型6(igdk)、类型7(dcab)、类型8(hhae)、类型10(ejal)、类型11(emih)、类型15(aldd)、类型16(lkji)、类型18(kbco)、类型19(bbbe)、类型22(fbbf)和类型25(fdbf),分别都是由1株菌组成。

表2 根瘤中细菌的分离频率和丰富度Table 2 Isolation frequency and richness of the nodule bacteria
注:s,同一种OUT的菌株数。
Note:s, number of strains with the same OUT。

表3 供试苦马豆根瘤分离菌及其16S rDNA PCR-RFLP图谱类型和16S rDNA相似菌株Table 3 Isolates obtained from Sphaerophysa salsula and results of 16S rDNA gene RFLP and 16S rRNA gene sequencing of the most similar strains
续表3(1)

菌株编号No.ofstrain图谱类型RFLPtype分离地 Location 最相似菌株 Themost similarstrain 登录号Accessionnumber相似性SimilarityZw-1115中卫ZhongweiQtx-215青铜峡QingtongxiaZy-2(GU201844)6(ijdk)张掖ZhangyeM.tianshanenseA-1BSAF04144799%Qtx-24(GU201840)7(dcab)青铜峡QingtongxiaM.mediterraneumUPM-Ca36L3882599%中华根瘤菌属SinorhizobiumQtx-8-1(GU129566),Qtx-828(hhae)青铜峡QingtongxiaS.melilotiLMG6133X67222)99%副球菌属ParacoccusZy-3H-(GU129567)9(cakn)张掖ZhangyeP.halophilusHN-182DQ42348296%Zw-119(cakn)中卫Zhongwei鞘脂单胞菌属SphingomonasGuashi-1(GU201847)10(ejal)瓜州GuazhouS.pruniIFO15498Y0963798%固氮螺菌属InquilinusMq-10(GU201848)11(emih)民勤MinqinI.limosusAU476NR_02904699%假单胞菌属PseudomonasZy-2-1(GU201849)12(ijek)张掖ZhangyeP.fluorescens2P24AY44704599%Zy-2212张掖ZhangyeZy-2312张掖ZhangyeGz-2012瓜州GuazhouQtx-25212青铜峡Qingtongxia沙雷氏菌属SerratiaZw-22(GU201850),Zw-221,Zw-8213(miha)中卫ZhongweiS.plymuthicaDSM4540AJ23343399%Gt-10113高台GaotaiGz-17113瓜州GuazhouQtx-3013青铜峡Qingtongxia分枝杆菌属MycobacteriumQtx-19(GU201853)14(afgg)青铜峡QingtongxiaM.sacrumBN3151AY235429100%Mq-2114民勤MinqinQtx-2514青铜峡Qingtongxia诺卡氏菌属NocardiaGt-25(GU201852)15(aldd)高台GaotaiN.uniformisDSM43136AF430044100%链雷菌属StreptomycesGt-10(GU201851),Gt-2016(lkji)高台GaotaiS.bottropensisMBRC13023AB184262100%
续表3(2)

菌株编号No.ofstrains图谱类型RFLPtype分离地 Location 最相似菌株 Themost similarstrains 登录号Accessionnumber相似性Similarity类芽孢杆菌属PaenibacillusGt-1(GU201854)17(cbej)高台GaotaiP.amylolyticusNRRLNRS-290NR_02588299%Mq-3117民勤MinqinGt-1117高台Gaotai短短芽孢杆菌属BrevibacillusMq-17(GU201855)18(kbco)民勤MinqinB.borstelensisLMG15536AF378230100%LysinibacillusZw-13-3(GU201856)19(bbbe)中卫ZhongweiL.fusiformisNBRC15717AB245423100%芽孢杆菌属BacillusQtx-11(GU201860),Qtx-1220(jbfe)青铜峡QingtongxiaB.simplexLMG21002(AJ628745)100%Gz-220瓜州GuazhouMq-10120民勤MinqinGt-191,Gt-5120高台GaotaiGz-22,Gz-120瓜州GuazhouZw-1320中卫ZhongweiGt-1920高台GaotaiGt-820高台GaotaiZw-22-2(GU201861)21(nmfd)中卫ZhongweiB.safensisKL-052AY030327)100%Gt-49,Gt-6,Gt-62,Gt-7221高台GaotaiZw-13221中卫ZhongweiGz-4121瓜州GuazhouMq-143,Mq-144,Mq-1921民勤MinqinZw-1121中卫ZhongweiGz-621瓜州GuazhouGt-61,Gt-13,Gt-23,Gt-4221高台GaotaiGz-20121瓜州GuazhouGt-12,Gt-41,Gt-21,Gt-71,Gt-31,Gt-5221高台GaotaiGzn-9-1(GU201863)22(fbbf)瓜州GuazhouB.licheniformisCICC10104DQ082997100%Gaoshi-1(GU201858)23(bebe)高台GaotaiB.cereusQD232EF488087100%Qtx-10(GU201862),Qtx-1624(ecfb)青铜峡QingtongxiaB.pumilusB402DQ523500100%葡萄球菌属StaphylococcusGaoshi-7(GU201857)25(fdbf)高台GaotaiS.saprophyticusATCC15305D83371100%
从宁夏青铜峡市分离获得29株菌,共发现有11种类型,其中有24株分别属于类型1(jhac)、类型2(bakn)、类型4(dcaa)、类型5(ecab)、类型12(ijek)、类型14(afgg)和类型20(jbfe),且这些类型在其它样地也有分布,剩余的5株分别属于类型3(ecam)、类型7(dcab)、类型8(hhae)和类型24(ecfb),且这几种类型在其它样地没有发现,为该样地的特有类型。从甘肃省高台县共分离得到30株菌,共发现有9种类型,其中类型20(jbfe)有9株菌,类型21(nmfd)有4株菌,类型4(dcaa)有4株菌,类型17(cbej)有2株菌,类型13(miha)有1株菌,剩余的为该样地的特有类型,包括类型15(aldd)、类型16(lkji)、类型25(fdbf)分别各有1株菌,类型23(bebe)有7株菌。从甘肃省瓜洲县分离获得14株菌,多样性比较丰富,共发现有8种类型,其中有12株分别属于类型5(ecab)、类型10(ejal)、类型12(ijek)、类型13(miha)、类型20(jbfe)和类型21(nmfd),且这些类型在其它样地也有分布,剩余的2株分别属于类型4(dcaa)和类型22(fbbf),且这2种类型在其它样地再没有发现,为该样地的特有类型。从甘肃省民勤县分离获得13株菌,共发现有7种类型,其中有11株分别属于类型1(jhac)、类型4(dcaa)、类型14(afgg)类型17(cbej)和类型18(kbco),且这些类型在其它样地也有分布,剩余的2株分别属于类型11(emih)和类型20(jbfe),且这2种类型在其它样地没有发现,为该样地的特有类型。从甘肃省张掖市分离获得9株菌,共发现有5种类型,其中有8株分别属于类型2(bakn)、类型9(cakn)、类型4(dcaa)和类型12(ijek),且这些类型在其它样地也有分布,剩余的1株属于类型6(igdk),为该样地的特有类型。从宁夏中卫市分离并获得20株菌,共发现有7种类型,其中有19株分别属于类型4(dcaa)、类型5(ecab)、类型9(cakn)、类型13(miha)、类型20(jbfe)和类型21(nmfd),且这些类型在其它样地也有分布,剩余的1株属于类型19(bbbe),为该样地的特有类型。
2.3 16S rDNA全序列相似性及系统发育分析
根据16S rDNA PCR-RFLP遗传图谱组合类型,115株内生细菌可以被分为25种RFLP类型。选取代表菌株(共25株分别代表25种RFLP类型)的16S rDNA扩增产物进行测序。由测序得到其近全长序列(1 337~1 491 bp),用BLAST软件与从Genbank(NCBI)中检索到的已知及相似度最高的菌种进行多重序列比对。结果表明,其中50株分别属于中华根瘤菌属、根瘤菌属、中慢生根瘤菌属;剩余65株明显区别于豆科植物结瘤细菌(legume-nodulating bacteria,LNB),分别属于副球菌属、鞘脂单胞菌属、固氮螺菌属、假单胞菌属、沙雷氏菌属、分枝杆菌属、诺卡氏菌属、链霉菌属、类芽孢杆菌属、短短芽孢杆菌属、Lysinibacillus、葡萄球菌属和芽孢杆菌属。
根据代表菌株的16S rDNA序列与相似度最高的菌株的序列构建出系统发育树(图1)。可知,25株代表菌株中有8株在系统分类上分别归属于中华根瘤菌属、根瘤菌属和中慢生根瘤菌属;剩余的17株分别归属于副球菌属、鞘脂单胞菌属、 固氮螺菌属、假单胞菌属、沙雷氏菌属、分枝杆菌属、诺卡氏菌属、链霉菌属、类芽孢杆菌属、短短芽孢杆菌属、Lysinibacillus、葡萄球菌属和芽孢杆菌属的系统发育分支上(图1)。
分支1为中华根瘤菌属,有1株菌。菌株Qtx-8-1与S.meliloti构成一个小的分支,Qtx-8-1与S.meliloti的相似率为99.78%。
分支2为根瘤菌属。菌株Qtx-3采自宁夏青铜峡市,经比对Qtx-3与属内其它参比菌株的最大相似率为97.36%,可能为潜在的新种。菌株Qtx-10-1与R.leguminosarum形成另一小分支,Qtx-10-1与R.leguminosarum的相似率为100%。说明Qtx-10-1与R.leguminosarum是同一种菌。Zy-3-3与R.galegae的相似率为99.61%。
分支3为中慢生根瘤菌属,共有Qtx-24、Zw-2-1、Zy-2和Gz-34四株菌。经比对,Qtx-24与M.mediterraneum相似率为99.71%;Zw-2-1与M.gobiense相似率为100%,说明它们为同一种菌;Zy-2与M.tianshanense的相似率为99.21%;Gz-34与M.amorphae相似率为99.88%; Zw-2-1与M.mediterraneum的相似率为99.85%。
分支4为副球菌属,由Zy-3与其它参比菌株组成。Zy-3形成一单独的小分支,它与分支内其它参比菌株的最大相似率为96.58%,可能为副球菌属内潜在的新种。
分支5为鞘脂单胞菌属,经比对,Guashi-1与S.pruni、S.macrogoltabida和S.ginsengisoli的相似率分别为99.18%、98.92%、98.63%。
分支6为固氮螺菌属,由Mq-10与I.limosus组成,Mq-10与I.limosus的相似率为99.69%。
分支7为假单胞菌属,经比对,Zy-2-1与P.brassicacearum、P.fluorescens和P.kilonensis的相似率依次为99.79%、99.72%、99.72%。
分支8为沙雷氏菌属,Zw-22与S.plymuthica构成一小分支,它们之间的相似率为99.98%,Zw-22与S.grimesii和S.ficaria的相似率分别为98.52%、98.45%。
分支9为链霉菌属,Gt-10与S.bottropensis形成一小支,相似率为100%。Gt-10与S.europaeiscabiei相似率为99.03%;与S.stelliscabiei相似率为98.75%。
分支10为诺卡氏菌属,Gt-25与N.uniformis组成一单独的小分支,经比对它们之间的相似率达到100%。
分支11为分枝杆菌属,Qtx-19与M.sacrum构成一小分支,它们之间相似度为100%。Qtx-19与M.diernhoferi、M.lacticola和M.wolinskyi的相似率依次为98.79%、98.93%和98.00%。
分支12为类芽孢杆菌属,Gt-1与P.amylolyticus组成一单独的小分支,其相似率为99.64%;与P.tylopili相似率为99.49%。
3 讨论与结论
经过比对16S rDNA近全长的序列(1 337~1 491 bp), 表明65株内生菌具有丰富的遗传多样性,并分别归属于3个不同的菌门:变形菌门(Proteobacteria,革兰氏阴性菌),放线菌门(Actinobacteria)和厚壁菌门(Firmicutes,革兰氏阳性菌)。在变形菌门中有4个菌株属于α-变形菌纲(Paracoccus,Sphingomonas和Inquilinus);11个属于γ-变形菌纲(Pseudomonas和Serratia);6个菌株分别归属于放线菌门中的Mycobacterium,Nocardia和Streptomyces;有45株分别归属于厚壁菌门中的Paenibacillus,Brevibacillus,Staphylococcus,Lysinibacillus和Bacillus,其中芽孢杆菌属是优势根瘤内生菌,占苦马豆根瘤中所有内生细菌的58.5%。基于16S rRNA基因序列的系统发育分析表明,38株芽孢杆菌分别归属于5个种(Bacillussimplex,Bacillussafensis,Bacilluscereus,Bacilluspumilus,Bacilluslicheniformis)。Serratiaplymuthica,Lysinibacillusfusiformis和Staphylococcussaprophyticus这几个菌种是没有从根瘤中分离到的内生菌。另外,Qtx-14 和Zy-3 与最近的已知种序列相似率小于97%,预示着它们为潜在的新种。
有研究者从黑龙江地区大豆根瘤中分离出的菌株经16S rDNA序列系统发育分析鉴定为泛菌属(Pantoea)、沙雷氏菌属、不动菌属(Acinetobacter)、芽孢杆菌属、土壤杆菌(Agrobacterium)和伯克氏菌属(Burkholderia),其中泛菌属(Pantoea)为大豆根瘤中的优势菌株[9]。沙雷氏菌属和芽孢杆菌属在本研究结果中也有发现,除此之外,本研究还发现了副球菌属、鞘脂单胞菌属、固氮螺菌属、假单胞菌属等其它内生菌,这些内生菌群体组成的差异可能是由于黑龙江地区与西北地区的环境条件及寄主植物的不同所引起的。已有研究从多种豆科植物中分离出土壤杆菌,并把它们鉴定为根瘤中非共生内生细菌[5]。然而,在本研究中,从苦马豆根瘤中没有分离到土壤杆菌,说明土壤杆菌对于西北地区苦马豆植物来说不是优势菌种。在豆科植物根瘤和其他植物组织中P.agglomerans是普遍存在的内生菌[7]。另外,有报道称P.agglomerans可以在Hedysarum上结瘤[4],但在本研究中也没有分离到。这些差异可能是因为根瘤中内生菌的群体组成受寄主植物、地理环境共同作用的结果,这与根瘤菌类似[22]。从不同的地理位置,同一寄主植物中分离到的内生菌归于不同的属或种,说明了环境条件对根瘤中细菌分类地位的影响大于寄主专一性的影响,这种现象已经普遍存在,在内生菌和根瘤菌的研究中都有过报道[4]。本研究从苦马豆根瘤中分离出的根瘤菌大多属于中慢生根瘤菌属,在中华根瘤菌属、根瘤菌属中也均有分布但很少,这点徐琳等[22]的研究结论一致。

图1 采用邻接法(Neighbor-joining method)绘制的代表菌株16S rRNA基因系统发育树状图Fig.1 16S rRNA gene sequence-based dendrogram generated by the neighbor-joining method.
注:分支上的数值表示大于70%的Bootstrap值。
Note:Bootstrap values(1 000 replicates) are indicated above the branches(>70%) showing the phylogenetic positions of representative strains(shown in bold) associated withSphaerophysasalsularoot nodules. Scale bar indicates 1% substitution of nucleotide
本研究结果表明,从同一样地苦马豆根瘤中分离的内生细菌基因型有可能比较相似,也可能差异很大,表现出丰富的多样性,不同样地分离的菌株可能各不相同,也可能有较好的一致性。每个样地中都分别存在明显的优势种和特有种群,样地间两两比较可以发现,有一部分基因型是样地间所共有的,特别是在性状上相近的样地间具有更多相似的基因型,而有些基因型则是有些样地所特有的。6个样地苦马豆根瘤中分离的内生细菌均具有明显的多样性,而且不同苦马豆根瘤中分离的内生细菌的遗传的多样性与其地理来源具有不同的相关性,此结果与前人[8-9]的研究结果一致。
本研究中,在一些根瘤中只分离出了内生菌,如:Gaoshi-1(B.cereus)和Gaoshi-7(Staphylococcussaprophyticus),此可能是在同一根瘤中由于内生菌生长的过快或者产生抗生素,从而影响了共生菌的生长[24-25];也可能是与根瘤菌有关的不可培养或难培养状态的存在导致了其不可培养,处于该状态下的根瘤菌可以通过直接PCR分析的方法进行检测,这还需进一步深入研究,以更好地深刻揭示其细菌遗传多样性的信息。
在野生豆科植物苦马豆的根瘤中,非共生的内生菌是普遍存在的;寄主植物的遗传背景和地理环境的共同作用是导致根瘤内生细菌遗传多样性的主要因素;在西北部分地区,苦马豆植物根瘤中的最优势内生细菌为芽孢杆菌属。本研究对确定根瘤内生菌特殊的系统发育地位及根瘤内生菌、根瘤菌、寄主植物和环境因子间互作关系的研究提供了基本信息。
References:
[1] Bai Y M,Daoust F,Smith D L,Driscoll B T.Isolation of plant-growth-promotingBacillusstrains from soybean root nodules.Candian Journal of Microbiology,2002,48(2):230-238.
[2] Bai Y,Zhou X,Smith D.Enhanced soybean plant growth due to coinoculation ofBacillusstrains withBradyrhizobiumjaponicum.Crop Science,2003,43(1):1774-1781.
[3] Barrett C F,Parker M A.Coexistence ofBurkholderia,Cupriavidus,andRhizobiumsp. nodule bacteria on twoMimosaspp. in Costa Rica.Applied and Environmental Microbiology,2006,72(7):1198-1206.
[4] Benhizia Y,Benhizia H,Benguedouar A,Muresu R,Giacomini A,Squartini A.Gamma proteobacteria can nodulate legumes of the genusHedysarum.Systematic and Applied Microbiology,2004,27(2):462-468.
[5] De Lajudie P,Willems A,Nick G,Mohamed T S,Torck U,Filai-Maltouf A,Kersters K,Dreyfus B,Lindström K,Gillis M.Agrobacteriumbv. 1 strains isolated from nodules of tropical legumes.System and Applied Microbiology,1999,22(1):119-132.
[7] Kan F L,Chen Z Y,Wang E T,Tian C F,Sui X H,Chen W X.Characterization of symbiotic and endophytic bacteria isolated from root nodules of herbaceous legumes grown in Qinghai-Tibet Plateau and in other zones of China.Archives of Microbiology,2007,188(10):103-115.
[8] Li L,Sinkko H,Montonen L,Wei G H,Lindström K,Räsänen L A.Biogeography of symbiotic and other endophytic bacteria isolated from medicinalGlycyrrhizaspecies in China.FEMS Microbiology Ecology,2012,79(2):46-68.
[9] Li J H,Wang E T,Chen W F,Chen W X.Genetic diversity and potential for promotion of plant growth detected in nodule endophytic bacteria of soybean grown in Heilongjiang Province of China.Soil Biology and Biochemistry,2008,40(7):238-246.
[10] Philipson M N,Blair I D.Bacteria in clover root tissue.Candian Journal of Microbiology,1957,3:125-129.
[11] Sturz A V,Christie B R,Matheson B G,Nowak J.Biodiversity of endophytic bacteria which colonize red clover nodules,roots,stems and foliage and their influence on growth.Biology and Fertility Soils,1997,25(3):13-19.
[12] Tokala R K,Strap J L,Jung C M,Crawford D L,Hamby Salove M,Deobald L A,Bailey F,Morra M J.Novel plantmicrobe rhizosphere interaction involvingStreptomyceslydicusWYEC108 and the pea plant(Pisumsativum).Applied and Environmental Microbiology,2002,68(1):2161-2171.
[13] Vandamme P,Goris J,Chen W M,De vos P,Willems A.Burkholderiatuberumsp. nov. andBurkholderiaphymatumsp.nov.,nodulate the roots of tropical legumes.Systematic and Applied Microbiology,2002,25(9):507-512.
[14] Zakhia F,Jeder H,Willems A,Gillis M,Dreyfus B,de Lajudie P.Diverse bacteria associated with root nodules of spontaneous legumes in Tunisia and first report fornifH-like gene within the generaMicrobacteriumandStarkeya.Microbiology and Ecology,2006,51(7):375-393.
[15] Zakhia F,de Lajudie P.Taxonomy ofRhizobia.Agronomie,2001,21(5):569-576.
[16] Sprent J I.Nodulation in Legumes.In:Janet S.(ed).London:Royal Botanic Gardens,2001.
[17] Terefework Z,Kaijalainen S,Lindström K.AFLP fingerprint as a tool to study the genetic diversity ofRhizobiumgalegaeisolated fromGalegaorientalisandGalegaofficinalis.Journal Biotechnology,2001,91(6):169-180.
[18] Weisburg W G,Barns S M,Pelletior D A,Lane D J.16S ribosomal DNA amplification for phylogenetic study.Journal of Bacteriology,1991,173(4):697-703.
[19] υan Berkum P,Beyene B,Eardly B D.Phylogenetic relationships amongRhizobiumspecies nodulating the common bean (PhaseolusvulgarisL.).International Journal Systematic Bacteriology,1996,46(3):240-244.
[20] Vinuesa P,Silva C,Lorite M J,Izaguirre-Mayoral M L,Bedmar E J,Martínez-Romero E.Molecular systematics of rhizobia based on maximum likelihood andBayesianphylogeniesinferred fromrrs,atpD,recAandnifHsequences,and their use in the classification ofSesbaniamicrosymbiontsfromVenezuelanwetlands.Systematic and Applied Microbiology,2005,28:702-716.
[21] Tlusty B,van Berkum P,Graham P H.Characteristics of the rhizobia associated withDaleaspp. in the Ordway,Kellogg-Weaver Dunes and Hayden prairies.Canadian Journal of Microbiology,2005,51(2):15-23.
[22] 徐琳,徐佳洁,刘巧莉,谢瑞美,韦革宏.中国西北不同地区苦马豆根瘤菌的遗传多样性.生物多样性,2009,17(1):69-75.
Xu L,Xu J J,Liu Q L,Xie R M,Wei G H.Genitic diversity in rhizobia isolated fromSphaerophysasalsulain several regions of northwestern of China.Biodiversity Science,2009,17(1):69-75.(in Chinese)
[23] 李振东,陈秀蓉,杨成德.珠芽蓼内生菌Z17抑菌能力测定及其鉴定.草业科学,2011,28(12):2096-2101.
Li Z D,Chen X R,Yang C D.Identification ofPolygonumviviparumendophytic bacteria Z17 and its capacity to antagonistic towards pathogenic fungi.Pratacutural Science,2011,28(12):2096-2101.(in Chinese)
[24] 毕江涛,王小霞,陈卫民,王静,贺达汉.甘草内生真菌分离及其抑菌活性初探.草业科学,2013,30(3):357-364.
Bi J T,Wang X X,Chen W M,Wang J,He D H.Isolation of endophytic fungi from medicinal plantGlycyrrhizauralensisand its microbial inhibition activity.Pratacutural Science,2013,30(3):357-364.(in Chinese)
[25] 邓振山,李军.豆科植物根瘤中非共生的内生菌遗传多样性研究进展.微生物学杂志,2012,32(4):63-68.
Deng Z S,Li J.Genentic diversity of nonsymbiosis endophytic bacteria in leguminous root nodules.Journal of microbiology,2012,32(4):63-68.(in Chinese)
(责任编辑 苟燕妮)
Genetic diversity of endophytic bacteria in nodule ofSphaerophysasalsula
Deng Zhen-shan
(College of Life Sciences, Yan’an University, Yan’an 716000, China)
Sphaerophysasalsulais an important legume distributed in northwestern China. In order to better understand the diversity and phylogeny of endophytic bacteria collected from nodules ofSwainsoniasalsulain different regions of northwestern China, genetic diversity of these isolated endophytic bacteria was estimated using 16S rDNA PCR-RFLP and 16S rDNA sequencing. The results showed that these 115 bacteria strains had 28 genetic types which were further identified by 16S rDNA sequencing. These strains belonged to the following genera:Mesorhizobium,Rhizobium,Sinorhizobium,Paracoccus,Sphingomonas,Inquilinus,Pseudomonas,Serratia,Mycobacterium,Nocardia,Streptomyces,Paenibacillus,Brevibacillus,Lysinibacillus,Staphylococcus,Bacillus. These results demonstrated that endophytic bacteria in nodule had rich genetic diversity inS.salsula.
Sphaerophysasalsula; 16S rDNA PCR-RFLP; genetic diversity; phylogeny
Deng Zhen-shan E-mail:zhenshandeng214@163.com
10.11829/j.issn.1001-0629.2015-0688
2015-12-04 接受日期:2016-05-24
陕西省科技统筹创新工程项目(2012CGX7);陕西省科技统筹创新工程项目(2012KTZB03-02-03);陕西省科技统筹创新工程项目 (2012KTZB03-02-03);陕西省科技统筹创新工程项(2016TTC-N-3-1);延安市科技局重大专项项目(2014CGZH-06);陕西省教育厅服务服务地方专项计划项(16JF029);延安大学“陕北微生物资源与利用研究中心”科研机构专项基金
邓振山(1969-),男,陕西黄陵人,副教授,博士,主要从事微生物资源与利用和环境微生物研究。E-mail:zhenshandeng214@163.com
S154.3+1;Q943
A
1001-0629(2016)10-1951-12*
邓振山.苦马豆根瘤中内生细菌遗传多样性分析.草业科学,2016,33(10):1951-1962.
Deng Z S.Genetic diversity of endophytic bacteria in nodule ofSphaerophysasalsula.Pratacultural Science,2016,33(10):1951-1962.
