黄酮与溶菌酶相互作用的强度衰减—基质辅助激光解吸离子化—质谱研究
2016-12-08唐君付强崔勐邢俊鹏刘志强刘淑莹
唐君+付强+崔勐+邢俊鹏+刘志强+刘淑莹

摘要 建立了黄酮与溶菌酶相互作用研究的强度衰减-基质辅助激光解吸离子化-质谱(Intensity fading matrix-assisted laser desorption/ionization mass spectrometry,IF-MALDI-MS)分析方法。在优化的基质DHB条件下,分别研究了木犀草素、染料木素、芹菜素、槲皮素和大豆黄素与溶菌酶相互作用,比较了溶菌酶加入前后黄酮的相对丰度的变化,并通过竞争实验的方法研究了5种黄酮与溶菌酶结合的亲和性大小。结果表明,5种黄酮与溶菌酶均存在相互作用,亲和性强弱为:木犀草素>芹菜素,染料木素>槲皮素>大豆黄素。结合5种黄酮的结构特征,讨论了黄酮的结构及其与溶菌酶亲和性的关系,发现C5位和C3′位的羟基有利于黄酮与溶菌酶的结合,C3位的羟基不利于黄酮与溶菌酶的结合。本方法具有简便,快速,高效等优点,也可以应用于其它天然产物与蛋白质的相互作用的研究。
关键词 黄酮; 溶菌酶; 相互作用; 基质辅助激光解吸离子化-质谱
2016-01-29收稿;2016-03-11接受
本文系吉林省科技发展计划项目(No. 201105032)资助
E-mail: cuimeng@ciac.ac.cn
1 引 言
黄酮是一类重要的天然产物,广泛存在于各种植物及植物制品中。很多研究报道了它们具有抗炎、抗氧化、抗过敏、抗血栓、抗菌和抗癌等生理活性[1~3]。而黄酮的诸多生理活性主要通过与体内的各种酶、受体、载体的结合而发挥作用[4~6]。溶菌酶是人体内的一种非特异性免疫蛋白,广泛存在于体液和血清中[7~10]。研究发现溶菌酶可以作为药物的载体,协助药物在体内的吸收和转运[11~13]。所以研究溶菌酶和黄酮的相互作用,不仅有助于了解黄酮的生理活性,更有助于了解黄酮在体内的吸收及代谢情况。
目前, 研究溶菌酶与黄酮相互作用的方法主要有紫外可见光谱、红外光谱、圆二色谱和荧光光谱等方法[14~17]。但是这些方法的灵敏度和分析通量等尚有不足。随着现代质谱的发展,质谱以其快速、灵敏、准确等特点在蛋白质与小分子配体研究中起着越来越重要的作用。但是有关黄酮与蛋白质相互作用的研究多局限于电喷雾质谱的方法,例如Chen等曾用电喷雾质谱研究了磷酰衍生化后的黄酮与溶菌酶的相互作用 [18,19]。2003年,Villanueva等[20]首次建立了蛋白质与小分子配体相互作用的强度衰减-基质辅助激光能吸电离-质谱研究方法,该方法主要是基于加入靶蛋白后,小分子配体的离子峰强度发生衰减以识别配体与蛋白质的相互作用。目前该方法已应用到蛋白质和小肽[21]或生物碱相互作用[22]以及配体筛选 [23]的研究中,而对于溶菌酶和黄酮相互作用的研究尚未见报道。本研究建立了黄酮和溶菌酶相互作用的强度衰减-基质辅助激光解吸离子化-质谱(IF-MALDI-MS)方法,不仅快速识别出与溶菌酶相互作用的黄酮化合物,还比较了不同的黄酮与溶菌酶的结合强弱。与电喷雾质谱方法相比,本方法不仅具有传统MALDI质谱方法的耐盐、分析速度快等优点,而且还能克服传统MALDI质谱分析中黄酮与溶菌酶复合物易解离等缺点。本方法不仅为研究其它天然产物与蛋白质的相互作用提供方法学参考,还可应用于与蛋白质相互作用的靶分子的快速筛选。
2 实验部分
2.1 仪器与试剂
MALDI-TOF MS:配有337 nm 脉冲N2激光的Voyager DE-STR仪器 (Applied Biosystems, Framingham, MA)。
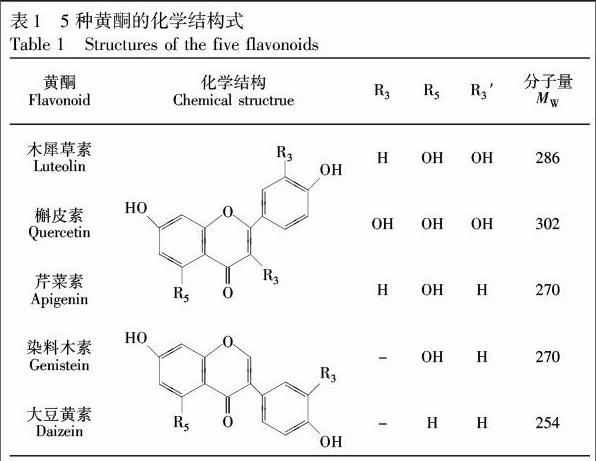
溶菌酶(Sigma-Aldrich公司),使用前未做进一步的纯化。黄酮:木犀草素、染料木素、芹菜素、槲皮素、和大豆黄素(纯度≥98%,上海源叶生物科技有限公司), 5种黄酮的结构如表1所示。 三氮乙酰葡糖胺Tri-N-acetylglucosamine (NAG3,纯度≥98%,加拿大Toronto Research Chemicals公司); 醋酸铵(分析纯)、棉籽糖(Fluka公司); 甲醇(HPLC级,Fisher Chemical公司); MALDI基质:2,5-Dihydroxybenzoic acid (DHB)、Sinapinic acid (SA)、α-Cyano-4-hydroxycinnamic acid (CHCA)、2,4,6-Trihydroxyacetophenone (THAP)、2,5-Dihydroxyacetophenone (DHAP)、Hyaluronan (HA)、3-Aminoquinoline (AQ)、3-Hydroxy picolinic acid (HPA)均购于Sigma Aldrich公司; 乙腈(色谱纯,Fisher公司)。超纯水(18.2 MΩ cm)由 MilliQ 设备(Millipore公司)制得。
2.2 样品制备
溶菌酶、NAG3和棉籽糖分别用Milli-Q水制备成1 mmol/L的储备液。黄酮:木犀草素,染料木素,芹菜素,槲皮素和大豆黄素分别用甲醇溶解,并用Milli-Q水稀释制成1 mmol/L的储备液。MALDI基质DHB、CHCA、DHAP、THAP、HPA、AQ、SA分别用乙腈-水(1∶1, V/V)配制成浓度为10 mg/mL的溶液。样品采用干滴法点样。基质低质量区干扰实验中,取1 L基质溶液点靶,室温干燥后进行MALDI-MS分析。模型体系的IF-MALDI-MS实验中,将NAG3、棉籽糖和水/溶菌酶以 3∶3∶1(V/V)混合制备成为待测溶液,在37℃条件下温浴5 min。将待测液与基质溶液等体积混合,取1 L混合液点靶,室温干燥后进行MALDI-MS分析。黄酮与溶菌酶的IF-MALDI-MS实验中,将黄酮,棉籽糖和水/溶菌酶以 3∶3∶1(V/V)混合制备成为待测溶液,在37℃下温浴5 min。将待测液与基质溶液等体积混合,取1 L混合液点靶,室温干燥后进行MALDI-MS分析。竞争实验是将黄酮A、黄酮B、棉籽糖、水/溶菌酶以1.5∶1.5∶3∶1(V/V)混合,在37℃下反应5 min后,将待测液和等体积的DHB溶液混合,取1 L混合液点靶,室温干燥后进行MALDI-MS分析,每个样品测定3次,取平均值。
2.3 质谱条件
采用正离子反射模式:激光源为337 nm的N2源,质量扫描范围m/z 200~1500,加速电压设置为22 kV,延长时间设置为100 ns,每张谱图均通过100次激光辐照累加平均得到。
3 结果与讨论
3.1 MALDI基质优化
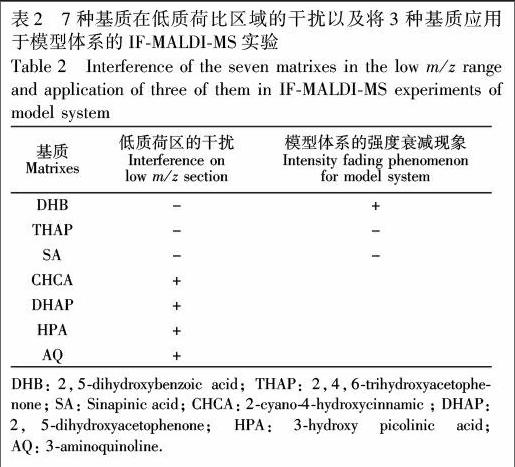
由于5种黄酮(木犀草素、芹菜素、槲皮素、染料木素和大豆黄素)的分子量均在200~400 之间(表1),选择在低质荷比区域(m/z 200~400)干扰较小的基质非常重要,因而对7种基质DHB、CHCA、DHAP、THAP、HPA、AQ和SA在低质荷比区域的出峰情况进行了MALDI-MS研究。实验结果如表2所示,对黄酮的MALDI-MS检测的影响较小,用“-”表示;而在低质荷比区域出峰较多,对黄酮的检测干扰较大的基质用“+”表示。结果表明,3种基质DHB、THAP和SA在低质荷比区域出峰较少。
3.2 模型体系的IF-MALDI-MS实验
利用3种在低质荷比区域干扰较小的基质(DHB、THAP、SA)对模型体系进行了IF-MALDI-MS实验。选择溶菌酶和与其特异性结合的NAG3[24]和内标棉籽糖为模型体系,分别对混合溶液进行MALDI-MS检测,比较了在加入溶菌酶前后NAG3与内标峰强度比的变化。由表2可知,只有当DHB作基质时,NAG3与内标峰强度比降低明显(数据未给出),用”+”表示。THAP和SA作基质时,未观察到模型体系的强度减弱现象,用“-”表示,因此,本实验中选择DHB作为基质。
3.3 溶菌酶与黄酮的IF-MALDI-MS实验
将5种黄酮分别与棉籽糖混合制备成为待测溶液,对待测溶液加入溶菌酶前后的混合液进行IF-MALDI-MS实验,结果如图1所示。其中,左栏为加入溶菌酶之前的混合液的MALDI的质谱图,右栏为加入溶菌酶之后的混合液的MALDI质谱图。从图1可见,加入溶菌酶后,5种黄酮与内标的峰强度比均减弱,说明这5种黄酮与溶菌酶均存在相互作用。图1b中离子峰m/z 481.7为基质DHB的加合离子峰[(DHB)3+H2O+H]+。由图1还可见,加入溶菌酶样品后,内标的[M+Na]+离子峰较[M+K]+有显著升高, 这很可能是由于溶菌酶样品在制备过程中会有少量的Na+引入,以及Na+与小寡糖分子的结合力要高于K+导致的[25]。
3.4 竞争实验
为了研究5种黄酮与溶菌酶的亲和性大小,采用竞争实验的方法,首先将黄酮A、黄酮B以及内标(IS)按一定比例混合成为待测液,对待测液加入溶菌酶前后的混合液进行MALDI-MS测定。比较加入溶菌酶前后的黄酮A和黄酮B相对于内标的离子峰强度比的变化(ΔRI(A)和ΔRI(B)),以下是计算公式:
其中,I(A)o, I(B)o和I(IS)o分别表示的是加入溶菌酶前的MALDI质谱图中黄酮A,黄酮B和内标IS的相对丰度。I(A),I(B) 和I(IS)分别表示加入溶菌酶后的MALDI质谱图中黄酮A,黄酮B和内标IS的相对丰度,其中内标的相对丰度均为[M+Na]+峰和[M+K]+峰的相对丰度的总和。以染料木素和木犀草素的竞争实验为例,图2a表示的是加入溶菌酶前的染料木素(MA)、木犀草素(MB)和内标(IS)的MALDI质谱图,图2b表示加入溶菌酶之后的染料木素(MA)、木犀草素(MB)和内标(IS)的MALDI质谱图。经过计算发现,ΔRI(木犀草素)>ΔRI(染料木素),说明木犀草素与溶菌酶的结合亲和性比染料木素更强。
其它黄酮的竞争实验的分析结果如表3所示。5种黄酮与溶菌酶结合亲和性顺序为:木犀草素>芹菜素,染料木素>槲皮素>大豆黄素。由于芹菜素和染料木素的分子量相同,所以没有对两者与溶菌酶的亲和性强弱进行比较。采用IF-MALDI-MS方法所得到的黄酮与溶菌酶的亲和性大小与荧光结果是一致的[26],进一步证实了本方法的可靠性。
3.5 黄酮结构和黄酮与溶菌酶亲和性关系的研究
Yang 等[26]采用荧光光谱方法发现,黄酮与溶菌酶结合的亲和性与黄酮的羟基的位置和数量有关。本研究结果表明:(1)黄酮苷元C3位的羟基不利于黄酮与溶菌酶的结合。槲皮素比木犀草素在C3位多一个羟基,其与溶菌酶的亲和性小于木犀草素。(2)黄酮苷元的C3′位的羟基有利于黄酮与溶菌酶的结合。木犀草素比芹菜素在B环的C3′位多一个羟基,其与溶菌酶的亲和性大于芹菜素。(3)黄酮苷元的C5位的羟基有利于黄酮与溶菌酶的结合。染料木素比大豆黄素在A环的C5位多一个羟基,其与溶菌酶的亲和性大于大豆黄素。
References
1 Mamadalieva N Z, Herrmann F, El-Readi M Z, Tahrani A, Hamoud R, Egamberdieva D R, Azimova S S,Wink M. J. Pharm. Pharmacol., 2011,
63(10): 1346-1357
2 Luo Y, Li X, He J, Su J, Peng L, Wu X, Du R, Zhao Q. Food Chem., 2014, 164: 30-35
3 Hoensch H, Oertel R. Dtsch. Med. Wochenschr., 2012, 137(51-52): 2738-2740
4 Middleton E, Kandaswami C, Theoharides T C. Pharmacol. Revi., 2000, 52(4): 673-751
5 Dangles O, Dufour C. Flavonoids: Chem. Biochem. Appl., 2006: 443-469
6 Havsteen B H. Pharmacol. Ther., 2002, 96(2): 67-202
7 Cohn Z A, Hirsch J G. J. Exp. Med., 1960, 112(6): 983-1004
8 Greenwald R A, Josephso A S, Diamond H S, Tsang A. J. Clin. Invest., 1972, 51(9): 2264-2270
9 Pinkus G S, Said J W. Am. J. Pathol., 1977, 89(2): 351-366
10 Jash C, Kumar G S. RSC Adv., 2014, 4(24): 12514-12525
11 Paramaguru G, Kathiravan A, Selvaraj S, Venuvanalingam P, Renganathan R. J. Hazard. Mater., 2010, 175(1-3): 985-991
12 Ding F, Zhao G, Huang J, Sun Y, Zhang L. Eur. J. Med. Chem., 2009, 44(10): 4083-4089
13 Ding F, Li X N, Diao J X, Sun Y, Zhang L, Ma, L, Yang, X L, Zhang L, Sun Y. Ecotox. Environ. Safe., 2012, 78: 41-49
14 Roy A S, Utreja J, Badhei S. J. Incl. Phenom. Macrocycl. Chem., 2015, 81(3-4): 385-394
15 Cheng L L, Wang M, Wu M H, Yao S D, Jiao Z, Wang S L. Spectrochim Acta A, 2012, 97: 209-214
16 Huang Y, Cui L J, Wang J M, Huo K, Chen C, Zhan W H, Dou Y H. Eur. J. Med. Chem., 2011, 46(12): 6039-6045
17 CHEN Lin, HUANG Ping, YANG Hui-Qing, DENG Ya-Bin, LI Dong-Hui. Chinese J. Anal. Chem., 2014, 42(7): 962-967
陈 林, 黄 萍, 杨惠卿, 邓雅斌, 李东辉. 分析化学, 2014, 42(7): 962-967
18 Chen X L, Qu L B, Zhang T, Liu H X, Yu F, Yu Y Z, Liao X C, Zhao Y F. Anal. Chem., 2004, 76(1): 211-217
19 Chen X L, Liu H X, Qu L B, Yu Y Z, Lu J S, Zhao Y F. Anal. Chim. Acta, 2004, 511(1): 175-182
20 Villanueva J, Yanes , Querol E, Serrano L, Aviles F X. Anal. Chem., 2003, 75(14): 3385-3395
21 Wang Z, Yu X, Cui M, Liu Z, Song F, Liu S. J. Am. Soc. Mass Spectrom., 2009, 20(4): 576-583
22 Liu W, Liu S, Li H, Song F, Liu Z, Liu S. Rapid Commun. Mass Spectrom., 2011, 25(7): 973-978
23 Yanes , Villanueva J, Querol E, Aviles F X. Drug Discovery Today Targets, 2004, 3(2): 23-30
24 Von Dreele R B. Acta Crystallogr. Sect. D-Biol. Crystallogr., 2005, 61: 22-32
25 Cancilla M T, Penn S G, Carroll J A, Lebrilla C B. J. Am. Chem. Soc., 1996, 118(28): 6736-6745
26 Yang R, Yu L, Zeng H, Liang R, Chen X, Qu L. J. Fluoresc., 2012, 22(6): 1449-1459
Abstract Flavonoids are reported to have a variety of biological activities. Investigations on the interactions of flavonoids with protein are essential to understand the biological activities of flavnoids. In this paper, by using the optimized matrix 2,5-dihydroxybenzoic acid (DHB), we investigated the interaction of five flavonoids (luteolin, apigenin, genisten, quercetin, dazein) with lyszoyme using intensity fading (IF)-MALDI-MS method. The changes of relative abundances of flavonoids before and after addition of lyszoyme were calculated, and the binding affinity order was obtained by competitive experiments. It was found that all of the five flavonoids bound specifically to lysozyme. And the order of binding affinities of the five flavonoids to the lysozyme was luteolin>apigenin, genisten>quercetin>dazein. Based on their structures, it was found that the hydroxyl on C5 and C3′ benefited the binding of flavonoid towards lyszoyme. However, the hydroxyl on C3 hindered the binding of flavonoid towards lysozyme. The results should be helpful to understand the biological activity of flavonoids. And the IF-MALDI-MS method could also be used to screening the interactions of other natural products and protein.
Keywords Flavonoid; Lysozyme; Interaction; Matrix-assisted laser desorption/ionization; Mass Spectrometry
