沟眶象转录组微卫星特征分析
2016-11-18武政梅温俊宝
武政梅,高 朋,温俊宝
(北京林业大学林木有害生物防治北京市重点实验室,北京100083)
沟眶象转录组微卫星特征分析
武政梅,高 朋,温俊宝*
(北京林业大学林木有害生物防治北京市重点实验室,北京100083)
本研究基于高通量测序获得的沟眶象Eucryptorrhynchuschinensis(Olivier)转录组数据,采用MISA(Micro Satellite)软件对其简单序列重复(simple sequence repeat, SSR)位点进行了高通量发掘。对筛选得到的1 kb以上的Unigene(17590条,占Unigene总数的26.98%)进行了SSR分析,结果发现含SSR的序列有1665条,共发现1823个SSR,平均32.52 kb出现一个SSR。沟眶象微卫星主要以三碱基重复类型为主(637,34.94%),其次为单碱基重复(576,31.60%)。共发现104种碱基重复基元,所占比例最高的为(A/T)n(30.39%),其次为(AT/AT)n(6.91%)。沟眶象SSR的平均长度为15.74 bp,长度大于20 bp的SSR仅占总数的12.70%。研究还发现沟眶象微卫星出现的频率和其长度呈极显著负相关(P<0.01),相关系数为-0.572。研究结果为开发高多态性微卫星引物来进行沟眶象功能基因组学、种群遗传结构、种群遗传多样性等研究奠定了基础。
沟眶象;微卫星;转录组;重复类型;重复基元
微卫星(microsatellite)又称简单序列重复(simple sequence repeat, SSR),一般以1-6个碱基为核心序列,其长度通常较短(何平,1998)。由于这些序列广泛存在于真核细胞的基因组(史洁等,2012),同时具有可重复性高、多态性强、数量丰富、共显性、对基因组有很好覆盖性等特点(Liuetal., 2013),因此微卫星已经成为不同物种遗传研究中使用最广泛的分子标记(王丽鸳等,2014),已被广泛应用于种群遗传多样性分析、动植物分类和进化、遗传图谱的构建、基因定位和克隆、分子标记辅助育种、品种鉴定等领域(Lietal., 2002; Varshneyetal., 2005)。SSR按来源分为基因组SSR(Genomic SSR, gSSR)和表达序列标签SSR(Expressed SSR,EST-SSR)(王东等,2014)。两种SSR相比较,因EST-SSR源于基因的转录区,其多态性与基因功能可能有直接关系(Eujayletal., 2002),因此通用性更高。新一代高通量测序技术的快速发展为利用转录组序列开发SSR标记带来了福音,它不仅具有EST-SSR标记的优点,而且能产生更为海量的转录组数据,因此为SSR标记的开发提供了更全面的信息,从而提高了遗传多样性研究的准确性。目前,研究者们已经对很多物种转录组中的微卫星进行了研究分析,鞘翅目昆虫中就有云南切梢小蠹Tomicusyunnanensis(袁远等,2014)、绿豆象Callosobruchuschinensis(Duanetal., 2014)、黄粉虫Tenebriomolitor(Zhuetal., 2013)等,结果显示在不同物种中微卫星的分布特征差异很大。
沟眶象EucryptorrhynchuschinensisOlivier 隶属鞘翅目Coleptera象甲科Curculioniclae隐喙象亚科Cryptorrhynchinae沟眶象属EucryptorrhynchusHdler,主要以幼虫蛀食臭椿根部为害。由于近10多年来检疫不严,沟眶象及其近缘种臭椿沟眶象Eucryptorrhynchusbrandti已传播扩散至西北多个省区并成片严重发生,整个宁夏二代农田林网中臭椿受害株率达15%以上,致死率达7%,严重影响了臭椿的环境生态效益和观赏经济价值(杨贵军等,2008a)。目前有关沟眶象的报道,主要集中在生物学、生态学以及防治措施等领域,几乎没有有关其遗传背景和分子生物学方面的研究。本文根据已构建的沟眶象转录组数据库,对其微卫星进行了高通量发掘,旨在为今后开发高多态性微卫星引物,对沟眶象进行功能基因组学、种群遗传结构、种群遗传多样性等研究打下基础。
1 材料与方法
1.1 试虫来源和转录组数据
沟眶象样品采自宁夏回族自治区银川市灵武市梧桐树乡一队,经液氮瞬时冷冻后放于-80℃的冰箱中保存备用。沟眶象总RNA样品检测合格后,利用Illumina HiSeq 2500测序平台进行高通量深度测序,共获得65186条Unigenes,转录组数据未发表。
1.2 微卫星的筛选
利用MISA软件(Micro Satellite identification tool)(http://www.pgrc.ipk-gatersleben.de/misa/)对沟眶象转录组中1 kb以上的Unigene进行SSR位点分析,筛选标准为:单碱基、二碱基、三碱基、四碱基、五碱基、六碱基的最少重复次数分别为12、6、5、5、5、5。复合SSR两个位点间最大间隔碱基数为(Maximal number of bases interrupting 2 SSRs in a compound microsatellite):100。
2 结果与分析
2.1 沟眶象转录组序列中SSR重复的分布
本研究利用MISA软件对筛选得到的1 kb以上的Unigene进行了SSR 分析,评估序列拼接总长度为59281851 bp,获得SSR数目为1823个(完整型SSR 1709个),这些SSR分布于1665条序列中,平均相隔32.52 kb出现一个SSR序列,SSR的出现频率为10.36%。本研究仅对沟眶象转录组中1-6碱基完整型SSR进行分析,部分SSR信息见表1。
2.2 沟眶象转录组重复基元的分布
在沟眶象所有碱基重复类型中,三碱基重复SSR含量最多,约占总数的37.27%,其次为单碱基重复SSR(33.70%)、二碱基重复SSR(26.04%),没有发现六碱基重复SSR。研究还发现在沟眶象104种重复基元中,(A/T)n、(AT/AT)n、(TTA/TAA)n、(ATAA/TTAT)n、(AGGTT/AACCT)n和(AAGTA/TACTT)n分别在单、二、三、四、五碱基中出现频率最多,它们在各自重复基元类型中的比例分别是96.18%、28.31%、14.76%、23.26%、50.00%(表2)。
在沟眶象的104种碱基重复基元中,单、二、三、四、五碱基重复基元分别有4、12、58、23、7种,出现频率最高的是(A/T)n(30.39%),其次是(AT/AT)n(6.91%)、(TA/TA)n(6.80%)、(TTA/TAA)n(5.16%)、(AAT/ATT)n(4.44%)等。不同类型重复基元的SSR分布见图1。

表1 沟眶象转录组中微卫星数据库的部分结果

表2 沟眶象转录组中不同微卫星优势重复基元出现的频率
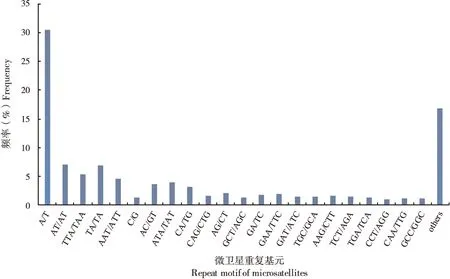
图1 基于重复基元类型的微卫星分布(考虑到碱基互补)Fig.1 Microsatellites distribution on different repeat motifs (considering sequence complementary)注:Other motifs 表示频率小于0.80%的重复基元类型。Note:Other motifs denote the repeat motifs with frequency below 0.80%.
2.3 沟眶象转录组序列中微卫星的长度分布
沟眶象转录组中所发现的1709个完整型SSR中,微卫星长度存在显著变异,从12-48个碱基不等,平均长度为15.74个碱基。如图2显示,沟眶象微卫星以重复长度小于20 bp的短重复序列最多,其中12-15 bp的数量最多(1098,64.25%),其次为16-20 bp(394,23.05%),长度大于20 bp的长序列重复仅占微卫星总数的12.70%。本研究进一步利用SPSS软件进行Person相关性分析,结果表明沟眶象微卫星出现的频率和其长度呈极显著负相关(P<0.01),相关系数为-0.572。

图2 沟眶象转录组中微卫星的长度分布Fig. 2 Length distribution of microsatellites in Eucryptorrhynchus chinensis transcriptome
3 结论与讨论
本研究从沟眶象转录组中发掘到1823个SSR位点,分布于1665条Unigene序列中。沟眶象SSR的出现频率为10.36%,这与黑翅土白蚁Odontotermesformosaanus(9.98%)(Huangetal., 2012)中SSR的出现频率差不多,高于烟粉虱Bemisiatabaci(5.07%)(Xieetal., 2012)、细梢小卷蛾Rhyacionialeptotubula(3.09%)(Zhuetal., 2013)、粘虫Mythimnaseparate(1.93%)(胡艳华等,2015)中SSR的出现频率。鞘翅目昆虫中,云南切梢小蠹Tomicusyunnanensis(袁远等,2014)和黄粉虫Tenebriomolitor(Zhuetal., 2013)转录组中检测出的SSR的出现频率分别为1.29%、1.79%,明显低于沟眶象转录组中SSR的出现频率,这说明沟眶象转录组中SSR数量相对来说十分丰富。分析其中的原因,除了SSR位点的搜索方法或计算标准有差异外,最根本的原因可能是物种特异性(黄海燕等,2013)。
在鉴定得到的沟眶象转录组SSR中,三碱基重复类型的数量最高,约占总数的33.70%。这与绿豆象Callosobruchuschinensis(Duanetal., 2014)、二点委夜蛾Athetislepigone(Lietal., 2013)、白背飞虱Sogatellafurcifera(Xuetal., 2012)、云南切梢小蠹Tomicusyunnanensis(袁远等,2014)等昆虫的SSR以三碱基重复为主类似。大部分昆虫中的微卫星都是以三碱基重复为主导重复类型,这有可能是为了防止蛋白质编码时发生移码突变现象(Sunetal., 2011)。本研究中沟眶象微卫星重复基元中所占比例最高的为(A/T)n,达到30.39%,这与黄粉虫、云南切梢小蠹、扶桑绵粉蚧Phenacoccussolenopsis(罗梅等,2014)的转录组中SSR的分析结果一致。沟眶象三碱基重复中,TTA/TAA为优势重复基元,而鞘翅目昆虫中的黄粉甲和云南切梢小蠹三碱基重复中都是以AAT/ATT为优势重复基元。这种差异可能与昆虫自身SSR特点以及转录组数据来源等紧密相关。
SSR分子标记的多态性是判断其可用性的重要依据,而其长度又是影响其多态性高低的重要因素。Temnykh等(2001)的研究表明,当SSR长度≥20 bp时多态性较高,长度在12-20 bp时多态性中等,而长度在12 bp以下时SSR多态性极低。本研究在SSR筛选过程中已经将长度在12 bp 以下的SSR过滤掉,最终发现沟眶象转录组中SSR长度在12-48个碱基不等,平均长度为15.74个碱基,具有高多态性的SSR所占比例仅为12.70%,这部分多态性潜能高的SSR在高效开发沟眶象微卫星标记方面应该具有较高的利用价值(杨华等,2011)。本研究还发现沟眶象微卫星出现的频率和其长度呈极显著负相关(P<0.01),相关系数为-0.572。根据Samadi等(1998)的研究可以得出,基因组中长度较短的SSR变异速率较快,而长度较长的SSR变异速率则较慢,相对来说较稳定。虽然基因序列中的SSR增加了基因的不稳定性,但正是这样的不稳定性为基因变异提供了动力。
本研究基于构建获得的沟眶象转录组数据库,对其SSR进行了高通量发掘,并对沟眶象SSR的特征进行了分析,表明利用转录组数据发掘SSR是高效可行的。研究结果为开发高多态性微卫星引物来进行沟眶象功能基因组学、种群遗传结构、种群遗传多样性等研究奠定了基础,这对于从分子角度探究沟眶象的扩散机制及综合防治等都具有非常重要的意义,同时为探索沟眶象和其近缘种臭椿沟眶象的起源进化关系打下基础。
References)
Duan CX, Li DD, Sun SL,etal. Rapid development of microsatellite markers forCallosobruchuschinensisusing Illumina paired-end sequencing[J].PLoSONE, 2014,9(5): e95458.
Eujayl I, Sorrells M, Banm M,etal. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat[J].TheoreticalandAppliedGenetics,2002,104(2): 399-407.
He P. Abundance, polymorphism and applications of microsatellite in eukaryote[J].Hereditas,1998,20(4):42-47.[何平.真核生物中的微卫星及其应用[J].遗传,1998, 20(4):42-47]
Huang HY, Du HY, Wuyun TN,etal.Development of SSR molecular markers based on transcriptome sequencing ofEucommiaulmoides[J].ScientiaSilvaeSinicae, 2013, 49(5): 176-181.[黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J]. 林业科学, 2013,49(5): 176-181]
Huang Q, Sun P, Zhou X,etal.Characterization of head transcriptome and analysis of gene expression involved in caste differentiation and aggression inOdontotermesformosanus(Shiraki)[J].PLoSONE,2012,7(11): e50383.
Hu YH, Li M, Zhang HF,etal. The information analysis of SSR loci in theMythimnaseparate(Walker) transcriptome[J].JournalofShanxiAgriculturalUniversity, 2015, 35(5): 484-489.[胡艳华,李敏,张虎芳,等.粘虫转录组中SSR位点的信息分析[J].山西农业大学学报, 2015, 35(5): 484-489]
Li LT, Zhu YB,Ma JF,etal.An analysis of theAthetislepigonetranscriptome from four developmental stages[J].PLoSONE, 2013, 8(9): e73911.
Li YC, Korol AB, Fahima T,etal.Microsatellites: Genomic distribution, putative functions and mutational mechanisms: A review[J].MolecularEcology, 2002,11(12): 2453-2465.
Liu T, Zhu S, Fu L,etal. Development and characterization of 1827 expressed sequence tag-derived simple sequence repeat markers for ramie (BoehmerianiveaL. Gaud)[J].PLoSONE,2013,8(4): e60346.
Luo M, Zhang H, Bin SY,etal. High-throughput discovery of SSR genetic markers in the mealybug,Phenacoccussolenopsis(Hemiptera:Pseudococcidae), from its transcriptome database[J].ActaEntomologicaSinica,2014,57(4): 395-400. [罗梅,张鹤,宾淑英,等.基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物[J].昆虫学报, 2014, 57(4): 395-400]
Samadi S, Artiguebielle E, Estoup A,etal. Density and variability of dinucleotide microsatellites in the parthenogenetic polyploid snailMelanoidestuberculata[J].MolecularEcology,1998,7(9):1233-1236.
Shi J,Yin TM,Guan HW,etal.Characteristic analysis of microsatellitesCamelliaspp.[J].JournalofNanjingForestryUniversity(Natural Sciences Edition),2012,36:47-52. [史洁,尹佟明,管宏伟,等.油茶基因组微卫星特征分析[J].南京林业大学学报(自然科学版), 2012,36:47-52]
Temnykh S, DeClerck G, Lukashova A,eta1. Computational and experimental analysis of microsatellites in rice (OryzasativaL.)[J].GenomeResearch, 2001,11:1441-1452.
Varshney RK, Graner A, Sorrells ME. Genic microsatellite markers in plants:Features and applications [J].TrendsinBiotechnology, 2005, 23(1):48-55.
Wang D, Cao LY, Gao JP. Data mining of simple sequence repeats inCodonopsispilosulatranscriptome[J].ChineseTraditionalandHerbalDrugs, 2014, 46(8):2390-2394.[王东,曹玲亚,高建平.党参转录组中SSR位点信息分析[J].中草药,2014,46(8): 2390-2394]
Wang LY, Wei K, Zhang CC,etal.Characterization of micorsatellites in tea (Camelliasinensis) floral transcriptome[J].ActaAgronomicaSinica, 2014, 40(1): 80-85. [王丽鸳,韦康,张成才,等.茶树花转录组微卫星分布特征[J].作物学报, 2014, 40(1): 80-85]
Xie W, Meng QS, Wu QJ,etal. Pyrosequencing theBemisiatabacitranscriptome reveals a highly diverse bacterial community and a robust system for insecticide resistance[J].PLoSONE, 2012, 7(4): e35181.
Xu Y, Zhou W, Zhou Y,etal.Transcriptome and comparative gene expression analysis ofSogatellafurcifera(Horvath) in response to southern rice black-streaked dwarf virus[J].PLoSONE, 2012, 7(4): e36238.
Yang GJ, Yong HL, Wang XP.The biological characters and behavior ofEucryptorrhynchuschinensis[J].ChineseBulletinofEntomology, 2008, 45(1): 65-69.[杨贵军,雍惠莉,王新谱.沟眶象的生物学特性及行为观察[J].昆虫知识, 2008, 45(1): 65-69]
Yang H, Chen Q, Wei ZL,etal.Analysis on SSR information inCamelliasinensistranscriptome[J].JournalofAnhuiAgriculturalUniversity,2011,38:882-886.[杨华,陈琪,韦朝领,等.茶树转录组中SSR位点的信息分析[J].安徽农业大学学报,2011,38:882-886]
Yuan Y, Zhang LF, Wu GX,etal. High-throughput discovery microsatellites inTomicusyunnanenss(Coleoptera: Scolytonae)[J].JournalofEnvironmentalEntomology,2014,36(2):166-170.[袁远,张丽芳,吴国星,等.云南切梢小蠹微卫星的高通量发掘[J].环境昆虫学报, 2014,36(2):166-170]
Zhu JY, Li YH, Yang S,etal.De novo assembly and characterization of the gobal transcriptome forRhyacionialeptotubulausing Illumina paired-end sequencing[J].PLoSONE, 2013, 8(11): e81096.
Zhu JY,Wu GX,Yang B.High-throughput discovery of SSR genetic markers in the yellow mealworm beetle,Tenebriomolitor(Coleoptera: Tenebrionidae), from its transcriptome database[J].ActaEntomologicaSinica,2013,56(7):724-728.
Characteristic analysis of microsatellite inEucryptorrhynchuschinensistranscriptome
WU Zheng-Mei, GAO Peng, WEN Jun-Bao*
(Beijing Key Laboratory for Forest Pest Control, Beijing Forestry University, Beijing 100083, China)
According to the constructed transcriptome database ofEucryptorrhynchuschinensis, the microsatellites have been explored at a high-throughput scale by using Micro Satellite identification tool. The microsatellites in the unigenes (the length ≥1 kb) (17590, 26.89%) were analyzed and a total of 1823 SSR were identified in 1665 unigenes, with one SSR per 32.52 kb. Tri-nucleotide repeats were the most abundant (637, 34.94%), followed by mono nucleotide repeats (576, 31.60%). Among all the 104 SSR motifs, (A/T) n was the most frequent repeat motif (30.39%), followed by (AT/AT) n (6.91%). The average length of microsatellites was 15.74 bp, the microsatellites over 20 bp were only 12.70%.There was significant negative correlation (P<0.01) between the frequency and the length of microsatellites ine.chinensistranscriptome and the correlation coefficient was -0.572. These results lay a foundation for developing high polymorphic microsatellites to research the functional genomics, population genetic structure and genetic diversity ofe.chinensis.
Eucryptorrhynchuschinensis; microsatellite; transcriptome; repeat type; repeat motif
林业公益性行业科研专项(201304412;201204501)
武政梅,女,1991年生,山东泰安人,硕士,研究方向为昆虫分子生物学,E-mail: zhengmeibest@163.com
*通讯作者Author for correspondence, E-mail:wenjb@bjfu.edu.cn
Received:2015-10-29;接受日期Accepted:2015-12-29
Q965;S433.5
A
1674-0858(2016)05-0979-05
