生长相关分子标记在翘嘴鳜五代中的富集
2016-11-12梁旭方田昌绪吕丽媛
宋 易 梁旭方 田昌绪 吕丽媛 赵 程
(华中农业大学水产学院,农业部淡水生物繁育重点实验室,湖北省淡水渔业协同创新中心,武汉 430070)
生长相关分子标记在翘嘴鳜五代中的富集
宋易梁旭方田昌绪吕丽媛赵程
(华中农业大学水产学院,农业部淡水生物繁育重点实验室,湖北省淡水渔业协同创新中心,武汉 430070)
为进一步了解人工选育对翘嘴鳜生长相关遗传标记的影响作用,研究以翘嘴鳜“华康1号”的5代选育群体为实验材料,对具有生长相关优势基因型的5个标记的6个位点进行扩增,通过直接测序和聚丙烯酰胺凝胶电泳两种方法分型后,统计其优势基因型个体数目在翘嘴鳜5代中的变化。结果显示,在5代群体中,2个单核苷酸多态性位点和4个微卫星位点优势基因型的数目的分布范围为0—4,从F1到F5代,这6个位点优势基因型的平均值分别为0.36、0.71、0.68、0.77和0.94,优势基因型的平均含量随选育世代的增加呈现递增趋势,从侧面反映了人工选育在一定程度上富集了优良基因。此外,对微卫星位点进行了遗传相似性和遗传距离分析,结果显示,随着选育的进行,后续世代与F1的遗传距离有明显的增大趋势,遗传相似性减小,这符合育种的客观规律。但相邻世代间的遗传距离则逐代减小,遗传相似性逐代增大,说明人工选育将遗传相似性较大的群体保留下来了,这种相似性表现在表型上包括生长快、体重大、体长增加等。F1到F5代处于中度遗传多样性的稳定状态,说明群体还存在选育空间。
优势基因型;富集;微卫星;单核苷酸多态性;翘嘴鳜
翘嘴鳜(Siniperca chuatsi Basilewsky),俗称季花、桂花,隶属鲈形目(Perciformes)、暖鲈科(Percichthyidea)、鳜属,是鳜属鱼类中体型最大、生长最快的一种鳜。其肉质坚实细嫩、味道鲜美、营养丰富、无肌间刺,是一种经济价值很高的名贵品种,在我国淡水养殖业中占重要经济地位[1]。随着鳜养殖业的发展,其养殖规模不断扩大,养殖密度不断提高,水域生态环境不断遭到人为破坏,加之饲养管理不善,使得鳜渔业资源大幅度减少,甚至成为偶见种[2]。因此,研究开发和保护翘嘴鳜资源越显重要和迫切,开展翘嘴鳜的分子生物学研究,以揭示翘嘴鳜的遗传特征,为渔业利用提供相关资料以及翘嘴鳜的人工养殖及资源保护提供科学依据。
标记辅助育种是分子生物学最有效的育种技术之一,近年来在越来越多的育种项目中,产业育种者已经通过这种技术获得了生长显著快的个体,从而减少了养殖群体一些经济性状的的丢失,包括生长速度、抗病率和存活率等[3,4]。但目前关于标记辅助育种的研究主要集中在序列位点的筛选,和筛选出的位点与性能的相关性分析方面[5—7],而对筛选出的与性状具有显著性相关的位点在选育过程中的一个变化趋势,却很少有人进行过研究。本研究在已经选育出了生长快的翘嘴鳜5代群体的基础上,对筛得的标记进行一个可靠性的验证,以分析其用于辅助育种的可靠性。同时为进一步了解人工选育对翘嘴鳜生长相关遗传标记的影响作用提供依据。实验中用到的5代群体从F1到F5代,其生长速率依次增加[8,9],在5代群体中对筛选到的分子标记的优势基因型数目进行分析,研究结果可为标记辅助育种提供依据。
此外,由于优势基因型的数量与相关性状的显著性存在正相关性,Kelly等[10]早在1995年就证实了基因聚合育种可以通过选育同时具有几种抗性基因的个体而使其在抗性和生长速度上同时得到提高。因此,有研究者希望通过基因聚合育种加快育种进程,Servin等[11]提出要提高物种的某一性状遗传价值就要通过增加控制这一性状的优势基因数目来实现,Lange和Whittaker[12]以及Hayes和Goddard[13]也指出,通过将所有的有效标记用于同一群体的选育可以提高分子标记辅助育种的效率和成功率。在鱼类中,徐磊等[14]通过对生长相关标记的优势基因型含量分析发现大口黑鲈的生长速率与优势基因型数目之间存在正相关性,而李红霞等[15]在建鲤中也发现了相同的结论。因此,通过将某一性状相关的基因或基因型聚合在一个群体上进行研究,能更好地反应出标记与性状的相关性,提高标记用于辅助育种的效率。本实验选取的2个多核苷酸多态性位点筛选自生长激素基因[4],4个微卫星位点筛选自鳜转录组生长相关基因邻近序列[16],将它们结合起来进行研究是基因聚合育种科学性的体现。
此次研究在翘嘴鳜养殖5代群体共155个个体中进行来自不同基因序列位点的生长相关优势基因型分析,在验证前期筛选到的位点用于辅助育种的可靠性的同时,又通过来自不同基因序列的微卫星位点、单核苷酸多态性位点来进行综合探究,得到的具有实际应用性的位点,可用于今后普通翘嘴鳜个体的选育工作中。
1 材料与方法
1.1材料
实验鱼来源于翘嘴鳜的5个连续选育世代,是湖北省淡水渔业协同创新中心从2008—2013年间连续选育出的世代群体,从F1到F5代的生长速率依次增加[8,9]。从每一个世代中随机挑选31尾翘嘴鳜个体,共计155尾。剪取鳍条用95%的酒精保存,放入-20℃冰箱储存。
1.2引物
实验中用于优势基因型分析的共有5对多态性引物均来自实验室已发表文献[4,16],运用引物设计软件Primer Premier 5.0设计引物,引物由上海英骏完成合成,引物的详细信息见表1。

表1 翘嘴鳜4对生长相关微卫星位点和1对生长相关SNPs位点的引物序列及特征Tab. 1 Primer sequences and characteristics of 4 growth-related microsatellites and growth-related SNPs of S. chuatsi(Basilewsky)
1.3方法
基因组DNA提取参照天根动物组织基因组DNA提取试剂盒(北京天根生物科技有限公司)说明书的介绍方法提取样品基因组DNA,1.2%的琼脂糖凝胶电泳和紫外分光光光度计检测DNA质量和浓度,调整DNA终浓度到100 ng/μL后,-20℃保存。
PCR反应程序PCR反应总体积为50 μL,含有10×buffer 5 μL,dNTP(10 μmol/L) 1 μL,上下游引物(20 μmol/L)各1 μL,基因组DNA 1 μL,rTaq酶(北京康为世纪生物科技有限公司) 0.5 μL。在Biometra070-851PCR仪进行扩增,扩增程序为94℃预变性4min,然后30个循环,94℃变性30s,53.5—55.5℃复性45s,72℃延伸30s,最后72℃延伸10min。
PCR产物的检测SSR产物检测:PCR产物采用8%非变性聚丙烯酰胺凝胶,0.5×TBE缓冲液,120 V/cm电压电泳。电泳结束后,取下凝胶用双蒸水漂洗30s,后转移至0.1%硝酸银染色10min,再用双蒸去离子水快速漂洗2次后,加入700 mL 显影液显色(含14 g氢氧化钠,0.28 g碳酸钠,2.8 mL 37%甲醛),待条带显现后,加入双蒸水终止显色。用Alpha Innotech402942凝胶成像系统扫描记录。并用Alpha View SA软件对每对微卫星引物扩增的等位基因分子量大小进行估算。
SNP产物检测:将特异性好(琼脂糖电泳条带单一)的PCR产物送公司测序,获得多态性位点序列信息。
1.4数据统计与分析
基因型的读取微卫星分位点分型:根据每个个体产生的条带位置确定其基因型,读取各个个体的基因型,并记录。
单核苷酸多态性位点分型:将获得的序列信息用SeqMan软件进行峰值读图,用MS-tools记录每个群体中各个个体的位点的基因型,并记录。
SNP位点优势基因型的分型及生长相关性分析参见田昌绪等[4]的研究,采用直接测序的方法对群体进行基因型分型后,对各基因型与体重、体长、体高进行显著性分析而得出其优势基因型;SSR位点优势基因型的分型及生长相关性分析参见易提林等[16]的研究,采用聚丙烯酰胺凝胶和银染的方法对群体分型后,对基因型进行体重、体长、体高的显著性分析而得出其优势基因型。SNP位点g.4940A>C、g.4948A>T的优势基因型为CC、TT,微卫星位点SC10、SC52、Sin135、AP34-23的优势基因型为230/256、211/220、275/290、238/255。
基因型数目的统计分别在每代31个个体,共155个个体中统计其基因型数目,计算单核苷酸多态性位点g.4940A>C、g.4948A>T和微卫星位点SC10、SC52、Sin135、AP34-23的优势基因型数目。
遗传距离和遗传相似性的计算基于微卫星标记的5个群体两两比较的遗传相似度(S)和遗传距离(D),按Nei[17]方法进行计算,运用软件Mega 6.0进行计算,并分析其变化趋势,计算公式:S= 2 Nxy/(Nx+ Ny)。
把电泳图谱中任何一个个体在任意一泳道垂直方向显示的带纹记做“1”,而其他个体对应的位点缺少此带则记做“0”,这样可以得到各个个体的二进制数据信息的方阵。将上述二进制数据方阵用PhylTools 软件计算获得个体间遗传距离矩阵;根据Nei 公式计算任意翘嘴鳜的遗传相似度S以及遗传距离D。
其中,Nxy为两个体共享的图带数,Nx和Ny分别为x和y 个体拥有的图带数。一个群体(或品种、品系)内多个个体两两配对比较得到的S 值的平均值就反映了该群体内DNA 指纹图相似程度或变异程度。
遗传多态性的计算利用Popgen32软件进行统计分析。计算出5个群体每个位点的等位基因数、有效等位基因数、基因观测杂合度、基因期望杂合度等遗传参数,分析其变化趋势;
多态信息含量(Polymorphism information content,PIC)按BOTSTEIN等[18]的公式计算。
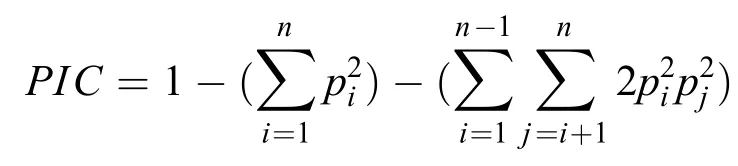
式中,Pi、Pj分别为第i个和第j个等位基因的频率;n为等位基因频率数目。
2 结果
2.1选育世代优势基因型的富集
每个选育世代优势基因型数目分布这5对引物在翘嘴鳜5代选育群体中表现出了良好的特异性和较高多态性,在155尾鳜DNA中扩增后,统计每代优势基因型数目(表2)。

表2 翘嘴鳜每代个体优势基因型数量的分布Tab. 2 The quantitative distribution of samples with different advantage genotypes of each generation
每个选育世代优势基因型频率分布根据上述统计的每个世代优势基因型数目,计算每个世代的优势基因型频率(表3),由统计结果可知,从F1到F5,其优势基因型频率呈上升趋势。

表3 翘嘴鳜每代优势基因型总数频率分布Tab. 3 The frequencies of total advantage genotypes in each generation of Siniperca chuatsi(Basilewsky)
2.2五代群体遗传距离分析
为研究选育对5代群体的遗传结构的影响,计算了各群体的微卫星标记间的遗传距离和遗传相似度(表4)。结果显示,随着选育的进行,F5与F1的遗传距离(黑体)有明显的增大,遗传相似性减小,这符合选育的客观规律。但相邻世代间的遗传距离(黑体)则逐代减小,遗传相似性逐代增大,说明人工选育将遗传相似性较大的群体保留下来了,这种相似性表现在表型上包括生长快、体重大、体长增加等。
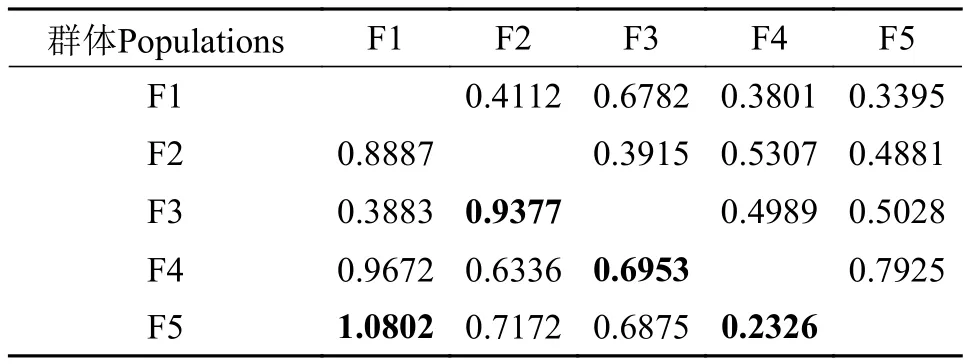
表4 基于微卫星标记的翘嘴鳜选育世代群体遗传相似度(右上角)和遗传距离(左下角)Tab. 4 The genetic similarity ang genetic ditance based on microsatellite of the breeding generations of Siniperca chuatsi(Basilewsky)
2.3五代群体遗传多样性分析
微卫星标记在5个群体中平均等位基因数、平均有效等位基因数、平均观测杂合度和平均期望杂合度、平均多态信息含量(表5)。F1到F5代的PIC分别为0.5317、0.5611、0.5191、0.4248和0.4716,并没有出现较大的波动,根据Botstein等[18]提出的当PIC>0.5时,该位点为高度多态,当0.25<PIC<0.5时,该位点为中度多态,当PIC<0.25时,则为低度多态。本研究随机群体PIC处于中等多态水平,说明群体能维持自身的稳态,并存在选育空间。
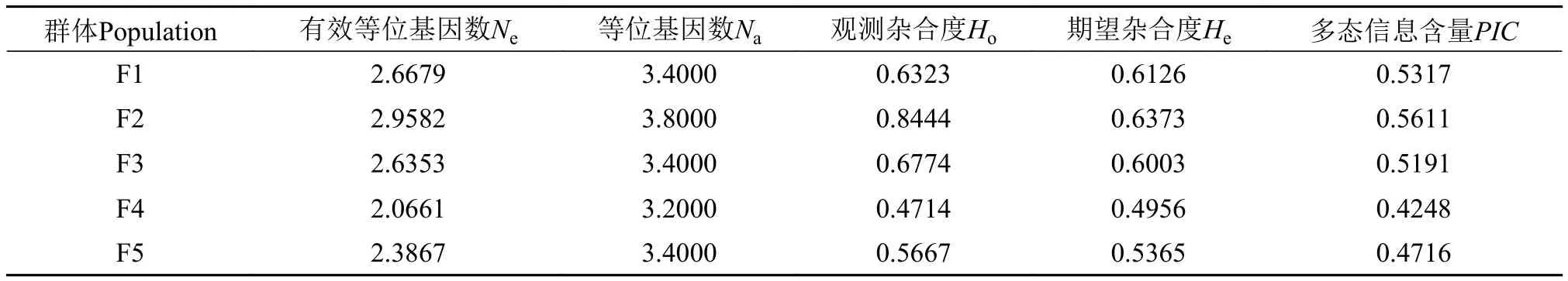
表5 基于微卫星标记的翘嘴鳜选育世代群体遗传多样性参数Tab. 5 The diversity parameters based on microsatellite of the breeding generations of Siniperca chuatsi(Basilewsky)
3 讨论
3.1生长相关分子标记优势基因型的富集
优势基因型在选育群体“华康1号”中的富集结果表明,这6个位点优势基因型平均值随着群体生长速率的增加而增大,这些位点可作为翘嘴鳜普通个体选育中的可靠性标记用于将来的辅助育种中。这一结果对于鱼类其他经济性状,比如肉质、适温性等的选育工作也是十分有借鉴作用的,研究者可以将前期筛得的与性状相关的位点在一个表型明显的世代群体中进行验证,若具有较好的特异性,且位点的性状相关优势基因型与世代群体的表型有显著的相关性,则可大大提高这些标记用于后期辅助育种的成功率,减少一些因标记的不实用性造成的损失。优势基因型以及基因聚合育种用于肉质、生长速率的选育工作前期已经有研究者在建鲤[19]和大口黑鲈[20]中进行过,也获得了有效的性状相关的标记,且徐磊等[14]也在其选育的大口黑鲈世代群体中对前期筛得的标记进行检测,获得了较好的检测结果。
另外,孙效文等[21]在研究中发现,生长相关标记的数目与个体大小之间存在正相关性,也就是说含有效生长相关标记越多的个体,其生长速度越快。在这个事实的基础上,本实验结果中筛选到的标记的生长相关优势基因型数目与世代群体的生长速度之间存在正相关性,就说明了这些位点与生长性状相关联的可靠性。类似的研究在其他水产动物中还不够深入,只是停留在位点的筛选和性状相关性分析层面。何焱等[22]在中华鲟的LPL 和HL基因上筛得了生长密切相关的SNP位点,彭敏燕等[23]在翘嘴鳜AMY基因上检测到了SNP和SSR位点,并分析了其单倍型与摄食性状的关联性,陶文静等[24]对生长激素受体基因进行了SNP位点筛选及增重相关性分析,但这些位点应用于后期辅助育种工作的仍十分少见,若对这些筛选到的位点进行类似的标记数目与个体表型之间的相关性分析,则可对其用于选育工作的可靠性进行验证,促进这些标记在生产实践中的应用,这对提高标记辅助育种的应用性是十分有意义的。
3.2生长相关基因的聚合育种
此外,本实验的2个多核苷酸多态性位点筛选自生长激素基因[4],4个微卫星位点筛选自鳜转录组生长相关基因邻近序列[16],这些位点优势基因型的平均含量是对多个生长相关基因的聚合效果,能更全面地反应标记与表型之间的相关性。研究表明,动物的生长性状都是受多个基因控制的[25],因此,在不同基因序列上进行位点筛选和优势基因型统计的多基因聚合分析,可以为了解多个基因作用于同一性状的综合作用奠定重要的基础。多基因聚合分析在真菌系统分类[26]、鸡肌肉脂肪性状[27]和猪繁殖性状[28]的研究中都已用到,但在鱼类中的应用还不多,郭立等[29]进行了线粒体多基因聚合分析在鲻科鱼类的谱系筛选及其效应探究,徐磊等[14]做了优势基因型在大口黑鲈中的富集分析,此外,类似的聚类研究主要集中在地理分布[30]和外形分析[31]等方面,深入分子水平的聚合分析还有待进一步研究。而微卫星遗传距离的分析说明人工选育将遗传相似性较大的群体保留下来了,这种相似性表现在表型上包括生长快、体重大、体长增加等,这是对人工选育作用的进一步验证。F1到F5代的遗传多样性并没有出现较大的波动,而是处于一个稳定的中等水平,说明群体既存在选育空间,又可以处于自身稳定的状态。
综上所述,分子标记辅助育种和基因聚合育种相结合的育种思路已经越来越受到关注。本研究中用到的生长激素基因已经得到较深入研究,许多研究者都在鱼类里验证了该基因的生长功能[32—35]。而翘嘴鳜“华康1号”5代选育群体具有从F1到F5生长速率依次增加的明显表现型,在这样的选育群体中对筛选到的分子标记的生长相关优势基因型平均值进行分析,可以验证标记用于辅助育种的可靠性,同时可以检测人工选育对翘嘴鳜生长相关遗传标记的影响作用。但是,选育出具有显著表型的群体是一个较庞大的工程,群体的选育和标记的筛选是一个互相检测的过程,若选育出表型显著的群体,类似本实验的研究便可开展,为筛选到更多可用于辅助育种的标记提供可靠的实验材料,同时大大减少了育种的工作量。另外,从研究较透彻的性状相关功能基因上去筛选标记,也可大大提高标记的可靠性[36],候选基因法近些年在人类疾病、鸟类迁徙适应性和谷物含锌量、抗病性等方面都得到应用[37—39],在水产动物上的发展也在推进[40—43]。多基因、多标记的聚合分析已经是育种的必然趋势,而获得表型显著的群体后,这种分析又可变得更快、更容易实现了。
[1]Liang X F. Study on mandarin fish and its culture home and abroad [J]. Fisheries Science & Technology,1996,23(1):13—17 [梁旭方. 国内外鳜类研究及养殖现状. 水产科技情报,1996,23(1):13—17]
[2]Zhao J L,Wang W W,Li S F,et al. Structure of the mitochondrial DNA control region of the sinipercine fishes and their phylogenetic relationship [J]. Acta Genetica Sinica,2006,33(9):793—799
[3]Liang X F,Oku H,Ogata H Y,et al. Weaning Chinese perch Siniperca chuatsi(Basilewsky) onto artificial diets based upon its specific sensory modality in feeding [J]. Aquaculture Research,2001,32(s1):76—82
[4]Tian C,Yang M,Lv L,et al. Single nucleotide polymorphisms in growth hormone gene and their association with growth traits in Siniperca chuatsi(Basilewsky) [J]. International Journal of Molecular Sciences,2014,15(4):7029—7036
[5]Zhang G,Chu W,Hu S,et al. Identification and analysis of muscle-related protein isoforms expressed in the white muscle of the mandarin fish(Siniperca chuatsi) [J]. Marine Biotechnology,2011,13(2):151—162
[6]Qu C,Liang X,Huang W,et al. Isolation and characterization of 46 novel polymorphic EST-simple sequence repeats(SSR) markers in two Sinipercine fishes(Siniperca)and cross-species amplification [J]. International Journal of Molecular Sciences,2012,13(8):9534—9544
[7]Sun D Q,Sun Y N,Wang R X,et al.The characteristic analysis of microsatellites from ESTs in Miichthys miiuy[J]. Acta Hydrobiologica Sinica,2011,35(5):753—760[孙典巧,孙悦娜,王日昕,等. 鱼EST序列中微卫星标记的初步筛选及特征分析. 水生生物学报,2011,35(5):753—760]
[8]Liang X F,Guo W J,Fang L. The Siniperca chuatsi‘Huakang No.1' [J]. China Fisheries,2015,(8):47—49[梁旭方,郭稳杰,方刘. 翘嘴鳜“华康 1 号”. 中国水产,2015,(8):47—49]
[9]Sun L F,Li J,Liang X F,et al. Evaluation on selective breeding effect of Siniperca chuatsi [J]. Journal of Guangdong Agriculture & Science,2014,41(13):114—118 [孙龙芳,李姣,梁旭方,等. 翘嘴鳜F3-F5群体选育效果分析. 广东农业科学,2014,41(13):114—118]
[10]Kelly J D,Afanador L,Haley S D. Pyramiding genes for resistance to bean common mosaic virus [J]. Euphytica,1995,82(3):207—212
[11]Servin B,Martin O C,Mézard M. Toward a theory of marker-assisted gene pyramiding [J]. Genetics,2004,168(1):513—523
[12]Lange C,Whittaker J C. On prediction of genetic values in marker-assisted selection [J]. Genetics,2001,159(3):1375—1381
[13]Hayes B J,Goddard M E. Prediction of total genetic value using genome-wide dense marker maps [J]. Genetics,2001,157(4):1819—1829
[14]Xu L,Bai J J,Li S J. Pyramiding of growth-related genotypes in breeding generations of largremouth bass ‘YoluNo. 1' [J]. Journal of Henan Agriculture University,2014,35(1):7—11 [徐磊,白俊杰,李胜杰,等. 生长相关优势基因型在大口黑鲈‘优鲈1号'选育世代中的聚合.华南农业大学学报,2014,35(1):7—11]
[15]Johnsson J I,Björnsson B T. Growth hormone increases growth rate,appetite and dominance in juvenile rainbow trout,Oncorhynchus mykiss [J]. Animal Behaviour,1994,48(1):177—186
[16]Yi T L,Fang L,Liang X F,et al. Characterization of microsatellite markers and their correlations with growth traits in Mandarin fish(Siniperca chuatsi) [J]. Genetics and Molecular Research,2014,14(3):8926—8934
[17]Lynch M. The similarity index and DNA fingerprinting[J]. Molecular Biological Evolution,1990,7(5):478—484
[18]Botstein D,White R L,Skolnick M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. American Journal of Human Genetics,1980,32(3):314—331
[19]Li H X,Li J L,Tang Y K,et al. Correlation analysis between body weight gain and ODC1 genotypes in Cyprinus carpio var. Jian [J]. Acta Hydrobiologica Sinica,2014,38(3):414—421 [李红霞,李建林,唐永凯,等. 建鲤ODC1基因型与增重的相关性分析. 水生生物学报,2014,38(3):414—421]
[20]Xu L,Bai J J,Li S J. The pyramiding effect of growth-related genotypes in largemouth bass(Micropterus salmoides) [J]. Journal of Fishery Sciences of China,2014,21(1):53—58 [徐磊,白俊杰,李胜杰. 大口黑鲈生长性状相关标记的聚合效果分析. 中国水产科学,2014,21(1):53—58]
[21]Sun X W,Lu C Y,Cao D C,et al. Molecular markers associated with body weight of mirror carp and selection and raising of progenies [J]. Journal of Fisheries of China,2009,33(2):177—181 [孙效文,鲁翠云,曹顶臣,等. 镜鲤体重相关分子标记与优良子代的筛选和培育.水产学报,2009,33(2):177—181]
[22]He Y,Shen D,Liang X F,et al. Genetic polymorphisms of LPL and HL and their association with the performance of Chinese sturgeons fed a formulated diet [J]. Genetic and Molecular Research,2013,12(4):4559—4566
[23]Peng M Y,Liang X F,Fang R,et al. Detection of SNPs and SSR of amylase(AMY) gene in Siniperca chuatsi [J]. Journal of Jinan University(Natural Science),2013,34(1):96—100 [彭敏燕,梁旭方,方荣,等. 翘嘴鳜淀粉酶基因SNPs 和微卫星位点多态性的检测. 暨南大学学报(自然科学版),2013,34(1):96—100]
[24]Tao W J,Ma L J,Ruan R X,et al. SNP loci associated with weight gain on growth hormone receptor genes in Cyprinus carpio var. Jian [J]. Acta Hydrobiologica Sinica,2011,35(4):622—629 [陶文静,马龙俊,阮瑞霞,等. 建鲤GHR基因多态性及与增重相关的SNP位点的筛选. 水生生物学报,2011,35(4):622—629]
[25]Li M Y,Chen D W,Zhang K Y. Genes related to meat quality of pig and their expression regulation [J]. Animal Husbandry and Veterinary Medicine,2008,40(4):95—99 [李梦云,陈代文,张克英,等. 影响猪肉质性状的基因及其表达调控. 畜牧与兽医,2008,40(4):95—99]
[26]Xie S N. Molecular Systematic Studies of Bipolaris and Its Similar Genera by Multiple Gene Aggregate Analysis[D]. Henan Agriculture University. 2012 [谢淑娜. 利用多基因聚合分析进行平脐蠕孢属及其近似属的分子系统学研究. 河南农业大学. 2012]
[27]Chang G B,Zhou Q,Lei L L,et al. Genetic Analysis of polygene pyramiding in intramuscular fat traits in chicken [J]. China Poultry,2009,31(19):25—28 [常国斌,周琼,雷黎立,等. 鸡肌内脂肪性状的多基因聚合效应分析. 中国家禽,2009,31(19):25—28]
[28]Liu X,Chamba Y Z,Wang Q,et al. Effects of multigenes for reproductive traits in Tibet pig [J]. Journal of Genetics and Genomics,2010,32(5):480—485 [刘轩,强巴央宗,王强,等. 藏猪繁殖性状多基因效应分析. 遗传,2010,32(5):480—485]
[29]Guo L,Tang W X,Xia R,et al. Research on effect of multiple mitochondrial genes to reveal incomplete pedigree of mugilidae fish and Chelon [A]. Collection of Abstracts in 2008 Chinese Ichthyological Society [C]. 2008[郭立,唐卫星,夏蓉,等. 线粒体多基因揭示鲻科鱼类的不完全谱系筛选与龟鲻属的有效性探讨. 中国鱼类学会2008学术研讨会论文摘要汇编. 2008]
[30]Wang L L,Zhao T Q. Cluster analysis on geographic distribution of fresh water fishes in Zhejiang Province,China[J]. Journal of Zhejiang Normal University(Natural Science),1999,22(4):86—89 [王丽丽,赵铁桥. 浙江省淡水鱼类地理分布的聚类分析. 浙江师大学报(自然科学版),1999,22(4):86—89]
[31]Zhou Y B,Jiang Q W,Zhao G M,et al. Cluster analysis of morphologic quantitative traits of Oncomelania hupensis from main-land of China [J]. Acta Zootaxonomica Sinica,2006,31(2):441—447 [周艺彪,姜庆五,赵根明,等. 中国大陆钉螺螺壳形态性状聚类分析. 动物分类学报,2006,31(2):441—447]
[32]Peng C,Peter R E. Neuroendocrine regulation of growth hormone secretion and growth in fish [J]. Zoological Studies-Taipei,1997,36:79—89
[33]Liu F,Lin Q S,Li Z,et al. The GH gene diversity among three Siniperca fish species [J]. Oceanologia et Limnologia Sinica,2009,40(4):470—478
[34]Ma X L,Zhang Y,Zhou L B,et al. Effects of LHRH-A on the growth and gene expressions of the growth axis in Nile tilapia Oreochromis niloticus [J]. Acta Hydrobiologica Sinica,2013,37(1):42—47 [马细兰,张勇,周立斌,等. LHRH-A对尼罗罗非鱼生长及生长轴相关基因表达的影响. 水生生物学报,2013,37(1):42—47]
[35]Shan X S,Yue H M,Chen X H,et al. cDNA cloning,expression and immunolocalization of growth hormonegene in Acipenser dabryanus [J]. Acta Hydrobiologica Sinica,2015,39(2):307—314 [单喜双,岳华梅,陈细华,等. 达氏鲟生长激素基因cDNA克隆,表达及免疫荧光定位研究. 水生生物学报,2015,39(2):307—314]
[36]Burgos M R,Chin J H,Koide Y,et al. Development and application of gene-specific markers for broad-spectrum blast resistance genes Pi2 and Pi9 [A]. Pest Management Council of the Philippines Anniversary and Annual Scientific Conference,Cagayan De Oro City,Philippines[C]. PMCP. 2012
[37]Levy-Lahad E,Wasco W,Poorkaj P,et al. Candidate gene for the chromosome 1 familial Alzheimer's disease locus [J]. Science,1995,269(5226):973—977
[38]Müller J C,Partecke J,Hatchwell B J,et al. Candidate gene polymorphisms for behavioural adaptations during urbanization in blackbirds [J]. Molecular Ecology,2013,22(13):3629—3637
[39]Gande N K,Kundur P J,Soman R,et al. Identification of putative candidate gene markers for grain zinc content using recombinant inbred lines(RIL) population of IRRI38 X Jeerigesanna [J]. African Journal of Biotechnology,2015,13(5):657—663
[40]Wei Y Z,Xie Y F,Li G H,et al. The status and prospect of fish gene engineering researches [J]. Acta Hydrobiologica Sinica,1992,16(1):71—78 [魏彦章,谢岳峰,李国华,等. 鱼类基因工程研究的现状和展望. 水生生物学报,1992,16(1):71—78]
[41]Thorgaard G H. Application of genetic technologies to rainbow trout [J]. Aquaculture,1992,100(1):85—97
[42]Wang Q,You F,Xin M J,et al. Correlation analysis between polymorphisms of GSTM gene and cold-tolerance trait in zebrafish,Danio rerio [J]. Marine Sciences,2015,39(1):1—7 [王倩,尤锋,辛梦娇,等. 斑马鱼GSTM单核苷酸多态性与低温耐受性的相关分析. 海洋科学,2015,39(1):1—7]
[43]Wang C X,Lu M X,Gao F Y,et al. Screening of single nucleotide polymorphisms(SNPs) related with growth in growth hormone secretagogue receptor gene(GHSR) of Nile Tilapia [J]. Journal of Agricultural Biotechnology,2015,23(6):762—771 [王春晓,卢迈新,高风英,等. 尼罗罗非鱼生长激素促分泌素受体基因(GHSR)生长相关单核苷酸多态性(SNPs)位点的筛选. 农业生物技术学报,2015,23(6):762—771]
PYRAMIDING OF GROWTH-RELATED MARKERS IN FIVE BREEDING GENERATIONS OF SINIPERCA CHUATSI BASILEWSKY
SONG Yi,LIANG Xu-Fang,TIAN Chang-Xu,LÜ Li-Yuan and ZHAO Cheng
(Key Lab of Freshwater Animal Breeding,College of Fisheries,Huazhong Agricultural University,Ministry of Agriculture,Freshwater Aquaculture Collaborative Innovation Center of Hubei Province,Wuhan 430070,China)
Gene pyramiding breeding is based on the effect of polygenic polymerization to change correlated character. In previous studies,growth-related markers were found in Siniperca chuatsi. Among them,five markers with six growth-favorable genotypes were selected,including two sites of single nucleotide polymorphism(SNP) located in GH gene and four sites of microsatellite(SSR):SC10,SC52,Sin135 and AP34-23. To get deeper insight into the effect of artificial breeding on growth-related markers identified in previous studies,we analyzed the number of favorable genotypes of these markers in five breeding generations of Siniperca chuatsi,‘Huakang No.1'. By direct-sequencing and polyacrylamide gel electrophoresis,we found that the sum of two SNP favorable genotypes and four SSR favorable genotypes appeared pyramiding. The result showed that the number of total favorable genotypes ranges from 0 to 4 in the tested fishes. The average contents were 0.36,0.71,0.68,0.77,0.94 in F1,F2,F3,F4 and F5 generations,respectively,showing the number of favorable genotypes increased synchronously with the growth rate of the populations,which reflects that,artificial breeding has aggregated favorable genotypes in some degree. The SSR analysis showed that,when compared with F1,the genetic distances increased from F2 to F5 while the genetic similarity decreased,which observed objective law. When compared with its contiguous generation,the genetic distances decreased from F1 to F5 while the genetic similarity increased. The analysis of the genetic distances indicated that artificial breeding has conserved individuals of higher genetic similarity,which in performance have phenotypes of fast growth,heavier body weight or longer body length. The genetic diversity shows no large fluctuations,but a state of stability,which indicated further space for breeding.
Favorable genotype; Pyramiding; SSR; SNP; Siniperca chuatsi Basilewsky
G344+.1
A
1000-3207(2016)05-0951-07
10.7541/2016.123
2016-03-30;
2016-05-05
国家自然科学基金项目(31272641); 珠江流域高产精养池塘健康养殖技术与示范(2012BAD25B04); 中央高校基本科研业务费(2662015PY041,2015BQ040)专项资助 [Supported by the Science Foundation of China(31272420); the Key Projects in the National Science & Technology Pillar Program(2012BAD25B04); the Fundamental Research Funds for the Central Universities(2662015PY041,2015BQ040)]
宋易(1991—),女,湖北咸宁人; 在读硕士生; 研究方向为鱼类遗传与育种。E-mail:15902792427@163.com
梁旭方(1965—),男,博士,教授; E-mail:xufang_liang@hotmail.com
