柠条锦鸡儿CkGR基因克隆及功能分析
2016-11-10张腾国毛玉珊聂亭亭刁志宏
张腾国 周 轲 毛玉珊 聂亭亭 李 萍 刁志宏 王 娟
(西北师范大学 生命科学学院,兰州 730070)
柠条锦鸡儿CkGR基因克隆及功能分析
张腾国 周 轲 毛玉珊 聂亭亭 李 萍 刁志宏 王 娟
(西北师范大学 生命科学学院,兰州 730070)
通过RACE技术从柠条锦鸡儿中克隆得到一个新的GR基因,全长2 122 bp,包括5′非翻译区(5′-UTR)57 bp,3′非翻译区(3′-UTR)415 bp,开放阅读框(ORF)1 650 bp,编码550个氨基酸,推测的蛋白质分子量为59.2 kDa,理论等电点为8.2,命名为CkGR。CkGR与鹰嘴豆CaGR的同源性较高,为90.1%。利用染色体步移法克隆得到CkGR起始密码子ATG上游648 bp的启动子序列,PlantCARE软件分析表明,该序列具有启动子的基本元件CAAT-box和TATA-box以及多种与逆境胁迫相关的顺式调控元件。实时荧光定量PCR分析表明,CkGR在柠条锦鸡儿的根、茎和叶中均有表达,没有组织特异性;CkGR的表达受低温、高盐和干旱胁迫的诱导,表明CkGR在柠条锦鸡儿适应低温、高盐和干旱胁迫的过程中发挥作用。
柠条锦鸡儿;CkGR基因;分子克隆;表达分析
植物体内细胞的生理生化反应都可产生活性氧(reactive oxygenspecies,ROS)。正常生长条件下,植物细胞中ROS的产生维持在较低的水平,需氧有机体能利用ROS调节代谢、进行信号传递和防御反应。然而,低温、高温、高盐、强光照、重金属、紫外辐射、空气中的O3和SO2、机械损伤等逆境条件都会导致植物体内产生大量的ROS[1]。ROS化学性质十分活泼,若不及时清除,可引起蛋白质、膜脂和其它细胞成分的损伤,进而导致细胞及组织的死亡[2]。处于逆境环境下的植物体本身常形成抗氧化系统,对胁迫造成的活性氧积累作出积极反应。植物细胞的抗氧化系统由抗氧化酶类和抗氧化剂类两部分组成,它们在植物体内都可消除ROS。抗氧化酶类主要有超氧化物歧化酶(SOD)、过氧化氢酶(CAT)、过氧化物酶(POD)、抗坏血酸过氧化物酶(APX)、谷胱甘肽还原酶(GR)、脱氢抗坏血酸还原酶(DHAR)等;抗氧化剂类主要有抗坏血酸(ASA)、还原型谷胱甘肽(GSH)和类胡萝卜素等[3~4]。其中,GR作为植物体内一种重要的抗氧化酶类,其主要功能是将氧化型谷胱甘肽(oxidaized glutathione disulfide,GSSG)还原成还原型谷胱甘肽(reduced glutathione,GSH),从而为ROS的清除提供还原力,保护植物免受伤害。GR是一种黄素蛋白还原酶,属于依赖型NADPH氧化还原酶家族,广泛存在于原核生物和真核生物中[5]。GR利用NADPH作为唯一的还原力和电子供体,催化氧化型谷胱甘肽(GSSG)还原成还原型谷胱甘肽(GSH),保持细胞内GSH/GSSG的高比值。GSH在减缓和清除由ROS、生物异源物质及重金属造成的环境氧化胁迫中起着重要作用,并且可有效还原-S-S键,稳定-SH族蛋白和膜蛋白结构,保护酶结构蛋白上的巯基,抵抗过氧化作用[6~7]。
当植物受到非生物逆境胁迫时,体内的ROS受体接受逆境信号,激活氧化还原敏感转录因子,如HSF(heat shock factor)和NPR1(non-expressor of pathogenesis-related gene 1),或者通过脱落酸(abscisic acid,ABA)依赖的信号转导途径,再经级联放大反应激活相应转录因子,从而激活或抑制GR和其它抗氧化防御基因的表达。Kaminaka等[8]通过克隆水稻胞质GR基因,获得了该基因上游的启动子序列,发现在394和1 320 bp存在2个ABA应答元件的核心序列(5′-ACGTGGC-3′),并且该基因的表达量在ABA及其它相关的非生物胁迫如低温、干旱、盐胁迫下明显增加。从而证实在逆境条件下,水稻胞质GR基因表达通过ABA信号转导途径被调控,受到ABA以及低温、干旱、盐胁迫应答元件的调控。Yousuf等[9]将克隆得到的拟南芥GR基因转入金丝桃中,使其过量表达,对比野生型金丝桃,转基因植株的抗冷性明显增强。这也说明,GR基因的表达调控效应受到低温等逆境胁迫的影响。此外,从烟草、豌豆、水稻、多枝赖草、不结球白菜等[10~13]植物中也已成功克隆得到GR基因,但在柠条锦鸡儿中未见报道。
柠条锦鸡儿是豆科(Leguminosae)锦鸡儿属(Caragana)植物,在中国分布于内蒙古西部、陕西北部以及宁夏等地区[14]。它枝叶繁茂,产草量高,营养丰富,适应性强,是家畜的优良饲料以及防风固沙,保持水土的重要材料,对环境具有广泛的适应性和很强的抗逆性[15~16]。本研究以柠条锦鸡儿为材料,克隆GR基因及其启动子,并对该基因进行组织特异性和低温、高盐以及干旱胁迫定量表达分析,为研究GR基因在植物抗逆胁迫中的作用机制提供一定参考依据。
1 材料与方法
1.1 实验材料
以柠条锦鸡儿(CaraganakorshinskiiKom.)为供试材料,挑选颗粒饱满,大小均匀柠条锦鸡儿种子,用75%酒精浸泡1 min,5%次氯酸钠溶液浸泡25 min,无菌水冲洗3~5次,无菌滤纸吸去种子表面水分,于1/2 MS培养基上培养,培养条件为25℃/22℃(昼/夜),光周期为16 h/8 h(光/暗),光照强度为130 μmol·m-2·s-1。培养约45 d后进行不同种类的非生物胁迫,剪取柠条锦鸡儿叶片液氮冷冻后用于RNA的提取。
1.2 实验方法
1.2.1 CkGR基因的克隆
选取生长良好的柠条锦鸡儿植株,以其幼嫩叶片为材料,按照Trizol试剂说明书进行总RNA的提取。提取的RNA用PrimeScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)反转录试剂盒(购自TaKaRa公司)进行反转录得到相应的cDNA,置于-20℃冰箱保存备用。
克隆CkGR基因的部分cDNA片段,从GenBank中下载已克隆得到的植物GR基因序列,用DNAstar软件Editsep模块对其编码区进行序列编辑,用DNAstar软件MegAlign模块进行序列比对,根据比对结果,设计简并性引物PrimerⅠ和PrimerⅡ,Primer Ⅲ和Primer Ⅳ(表1)。以反转录得到的柠条锦鸡儿cDNA为模板,分别以PrimerⅠ和PrimerⅡ,Primer Ⅲ和Primer Ⅳ为引物,按照宝生物工程公司的Premix Ex Taq DNA Polymerase操作说明书于25 μL反应体系进行PCR扩增。反应体系:预混酶12.5 μL,Primer Ⅰ/Primer Ⅱ 1 μL,Primer Ⅲ/Primer Ⅳ 1 μL,模板1 μL,ddH2O 9.5 μL。扩增产物按照Bioteke Corporation回收试剂盒(进口离心柱型,购自甘肃智信达公司)回收,连入pTG19-T载体。反应体系:T4DNA Ligase Buffer 0.5 μL,pTG19-T载体0.5 μL,T4DNA Ligase 0.5 μL,回收产物3.5 μL。4℃过夜连接。连接产物转化大肠杆菌Trans 5α感受态细胞,37℃过夜培养,蓝白斑筛选后挑取白斑进行菌液PCR检测,挑选阳性克隆,送北京六合华大基因有限公司测序。
按照MARTerTMRACE cDNA Amplification Kit方法分别进行3′-RACE和5′-RACE扩增。将提取的柠条锦鸡儿总RNA按照MARTerTMRACE cDNA Amplification Kit方法修饰后用试剂盒内特异性引物反转录,进行3′-RACE扩增。根据扩增得到的CkGR中间片段序列设计两对上游特异性引物3′-RACE U1和3′-RACE U2(表1),试剂盒中自带的short primer作为下游引物3′-RACE D1和3′-RACE D2(表1)。以3′-RACE U1,3′-RACE D1为引物进行第一次PCR扩增,以一扩产物为模板,以3′-RACE U2,3′-RACE D2为引物进行第二次PCR扩增,得到预期大小的片段,切胶回收,连接pTG19-T载体,转化大肠杆菌Trans 5α,蓝白斑筛选阳性克隆、测序。5′-RACE根据扩增得到的CkGR中间片段序列设计两对特异性引物5′-RACEU1和5′-RACE D1,5′-RACEU2和5′-RACED2(表1),以5′-RACEU1和5′-RACE D1为引物进行一次PCR扩增,再以一扩产物为模板,以5′-RACEU2和5′-RACED2为引物进行二次PCR扩增,得到预期大小的片段,切胶回收,连接pTG19-T载体,转化大肠杆菌Trans 5α,蓝白斑筛选阳性克隆、测序。
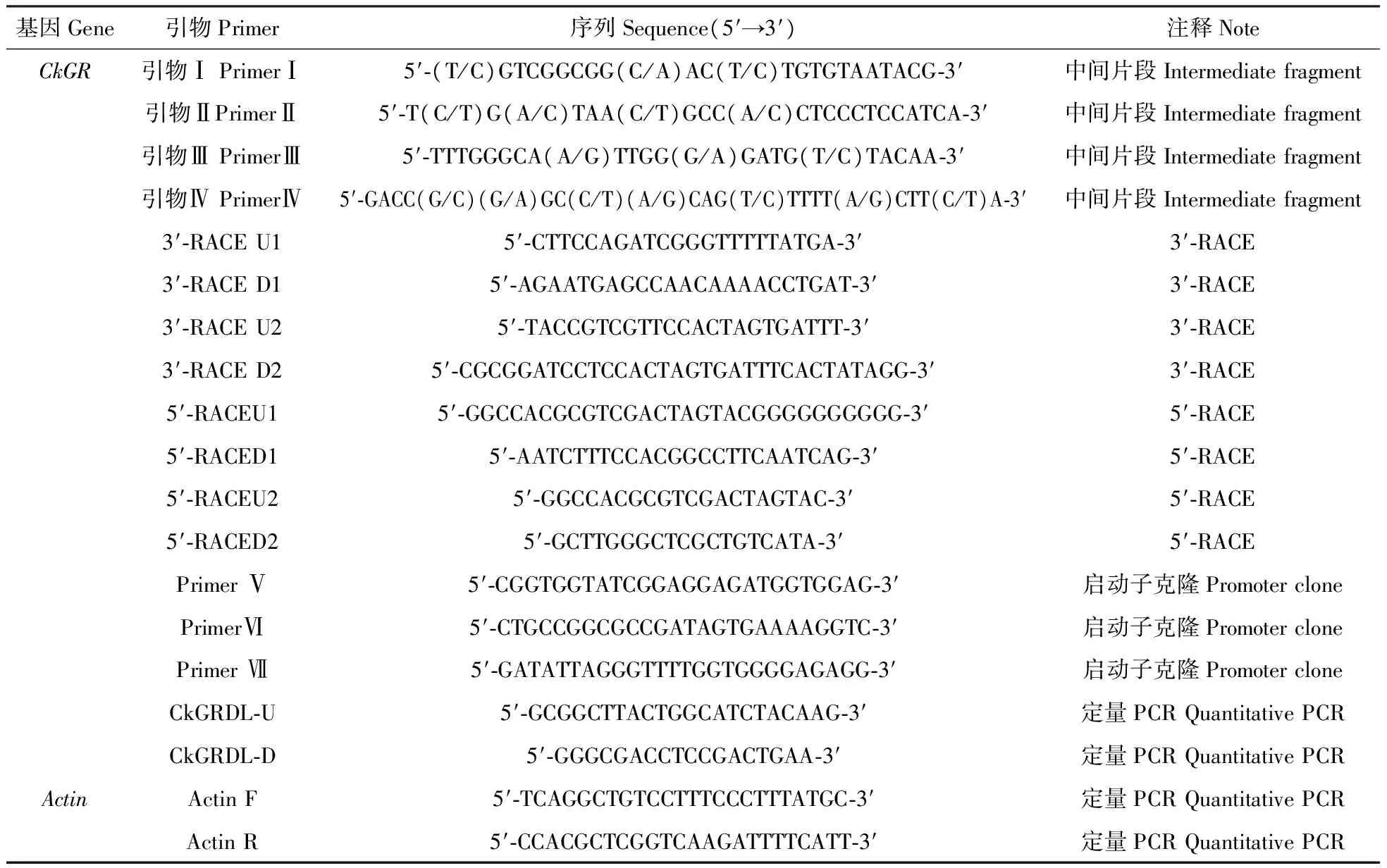
表1 PCR引物序列
1.2.2 CkGR预测蛋白的生物信息学分析
应用DNAstar软件的Protean模块进行氨基酸组成成分、蛋白基本特征分析;采用Clustal方法构建以氨基酸序列为基础的序列比对;
用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa-automat.pl?page=/NPSA/npsa-sopma.html)在线预测蛋白质的二级结构;根据生物信息学软件TopPred(http://services.cbib.U-bordeaux2.fr/pise/Toppred.htm1)在线分析蛋白质的亲水/疏水性。
1.2.3 CkGR基因启动子的克隆
选取生长良好的柠条锦鸡儿植株,以其幼嫩叶片为材料,按照TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0试剂盒说明书提取总DNA,置于-20℃冰箱备用。
根据已经得到的CkGR基因序列,按照染色体步移技术中特异性引物(Specific Primer,SP)的设计原则,设计三条同向且退火温度较高的特异性下游引物Primer Ⅴ、Primer Ⅵ和Primer Ⅶ(表1),使得Primer Ⅵ位于Primer Ⅴ内侧,Primer Ⅶ位于Primer Ⅵ内侧,以试剂盒中自带的退火温度较低的兼并引物(Annex Primer,AP)为上游引物,即AP1,AP2,AP3,AP4,按照宝生物工程公司的Genome Walking Kit试剂盒操作说明书于25 μL反应体系进行热不对称PCR。反应体系:DNA模板0.5 μL,PCR BufferII 2.5 μL,dNTP MT 4 μL,AP 0.5 μL,SP 0.5 μL,TaKaRa LA Taq 0.25 μL,ddH2O 16.75 μL。反应程序:94℃ 1 min,98℃ 1 min(94℃ 30 s,65℃ 1 min,72℃ 2 min)5个循环,72℃ 2 min(94℃ 30 s,25℃ 3 min,72℃ 2 min,94℃ 30 s,65℃ 1 min,72℃ 2 min,94℃ 30 s,65℃ 1 min,72℃ 2 min,94℃ 30 s,44℃ 1 min,72℃ 2 min)15个循环,72℃ 10 min。扩增得到预期大小的片段,切胶回收,连接pTG19-T载体,转化大肠杆菌Trans 5α,蓝白斑筛选阳性克隆、测序。
1.2.4 实时荧光定量PCR分析CkGR基因表达
组织特异性表达分析,从柠条锦鸡儿根、茎和叶中提取总RNA,反转录后用实时荧光定量PCR检测。选取长势良好的柠条锦鸡儿幼苗,分别对其进行以下处理:低温处理,4℃培养箱中处理0、12、24、36和48 h后提取叶片总RNA;盐处理,在NaCl浓度为0、50、100、150和200 mmol·L-1的Hoagland营养液中浸泡24 h后提取叶片总RNA;干旱处理,在PEG6000含量为0%、5%、10%、15%和20%的Hoagland营养液中浸泡48 h后提取叶片总RNA。以上不同处理后提取的总RNA,经反转录得到cDNA作为实时荧光定量PCR模板,分别以ActinF和ActinR,CkGRDL-U和CkGRDL-D(表1)为管家基因引物和CkGR基因引物,按照宝生物工程公司的SYBR Premix Ex TaqTM操作说明书于25 μL反应体系在实时荧光定量PCR自动扩增仪(iQTM5 Optical Module,购自美国Bio-Rad公司)中扩增,每个样品做3个重复。扩增体系:SYBR Premix Ex TaqTM12.5 μL,ActinF/CkGRDL-U、ActinR/CkGRDL-D(10 μmol·L-1)0.5 μL,cDNA 2 μL,ddH2O 9.5 μL。扩增程序:95℃ 30 s;95℃ 5 s,60℃ 20 s,40个循环;55~95℃每30 s渐进升高0.5℃,81个循环。扩增完成后用2-△△Ct[17]法分析数据,确定CkGR基因相对表达量。
2 结果与分析
2.1 CkGR基因的克隆
根据GenBank上已克隆得到的植物GR基因序列,设计两对简并引物,以从柠条锦鸡儿叶片中提取的总RNA反转录产物为模板,扩增得到两条530和860 bp左右的单一条带(图1),和预期大小相符。将这两条带拼接后得到一条长度为1 309 bp的中间片段。将此中间片段的测序结果与其他植物GR基因序列进行比对,发现与鹰嘴豆CaGR的同源性较高。因此,初步预测该扩增产物为柠条锦鸡儿GR基因的片段。
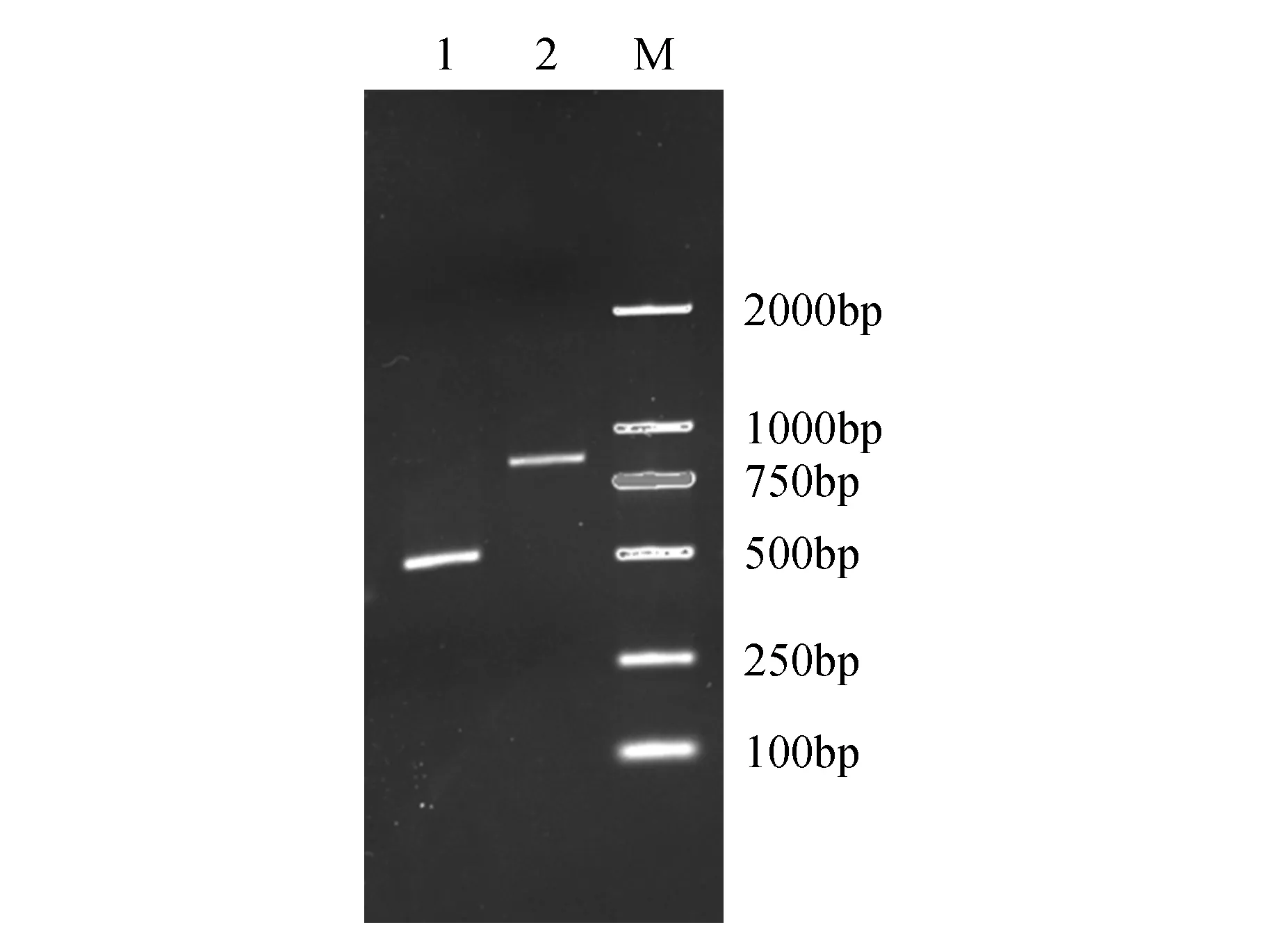
图1 CkGR基因片段RT-PCR扩增 M.分子量标准;1~2.CkGR基因片段Fig.1 RT-PCR amplification of CkGR fragment in C.korshinskii Kom. M.Marker; 1-2.Amplification of CkGR fragmen
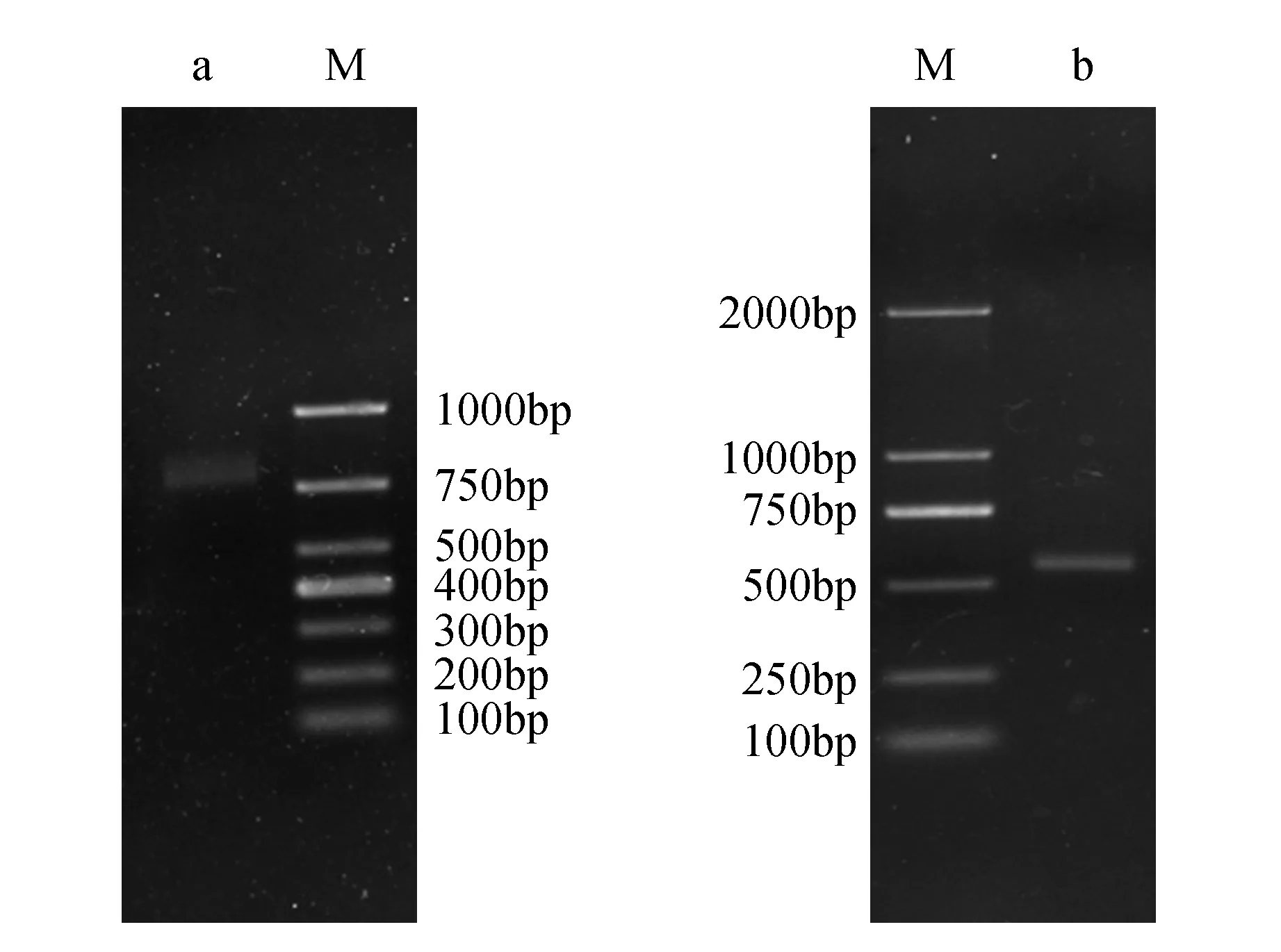
图2 3′-RACE和5′-RACE 扩增 M.分子量标准;a. 3′-RACE扩增;b. 5′-RACE扩增Fig.2 Amplification of 3′-RACE and 5′-RACE M. Marker; a. Amplification result of 3′-RACE; b. Amplification result of 5′-RACE
3′-RACE扩增得到760 bp大小的片段(图2:a),与预期片段大小相符。测序结果经核苷酸序列比对发现与前面扩增得到的CkGR部分片段的重叠部分的核苷酸序列完全相同,说明3′-RACE克隆得到的片段与前面获得的CkGR的部分片段属于同一条基因。5′-RACE扩增得到520 bp大小的片段(图2:b),与预期片段大小相符。测序结果经核苷酸序列比对发现与前面扩增得到的CkGR部分片段的重叠部分的核苷酸序列完全相同,说明5′-RACE获得的片段与前面克隆得到的部分片段属于同一条基因。
将扩增得到的中间片段序列以及3′-RACE和5′-RACE扩增得到的序列进行拼接,得到一条2 122 bp的基因序列,包含一个1 650 bp的开放阅读框(ORF),起始密码子ATG前有一段57 bp的5′非翻译区(5′-UTR),终止密码子TGA后有一段415 bp的3′非翻译区(3′-UTR),包含13 bp的poly A。此外,该基因编码550个氨基酸(图3),蛋白质分子量为59.2 kDa,理论等电点为8.2,命名为CkGR。根据3′-RACE和5′-RACE的扩增序列设计引物进行PCR扩增,获得了与拼接序列的核苷酸组成完全相同的片段,进一步确认了以上获得的拼接序列就是CkGR的基因序列。

图3 CkGR基因cDNA序列及预测氨基酸序列Fig.3 The cDNA sequence and deduced amino acid sequence of CkGR gene
2.2CkGR预测氨基酸序列与其他植物氨基酸序列比对分析
从Genbank中下载已克隆得到的植物GR基因序列,用DNAStar软件Megalign模块中的ClustalW方法进行氨基酸序列比对(图4)。结果表明,CkGR蛋白与许多植物的GR蛋白同源性较高,与鹰嘴豆CaGR(GenBank登录号:XM_004495086),大豆GmGR(GenBank登录号:XM_003536789),豌豆PsGR(GenBank登录号:X60373),毛果杨PtGR(GenBank登录号:XM_002321479),菜豆PvGR(GenBank登录号:DQ459505),蓖麻RcGR(GenBank登录号:XM_002520940)的同源性分别为90.1%,87.5%,89.0%,77.4%,85.3%,76.8%。
CkGR预测蛋白的疏水性在线分析表明,CkGR蛋白为疏水蛋白。二级结构在线预测显示,CkGR蛋白包含21.31% α-螺旋、28.42%延伸链、10.02% β-转角(beta turn)和40.26%不规则卷曲。
2.3 CkGR基因启动子的克隆及分析
以柠条锦鸡儿中提取到的总DNA为模板,进行巢式PCR扩增,经过AP3,SP3引物三扩后在770 bp处有一明显条带(图5),回收后测序。结果表明,成功克隆得到CkGR基因起始密码子ATG上游648 bp的启动子序列。采用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)软件进行基础启动子区及调控元件分析(图6),发现该序列具有启动子基本元件CAAT-box和TATA-box,还包括1个ABA应答元件ABRE(位于+105 bp处),1个茉莉酸甲酯响应元件CGTCA-motif(位于-508 bp处),一个光应答元件ACE(位于+106 bp处),1个赤霉素敏感元件GARE-motif(位于+319 bp处),两个厌氧诱导所需的顺式调控元件G-Box(分别位于-105 bp,+534 bp处),一个光应答元件GT1-motif(位于-140 bp处),一个热应答元件HSE(位于+479 bp处),一个干旱诱导的MBY结合位点MBSI(位于+221 bp处),两个胚乳表达相关顺式作用调控元件Skn-1_motif(分别位于-245 bp,-261 bp处),一个逆境应答顺式作用元件TC-rich repeats(位于+208 bp处),一个水杨酸应答顺式作用元TCA-element(位于+361 bp处),一个昼夜节律顺式调控元件circadian(位于-463 bp处)。此外,通过软件分析发现,该启动子还具有AAGAA-motif,GCN4_motif,TGACG-motif,TGG-motif等高效应元件。

图4 CkGR与其它植物GR氨基酸序列比对Fig.4 Alignment of the CkGR with GR from other plant species
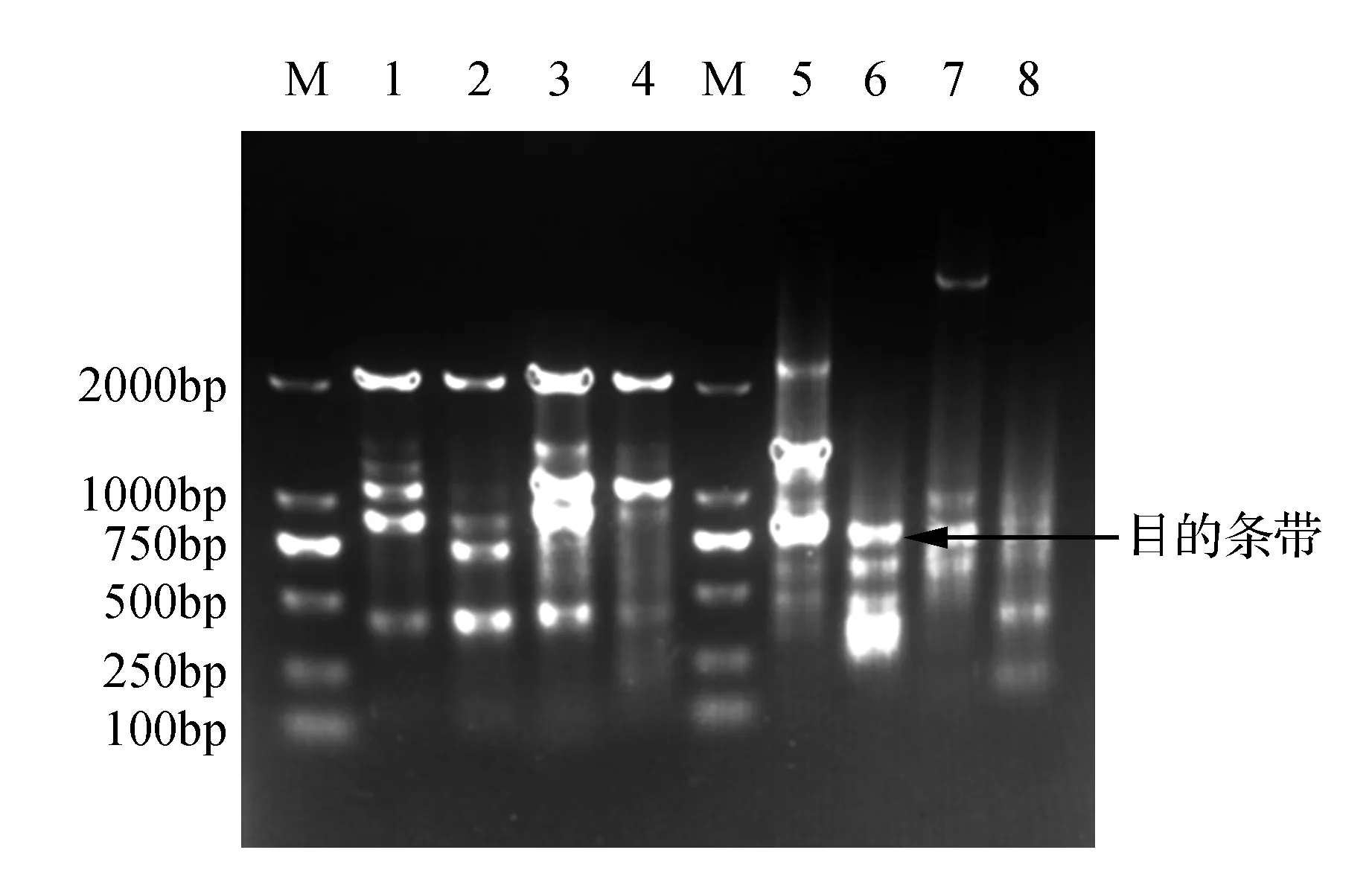
图5 三扩步移PCR产物电泳图 M.分子量标准;1~9.三次PCR产物Fig.5 Results of walking PCR M.Marker; 1-9.three times of PCR products
2.4 CkGR基因表达分析
以柠条锦鸡儿根、茎和叶中提取的mRNA反转录得到的cDNA为模板,进行实时荧光定量PCR,分析CkGR基因在柠条锦鸡儿不同组织的表达。结果表明,CkGR基因在柠条锦鸡儿的根、茎和叶中均有表达,没有组织特异性,叶中表达量最丰富(图7)。
4℃低温处理结果表明,CkGR基因的表达受4℃低温胁迫诱导。4℃处理12、24、36和48 h后,CkGR基因转录水平分别提高到对照(0 h)的1.64、11.39、23.10和3.73倍(图8)。CkGR基因的表达量呈现先升高后下降的趋势,在36 h达到最大,转录水平最高。
NaCl处理结果表明,高浓度的NaCl可诱导CkGR基因表达。当NaCl浓度为50、150和200 mmol·L-1时,CkGR基因的表达量分别提高到对照的5.86、7.06和9.45倍,CkGR基因的转录水平明显增强,NaCl浓度为100 mmol·L-1时,CkGR基因的表达量提高到对照的21.86倍,CkGR基因的转录水平最高(图9)。

图6 Plant CARE分析CkGR基因启动子序列的结果Fig.6 Nucleotide sequence of cloned CkGR gene promoter by Plant CARE

图7 CkGR基因在不同组织中的表达Fig.7 The expression of CkGR gene in different tissues

图8 低温(4℃)胁迫下CkGR基因在柠条锦鸡儿中的表达Fig.8 The expression of CkGR gene in C.korshinskii Kom. under cold(4℃) treatment
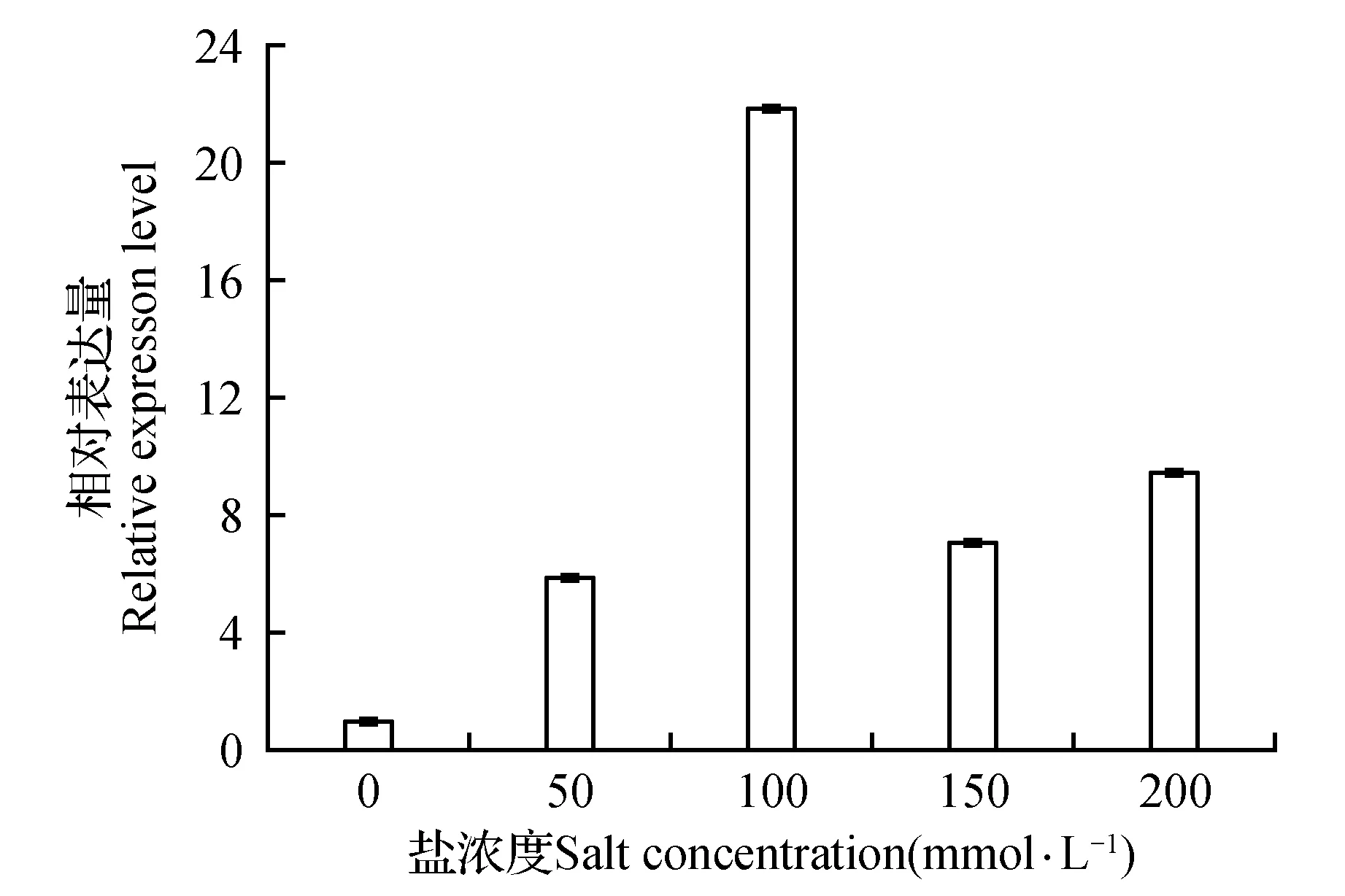
图9 盐胁迫下CkGR基因在柠条锦鸡儿中的表达Fig.9 The expression of CkGR gene in C.korshinskii Kom. under salt treatment

图10 干旱迫下CkGR基因在柠条锦鸡儿中的表达Fig.10 The expression of CkGR gene in C.korshinskii Kom. under drought treatment
干旱胁迫结果表明,用5%、10%、15%和20%的PEG6000处理后,CkGR基因转录水平分别提高到对照的2.95、2.73、9.00和7.78倍,CkGR基因表达量呈现出先升高后下降的趋势,在PEG6000质量分数为15%时达到最高(图10)。说明柠条锦鸡儿CkGR基因在适应干旱胁迫的过程中发挥作用。
3 讨论
对植物来说,环境因子如干旱、极端温度、盐害、氧化剂、大气污染物等都能导致氧化胁迫。GR参与植物逆境响应代谢途径,通过改变基因转录水平及酶活性水平,降低植物体内ROS含量,从而保护植物不受氧化胁迫的危害[18]。在用NaCl处理玉米幼苗的过程中,GR表达量在处理初期上升,而在处理后期开始下降[19]。用不同浓度的NaCl处理多枝赖草幼苗,随着NaCl浓度的增加,GR基因表达也逐渐加强[13]。用250 mmol·L-1NaCl处理油菜幼苗,随着处理时间的增加,GR基因表达也逐渐加强,24 h的GR基因表达最强,然后下降;采用1 mmol·L-1ABA处理,GR的表达量先增加,4 h达到最高值,然后下降,但均比对照表达量高[20],这与本研究的结果一致。Rebecca等[21]研究表明,将菜豆细胞质GR基因转入烟草,GR的总活性明显提高;Barry等[22]的研究结果表明,将GR基因转入棉花,冷处理由28℃降到14℃,转基因棉花的GR活性比野生型棉花高30~40倍。这说明GR在氧化胁迫中通过活性的升高对清除ROS起积极作用。本研究从柠条锦鸡儿中克隆得到了含完整编码序列的CkGR基因,序列分析表明CkGR和其他植物中的GR同源性很高。利用染色体步移技术得到CkGR基因起始密码子ATG上游648 bp的启动子序列,用PlantCARE软件分析表明,该启动子序列除包含启动子基本作用元件CAAT-box和TATA-box外,还包含与非生物胁迫如ABA,干旱,高温等相关的多种顺式调控元件。CkGR基因的组织特异性表达结果表明,该基因在柠条锦鸡儿根、茎和叶中都有表达,无组织特异性,叶中表达量最高。对低温、高盐和干旱胁迫下CkGR基因的表达量进行定量分析,结果表明,低温、高盐和干旱胁迫都会使CkGR基因的表达量发生先升高后下降的变化,但均比对照表达量高,说明CkGR基因在柠条锦鸡儿抵御低温、高盐和干旱胁迫过程中发挥了作用,为进一步研究GR在植物抗逆胁迫中的作用机制提供了参考依据。
1.Mullineaux P M,Cressen G P.Glutathione reductase:regulation and role in oxidative stress[M].//John G.Oxidative stress and the molecular biology of antioxidant defenses.United Kingdom:Cold Spring Harbor Laboratory Press,1997.
2.Gill S S,Tuteja N.Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants[J].Plant Physiology and Biochemistry,2010,48(12):909-930.
3.张锐,兰文升,贺秀媛,等.贝类毒素大田软海绵酸OA对小鼠肝脏还原型谷胱甘肽GSH、过氧化氢CAT、超氧化物歧化酶SOD的影响[J].生物学杂志,2014,31(1):11-14.
Zhang R,Lan W S,He X Y,et al.Influence of reduced glutathione GSH,catalase CAT,superoxide dismutase SOD in liver of mice by exposure to shellfish toxin OA[J].Journal of Biology,2014,31(1):11-14.
4.Belozerskaya T A,Gessler N N.Reactive oxygen species and the strategy of antioxidant defense in fungi:a review[J].Applied Biochemistry and Microbiology,2007,43(5):506-515.
5.Edwardse A,Rawsthorne S,Mullineauxp M.Subcellular distribution of multiple forms of glutathione reductase in leaves of pea(PisumsativumL.)[J].Planta,1990,180(2):278-284.
6.Meister A,Anderson M E.Glutathione[J].Annual Review of Biochemistry,1983,52(1):711-760.
7.Alscher R G.Biosynthesis and antioxidant function of glutathione in plants[J].Physiologia Plantarum,1989,77(3):457-464.
8.Kaminaka H,Morita S,Nakajima M,et al.Gene cloning and expression of cytosolic glutathione reductase in rice(OryzasativaL.)[J].Plant & Cell Physiology,1998,39(12):1269-1280.
9.Yousuf P Y,Hakeem K U R,Chandna R,et al.Role of glutathione reductase in plant abiotic stress[M].//Ahmad P,Prasad M N V.Abiotic stress responses in plants.New York:Springer-Verlag,2012:149-158.
10.Creissen G P,Mullineaux P M.Cloning and characterisation of glutathione reductase cDNAs and identification of two genes encoding the tobacco enzyme[J].Planla,1995,197(2):422-425.
11.Stevens R G,Creissen G P,Mullineaux P M.Cloning and characterisation of a cytosolic glutathione reductase cDNA from pea(PisumsativumL.)and its expression in response to stress[J].Plant Molecular Biology,1997,35(5):641-654.
12.史仁玖,赵茂林,杨清.多枝赖草谷胱甘肽还原酶基因的克隆及分析[J].西北农林科技大学学报:自然科学版,2006,34(2):6l-67.
Shi R J,Zhao M L,Yang Q.Cloning and analysis of glutathione reductase gene fromLeymusmulticaulis[J].Journal of Northwest Sci-Tech University of Agriculture and Forestry:Natural Science Edition,2006,34(2):61-67.
13.李彦肖,彭海涛,管苇,等.不结球白菜谷胱甘肽还原酶基因NhccGR1的克隆与表达分析[J].南京农业大学学报,2012,35(3):7-12.
Li Y X,Peng H T,Guan W,et al.Cloning and expression analysis of glutathione reductase geneNhccGR1 in non-heading Chinese cabbage[J].Journal of Nanjing Agricultural University,2012,35(3):7-12.
14.李高,杨杞,张烨,等.柠条锦鸡儿香豆酸-3-羟化酶基因克隆及其功能初步研究[J].中国生物工程杂志,2013,33(4):61-67.
Li G,Yang Q,Zhang Y,et al.Cloning and sequence analysis of the CkC3H gene fromCaraganakorshinskiiKom. and preliminary studies of its function[J].China Biotechnology,2013,33(4):61-67.
15.刘龙会,古松,郭亚娇,等.柠条锦鸡儿小孢子发生和雄配子体发育[J].西北植物学报,2012,32(1):81-84.
Liu L H,Gu S,Guo Y J,et al.Microsporogenesisand male gametophyte development ofCaraganakorshinskiiKom.[J].Acta Botanica Boreali-Occidentalia Sinica,2012,32(1):81-84.
16.杨杞,张涛,王颖,等.干旱胁迫下柠条锦鸡儿叶片SSH文库构建及CkWRKY1基因克隆[J].林业科学,2013,49(7):62-68.
Yang Q,Zhang T,Wang Y,et al.Construction of a suppression subtractive hybridization library ofCaraganakorshinskiiKom. under drought stress and cloning ofCkWRKY1 gene[J].Scientia Silwae Sinicae,2013,49(7):62-68.
17.杨慧萍,刘雅莉,娄倩,等.葡萄风信子谷胱甘肽S转移酶基因克隆与表达分析[J].植物研究,2016,36(1):134-140.
Yang H P,Liu Y L,Lou Q,et al.Clone and characterization of a glutathione-S-transferase gene inMuscariarmeniacum[J].Bulletin of Botanical Research,2016,36(1):134-140.
18.林源秀,顾欣昕,汤浩茹.植物谷胱甘肽还原酶的生物学特性及功能[J].中国生物化学与分子生物学报,2013,29(6):534-542.
Lin Y X,Gu X X,Tang H R.Characteristics and biological functions of glutathione reductase in plants[J].Chinese Journal of Biochemistry and Molecular Biology,2013,29(6):534-542.
19.郭丽红,陈善娜,龚明.钙对玉米幼苗谷胱甘肽还原酶活性的影响[J].植物生理学通讯,2002,38(2):115-117.
Guo L H,Chen S N,Gong M.Effect of calcium on glutathione reductase of maize seedlings[J].Plant Physiology Communications,2002,38(2):115-117.
20.Lee H,Won S H,Lee B H,et al.Genomic cloning and characterization of glutathione reductase gene fromBrassicacampestrisvar.pekinensis[J].Molecules and Cells,2002,13(2):245-251.
21.Stevensr G,Creissen G P,Mullineaux P M.Characterisation of pea cytosolic glutathione reductase expressed in transgenic tobacco[J].Planta,2000,211(4):537-545.
22.Loganb A,Monteiro G,Kornyeyev D,et al.Transgenic overproduction of glutathione reductase does not protect cotton,Gossypiumhirsutum(Malvaceae),from photoinhibition during growth under chilling conditions[J].American Journal of Botany,2003,90(9):1400-1403.
The National Natural Science Foundation of China(31460099,31160089)
introduction:ZHANG Teng-Guo(1971—),male,Dr.,professor,Mainly engaged in the study of physiological and molecular biology.
date:2016-03-22
CloningandExpressionAnalysisofCkGRGeneinCaraganakorshinskiiKom.
ZHANG Teng-Guo ZHOU Ke MAO Yu-Shan NIE Ting-Ting LI Ping DIAO Zhi-Hong WANG Juan
(School of Life Science,Northwest Normal University,Lanzhou 730070)
A novelGRgenewas isolated fromCaraganakorshinskiiKom. by RACE. The full-length cDNA ofGRwas 2 122 bp, containing a 5′-UTR of 57 bp, a 3′-UTR of 415 bp, and a 1 650 bp opening reading frame(ORF). The deduced protein was 550 amino acids with molecular weight 59.2 kDa and isoelectric point 8.2, namedCkGR. ThisCkGRshowed high identities with theCicerarietinumCaGR(90.6%). The promoter ofCkGRgene was isolated by chromosomal walking and 648 bp sequence was obtained by sequencing. Plant CARE analysis of this sequence showed that the peomoter contained some typical elements CAAT-box and TATA-boxand kinds of Cis-acting elements involved in defense and stress responsiveness. RT-PCR analysis revealed thatCkGRwas expressed in roofs, stems, and leaves with almost no tissue specificity. The transcript level ofCkGRwas increased in response to cold, high salt and drought stress.CkGRplayed an important role during cold, high salt and drought stress inCaraganakorshinskiiKom..
CaraganakorshinskiiKom.;CkGRgene;molecular cloning;expression analysis
国家自然科学基金(31460099,31160089)
张腾国(1971—),男,博士,教授,主要从事抗逆生理及分子生物学研究。
2016-03-22
S793.3
A
10.7525/j.issn.1673-5102.2016.04.005
