橡胶树MSAP反应体系优化及其不同开割高度单株DNA甲基化分析
2016-11-10吴春太曾日中
吴春太 班 硕,2 黎 瑜 曾日中*
(1.中国热带农业科学院橡胶研究所/农业部橡胶树生物学与遗传资源利用重点实验室/国家橡胶树育种中心/海南省热带作物栽培生理学重点实验室,儋州 571737; 2.华中农业大学植物科学技术学院,武汉 430070)
橡胶树MSAP反应体系优化及其不同开割高度单株DNA甲基化分析
吴春太1班 硕1,2黎 瑜1曾日中1*
(1.中国热带农业科学院橡胶研究所/农业部橡胶树生物学与遗传资源利用重点实验室/国家橡胶树育种中心/海南省热带作物栽培生理学重点实验室,儋州 571737;2.华中农业大学植物科学技术学院,武汉 430070)
以‘热研7-33-97’橡胶树无性系幼嫩叶片为材料,通过利用单因素和正交试验相结合的方法,对酶切、预扩增和选择性扩增3个影响甲基化敏感扩增多态性(MSAP)分析的关键步骤的反应体系中关键影响因素进行了优化,建立橡胶树MSAP反应最佳体系,并用于高低割线橡胶树基因组DNA甲基化差异分析。结果表明:在50 μL反应体系中,750 ng基因组DNA用EcoRⅠ 20 U,HpaⅡ 20 U或MspⅠ 10 U于37℃恒温同步酶切10 h,酶切完全。最佳预扩增体系(20 μL)为:连接产物4 μL,MgCl2(25 mmol·L-1)0.15 μL,dNTPs(2.5 mmol·L-1)0.1 μL,上下游引物E-00/HM-00(10 μmol·L-1)各0.3 μL,Taq酶(5 U·μL-1)0.1 μL,10×PCR Buffer 2 μL。最佳选择性扩增反应体系(20 μL)为:稀释20倍的预扩增产物2 μL,MgCl2(25 mmol·L-1)0.1 μL,dNTPs(2.5 mmol·L-1)0.125 μL,上下游引物E+3/HM+3(10 μmol·L-1)各0.4 μL,Taq酶(5 U·μL-1)0.1 μL,10×PCR Buffer 2 μL。高低割线树DNA的甲基化比例分别为37.22%和36.43%,2种开割胶树基因组CCGG位点胞嘧啶全甲基化率明显高于半甲基化率,推测橡胶树基因组甲基化主要模式可能是CpG型。综上表明,建立的MSAP反应体系稳定可靠且重复性好,为后续橡胶树不同胁迫(割胶)程度DNA甲基化的研究奠定了基础。
高低割线橡胶树;DNA甲基化;甲基化敏感扩增多态性(MSAP);体系优化
DNA甲基化(DNA methylation)是基因组DNA主要的表观遗传(epigenetics)修饰形式[1],在高等植物基因组中残基发生甲基化的胞嘧啶约有20%~50%,其中约有90%的5mC位于CpG岛或CpNpG三核苷酸对区域[2~4]。其在植物基因表达、生长发育、抵御环境胁迫等方面发挥着至关重要的调控作用[4~7]。目前检测DNA甲基化的方法有多种,其中甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)是检测基因组DNA胞嘧啶甲基化位点最常用的方法。自1999年Xiong等[8]首次证明植物DNA甲基化MSAP检测方法的可靠性以来,大量的研究表明,其已广泛应用于伴随树木生长发育和环境胁迫响应过程的表观遗传变异研究领域。目前,在毛竹(PhyllostachysheterocyclaVar)[9]、五月季竹(Phyllostachysbambusoides)[10]、毛白杨(Populustomentosa)[11~13]、杉木(Cunninghamialaneeolata)[14]、红豆杉(Taxusmedia)[15]、辐射松(Pinusradiata)[16~19]、落叶松(Larixleptolepis)[20~22]、泡桐(Paulowniafortunei)[23~26]等多种乔木中都有应用,因此对橡胶树木生长发育过程中和割胶胁迫等不良环境中时DNA甲基化模式和水平变化的研究十分必要。
橡胶树是产胶作物中最主要的树种,是常见热带木本经济作物,胶乳量丰富,可作上万种工业制品的原料,应用领域主要集中于商业、防御和运输等行业[27]。中国是世界第五大天然橡胶生产国,在海南、云南、广东等地均有栽培,对热区农村经济发展和生态环境改善有十分重要的影响。近年来针对橡胶树的机械伤害适应性机制进行了许多研究,但研究多集中于基因和蛋白表达等方面[28~30]。尽管吴春太等于2011年建立了橡胶树的MSAP荧光标记反应体系,并在种植材料类型不同的‘热研7-33-97’老幼态无性系中得到验证[31],同年,李海林等通过单因素和L9(34)正交试验法分别对酶切体系和选择性扩增体系进行优化,建立了适宜橡胶树MSAP分析的技术体系[32~33],其后利用该体系分析种植材料类型不同的‘热研8-79’老幼态无性系的DNA甲基化[34],但关于同一林段范围内单一品种割线高度不同单株间橡胶树DNA甲基化模式和水平的研究尚未见报道。探究橡胶树DNA甲基化模式和水平对了解橡胶树生长发育和环境胁迫适应机理有重大意义。本研究以‘热研7-33-97’橡胶树品种幼嫩叶片为材料,通过优化主要影响因素,建立橡胶树MSAP分析的技术体系,并初步应用于割线高度不同橡胶树基因组DNA甲基化的变化分析,旨在为阐明橡胶产量形成的分子机制,以及从分子层次为橡胶树的割面规划、割制改革等应用基础研究提供技术依据。
1 材料与方法
1.1 试验材料
高割线和低割线橡胶树选自中国热带农业科学院(儋州)试验场金富队科技示范园8年生‘热研7-33-97’,割龄约2年,高低割线树开割高度分别为1.2和0.9 m。每种割线树随机选择3株,于2014年3月采集健康的古铜期嫩叶,采集后装入冰盒迅速带回实验室,置于-80℃冰箱备用。
1.2 基因组DNA提取
首先从两种割线树各随机抽取3个单株,共6个单株,采用改良CTAB法[35]分别提取各单株基因组DNA,RNases处理后,利用1%琼脂糖凝胶电泳和核酸蛋白测定仪检测DNA的质量和浓度;然后从每个样品中取等量的DNA混合,用于橡胶树MSAP分析技术反应体系的建立,剩余的DNA样品按割线类别混合后用于不同割线橡胶树DNA甲基化模式与水平分析。
1.3 MSAP技术流程
MSAP基本程序按照Xiong等[8]报道的方法进行,并针对DNA酶切体系、预扩增体系、选择性扩增体系等反应条件分别进行筛选优化。
1.3.1 酶切体系
为确定最佳酶切方式,利用对甲基化敏感的同裂酶HpaⅡ和MspⅠ与限制性内切酶EcoRⅠ对基因组DNA分别进行单酶切、分步双酶切和同步双酶切:①选用EcoRⅠ、HpaⅡ和MspⅠ三种内切酶分别对基因组DNA进行单酶切。单酶切反应体系中分别加入EcoRⅠ 20 U、HpaⅡ 10 U、MspⅠ 20 U,浓度为250 ng·μL-1的模板DNA 3 μL,10×CutSmart Buffer缓冲液5 μL,加灭菌去离子水至50 μL,混匀后放入水浴锅中37℃恒温酶切,将EcoRⅠ、HpaⅡ和MspⅠ的酶切时间分别设定为4、10、10 h。②选用HpaⅡ/EcoRⅠ和MspⅠ/EcoRⅠ两组内切酶分别对基因组DNA进行分步双酶切。50 μL体系中,先加入10 UEcoRⅠ,5 μL 10×CutSmart Buffer,基因组DNA模板750 ng,再加入加灭菌ddH2O至50 μL,混匀后用水浴锅37℃恒温酶切4 h,最后分别加入HpaⅠ 20 U或MspⅠ 10 U酶切10 h。③选取HpaⅡ/EcoRⅠ和MspⅠ/EcoRⅠ两组内切酶分别对基因组DNA进行同步双酶切。50 μL体系中包括:750 ng基因组DNA,10×CutSmart Buffer缓冲液5 μL,MspⅠ 10 U,HpaⅡ和EcoRⅠ各20 U,ddH2O补至50 μL,37℃恒温酶切10 h。所有体系酶切完毕后,均65℃灭活20 min,并取6 μL酶切产物于1.2%琼脂糖凝胶电泳检测。
1.3.2 连接体系
连接反应体系包括24 μL上述完全双酶切产物,以EcoRⅠ/HpaⅡ同步酶切组合为标准(下同),1 μLEcoRⅠ(5 μmol·L-1)或HpaⅡ(50 μmol· L-1)接头,接头在加入反应体系之前先合为双链(95℃变性5 min,缓慢降至室温),3 μL 10×T4ligase Buffer,T4连接酶(350 U·μL-1)用量为350 U,加灭菌ddH2O至30 μL,混匀后于水浴锅中16℃连接过夜(12~16 h)。连接产物70℃灭活15 min,-20℃保存备用。
1.3.3 预扩增正交反应体系
用E-00/HM-00引物组合对连接产物进行预扩增,反应总体积为20 μL。为确定MSAP预扩增体系中连接产物用量和Mg2+、dNTPs、引物、TaqDNA聚合酶的浓度5个因素的最佳水平,根据前期预实验单因子考察结果,初步设定预扩增的各因素水平范围,试验采用L16(45)正交设计,各因素反应条件和L16(45)设计方案分别见表1~2。PCR扩增程序为:95℃预变性5 min;94℃变性30 s,56℃退火1 min,72℃延伸1 min,共30个循环;最后72℃延伸10 min。预扩后取5 μL预扩增产物进行1.2%琼脂糖凝胶电泳检测,凝胶分析系统记录保存结果。剩余产物-20℃冰箱保存备用。
表1橡胶树MSAP预扩增反应体系各因素体积水平
Table1FactorsandtheirvolumelevelsofMSAPpre-amplificationreactionsystemforH.brasiliensis(μL)
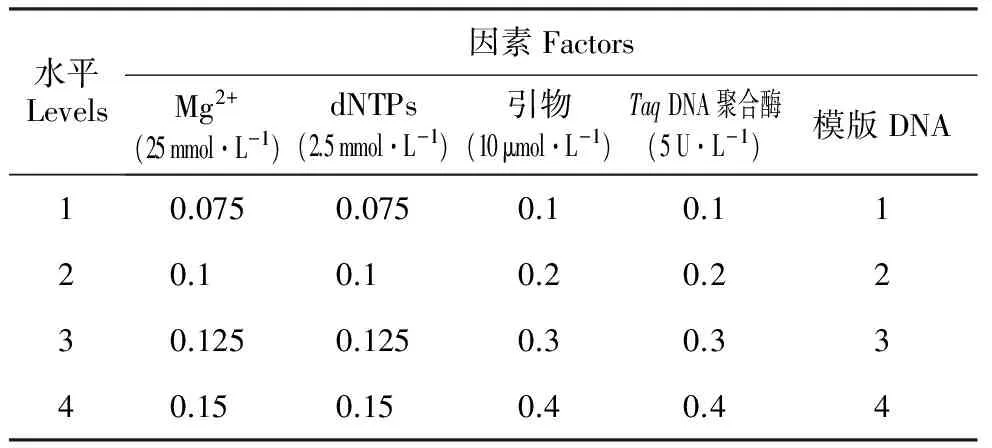
水平Levels因素FactorsMg2+(25mmol·L-1)dNTPs(2.5mmol·L-1)引物(10μmol·L-1)TaqDNA聚合酶(5U·L-1)模版DNA10.0750.0750.10.1120.10.10.20.2230.1250.1250.30.3340.150.150.40.44
表2橡胶树MSAP预扩增反应体系各因素水平L16(45)正交试验设计
Table2L16(45)orthogonaldesignoffactorsandtheirvolumelevelsinreactionsystemforMSAPpre-amplificationfromH.brasiliensis

编号Codes因素FactorsMg2+(25mmol·L-1)dNTPs(2.5mmol·L-1)引物(10μmol·L-1)Taq聚合酶(5U·L-1)模版DNA10.0750.0750.10.1120.0750.10.20.2230.0750.1250.30.3340.0750.150.40.4450.10.0750.20.3460.10.10.10.4370.10.1250.40.1280.10.150.30.2190.1250.0750.30.42100.1250.10.40.31110.1250.1250.10.24120.1250.150.20.13130.150.0750.40.23140.150.10.30.14150.150.1250.20.41160.150.150.10.32
表3橡胶树MSAP选择性扩增反应体系因素及水平数
Table3FactorsandtheirlevelnumbersofMSAPselectiveamplificationreactionsystemforH.brasiliensis

水平Levels因素FactorsMg2+(25mmol·L-1)dNTPs(2.5mmol·L-1)引物(10μmol·L-1)TaqDNA聚合酶(5U·L-1)预扩增产物稀释倍数10.0750.0750.10.11020.10.10.20.22030.1250.1250.30.33040.150.150.40.440
表4橡胶树MSAP选择性扩增反应体系各因素水平L16(45)正交试验设计
Table4L16(45)orthogonaldesignoffactorsandtheirlevelsinreactionsystemforMSAPselectiveamplificationfromH.brasiliensis

编号Codes因素FactorsMg2+(25mmol·L-1)dNTPs(2.5mmol·L-1)引物(10μmol·L-1)TaqDNA聚合酶(5U·L-1)预扩增产物稀释倍数10.0750.0750.10.11020.0750.10.20.22030.0750.1250.30.33040.0750.150.40.44050.10.0750.20.34060.10.10.10.43070.10.1250.40.12080.10.150.30.21090.1250.0750.30.420100.1250.10.40.310110.1250.1250.10.240120.1250.150.20.130130.150.0750.40.230140.150.10.30.140150.150.1250.20.410160.150.150.10.320
1.3.4 选择性扩增正交反应体系
在前期预实验单因子考察的基础上,选用E+3/HM+3引物组合对稀释后的预扩增最佳组合的产物进行选择性扩增,反应总体积为20 μL,包含不同稀释倍数的预扩增产物1.0 μL。并仍采用L16(45)正交试验确定选择性扩增反应中的预扩增产物稀释倍数和Mg2+、dNTPs、引物、TaqDNA聚合酶的浓度5个因素的最佳反应条件。各因素水平和正交实验设计分别见表3~4。PCR反应采用Touchdown程序:95℃ 5 min;94℃ 30 s,65℃ 30 s(每循环下降0.7℃),72℃ 1 min,共12个循环;94℃ 30 s,55℃ 30 s,72℃ 1 min,共24个循环;最后于72℃延伸10 min。取5 μL选择性扩增产物用1.2%琼脂糖凝胶电泳检测。
1.4 橡胶树基因组DNA的MSAP分析
选用E-ATT+H/M-TAA,E-ATT+H/M-TAT,E-ATT+H/M-TAG,E-ATT+H/M-TAC,E-ATT+H/M-TTA,E-ATT+H/M-TTT,E-ATT+H/M-TTG,E-ATT+H/M-TTC,E-ATT+H/M-TGA,E-ATT+H/M-TGT,E-ATT+H/M-TGG,E-ATT+H/M-TGC,E-ATT+H/M-TCA,E-ATT+H/M-TCT,E-ATT+H/M-TCG和E-ATT+H/M-TCC 12对选择性扩增引物,利用优化后的选择性扩增体系分别对高割线树和低割线树叶片进行MSAP分析,选择性扩增产物变性后取5.0~8.0 μL进行6%变性聚丙烯酰胺凝胶电泳检测,银染后获得指纹图谱,用以研究2种不同割线橡胶树之间DNA甲基化的变化情况。
2 结果与分析
2.1 橡胶树叶片基因组DNA质量检测
通过1%琼脂糖凝胶电泳和核酸蛋白测定仪对采用改良CTAB法提取的叶片基因组DNA的完整性和纯度进行检测。从图1可以看出,基因组DNA带型完整,锐度好,上样孔处无蛋白质和多糖残留,泳道无拖尾,无RNA,没有DNA降解;纯度检测结果显示,D260/D280值在1.78~1.80,D320值在0.002~0.004,说明提取的DNA完整性好、纯度高,完全符合橡胶树MSAP技术对DNA质量的要求。
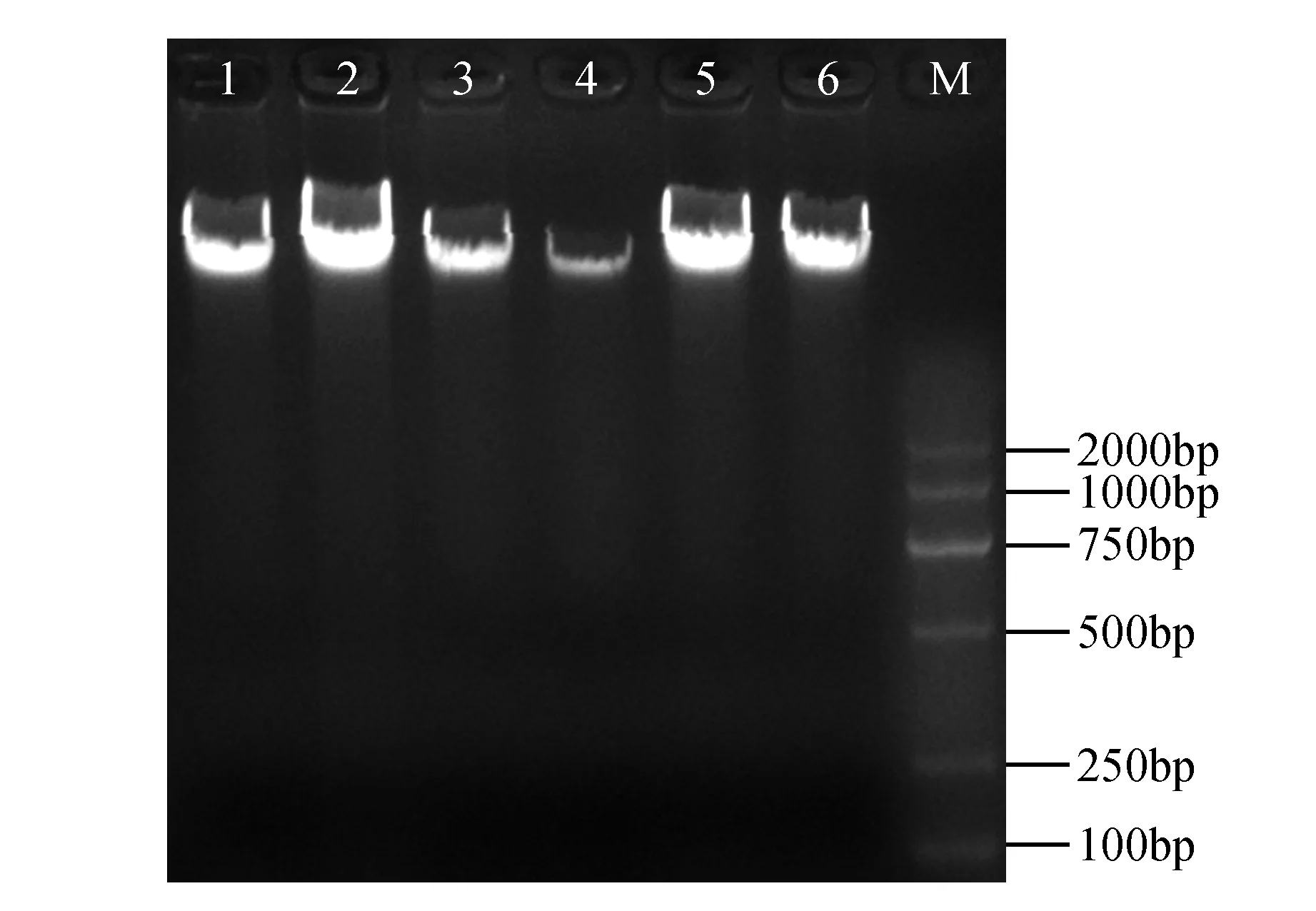
图1 橡胶树全基因组DNA琼脂糖凝胶电泳检测图 1~6.橡胶树基因组总DNA;M. 2000的分子量标准Fig.1 Agarose gel electrophoresis of total genomic DNA extraction from H.brasiliensis 1-6.Total genomic DNA of H.brasiliensis; M. Molecular weight standard
2.2 橡胶树基因组DNA的MSAP体系优化2.2.1 酶切体系
为考察EcoRⅠ、MspⅠ和HpaⅡ酶切的效果,分别用此三种限制性内切酶对基因组DNA进行单酶切、分步酶切和同步酶切。酶切产物电泳检测结果见图2。从图2可看出,EcoRⅠ、MspⅠ和HpaⅡ单酶切后的泳道上主条带消失,条带皆为弥散状态,且分布特征与高频酶和低频酶相符,表明酶切完全。而且分步酶切和同步酶切泳道内均呈弥散状,没有单一分子量的DNA条带出现,表明酶切充分,其片段大小均满足MSAP技术对DNA的要求,可进行后续的连接反应。但由于同步酶切较分步酶切节约酶切时间4 h,避免因水浴时间过长而导致的模板DNA的降解,故选择同步酶切为最佳酶切体系,最终的50 μL同步酶切体系中含有模板DNA 750 ng、EcoRⅠ 20 U、MspⅠ 10 U/HpaⅡ 20 U,酶切时间为10 h。

图2 不同方式酶切的橡胶树基因组DNA电泳图谱 1.EcoRⅠ/HpaⅡ分步酶切;2.EcoRⅠ/MspⅠ分步酶切;3.阳性对照;4.HpaⅡ单酶切;5.MspⅠ单酶切;6.EcoRⅠ单酶切;7.EcoRⅠ/HpaⅡ同步酶切;8:EcoRⅠ/MspⅠ同步酶切;M. 2000的分子量标准Fig.2 Agarose gel electrophoresis of Hevea brasiliensis genomic DNA digested with enzyme by different ways 1.Fractional digestion of EcoRⅠ/HpaⅡ; 2.Fractional digestion of EcoRⅠ/MspⅠ; 3.Positive control; 4.HpaⅡ single enzyme digestion; 5.MspⅠ single enzyme digestion; 6.EcoRⅠ single enzyme digestion; 7.Synchronous digestion of EcoRⅠ/HpaⅡ; 8.Synchronous digestion of EcoRⅠ/MspⅠ; M.Molecular weight standard
2.2.2 预扩增
对16个处理的预扩增产物电泳(图3)分析表明,PCR扩增结果存在明显差异。从图3中可见,1~16泳道中第1、2、6、8、11、12、15、16泳道的预扩增产物主要集中在500~1 000 bp,产物片段过大,不符合MSAP对预扩增产物的要求。而第5、9、10、13泳道的预扩增产物的量相对较少,可能会导致后续的选择性扩增不能顺利进行。尽管第3、7泳道的预扩增产物片段主要集中在100~750 bp,条带弥散且产物的量足以进行选择性扩增,但是这两个体系在100~500 bp扩增产物量太少,同样也不利于后续试验。处理4和14预扩增体系的条带主要集中在100~750 bp,呈弥散状,预扩增产物浓度相对较高,符合MSAP对预扩增产物的要求。最终确定处理14为最佳预扩增体系,即20 μL总体积中,含连接产物4 μL,MgCl2(25 mmol·L-1)0.15 μL,dNTPs(2.5 mmol·L-1)0.1 μL,预扩增引物(10 μmol·L-1)0.3 μL,TaqDNA聚合酶(5 U·μL-1) 0.1 μL,10×PCR Buffer 2 μL。
2.2.3 选择性扩增
对所有正交处理选择性扩增的产物电泳(图4)分析表明,PCR扩增结果存在明显差异。从图3中可看出,第1、2、6、9、15、16泳道片段主要集中在250~2 000 bp,背景涂布较明显;第5、8、10、11、12、13、14泳道存在少量的非特异性扩增条带;处理3、4、7选择性扩增体系扩增效果好,背景无涂布现象,条带清晰,主条带亮度较高,无明显的非特异性扩增且扩增的多态性条带主要集中在100~500 bp,符合MSAP中选择性扩增对产物的要求。由于在考虑结果的同时也要兼顾试验成本的节约,所以最终选择第7号体系为选择性扩增的最佳体系比较合理,即20 μL总体积中,含稀释20倍的预扩增产物2 μL,MgCl2(25 mmol·L-1) 0.1 μL,dNTPs(2.5 mmol·L-1) 0.125 μL,选择性扩增引物(10 μmol·L-1)各0.4 μL,TaqDNA聚合酶(5 U·μL-1)0.1 μL,10×PCR Buffer 2 μL。
2.3高低割线橡胶树株间DNA甲基化模式与水平分析
利用优化后的橡胶树MSAP反应体系,对高割线和低割线橡胶树单株进行了DNA甲基化多态性分析,通过6%尿素变性聚丙烯酰胺凝胶电泳对12对引物扩增产物进行检测,结果如图4所示。对图4进行直观分析可见(图5),每对引物在2种割株中的扩增均获得清晰的扩增谱带。由MSAP电泳图谱分析显示(表5),在高割线树和低割线树中扩增出的条带数分别为266和291。高割线树和低割线树中均有3种甲基化模式存在,I型条带(去甲基化位点)数目最多,约占扩增产物总数的63.18%以上,Ⅲ型条带(全甲基化位点)占31.63%左右,Ⅱ型条带(半甲基化位点)相对较少约5.19%。但在2种胶树间未检测出有甲基化模式差异的多态性位点。

图3 正交优化预扩增产物琼脂糖凝胶电泳图谱 1~16. L16(45)正交试验中不同处理编号;M. 2000的分子量标准 下同。Fig.3 Agarose gel electrophoresis map of optimized pre-amplification products using orthogonal designs 1-16. Treatments of orthogonal designs number 1 to 16; M. Molecular weight standard The same as below.

图4 正交优化选择性扩增产物电泳图谱Fig.4 Electrophoresis map of optimized selective amplification products by orthogonal designs

图5 高割线橡胶树和低割线橡胶树基因组DNA的MSAP扩增图谱 1、2、5、6、9、10、13、14、17、18、21、22、25、26、29、30、33、34、37、38、41、42、45、46. 高割线橡胶树基因组DNA;3、 4、7、8、11、12、15、16、19、20、23、24、27、28、31、32、35、36、39、40、43、44、47、48. 低割线橡胶树基因组DNA;奇数泳道. EcoRⅠ/HpaⅡ酶切与扩增产物;偶数泳道. EcoRⅠ/MspⅠ酶切与扩增产物;1~4.E-ATT/HM-TAA;5~8.E-ATT/HM-TAT;9~12.E-ATT/HM-TAG;13~16.E-ATT/HM-TAC;17~20.E-ATT/HM-TTA;21~24.E-ATT/HM-TTT;25~28.E-ATT/HM-TTG;29~32.E-ATT/HM-TTC;33~36.E-ATT/HM-TGA;37~40.E-ATT/HM-TGT;41~44.E-ATT/HM-TGG;45~48.E-ATT/HM-TGC;M. 2000的分子量标准(变性为单链)Fig.5 MSAP profiles of H.brasiliensis genomic DNA between high tapping cut and low tapping cut 1,2,5,6,9,10,13,14,17,18,21,22,25,26,29,30,33,34,37,38,41,42,45,46. High tapping cut H.brasiliensis genomic DNA; 3,4,7,8,11,12,15,16,19,20,23,24,27,28,31,32,35,36,39,40,43,44,47,48. Low tapping cut H.brasiliensis genomic DNA; Odd number of lanes. Fragments obtained after digestion with EcoRⅠ/HpaⅡ and amplification by PCR; Even number of lanes. Fragments obtained after digestion with EcoRⅠ/MspⅠ and amplification by PCR; 1~4.E-ATT/HM-TAA; 5~8.E-ATT/HM-TAT; 9~12.E-ATT/HM-TAG; 13~16.E-ATT/HM-TAC; 17~20.E-ATT/HM-TTA; 21~24.E-ATT/HM-TTT; 25~28.E-ATT/HM-TTG; 29~32.E-ATT/HM-TTC; 33~36.E-ATT/HM-TGA; 37~40.E-ATT/HM-TGT; 41~44.E-ATT/HM-TGG; 45~48.E-ATT/HM-TGC; M. Molecular weight standard(denatured single-stranded DNA)
Table5ComparisonofgenomicDNAmethylationlevelintwokindsofH.brasiliensisofdifferenttappingcuts

类型Type高割线树Hightappingcutplants低割线树Lowtappingcutplants总扩增条带数Totalamplifiedbands266291半甲基化条带数Hemimethylationbands1316全甲基化条带数Fullmetylationbands8690半甲基化率Hemimetylatedrate4.89%5.50%全甲基化率Fullmetylationrate32.33%30.93%总甲基化率Totalmethylationrate37.22%36.43%
MSAP图谱分析还显示,高割线树和低割线树甲基化总带数分别为99和106,总甲基化率分别为37.22%和36.43%。高割线树中,4.89%为半甲基化位点,32.33%为全甲基化位点。低割线树中,5.50%为半甲基化位点,30.93%为全甲基化位点(表5)。在2种开割胶树中,全甲基化的比例均比半甲基化的高。因此推断高低割线树中胞嘧啶甲基化的主要模式可能是CpG型。上述结果说明本研究建立的MSAP优化体系可用于橡胶树DNA甲基化多样性分析。
3 讨论
本试验基于MSAP反应体系预实验中了解的各因素的用量或浓度水平范围,结合单因素和正交试验法着重对橡胶树MSAP反应体系中酶切和预扩增及选择性扩增体系进行了优化,成功建立了橡胶树MSAP分析技术体系,即:50 μL双酶切体系中,750 ng橡胶树模板DNA经10 h同步酶切和12 h以上16℃连接后,取4 μL酶连产物进行预扩增,为减少非特异性扩增和保证足够的选择性扩增模板,以稀释20~40倍的预扩增产物用于选扩,选择性扩增产物图谱通过6%聚丙烯酰胺凝胶显示,图谱稳定、带型完整清晰、多态性丰富。
在优化体系的过程中同时发现,MSAP技术体系对DNA模板质量和试样质量的要求都非常高,首先要求样品采集应尽量选用同一发育时期的嫩叶片,采集古铜期大小叶最佳,其原因是橡胶树叶片的形成经历小叶古铜期、大叶古铜期、淡绿期、变色期和稳定期5个阶段,以及DNA甲基化水平易受环境的影响。然后要求提取的DNA既要保证完整性,又要保证无糖、蛋白质和RNA的残留,其理由是提取的DNA在含有多糖、多酚和蛋白质等物质的情况下,甚至有残留的乙醇和TE缓冲液,都容易造成酶切不完全,结果导致较多大片段带型出现[36]。可见制备高质量的基因组DNA是MSAP技术体系分析成败的首要前提。因此本文采用改进的CTAB提取DNA,所提取的DNA的质量和浓度均符合MSAP对DNA模板所要求的标准。
基因组DNA的酶切产物质量是决定MSAP成败的关键。本实验通过在50 μL反应体系中加入EcoRⅠ和HpaⅡ各10 U,或EcoRⅠ20 U和MspⅠ 10 U同步酶切基因组DNA 10 h,实现双酶酶切方法的摸索,保证了酶切反应完全的同时又防止模板DNA降解和节省试验时间;避免了高分子量片段的扩增,使多态性条带集中在100~750 bp,清晰可辨。酶切体系应适宜,体系过小,酶切反应不充分;HapⅡ、MspⅠ宜用5倍的高浓度酶,但酶量不得超过反应总体系的10%。
预扩增和选择性扩增也是影响MSAP的关键步骤。对预扩增正交试验的单因素分析表明,模板用量和引物用量是影响预扩增实验的关键因素。模板用量和引物用量同时或单个过低都会导致预扩增片段分子量范围整体偏上,如1、2、6、8、11、12、15、16号体系,各体系均无法达到预扩增对产物的要求,反之,随着模板用量和引物用量的一同升高,预扩增片段分子量范围整体下移,如体系4和14,两体系均达到预扩增对产物的要求。dNTP浓度和TaqDNA聚合酶浓度对预扩增结果有较大的影响,当反应体系中的dNTPs浓度较高而Mg2+浓度较低时,多余的dNTPs会与Mg2+结合,致使Mg2+浓度降低,进而显著降低Taq酶的酶活力[37],鉴于这一现象,需考虑提高Taq聚合酶浓度与dNTPs竞争Mg2+,避免PCR反应的催化不足,如体系4。而当反应体系的dNTPs浓度和Taq聚合酶浓度都较低且前高后低失衡时,可通过提高Mg2+浓度来满足Taq酶对Mg2+的需求,如体系14,选择性扩增产物分析表明,以本体系实验产物为模板扩增的多态性条带充足,试验结果真实、可靠,且节约试验成本。但当dNTPs浓度过低时,由于dNTPs不能为PCR反应提供充足的能量,为此会降低产物的产量[38],如1、5、9、13号体系,即使引物、Mg2+和Taq聚合酶浓度较高,仍会降低产物的产量,如体系9和13。此外,当dNTPs浓度较低时,DNA模板量过少也会降低PCR产物的量,如体系10。从选择性扩增分析中发现模板稀释倍数和引物用量是影响选扩增实验的关键因素,模板稀释倍数和引物用量同时或单个过低都会导致非特异扩增明显增多,如1、2、6、15、16号体系。dNTPs浓度过低,即使模板稀释倍数和引物用量较高,非特异扩增也会明显增多,如体系9。随着稀释倍数、引物用量和dNTPs浓度的增大,选扩增产物中的非特异扩增明显减少,如3、4、7号体系。
本研究首次采用优化后的橡胶树MSAP反应体系对高割线和低割线橡胶树单株进行甲基化模式分析,即使使用少部分引物组合,两种胶树中的I/Ⅱ/Ⅲ型甲基化模式均能被该方法检测到,并对高割线树和低割线树的全基因组甲基化水平做了对比评估,高低割线树叶片基因组甲基化水平无明显差异。可能由于所用引物组合数量有限和割线变化对叶片DNA甲基化影响较小的原因,未能够检测出两种胶树间可能存在的甲基化模式不同的多态性位点。仍可进一步证实本实验优化后建立的MSAP技术反应体系适于橡胶树DNA甲基化分析,且能区分不明显的甲基化水平差异,为橡胶树对环境的适应性表观遗传研究奠定了基础。
尽管吴春太等于2011年建立的橡胶树MSAP荧光标记技术体系具有检测位点多,带型易辨、多态性高和稳定性好等优点,且操作简便、快捷、高效,但在克隆多态性片段进行测序方面存在一定缺陷。同年,李海林等优化并建立了橡胶树MSAP银染反应体系,其重点是对选择性扩增体系进行优化。针对橡胶树基因组MSAP银染反应体系而论,本研究在选择性扩增体系中优选出的Mg2+、dNTPs、引物浓度分别比李海林等最佳组合选择性扩增体系中确定的量超出66.67%、25.00%、166.67%,而Taq聚合酶用量减半,由于Taq酶在费用项目中所占权重大,实则节约DNA甲基化检测成本[32]。此外,‘热研7-33-97’高低割线2种树的总甲基化平均率为36.83%,比接穗芽片来自不同部位的‘热研8-79’老幼态无性系的均值高出9.72%[33~34],但比繁殖方式不同的‘热研7-33-97’老幼态无性系的均值降低17.38%[31],其原因可能与品种基因型及其树龄的差异有关。除‘热研7-33-97’老幼态无性系全甲基化的平均比例略低于半甲基化的值外,‘热研7-33-97’高低割线树全甲基化的平均比例和‘热研8-79’老幼态无性系的平均比例均比各自半甲基化的值高。综上可知,橡胶树基因组甲基化程度与状态是丰富多变的。
1.Jablonka E,Lamb M J.The changing concept of epigenetics[J].Annals of the New York Academy of Sciences,2002,981:82-96.
2.Chan S W L,Henderson I R,Jacobsen S E.Gardening the genome:DNA methylationArabidopsisthaliana[J].Nature Reviews Genetics,2005,6(5):351-360.
3.Zhang X Y,Yazaki J,Sundaresan A,et al.Genome-wide high-resolution mapping and functional analysis of DNA methylation inArabidopsis[J].Cell,2006,126(6):1189-1201.
4.Zilberman D,Gehring M,Tran R K,et al.Genome-wide analysis ofArabidopsisthalianaDNA methylation uncovers an interdependence between methylation and transcription[J].Nature Genetics,2007,39(1):61-69.
5.Tariq M,Paszkowski J.DNA and histone methylation in plants[J].Trends in Genetics,2004,20(6):244-251.
6.Lu G Y,Wu X M,Chen B Y,et al.Detection of DNA methylation changes during seed germination in rapeseed(Brassicanapus)[J].Chinese Science Bulletin,2006,51(2):182-190.
7.Uthup T K,Ravindran M,Bini K,et al.Divergent DNA methylation patterns associated with abiotic stress inHeveabrasiliensis[J].Molecular Plant,2011,4(6):996-1013.
8.Xiong L Z,Xu C G,Maroof M A S,et al.Patterns of cytosine methylation pattern in an elite rice hybrid and its parental lines,detected by a methylation-sensitive amplification polymorphism technique[J].Molecular and General Genetics MGG,1999,261(3):439-446.
9.郭广平,顾小平,袁金玲,等.不同生理年龄毛竹DNA甲基化的MSAP分析[J].遗传,2011,33(7):794-800.
Guo G P,Gu X P,Yuan J L,et al.Research on the features of DNA methylation in leaves of different chronological ages ofPhyllostachysheterocyclavar.pubescensbased on the method of MSAP[J].Hereditas,2011,33(7):794-800.
10.孙慧敏,顾小平,袁金玲,等.五月季竹开花及复壮过程中DNA甲基化的MSAP分析[J].植物研究,2013,33(6):723-730.
Sun H M,Gu X P,Yuan J L,et al.Analysis on DNA methylation-sensitive amplified polymorphism inPhyllostachysbambusoidesduring flowering and rejuvenation[J].Bulletin of Botanical Research,2013,33(6):723-730.
11.马开峰,张志毅,王斯琪,等.毛白杨MSAP体系优化及DNA甲基化的初步分析[J].东北林业大学学报,2012,40(12):1-7.
Ma K F,Zhang Z Y,Wang S Q,et al.Optimization of MSAP system and preliminary analysis of DNA methylation inPopulustomentosa[J].Journal of Northeast Forestry University,2012,40(12):1-7.
12.Song Y P,Ma K F,Ci D,et al.Sexual dimorphic floral development in dioecious plants revealed by transcriptome,phytohormone,and DNA methylation analysis inPopulustomentosa[J].Plant Molecular Biology,2013,83(6):559-576.
13.Ma K F,Song Y P,Yang X H,et al.Variation in genomic methylation in natural populations of chinese white poplar[J].PLoS One,2013,8(5):e63977.
14.洪舟,施季森,郑仁华,等.杉木亲本自交系及其杂交种DNA甲基化和表观遗传变异[J].分子植物育种,2009,7(3):591-598.
Hong Z,Shi J S,Zheng R H,et al.Epigenetic inheritance and variation of DNA methylation in chinese fir(Cunninghamialanceolata) intraspecific hybrids[J].Molecular Plant Breeding,2009,7(3):591-598.
15.Fu C H,Li L Q,Wu W J,et al.Assessment of genetic and epigenetic variation during long-termTaxuscell culture[J].Plant Cell Reports,2012,31(7):1321-1331.
16.Valledor L,Meijón M,Hasbún R,et al.Variations in DNA methylation,acetylated histone H4,and methylated histone H3 duringPinusradiataneedle maturation in relation to the loss ofinvitroorganogenic capability[J].Journal of Plant Physiology,2010,167(5):351-357.
17.Fraga M F,Rodríguez R,Caal M J.Genomic DNA methylation-demethylation during aging and reinvigoration ofPinusradiata[J].Tree Physiology,2002,22(11):813-816.
18.Fraga M F,Caal M,Rodríguez R.Phase-change related epigenetic and physiological changes inPinusradiataD.Don[J].Planta,2002,215(4):672-678.
19.Valledor L,Pascual J,Meijón M,et al.Conserved epigenetic mechanisms could play a key role in regulation of photosynthesis and development-related genes during needle development ofPinusradiata[J].PLoS One,2015,10(5):e0126405.
20.魏华丽,吴涛,杨文华,等.落叶松体细胞胚胎发生过程中DNA甲基化模式变化分析[J].东北林业大学学报,2011,39(2):33-37.
Wei H L,Wu T,Yang W H,et al.DNA methylation pattern changes during somatic embryogenesis ofLarix[J].Journal of Northeast Forestry University,2011,39(2):33-37.
21.李爱,刘超,韩春乐,等.落叶松优势杂交子代与亲本间基因组甲基化变异研究[J].南开大学学报:自然科学版,2012,45(5):65-71.
Li A,Liu C,Han CL,et al.Variation in cytosine methylation patterns between reciprocal hybrids and their patental line inLarix[J].Acta Scientiarum Naturalium Universitatis Nankaiensis,2012,45(5):65-71.
22.Teyssier C,Maury S,Beaufour M,et al.In search of markers for somatic embryo maturation in hybrid larch(Larix×eurolepis):global DNA methylation and proteomic analyses[J].Physiologia Plantarum,2014,150(2):271-291.
23.黎明,翟晓巧,范国强,等.土霉素对豫杂一号泡桐丛枝病幼苗形态和DNA甲基化水平的影响[J].林业科学,2008,44(9):152-156.
Li M,Zhai X Q,Fan G Q,et al.Effect of oxytetracycline on the morphology of seedling with witches’ broom and DNA methylation level ofPaulowniatomentosa×P.fortunei[J].Scientia Silvae Sinicae,2008,44(9):152-156.
24.Cao X B,Fan G Q,Zhao Z L,et al.Morphological changes ofPaulowniaseedlings infected phytoplasmas reveal the genes associated with witches’ broom through AFLP and MSAP[J].PLoS One,2014,9(11):e112533.
25.Cao X B,Fan G Q,Deng M J,et al.Identification of genes related toPaulowniawitches’ broom by AFLP and MSAP[J].International Journal of Molecular Sciences,2014,15(8):14669-14683.
26.曹喜兵,赵振利,范国强,等.甲基磺酸甲酯对毛泡桐丛枝病苗DNA甲基化的影响[J].林业科学,2014,50(3):99-108.
Cao X B,Zhao Z L,Fan G Q,et al.Effect of methyl methanesulphonate on DNA methylation of witches’ broom seedlings ofPaulowniatomentosa[J].Scientia Silvae Sinicae,2014,50(3):99-108.
27.Ko J H,Chow K S,Han K H.Transcriptome analysis reveals novel features of the molecular events occurring in the laticifers ofHeveabrasiliensis(para rubber tree)[J].Plant Molecular Biology,2003,53(4):479-492.
28.杨署光,于俊红,史敏晶,等.割胶、机械伤害和外源茉莉酸对橡胶树乳管细胞防卫蛋白基因表达的影响[J].热带作物学报,2008,29(5):535-540.
Yang S G,Yu J H,Shi M J,et al.Effects of tapping,mechanical wounding and exogenous jasmonates on the expression of genes encoding for defense proteins in the latex ofHeveabrasiliensis[J].Chinese Journal of Tropical Crops,2008,29(5):535-540.
29.吴春太,黎瑜,代龙军,等.橡胶树甾醇14α-去甲基化酶基因(HbCYP51)的克隆及机械损伤和外源激素对其表达的影响[J].农业生物技术学报,2014,22(12):1525-1535.
Wu C T,Li Yu,Dai L J,et al.Cloning of sterol 14α-demethylase gene(HbCYP51) and effects of mechanical damage and exogenous phytohormones on its mRNA expression inHeveabrasiliensis[J].农业生物技术学报,2014,22(12):1525-1535.
30.Sookmark U,Pujade-renaud V,Chrestin H,et al.Characterization of polypeptides accumulated in the latex cytosol of rubber trees affected by the tapping panel dryness syndrome[J].Plant & Cell Physiology,2002,43(11):1323-1333.
31.吴春太,李维国,黄华孙.橡胶树MSAP反应体系的建立及其对无性系DNA的甲基化分析[J].广东农业科学,2011,38(9):141-144.
Wu C T,Li W G,Huang H S.Establishment of MSAP analysis system for rubber tree and its preliminary application in juvenile-type clone[J].Guangdong Agricultural Sciences,2011,38(9):141-144.
32.李海林,李维国,陈组兴,等.橡胶树MSAP技术体系的优化与建立[J].热带作物学报,2011,32(4):644-647.
Li H L,Li W G,Chen Z X,et al.Optimization and establishment of MSAP reaction system forHeveabrasiliensis[J].Chinese Journal of Tropical Crops,2011,32(4):644-647.
33.李海林,吴春太,李维国.巴西橡胶树DNA甲基化的MSAP分析[J].分子植物育种,2011,9(1):69-73.
Li H L,Wu C T,Li W G.Analysis on DNA methylation of rubber by methylation sensitive amplification polymorphism[J].Molecular Plant Breeding,2011,9(1):69-73.
34.李海林,李维国,杨朝渠.橡胶树幼态与老态无性系间的MSAP分析[J].热带作物学报,2014,35(4):617-621.
Li H L,Li W G,Yang Z Q.MSAP analysis of juvenile clones and mature clones ofHeveabrasiliensis[J].Chinese Journal of Tropical Crops,2014,35(4):617-621.
35.安泽伟,黄华孙.一种提取橡胶树叶中总DNA的方法[J].植物生理学通讯,2005,41(4):513-515.
An Z W,Huang H S.A method for genomic DNA extraction from leaves of rubber tree(HeveabrasiliensisMuell.Arg.)[J].Plant Physiology Communications,2005,41(4):513-515.
36.曹喜兵,赵改丽,范国强.泡桐MSAP体系建立及引物筛选[J].河南农业大学学报,2012,46(5):535-541.
Cao X B,Zhao G L,Fan G Q.Establishment ofPaulowniaMSAP reaction systems and primers screening[J].Journal of Henan Agricultural University,2012,46(5):535-541.
37.高寰,张铮,周婷,等.三叶木通MSAP反应体系的优化及引物筛选[J].中草药,2012,43(3):572-576.
Gao H,Zhang Z,Zhou T,et al.Optimization of MSAP reaction system forAkebiatrifoliataand primers screening[J].Chinese Traditional and Herbal Drugs,2012,43(3):572-576.
38.周贺,徐雯,李雅娟,等.泥鳅MSAP技术反应体系的建立及优化[J].大连海洋大学学报,2014,29(6):550-555.
Zhou H,Xu W,Li Y J,et al.Establishment and optimization of MSAP reaction system for loachMisgurnusanguillicaudatus[J].Journal of Dalian Ocean University,2014,29(6):550-555.
National Natural Science Foundation of China(No.31270713);Fundamental Research Funds for Rubber Research Institute,CATAS(No.1630022012008)
introduction:WU Chun-Tai(1972—),male,Ph.D.,associate researcher, experimental area engaged primarily in study on the characteristic of producing-rubber ofHeveabrasiliensis.
date:2016-03-25
OptimizationofMSAPAnalysisSystemforHeveabrasiliensisandItsApplicationinPlantofDifferentHeightsofOpeningforTapping
WU Chun-Tai1BAN Shuo1,2LI Yu1ZENG Ri-Zhong1*
(1.Rubber Research Institute,Chinese Academy of Tropical Agriculture Sciences/Key Laboratory of Biology and Genetic Resources of Rubber Tree,Ministry of Agriculture,the People’s Republican of China/State Center for Rubber Breeding/Hainan Provincial Key Laboratory of Physiology for Tropical Crops,Danzhou 571737;2.College of Plant Science and Technology,Huazhong Agricultural University,Hubei,Wuhan 430070)
Using the tender leaves ofHeveabrasiliensisvariety ‘CATAS 7-33-97’ as materials, several key influencing factors in leaf genomic DNA restriction enzyme digestion, pre-amplification and selective amplification system which affected the system quality of MSAP were optimized through the single factor and orthogonal design method in order to obtain the optimal MSAP reaction system of rubber tree. The optimized reaction system of MSAP was used for difference analysis of DNA methylation in two kinds ofH.brasiliensisof high and low tapping cuts. The results showed that 750 ng genomic DNA in a reaction volume of 50 μL could be fully digested byEcoRⅠ 20 U,HpaⅡ 20 U orMspⅠ 10 U at 37℃ for 10 h. MSAP-PCR pre-amplification was performed in 20 μL of reaction volume with the following mix: ligation products 4 μL, MgCl2(25 mmol·L-1)0.15 μL, dNTPs(2.5 mmol·L-1)0.1 μL,Taqpolymerase(5 U·μL-1) 0.1 μL, 10×PCR Buffer 2 μL, primer E-00/HM-00(10 μmol·L-1) 0.3 μL, respectively. The 20 μL selective reaction mixture contained 20 times diluted amplification products 2 μL, MgCl2(25 mmol·L-1) 0.1 μL, dNTPs(2.5 mmol·L-1) 0.125 μL,Taqpolymerase(5 U·μL-1) 0.1 μL, 10×PCR Buffer 2 μL, primer E+3/HM+3(10 μmol·L-1) 0.4 μL respectively. The total methylation rates of two samples were 37.22% and 36.43%, respectively. The full methylation rates were more than the hemi-methylation rates inH.brasiliensisof different tapping cuts. Therefore, CpG methylation may represent the major form of DNA methylation in rubber tree. Our data indicated that this MSAP reaction system with high stability and reliablity and good reproducibility have provided the foundation for different degree tapping stress of rubber tree associated research with the MSAP technology.
Heveabrasiliensisof high and low tapping cuts;DNA methylation;methylation sensitive amplified polymorphism(MSAP);system optimization
国家自然科学基金资助项目(31270713);橡胶研究所基本科研业务费资助项目(1630022012008)
吴春太(1972—),男,博士,副研究员,主要从事橡胶树产胶特性研究。
* 通信作者:E-mail:hnzrz@aliyun.com
2016-03-25
* Corresponding author:E-mail:hnzrz@aliyun.com
S794.1
A
10.7525/j.issn.1673-5102.2016.06.009
