普通小麦TaNAC1基因的表达及在拟南芥中的遗传转化
2016-11-10艾可筠侯和胜佟少明
艾可筠 朱 尧 侯和胜 佟少明*
(1.辽宁师范大学生命科学学院,大连 116081; 2.辽宁省植物生物工程重点实验室,大连 116081)
普通小麦TaNAC1基因的表达及在拟南芥中的遗传转化
艾可筠1,2朱 尧1,2侯和胜1,2佟少明1,2*
(1.辽宁师范大学生命科学学院,大连 116081;2.辽宁省植物生物工程重点实验室,大连 116081)
NAC类转录因子是植物特有的转录因子家族,在调节植物生长发育及逆境胁迫应答反应中起着重要作用。本文从普通小麦幼叶中获得了一个编码NAC结构域的转录因子基因,命名为TaNAC1;氨基酸序列分析表明,TaNAC1具有典型的NAC类转录因子所具有的五个亚结构域,隶属于NAC类转录因子的ATAF亚类;亚细胞定位实验表明,TaNAC1蛋白在细胞核内表达;转录水平上,TaNAC1基因的表达受到PEG、ABA、低温及高盐等非生物胁迫条件的诱导;将TaNAC1转化拟南芥后,与野生型比较发现,TaNAC1基因的过量表达会使转基因植株出现叶片发育畸形且生长缓慢,植株矮化及茎部融合等表型,表明TaNAC1基因可能在参与小麦叶片及茎的发育中起着重要的调控作用。
普通小麦;TaNAC1基因;拟南芥;表型
NAC类转录因子是植物特有的转录因子基因家族的重要成员之一,首先在矮牵牛中发现NAM基因编码蛋白的N端存在着一个保守的结构域,称为NAM结构域,随后在拟南芥中也发现存在着四个基因(ATAF1/ATAF2和CUC1/CUC2)编码蛋白的N端存在着与NAM高度相似的结构域[1~2]。目前,已经把编码具有与矮牵牛的NAM、拟南芥的ATAF1/2和CUC1/2基因相同结构域的基因统称为NAC基因。序列分析表明,所有NAC蛋白的N端都存在一个保守的NAC结构域,由A、B、C、D、E五个亚结构域组成,也有些NAC蛋白包含两个串联的NAC结构域[3];NAC作为转录因子,可以结合在烟草花叶病毒的35S启动子区域[4~7]及ERD1基因的启动子区域[8]。一般情况下,NAC蛋白结构域能促使其形成同源或异源的二聚体,但同时会也受到C端转录激活域的影响[3,6~7]。NAC蛋白的C端氨基酸序列变化多样,不包含任何已知蛋白的功能结构域,但具有转录激活活性的特征,即一些简单重复的氨基酸以及富含丝氨酸、苏氨酸、脯氨酸、谷氨酸等氨基酸组成的区域[9~10]。
NAC转录因子参与植物不同组织及器官的生长发育等多个进程的调控。在茎的发育过程中,NAC基因的突变会导致矮牵牛植株缺少芽顶端分生组织,并在幼苗期逐渐死亡,少部分形成芽顶端分生组织的突变体在幼苗期也会出现子叶融合,成苗期花发育异常的现象[2],同时NAC蛋白还参与植物细胞壁次生壁的形成及导管的分化,在水生植物向陆生演化过程中对茎杆的支持,以及输导组织的形成中都发挥了重要的作用[11~13];在根的发育中,拟南芥的NAC1基因作为TIR1下游的靶基因,可以诱导侧根的形成[7];在叶片发育过程中,拟南芥中有6个NAC类转录因子协同EIN3蛋白参与由EIN2蛋白介导的叶片衰老过程的调控[14],47个大麦的NAC基因中有15个参与大麦旗叶衰老进程的调控[15],小麦的TtNAM基因能够延迟叶片衰老,提高麦粒中的蛋白质及铁、锌等微量元素的含量[16];除此之外,NAC类转录因子还参与植物激素代谢,如番茄的SlNAC4基因的沉默会通过抑制乙烯的生物合成来延迟果实的成熟[17],大豆的NAC基因SHAT1-5参与大豆果实开裂的分子调控[18];另外,很多证据表明NAC类的转录因子还参与多方面的非生物胁迫[19]及植物免疫[20~21]等生理过程的调节,如干旱[8,22]、高盐[23]、高温[24]、水涝[25]、氧化[26]及低温[27]等。
普通小麦(TriticumaestivumL.)是我国主要的农作物,其功能基因的挖掘和注释一直是小麦分子生物学研究的重要内容之一,尤其是在小麦基因组测序完成后,为基因功能的注释提供了便利条件,根据预测,小麦的基因组中存在着134个NAC类型的转录因子(http://planttfdb.cbi.pku.edu.cn/index.php),本研究利用电子克隆等手段,从小麦幼叶中分离了受到PEG、ABA、低温及高盐诱导的TaNAC1基因,该基因编码的蛋白定位于细胞核内,超表达TaNAC1基因的拟南芥植株的茎及叶片的生长发育出现了异常,表明该基因可能在小麦茎及叶片的发育过程中起到调节作用。
1 材料和方法
1.1 材料
普通小麦(T.aestivumL.)种子采用5%的次氯酸钠消毒10 min,用蒸馏水冲洗4遍,置于4℃冰箱中暗培养1周后,将种子移入培养瓶,每瓶5粒左右,置于培养室中(温度25℃,相对湿度60%~70%,光强100 μmol·m-2·s-1)培养。当幼苗生长至两叶一心期时进行相应的处理,整个处理参照Mukhopadhyay[28]的方法在黑暗中进行。
高盐处理:在培养瓶中加入浓度为1 mol·L-1的NaCl溶液至终浓度为250 mmol·L-1。
PEG处理:在培养瓶中加入50% PEG溶液至终浓度为25%。
ABA处理:把ABA溶解在DMSO中制成10 mmol·L-1贮存液,然后加入到培养瓶中至终浓度为100 μmol·L-1。
在加入上述溶液以后,分别在0.5、1、2、12,24 h时,取小麦幼苗的叶片,用吸水纸吸干残余溶液后迅速置于液氮中,于-80℃冰箱中保存,待用;同时将生长在蒸馏水中的小麦幼苗作为对照组,在上述相应的时间取样。
低温处理是将小麦的幼苗置于4℃光照培养箱中,处理的时间和取材方法同上,对照组为在正常培养室的温度(25℃和光强为100 μmol·m-2·s-1)。
1.2 小麦幼叶总RNA提取及cDNA的合成
利用Trizol试剂盒(TaKaRa,大连)提取小麦幼叶的总RNA,经DNaseI处理后,用NanoDrop确定RNA的浓度和纯度,稀释成相同浓度后,通过1%的琼脂糖凝胶电泳确定RNA的完整性。取1 μg纯化后的RNA,按照PrimeScript One Step RT-PCR Kit试剂盒(TaKaRa,大连)说明书中的操作程序进行第1链cDNA的合成,-20℃保存备用。
1.3 基因的克隆及序列分析
以NAM(Accession no. X92204)和CUC2(Accession no. AB002560)为查询序列,在GenBank的nr数库中采用Blastn程序(http://www.ncbi.nlm.nih.gov/BLAST/),物种限定为T.aestivum,搜索与之高度相似的小麦的EST序列,将返回的结果拼接,以拼接序列为模板设计引物,上下游的引物序列分别为CGACGGAGAAGCAGGAAGC(5′-3′)及AGTCCTCTTCTTACCCGACTGC(5′-3′)。
以小麦幼叶的cDNA为模板进行PCR扩增,将扩增产物回收纯化后,与pMD-19T载体(TaKaRa,大连)连接,转化E.coliDH5α感受态细胞,经酶切和PCR鉴定后,送至上海生工生物工程有限公司进行双向测序;测序结果利用Sequencher软件进行拼接,并通过NCBI的ORF Finder程序进行开放阅读框及其编码氨基酸序列的预测;采用Interpro和predictNLS程序分别对该基因编码的蛋白的结构域进行搜索和核定位信号的预测。
1.4 氨基酸序列比对及进化树构建
以克隆到的基因编码的蛋白序列为查询序列,登录NCBI数据库,采用Blastp程序查找与其蛋白序列高度相似的序列,对结果进行筛选后,相似序列以FASTA格式下载存于本地机上。多序列比对采用ClustalX程序进行,并利用MEGA5.1进化分析软件,采用最大似然法(Maximum Likelihood,ML)构建系统发生树[29]。
1.5 胁迫条件下的基因转录水平的表达
利用实时荧光定量PCR检测不同胁迫处理条件下的TaNAC1基因在各个阶段的表达情况。以克隆到的目的基因序列为模板设计实时定量引物,上下游序列分别为CGCGAATTCACTACCCGATT(5′-3′)及ACTGCCAAAGGAGGAGGTGC(5′-3′);以小麦的Tubulin基因为内标设计实时定量引物,上下游引物序列分别为TGAGGGTCCTACACTTCG(5′-3′)及TCCTCTGGATTCCGTGCC(5′-3′)。实时定量PCR反应采用SYBR® Premix ExTaqTM试剂盒(TaKaRa,大连)在TP800实时定量PCR仪中进行。
以稀释10倍的不同胁迫条件下的cDNA为模板,反应体积为25 μL,包括2×SYBR Premix Ex TaqTM12.5 μL,正反向引物(10 μmol·L-1)各0.5 μL,cDNA 1.0 μL,ddH2O 10.5 μL,每个反应重复3次。反应程序为:95℃ 30 s;95℃ 5 s,60℃ 30 s,40个循环。实验结束后根据荧光实时定量PCR的分析曲线,自动设定荧光域值,分别计算出目标基因和内标基因各处理3次重复的Ct值的平均值,通过计算不同的2-ΔCt值,来确定目标基因在不同处理条件下各个阶段的转录水平的表达情况。
1.6 目标蛋白的细胞定位
将目标基因的5′及3′端分别引入EcoRⅠ及HindⅢ酶切位点,插入到pEGAD载体中,经含有60 mg·L-1氨苄青霉素的LB平板筛选后,挑取阳性克隆,并经过酶切及测序验证,获得经CaMV35S启动子驱动的绿色荧光蛋白(GFP)与目标蛋白的融合蛋白的表达载体。采用基因枪轰击洋葱表皮细胞,将表达载体导入其中,并置于22℃培养箱中培养14~16 h后,利用激光共聚焦显微镜(Confocal Laser Scanning Fluorescence Microscope,Carl Zeiss)观察荧光蛋白在洋葱表皮细胞中的位置。
1.7 转基因拟南芥的表型鉴定
将目标基因插入到pCAMBIA 1300载体的多克隆位点中构建双元表达载体,利用农杆菌介导方法转化野生型拟南芥(Columbia型),转化程序参照Bechtold等[30]进行,收获T0代种子后,在选择培养基上(含30 mg·L-1的潮霉素)进行筛选,选择T2代为3∶1分离比的转基因植株进行T3代筛选,同时以T3代转基因植株和野生型的叶片为材料,采用Trizol法提取RNA,反转录成cDNA后,以拟南芥的Actin及TaNAC1基因为模板设计引物,进行PCR扩增,选择阳性扩增结果的转基因植株进行表型鉴定。
将经过筛选的阳性株系及野生型的种子消毒并用无菌水清洗后,将种子平铺在MS培养基上,4℃下春化5天后,移入光照箱中(22℃恒温,24 h光照,光强为30~40 μmol·m-2·s-1)培养10 h后移入营养土中,每天观察并照相记录转基因植株各株系及野生型的叶片、茎以及种荚的发育情况。
2 结果与分析
2.1 小麦TaNAC1基因的克隆及序列分析
采用表1中的引物,以小麦幼叶的cDNA为模板,经过PCR扩增获得一个长为900 bp的条带,该序列的ORF长度为858 bp,预测编码285个氨基酸,分子量31.9 KDa,基因组PCR扩增的结果显示此基因不含有内含子;InterPro程序分析在该序列编码蛋白的N端18-168氨基酸残基处存在着NAC结构域,并具有A、B、C、D和E五个亚结构域(图1),predictNLS程序预测该蛋白序列包含着两个保守的核定位信号序列(图1)。将该基因命名为TaNAC1后提交到NCBI数据库中,返回的登录号为KR814823。
2.2小麦TaNAC1氨基酸序列的多序列比对及进化分析
多序列比对的结果表明,除了麦属植物外,TaNAC1蛋白的氨基酸序列与其它物种的相似性相对较高,如与Brachypodiumdistachyon(XP_003557366.1)氨基酸序列的一致性达到76%,与Zeamays(001141629.2),Sorghumbicolor(XP_002459566.1)和Oryzasativa(NP_001059213.1),氨基酸序列的一致性分别为70%,69%和68%。(图1),进一步说明NAC类的转录因子是植物中高度保守的蛋白质家族之一。

图1 TaNAC1蛋白的氨基酸序列以及与其它NAC蛋白的氨基酸序列的比对分析 “*”代表相同的氨基酸残基;“:”及“.”代表相似的氨基酸残基 A,B,C,D及E代表NAC结构域所包含的五个保守的亚结构域;NLS1,NLS2为预测到的两个核定位信号Fig.1 Multiple alignments of TaNAC1 and other NACs “*” represents the identical amino acid residues; “:” and “.” represent similar amino acid residues; A,B,C,D and E represent five conservative sub-domains of NAC respectively; NLS1 and NLS2 are two predicted nuclear localization signals.

图2 TaNAC1蛋白与其它物种NAC蛋白的聚类分析 图中黑色箭头表示TaNAC1蛋白所在位置Fig.2 The phylogenetic analysis of TaNAC1 and other NACs The black arrow indicates the location of TaNAC1
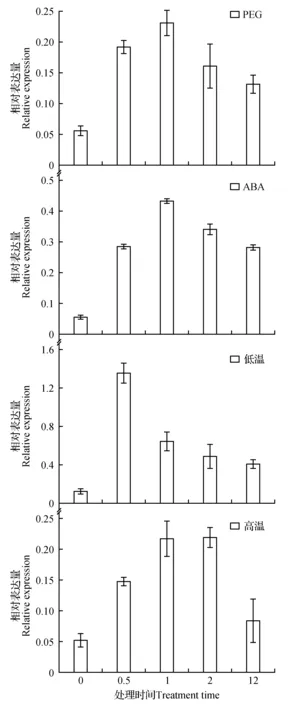
图3 TaNAC1基因在不同胁迫及植物激素ABA处理下,不同时间(0.5、1、2和12 h)的表达变化模式Fig.3 The transcription levels of TaNAC1 at different time points (0.5,1,2 and 12 h)by the treatment of various stresses and ABA
将搜索到的所有与TaNAC1相似的蛋白序列进行进化树的构建(物种名称及登录号见图2),结果表明,单子叶植物,双子叶植物各聚成单独的一枝,所有不同分支的NAC蛋白都是单系同源的,并不存在物种间的水平基因转移,说明此类NAC蛋白在一定程度上反映了物种进化的历史。导入到ClustalX软件中进行多序列比对,利用MEGA5.1进化分析软件采用最大似然法(Maximum Likelihood,ML)构建系统发生树。
2.3 TaNAC1基因在胁迫条件下的表达分析
采用实时定量PCR方法,检测小麦幼叶中TaNAC1基因在不同胁迫处理条件下的转录水平的表达变化。结果表明:PEG(25%)、ABA(100 μmol·L-1)和NaCl(250 mmol·L-1)的处理都使TaNAC1基因的转录在1 h内就达到最高,然后逐渐下降(图3:A,B,D);4℃低温处理使TaNAC1基因的转录在0.5 h后就迅速达到最高,然后下降(图3:C)。
2.4 TaNAC1基因的亚细胞定位分析
为验证TaNAC1蛋白在细胞中的作用部位,将TaNAC1基因插入35S启动子和pEGAD基因之间形成GFP-TaNAC1的融合基因,未插入TaNAC1基因的pEGAD空载体用作对照,分别用基因枪转化洋葱表皮细胞。结果显示GFP-TaNAC1融合蛋白在细胞核中表达(图4:A,B,C),而对照的GFP蛋白均匀分布在洋葱表皮细胞的每个部位,证明了TaNAC1基因编码的蛋白是核蛋白。

图4 GFP-TaNAC1融合蛋白在洋葱表皮细胞中定位分析 A.融合蛋白在荧光场下的照片;B.融合蛋白在光场下的照片;C.光场和荧光场叠加后的照片;D.对照GFP蛋白在荧光激发时的照片Fig.4 Location of GFP-TaNAC1 in the onion epidermis cell A.Image of GFP-TaNAC1 in fluorescence field; B.Image of GFP-TaNAC1 in light field; C.Image of GFP-TaNAC1 in fluorescence and light field; D.Image of GFP(control) in fluorescence field
2.5 转TaNAC1基因的拟南芥的表型鉴定
将T3代转基因植株各株系及野生型的RNA,反转录成cDNA后,采用拟南芥的Actin引物及TaNAC1的特异性引物进行转录水平的表达检测,如图5结果所示,所有的转基因植株都含有目的基因,表明外源基因已经整合到基因组中并在拟南芥中实现了表达。
在获得TaNAC1的转基因拟南芥后,对转基因植物的叶片发育情况和野生型(图6)进行了对比观察。在种子萌发期,转基因植株和野生型并没有差别;在子叶形成期,野生型的子叶对生分布(图6:A箭头所指处),而部分转基因株系的两片子叶发生融合(图6:A2-A3箭头所指处),根系短小, 数目少,发生子叶融合的转基因植株一般不能正常发育到成株,在幼苗期就会逐渐死亡;在莲座叶形成后,野生型莲座叶的叶形由卵形,边缘整齐,逐渐转为椭圆形(图6:B1),而大部分转基因叶片发育畸形,主要表现为裂叶及叶片融合(图6:B2-B3,C1-C3,D1-D3)。

图5 转基因拟南芥植株的分子鉴定 1~10.不同转基因植株的PCR扩增结果;11.野生型;12.阳性对照;13.水对照Fig.5 Molecular identification of transgenic Arabidopsis 1-10. The PCR amplification results of different transgenic plants; 11. Wild type; 12. Positive control; 13. ddH2O

图6 小麦TaNAC1的转基因拟南芥子叶和叶片发育的表型 A1-A3.转基因T1代植株的子叶;B1.野生型叶片发育表型;B2,B3,C1-C3.转基因T3代植株的叶片表型;D1-D3.发育畸形的叶片表型Fig.6 Phenotype of cotyledon and leaf development in transgenic Arabidopsis A1-A3. The cotyledon of T1 transgenic lines; B1. The leaf phenotype of wild type; B2,B3,C1-C3. The leaf phenotype of T3 transgenic lines; D1-D3. The phenotype of abnormal leaves

图7 小麦TaNAC1的转基因拟南芥的种荚发育的表型 A.野生型的茎及种荚表型;B~D.代表不同的转基因株系的茎及种荚发育的表型Fig.7 Phenotype of pod development in transgenic Arabidopsis A.The phenotype of stem and pod in wild type; B-D. The phenotype of stem and pod in transgenic Arabidopsis
转基因植株抽薹进入生殖生长后,与野生型(图7:A)相比,部分转基因株系的茎矮化,开花数量不一,种荚无序分布,集中在花茎的顶端(图7:B)或下部(图7:C),部分转基因植物的茎部发生融合(图7:D)。
3 讨论
本研究从普通小麦中克隆了一个NAC类转录因子基因TaNAC1,该基因编码蛋白属于NAC家族基因的ATAF类。在基因的分子进化分析中,我们采用了最大似然法(ML)、最大简约法(MP)以及邻接法(NJ)对所选取的序列进行了进化树的构建,3种算法都得到了相同的进化树的拓扑结构,并与传统的物种分类及进化的结果保持一致,说明NAC类转录因子可以作为基因进化研究的侯选基因之一。分析结果也显示,与此类NAC蛋白亲缘关系最远的为裸子植物Piceasitchensis,并没有在已经完成基因组测序的藻类等水生植物中找到编码NAC结构域的基因,因此NAC蛋白作为植物特有的转录因子,还缺少从水生植物到陆生植物进行转移的证据。另外,植物NAC蛋白在不同物种中序列的相似程度较高,与TaNAC1蛋白距离最远的S.moellendorffii,其氨基酸序列的相似性也已经达到41%;对于NAC蛋白的结构域,尤其是DNA结合域在不同物种间的相似程度更高,其保守性也确保了其结构的稳定性,也暗示了其在植物生长发育及逆境胁迫中的相似功能。
TaNAC1基因在转录水平的表达受到PEG、高盐、低温及植物激素ABA的诱导,很多研究都表明,NAC家族的AFAF类基因参与广泛的逆境胁迫应答反应[31~33],是植物逆境胁迫的重要调节因子之一。在拟南芥中,ANAC019,ANAC055及ANAC072基因在转录水平的表达受到干旱、高盐以及植物激素ABA及MeJA的诱导[8],以及其编码的NAC结构域的RD26基因也受到干旱和ABA的诱导[34];一些与TaNAC1蛋白在序列上相似程度很高的其它物种的NAC蛋白,如O.sativaIndica(ACX71077.1)、S.bicolor(AGG40203.1)及H.vulgare(AEG21060.1),其转录表达都不同程度地受到各种胁迫因子的诱导,尤其是GRAB1(CAA09371)蛋白与TaNAC1相似性达到93%,可以通过与小麦的矮化双生病毒复制蛋白RepA相互作用来抑制双生病毒的复制[21],但由于在拟南芥中超表达TaNAC1基因后,会造成转基因植物的茎、叶等器官发育异常,甚至产生致死的效应,故无法对其进行抗性分析,因此对于TaNAC1基因参与的逆境调控机理还有待于进一步研究。
超表达TaNAC1基因的转基因植株,在叶片发育初期出现了子叶及幼叶融合、幼叶黄化,后期部分叶片变大,短叶柄及叶片发育畸形,并出现开花数目减少,植株矮化等表型。与Huh[35]等人报道相一致,拟南芥的ATAF2基因超表达后也导致了转基因植株的叶片黄化及植株矮化,原因可能是ATAF2基因激活了NIT基因(编码催化IAN转变IAA和IAM的关键酶)的表达,使转基因植株生长素的合成发生变化,从而影响植株的发育;超表达RD26基因的转基因拟南芥也出现开花数目减少、短叶柄及叶片变大等表型[35];但是,拟南芥的NAC类基因的另一成员ANAC102受到低温、干旱、高盐及低氧的诱导,过表达或缺失该基因对拟南芥正常的生长和发育均没有影响,但是在低氧条件下,该基因的缺失表达会显著降低种子的发芽率[36]。在本研究中,尽管TaNAC1转基因株系都不同程度出现了叶片、茎等组织及器官发育异常的情况,但由于单子叶及双子叶植物在叶片、茎等器官形态建成上存在本质的差别,因此TaNAC1基因是否真正参与小麦叶片及茎等器官的形态建成还需要进一步得到证实。
综上所述,本研究从普通小麦幼叶中克隆了在细胞核中表达的NAC类转录因子TaNAC1基因,该基因转录水平的表达受到PEG、ABA、低温及高盐等非生物胁迫条件的诱导;在拟南芥中实现遗传转化后,转基因植株出现叶片发育畸形且生长缓慢,植株矮化及茎部融合等表型,表明该基因可能在参与小麦叶片及茎的发育中起着重要的调控作用。
1.Aida M,Ishida T,Tasaka M.Shoot apical meristem and cotyledon formation duringArabidopsisembryogenesis:interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes[J].Development,1999,126(8):1563-1570.
2.Souer E,van Houwelingen A,Kloos D,et al.The no apical meristem gene ofPetuniais required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell,1996,85(2):159-170.
3.Olsen A N,Ernst H A,LEGGIO L L,et al.NAC transcription factors:structurally distinct,functionally diverse[J].Trends in Plant Science,2005,10(2):79-87.
4.Duval M,Hsieh T F,Kim S Y,et al.Molecular characterization ofAtNAM:a member of theArabidopsisNAC domain superfamily[J].Plant Molecular Biology,2002,50(2):237-248.
5.Ernst H A,Olsen A N,Skriver K,et al.Structure of the conserved domain of ANAC,a member of the NAC family of transcription factors[J].EMBO Reports,2004,5(3):297-303.
6.Hegedus D,Yu M,Baldwin D,et al.Molecular characterization ofBrassicanapusNAC domain transcriptional activators induced in response to biotic and abiotic stress[J].Plant Molecular Biology,2003,53(3):383-397.
7.Xie Q,Frugis G,Colgan D,et al.ArabidopsisNAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J].Genes & Development,2000,14(23):3024-3036.
8.Tran L S P,Nakashima K,Sakuma Y,et al.Isolation and functional analysis ofArabidopsisstress-inducible NAC transcription factors that bind to a drought-responsive cis-element in the early responsive to dehydration stress 1 promoter[J].The Plant Cell,2004,16(9):2481-2498.
9.Ren T,Qu F,Morris T J.HRTgene function requires interaction between a NAC protein and viral capsid protein to confer resistance to turnip crinkle virus[J].The Plant Cell,2000,12(10):1917-1926.
10.Robertson M.Two transcription factors are negative regulators of gibberellin response in the HvSPY-signaling pathway in barley aleurone[J].Plant Physiology,2004,136(1):2747-2761.
11.Xu B,Ohtani M,Yamaguchi M,et al.Contribution of NAC transcription factors to plant adaptation to land[J].Science,2014,343(6178):1505-1508.
12.Nakano Y,Yamaguchi M,Endo H,et al.NAC-MYB-based transcriptional regulation of secondary cell wall biosynthesis in land plants[J].Frontiers in Plant Science,2015,6:288.
13.Endo H,Yamaguchi M,Tamura T,et al.Multiple classes of transcription factors regulate the expression of VASCULAR-RELATED NAC-DOMAIN7,a master switch of xylem vessel differentiation[J].Plant and Cell Physiology,2015,56(2):242-254.
14.Kim H J,Hong S H,Kim Y W,et al.Gene regulatory cascade of senescence-associated NAC transcription factors activated by ETHYLENE-INSENSITIVE2-mediated leaf senescence signalling inArabidopsis[J].Journal of Experimental Botany,2014,65(14):4023-4036.
15.Christiansen M W,Gregersen P L.Members of the barley NAC transcription factor gene family show differential co-regulation with senescence-associated genes during senescence of flag leaves[J].Journal of Experimental Botany,2014,65(14):4009-4022.
16.Uauy C,Distelfeld A,Fahima T,et al.A NAC Gene regulating senescence improves grain protein,zinc,and iron content in wheat[J].Science,2006,314(5803):1298-12301.
17.Zhu M K,Chen G P,Zhou S,et al.A new tomato NAC(NAM/ATAF1/2/CUC2) transcription factor,SlNAC4,functions as a positive regulator of fruit ripening and carotenoid accumulation[J].Plant and Cell Physiology,2014,55(1):119-135.
18.Dong Y,Yang X,Liu J,et al.Pod shattering resistance associated with domestication is mediated by a NAC gene in soybean[J].Nature Communications,2014,5:3352.
19.Mao X G,Zhang H Y,Qian X Y,et al.TaNAC2,a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances inArabidopsis[J].Journal of Experimental Botany,2012,63(8):2933-2946.
20.Donze T,Qu F,Twigg P,et al.Turnip crinkle virus coat protein inhibits the basal immune response to virus invasion inArabidopsisby binding to the NAC transcription factor TIP[J].Virology,2014,449:207-214.
21.Xie Q,Sanz-burgos A P,Guo H S,et al.GRAB proteins,novel members of the NAC domain family,isolated by their interaction with a geminivirus protein[J].Plant Molecular Biology,1999,39(4):647-656.
22.Sakuraba Y,Kim Y S,Han S H,et al.TheArabidopsistranscription factor NAC016 promotes drought stress responses by repressingAREB1 transcription through a trifurcate feed-forward regulatory loop involving NAP[J].The Plant Cell,2015,27(6):1771-1787.
23.Rahman H,Ramanathan V,Nallathambi J,et al.Over-expression of a NAC 67 transcription factor from finger millet(EleusinecoracanaL.) confers tolerance against salinity and drought stress in rice[J].BMC Biotechnology,2016,16(S1):35.
24.Lee S,Lee H J,Huh S U,et al.TheArabidopsisNAC transcription factor NTL4 participates in a positive feedback loop that induces programmed cell death under heat stress conditions[J].Plant Science,2014,227:76-83.
25.Rauf M,Arif M,Fisahn J,et al.NAC transcription factor speedy hyponastic growth regulates flooding-induced leaf movement inArabidopsis[J].The Plant Cell,2013,25(12):4941-4955.
26.Wang D X,Yu Y C,Liu Z H,et al.Membrane-bound NAC transcription factors in maize and their contribution to the oxidative stress response[J].Plant Science,2016,250:30-39.
27.Shan W,Kuang J F,Lu W J,et al.Banana fruit NAC transcription factor MaNAC1 is a direct target of MaICE1 and involved in cold stress through interacting with MaCBF1[J].Plant,Cell & Environment,2014,37(9):2116-2127.
28.Mukhopadhyay A,Vij S,Tyagi A K.Overexpression of a zinc-finger protein gene from rice confers tolerance to cold,dehydration,and salt stress in transgenic tobacco[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(16):6309-6314.
29.Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
30.Bechtold N,Pelletier G.In planta AgrobacteriumMediated transformation of adultArabidopsisthalianaplants by vacuum infiltration[M].//Martinez-zapater J M,Salinas J.Arabidopsis protocols.Totowa,NJ:Human Press Inc,1998:259-266.
31.Christianson J A,Dennis E S,Llewellyn D J,et al.ATAF NAC transcription factors:regulators of plant stress signaling[J].Plant Signaling & Behavior,2010,5(4):428-432.
32.Hu H H,Dai M Q,Yao J L,et al.Overexpressing a NAM,ATAF,and CUC(NAC) transcription factor enhances drought resistance and salt tolerance in rice[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(35):12987-12992.
33.Delessert C,Kazan K,Wilson I W,et al.The transcription factor ATAF2 represses the expression of pathogenesis-related genes inArabidopsis[J].The Plant Journal,2005,43(5):745-757.
34.Fujita M,Fujita Y,Maruyama K,et al.A dehydration-induced NAC protein,RD26,is involved in a novel ABA-dependent stress-signaling pathway[J].The Plant Journal,2004,39(6):863-876.
35.Huh S U,Lee S B,Kim H H,et al.ATAF2,a NAC transcription factor,binds to the promoter and regulatesNIT2 gene expression involved in auxin biosynthesis[J].Molecules and Cells,2012,34(3):305-313.
36.Christianson J A,Wilson I W,Llewellyn D J,et al.The low-oxygen-induced NAC domain transcription factorANAC102 affects viability ofArabidopsisseeds following low-oxygen treatment[J].Plant Physiology,2009,149(4):1724-1738.
introduction:AI Ke-Jun(1991—),female,Master student,is mainly engaged in plant molecular biology.
date:2016-07-26
ExpressionofTaNAC1GeneinCommonWheatandGeneticTransformationinArabidopsis
AI Ke-Jun1,2ZHU Yao1,2HOU He-Sheng1,2TONG Shao-Ming1,2*
(1.College of Life Sciences,Liaoning Normal University,Dalian 116081;2.Key Laboratory of Plant Biotechnology of Liaoning Province,Dalian 116081)
NAC-type transcription factor is one of the plant-specific gene families, which plays key roles in regulating the development and response to environmental stress in plants. A NAC-type transcription factor, designatedTaNAC1, was cloned from leaves in wheat. The encoding sequence of TaNAC1 contained five typical NAC conserved domains and belonged to ATAF sub-type NAC family. The TaNAC1 protein was located in the nucleus by transient expression in onion epidermis cell. The transcriptional expression was induced by environmental stresses such as ABA, PEG, low temperature and high salt. Phenotypic analysis in the transgenicArabidopsisshowed that over-expression ofTaNAC1 caused abnormal leaves, dwarfism and fused stem in transgenicArabidopsis.TaNAC1 maybe play important roles in regulation during the development of leaves and stems in wheat.
wheat;TaNAC1;Arabidopsis;phenotype
艾可筠(1991—),女,硕士研究生,主要从事植物分子生物学研究。
* 通信作者:E-mail:tongsm@163.com
2016-07-26
* Corresponding author:E-mail:tongsm@163.com
S512.1
A
10.7525/j.issn.1673-5102.2016.06.012
