大肠杆菌O157中肽聚糖水解酶MpaA的克隆、表达、纯化及晶体学研究
2016-11-07马银良刘爽崔亚琦康现江刘秀华
马银良,刘爽,崔亚琦,康现江,刘秀华
(河北大学生命科学学院,河北保定 071002 )
大肠杆菌O157中肽聚糖水解酶MpaA的克隆、表达、纯化及晶体学研究
马银良,刘爽,崔亚琦,康现江,刘秀华
(河北大学生命科学学院,河北保定071002 )
采用分子克隆方法将来源于大肠杆菌O157的mpaA基因构建入pET-21b(+)表达载体.诱导MpaA蛋白在大肠杆菌BL21(DE3)感受态细胞中高效表达,通过Ni亲和层析、离子交换层析和凝胶过滤层析三步法纯化得到高纯度蛋白.使用气象扩散法进行蛋白质晶体生长的初步筛选与优化,得到MpaA蛋白质单晶,并使用X射线衍射仪进行衍射数据的收集与处理.MpaA晶体分辨率为0.26 nm,属于P31空间群,晶胞参数为a=5.989 3 nm,b=5.989 3 nm,c=12.987 0 nm,β=90.000 °.MpaA晶体衍射数据的收集为后续结构解析和催化机制的阐明提供了基础.
大肠杆菌O157;肽聚糖水解酶;MpaA;晶体
肽聚糖是微生物细胞壁不可缺少的组成成分,在维持细胞形态、保持细胞完整性和抗高渗透压等方面具有重要作用[1-3].肽聚糖骨架是由N-乙酰胞壁酸(N-acetylmuramic acid,MurNAc)和N-乙酰葡糖胺(N-acetylglucosamine,GlcNAc)以β-1,4糖苷键交替连接而形成的线状聚糖链.在MurNAc上以共价键形式连接一短肽侧链L-Ala-γ-D-Glu-meso-DAP(或L-Lys)-D-Ala.一般而言,在革兰氏阳性细菌中,短肽分子上的第3个氨基酸是Lys,而在革兰氏阴性细菌中,短肽上的第3个氨基酸残基往往是meso-DAP(2,6-diaminopimelic acid,2,6-二氨基庚二酸)[2].
细菌肽聚糖处于不断组装与解聚的动态过程,在细胞生长、分裂和发育过程中进行重建[4].对于大多数革兰氏阴性菌而言,在每一代细胞中40%~50%的肽聚糖在水解之后,其产物都能够被循环再利用重新形成肽聚糖[5-6].在营养元素匮乏时,肽聚糖的循环再生则可为整个细胞分裂周期的完成提供必须的基础营养元素,并使之进入静止生长期.此外,在微生物之间存在碳氮源竞争时,肽聚糖的循环再利用对于微生物生存具有重要意义[5].
在大肠杆菌的肽聚糖循环再生过程中共涉及11种肽聚糖水解酶.这些酶能够回收肽聚糖降解产物,用于肽聚糖的重新合成,或被细胞用作能源[5].肽聚糖水解酶MpaA含有262个氨基酸,负责切割中间降解产物胞壁质三肽(L-Ala-γ-D-Glu-meso-DAP)中γ-D-Glu与meso-DAP之间的酰胺键,释放L-Ala-D-Glu和DAP[3,7].二肽产物L-Ala-D-Glu在差向异构酶YcjG和肽酶PepD作用下转化成L-Ala和L-Glu[8-9].在营养元素匮乏时,这些氨基酸产物能够被细胞作为能源使用,从而能够存活下来[5,10].MpaA专一性底物是胞壁质三肽(L-Ala-γ-D-Glu-meso-DAP),非特异性底物是L-Ala-γ-D-Glu-L-Lys-MurNAC三肽和γ-D-Glu-(L)- meso-DAP二肽;MpaA并不能够识别胞壁质四肽、二糖-肽或UDP-MurNAc-肽等前体分子[10].到目前为止,MpaA的底物特异性已被广泛研究,但差异性产生的原因是未知的,分子水平的催化反应机制也是未知的.鉴于此,本研究成功克隆、表达、纯化了大肠杆菌O157中的MpaA,对其进行了初步晶体学研究,从而为其三维结构的解析、催化机理的阐明奠定了基础.
1 材料与方法
1.1材料
pET-21b(+)质粒,大肠杆菌O157:H7str.Sakai基因组,大肠杆菌表达菌株BL21(DE3)为本实验室保存.质粒提取试剂盒和胶回收试剂盒购于TIANGEN公司;pfu DNA聚合酶和T4 DNA连接酶购于宝生物(大连)有限公司;限制性内切酶购于NEB公司和宝生物(大连)有限公司;PCR引物 (表1)、重组质粒测序由北京宝锐通公司完成;蛋白胨和酵母提取物购自OXOID公司.蛋白质纯化系统 AKTA explorer 100、亲和层析基质Ni-NTA His-Band resin、离子交换柱Source 15Q及分子筛预装柱 Superdex-200 increase 均购于GE Healthcare公司.

表1 引物设计
1.2mpaA基因的克隆与表达载体构建
目标基因(Gene ID:912455)使用PCR方法扩增后,经琼脂糖凝胶电泳检测及胶回收,使用限制性内切酶NdeI和XhoI双酶切,与经同样酶切后的pET-21b(+)表达载体进行连接,并转化至大肠杆菌BL21(DE3)感受态细胞.挑取多个转化子进行重组蛋白表达检测,将蛋白有阳性表达的转化子提取质粒,进行质粒PCR检测,当蛋白质表达及质粒PCR检测均为阳性时可确定为阳性克隆子,并送测序.
1.3MpaA蛋白质的大量表达与纯化
挑取阳性克隆接种于50 mL含有氨苄青霉素的液体LB培养基中,37 ℃、200 r/min过夜培养;按照1%的接种比例将菌液接入1 000 mL含有相应抗生素的液体LB培养基,37 ℃、200 r/min培养;当菌体密度达到 OD600为0.8时,降温至15 ℃,1 h后,加入IPTG至终浓度为0.15 mmol/L,诱导重组蛋白表达;诱导10 h之后,4 ℃、4 500 r/min离心15 min,收集菌体.将菌体细胞悬浮在40 mL 重悬溶液(20 mmol/L Tris-HCl buffer pH 8.0,200 mmol/L NaCl)中,在冰水混合物条件下用超声波法破碎细胞.细胞裂解液经14 500 r/min、4 ℃下离心45 min,收集上清液.首先,含有可溶性蛋白的上清液经Ni亲和层析柱进行纯化.Ni亲和层析柱预先用重悬溶液平衡,上样后用wash buffer(25 mmol/L Tris-HCl,pH 8.0,100 mmol/L NaCl,15 mmol/L imidazole)除去非特异性吸附的杂蛋白;使用elution buffer(25 mmol/L Tris-HCl,pH 8.0,100 mmol/L NaCl,250 mmol/L imidazole)洗脱目的蛋白MpaA.然后,将可溶性的蛋白使用溶液A(25 mmol/L Tris-HCl,pH 8.0)稀释4倍,上样到已使用溶液 A平衡好的离子交换柱 Source 15Q上,使用溶液A与溶液B(25 mmol/L Tris-HCl,pH 8.0,1 mol/L NaCl)进行150 mL的线性梯度洗脱.收集目标蛋白并浓缩.凝胶过滤层析柱 Superdex-200 increase 预先用溶液C(25 mmol/L Tris-HCl,pH 8.0,100 mmol/L NaCl)平衡,将浓缩蛋白样品上样,收集目标蛋白峰.SDS-PAGE电泳检测蛋白质的纯度及性状.所有的蛋白纯化步骤均在4 ℃进行.
1.4MpaA蛋白结晶、衍射、数据分析
蛋白质晶体生长条件的初筛采用20 ℃坐滴气象扩散法,使用Hampton research 公司的Index1-96、Crystal Screen、Crystal Screen 2试剂盒和Emerald公司的Wizard I、WizardII、WizardIII试剂盒,共336个条件.使用48孔坐滴板,池液100 μL,1 μL蛋白与1 μL 池液混合.在获得晶体初步生长条件基础上,通过改变结晶缓冲液中沉淀剂的浓度对MpaA晶体进行优化,得到衍射分辨率较高的蛋白质单晶,从而进行衍射数据的收集、处理及结构解析.在收集衍射数据时,MpaA蛋白质单晶在晶体防冻液(体积分数为20%的甘油)中浸泡30 s,置于X光机,于-173 ℃条件下收集衍射数据.衍射数据的收集在上海同步辐射光源(SSRF)线站BL17U1完成.衍射数据使用软件HKL2000进行处理[11].
2 结果
2.1pET-21b-MpaA表达载体构建
mpaA基因的PCR扩增产物经质量分数为1%的琼脂糖凝胶电泳检测分析,在750 bp处可见特异性的PCR产物条带,与mpaA基因理论值吻合,且无其他杂带.该PCR产物经DNA凝胶回收、NdeI和XhoI双酶切,连接至同样双酶切的pET-21b(+)载体上,并转化至大肠杆菌BL21(DE3)中.筛选出蛋白表达、质粒PCR检测均为阳性的转化子送出测序,测序结果表明插入基因序列正确、无突变.
2.2重组蛋白MpaA的表达与纯化
通过在液体LB培养基中大规模培养阳性克隆子pET-21b-mpaA,并诱导目的蛋白表达.在15 ℃条件下,MpaA可溶性表达.使用Ni亲和层析、离子交换层析和凝胶过滤层析三步法来纯化MpaA蛋白.经Ni亲和层析柱洗脱下来的蛋白质溶液,进一步使用离子交换柱Source 15Q进行纯化,呈现单一蛋白质洗脱峰,峰形对称且尖锐,无肩膀峰的存在,蛋白无明显损失(数据未显示).收集该蛋白质洗脱峰,浓缩至2 mL,高速离心后上样至Superdex-200 increase层析柱,洗脱结果如图1a所示,该蛋白质洗脱峰平滑、对称,说明MpaA蛋白性状良好、处于均一状态.SDS-PAGE凝胶电泳分析结果如图1b所示.SDS-PAGE凝胶电泳检测显示蛋白分子质量为29 ku,与预测理论值大小相等,MpaA蛋白纯度达到98%以上,质量浓度达到10 mg/L,可用于蛋白晶体的生长.

图1 MpaA蛋白的分子筛层析图谱(a)与SDS-PAGE检测(b)Fig.1 Gel-filtration chromatography profile of MpaA(a)and SDS-PAGE analysis of MpaA after chromatography(b)
2.3MpaA蛋白结晶、衍射及数据分析
MpaA晶体初筛2 d后,在Crystal screen 的18号条件(0.2 mol/L 乙酸镁、0.1 mol/L 二钾砷酸钠和200 g/L PEG 8 000)长出小块状晶体.调整优化沉淀剂和盐浓度,在条件为0.15 mol/L乙酸镁、0.1 mol/L二钾砷酸钠和170 g/L PEG 8 000时,晶体长成0.15 mm×0.15 mm×0.1 mm,如图2a所示,能够用于X射线衍射.MpaA晶体分辨率为0.26 nm(图2b),晶体衍射数据分析结果见表2.晶体空间群是P31,晶胞参数为a=5.989 3 nm,b=5.989 3 nm,c=12.987 0 nm,β=90.000 °.

图2 MpaA蛋白质晶体(a)和X光衍射图谱(b)Fig.2 Crystallization(a) and diffraction pattern(b) of MpaA
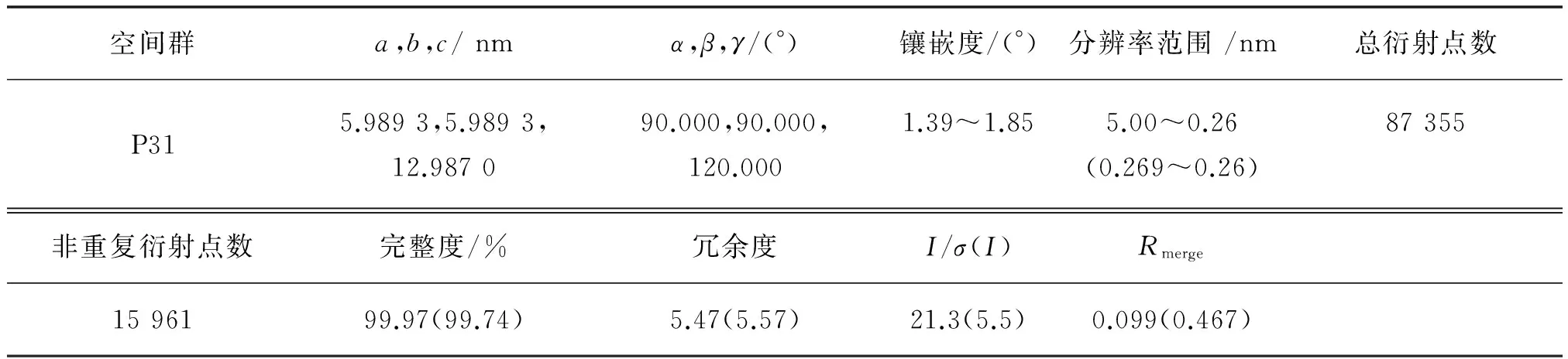
空间群a,b,c/nmα,β,γ/(°)镶嵌度/(°)分辨率范围/nm总衍射点数P315.9893,5.9893,12.987090.000,90.000,120.0001.39~1.855.00~0.26(0.269~0.26)87355非重复衍射点数完整度/%冗余度I/σ(I)Rmerge1596199.97(99.74)5.47(5.57)21.3(5.5)0.099(0.467)
括号内的数据代表的是最高分辨率壳层的数据.
3 讨论
肽聚糖水解酶MpaA在肽聚糖循环再生系统发挥功能,负责切割中间产物胞壁质三肽中γ-D-Glu与meso-DAP之间的酰胺键.释放的氨基酸产物或被用作原料重新合成肽聚糖,或被微生物在营养元素匮乏时用作能源.本研究克隆和表达了来源于致病微生物大肠杆菌O157中的肽聚糖水解酶MpaA,经三步骤纯化后对其进行晶体生长研究和初步晶体学分析,期望能够解析MpaA的三维结构,进而从结构生物学角度来揭示MpaA的底物特异性原因和催化反应机制.在克隆mpaA基因时,考虑到mpaA基因来源于大肠杆菌O157,使用NCBI中的Blast软件进行搜索验证,在大肠杆菌感受态细胞BL21(DE3)中也含有该基因.为避免假阳性结果的产生,在筛选基因mpaA阳性克隆子时,先进行重组蛋白的诱导表达检测,对蛋白诱导表达呈现阳性的克隆子提取其质粒,当质粒的PCR检测也呈现阳性时即可确认为阳性克隆子,送出测序确认克隆成功.在蛋白质MpaA分离纯化过程中,依次使用Ni亲和层析、离子交换及凝胶过滤层析三步骤.离子交换和凝胶过滤层析结果均显示出MpaA蛋白洗脱峰单一,对称且平滑,说明了MpaA蛋白性状良好、状态均一;结合SDS-PAGE检测结果表明,MpaA蛋白能够用于后继的晶体生长实验.在晶体生长条件初步筛选过程中,336个结晶溶液条件中仅有少数几个条件有蛋白质晶体长出,充分说明了蛋白质晶体生长需要做大量筛选的必要性.通过对结晶溶液中乙酸镁和PEG 8000的浓度分别优化至0.15 mol/L和 170 g/L时,得到大小适中、能够用于X线衍射的蛋白质单晶,分辨率为0.26 nm.在使用液氮速冻晶体的过程中,为防止冰晶的产生,在结晶溶液中添加甘油作为防冻剂,其终体积分数为20%.经衍射实验证实,20%的甘油不影响晶体的分辨率.此前,国外研究学者Maqbool等人已克隆、表达、纯化了来源于大肠杆菌O157的MpaA,遗憾的是该MpaA蛋白质不长晶体.不得已,Maqbool课题组成员对MpaA的同源蛋白Vh_MpaA进行了结构生物学研究[10].尽管具有近似催化功能的同源蛋白质结构可以为解释MpaA的催化机理提供一定的帮助,但最终需要测定其自身的结构才能够更好地回答重要的科学问题.这是因为蛋白质功能的行使必须依赖于其正确的三维结构的形成.本文的研究工作为正确解释MpaA的催化机理奠定了良好的基础.目前,本课题组正尝试利用分子置换法解析MpaA三维结构.MpaA三维结构的解析不仅能够在分子水平阐明其催化反应机制,而且也能够为新型抗感染小分子药物的设计提供结构学数据.
[1]WALDEMAR V,BERNARD J,PAULETTE C,et al.Bacterial peptidoglycan(murein)hydrolases [J].FEMS Microbiol Rev,2008a,32:259-286.DOI:10.1111/j.1574-6976.2007.00099.x.
[2]WALDEMAR V,DIDIER B,MIGUEL A D P.Peptidoglycan structure and architecture [J].FEMS Microbiol Rev,2008b,32:149-167.DOI:10.1111/j.1574-6976.2007.00094.x.
[3]HEIJENOORT J V.Peptidoglycan hydrolases of escherichia coli [J].Microbiol Mol Biol,2011,75(4):636-663.DOI:10.1128/mmbr.00022-11.
[4]ANANTHARAMAN V,ARAVIND L.Evolutionary history,structural features and biochemical diversity of the NlpC/P60 superfamily of enzymes [J].Genome Biology,2003,4:R11.DOI:10.1186/gb-2003-4-2-r11.
[5]JAMES T P,TSUYOSHI U.How bacteria consume their own exoskeletons(Turnover and recycling of cell wall peptidoglycan)[J].Microbiol Mol Biol,2008,72(2):211-227.DOI:10.1128/mmbr.00027-07.
[6]JACOBS C,HUANG LI-JUN,BARTOWSKY E,et al.Bacterial cell wall recycling provides cytosolic muropeptides as effectors for beta-lactamase induction [J].The EMBO Journal,1994,13(19):4684-4694.
[7]TSUYOSHI U,JAMES T P.Identification of MpaA,an amidase in Escherichia coli that hydrolyzes the gamma-D-glutamyl-meso-diaminopimelate bond in murein peptides [J].Journal of Bacteriology,2003,185(2):679-682.DOI:10.1128/jb.185.2.679-682.2003.
[8]SCHROEDER U,HENRICH B,FINK J,et al.Peptidase D of Escherichia coli K-12,a metallopeptidase of low substrate specificity [J].FEMS Microbiology Letters,1994,123(1-2):153-159.DOI:http://dx.doi.org/10.1111/j.1574-6968.1994.tb07215.x.
[9]GULICK A M,SCHMIDT D M Z,GERLT JOHN A,et al.Evolution of enzymatic activities in the enolase superfamily:crystal structures of the L-Ala-D/L-Glu epimerases fromEscherichiacoliandBacillussubtilis[J].Biochemistry,2001,40(51):15716-15724.DOI:10.1021/bi011641p.
[10]MAQBOOL A,HERVE M,LECREULX D M,et al.MpaA is a murein-tripeptide-specific zinc carboxypeptidase that functions as part of a catabolic pathway for peptidoglycan-derived peptides in gamma-proteobacteria [J].Biochem J,2012,448:329-341.DOI:10.1042/BJ20121164.
[11]OTWINOWSKI Z,WLANEK M.Processing of X-ray diffraction data collected in oscillation mode [J].Methods Enzymol,1997,276:307-326.DOI:10.1016/S0076-6879(97)76066-X.
(责任编辑:赵藏赏)
Cloning,expression,purification and crystallization of murein-tripeptide amidase MpaA from Escherichia coli O157
MA Yinliang,LIU Shuang,CUI Yaqi,KANG Xianjiang,LIU Xiuhua
(College of Life Sciences,Hebei University,Baoding 071002,China)
The genempaAfromEscherichiacoliO157 was inserted into pET-21b(+)vector using molecular cloning method.The recombinant protein MpaA was overexpressed inE.coliBL21(DE3)competent cells,and purified to homogeneity sequentially via a three-step protocol consisting of Ni-chelating affinity column,ion-exchange column and size-exclusion column chromatography.Crystallization search and optimization for MpaA were carried out using the sitting-drop vapour-diffusion method.The MpaA crystal diffracted to 0.26 nm resolution and belonged to the space group P31,with unit-cell parametersa=5.989 3 nm,b=5.989 3 nm,c=12.987 0 nm andβ=90.000 °.Preliminary crystallographic analysis builds up a good foundation for the structural determination and catalytic mechanism of MpaA.
EscherichiacoliO157;peptidoglycan hydrolase;MpaA;crystal
10.3969/j.issn.1000-1565.2016.04.013
2015-10-16
国家自然科学基金资助项目(31300056);河北省自然科学基金资助项目(C2014201039);河北省教育厅优秀青年基金资助项目(YQ2014007);河北大学自然科学基金资助项目(2011-220)
马银良(1989—),男,河北邢台人,河北大学在读硕士研究生.E-mail:mayinliang@outlook.com
刘秀华(1981—),女,河北泊头人,河北大学副教授,主要从事蛋白质结构与功能研究.
E-mail:xiuhualiu@hbu.edu.cn
Q71
A
1000-1565(2016)04-0412-05
