鲑鱼甲病毒阳性核酸物质制备及RT-PCR检测方法的建立
2016-10-26刘敏杜航宋傲臣高帅唐丽杰蒋烨李一经
刘敏,杜航,宋傲臣,高帅,唐丽杰,蒋烨,李一经
(1.东北农业大学动物科学与技术学院,哈尔滨 150030;2.东北农业大学动物医学学院,哈尔滨 150030)
鲑鱼甲病毒阳性核酸物质制备及RT-PCR检测方法的建立
刘敏1,杜航1,宋傲臣1,高帅1,唐丽杰2,蒋烨1,李一经2
(1.东北农业大学动物科学与技术学院,哈尔滨150030;2.东北农业大学动物医学学院,哈尔滨150030)
为建立快速检测鲑鱼甲病毒(Salmonid alphavirus,SAV)RT-PCR方法,根据GenBank公布的E2基因序列(1 375 bp)设计引物,扩增SAV E2全长基因,利用假病毒制备技术获得包裹E2核酸物质(RNA)的SAV假病毒溶液。以SAV假病毒作为阳性核酸物质质控品,以E2F:5'CCG-TTG-CGG-CCA-CAC-TGG-ATG 3',E2R:5'CCTCAT-AGG-TGA-TCG-ACG-GCA-G 3'为引物,优化反应条件,建立SAV的RT-PCR检测方法。结果表明,该方法可扩增SAV E2特异性516 bp的DNA片段,引物最适反应浓度为1.0 μmol·L-1,最佳退火温度为57.5℃,对SAV的三种亚型SAV1(V4640)、SAV2(V4619)、SAV5(V4638)检测结果均为阳性,对SVCV、IHNV和IPNV的PCR扩增结果均为阴性;并确定对SAV的核酸最低检出量达1.59 pg;以RT-PCR方法在不同时间对每份样品作3次重复检测,检测结果一致。表明此方法特异性强,敏感性高,稳定性和重复性较好,可用于SAV临床诊断和检测。
鲑鱼甲病毒;SAV E2;核酸物质;RT-PCR;检测方法
网络出版时间2016-7-21 14:09:34[URL]http://www.cnki.net/kcms/detail/23.1391.S.20160721.1409.004.html
刘敏,杜航,宋傲臣,等.鲑鱼甲病毒阳性核酸物质制备及RT-PCR检测方法的建立[J].东北农业大学学报,2016,47(7):56-62.
Liu Min,Du Hang,Song Aochen,et al.Positive nucleic acid material preparation and establishment of a RT-PCR for salmonid alphavirus[J].Journal of Northeast Agricultural University,2016,47(7):56-62.(in Chinese with English abstract)
鲑鱼甲病毒(Salmonid alphavirus,SAV)隶属于披膜病毒科(Togaviridae),甲病毒属(Alphavirus)[1],主要感染大西洋鲑(Salmo salar)和虹鳟(Oncorhynchus mykiss)等鲑科鱼类,引起胰腺、心肌炎症和昏睡等症状,致死率达1%~48%,在鱼类养殖各生长阶段均有爆发[2-3]。目前该病广泛流行于苏格兰、挪威、爱尔兰、法国等欧洲国家[4-6],2013年鲑鱼甲病毒感染被列入OIE水生动物疫病名录中。我国目前尚未发现该病,但随鲑科鱼类引进,该病传入我国风险增加。
SAV病毒粒子呈球形,有囊膜,直径为65.5± 4.3 nm[7],易在CHSE-214[8]、BF-2和FHM等细胞株上复制生长,以出芽方式释放病毒粒子,最适复制温度为15℃[9],对三氯甲烷敏感,在pH 3.0且50℃时迅速失活,在氯化铯中的浮力密度1.20 g·mL-1。SAV是单股正链RNA病毒,基因组长11~12 kb[10],含有两个开放阅读框,类似于真核生物mRNA,具有5'端帽子和3'端polyA尾巴结构。5'端开放阅读框占基因组全长三分之二,大小为8 kb,在复制初期快速编码多聚蛋白,多聚蛋白裂解形成四个非结构蛋白,依次是NSP1、NSP2、NSP3和NSP4,可作为基因组RNA形成模板。3'端开放阅读框占基因组全长三分之一,大小为4 kb,编码26S次级基因组mRNA,最终产生5个结构蛋白依次是衣壳蛋白、糖蛋白E3和E2、6K和E1结构蛋白[11-12],共同构成病毒包膜糖蛋白。E2和E1为跨膜蛋白暴露于病毒粒子表面,E1和E2形成异源二聚体[13-14]。根据E2和NSP3核苷酸序列差异将SAV分为6个基因型(SAV1-SAV6)[15-16],六个基因型的基因组和蛋白质组序列保守度较高,根据GenBank中公布基因序列比较,SAV1、SAV2和SAV3基因组和蛋白质组序列保守度分别为90%、95%,SAV1型不同毒株核苷酸序列保守度为95%,SAV2型不同毒株核苷酸序列保守度为93.6%。SAV 2、SAV 3可感染虹鳟引起睡病(Sleeping disease,SD);SAV 1、SAV 2、 SAV 4、SAV 5、SAV 6基因型可感染大西洋鲑引起胰腺病(Salmon pancreas disease,PD)[17]。
目前,我国尚未建立鲑鱼甲病毒检测方法。反转录PCR(Reverse transcription-polymerase chain reaction,RT-PCR)检测RNA病毒,耗时短、特异性强、灵敏度高、便于应用。本文根据GenBank公布SAV E2基因序列设计引物,扩增E2全长基因,拟制备包裹着E2核酸物质(RNA)SAV假病毒溶液。以SAV假病毒溶液作为阳性核酸物质的质控品,优化反应条件,建立SAV RT-PCR检测方法,为鲑鱼甲病毒临床诊断、流行病学调查、疫病监测和防控奠定基础。
1 材料与方法
1.1材料
SAV1(V4640)、SAV2(V4619)和SAV5(V4638)三种不同基因型毒株由英国苏格兰海洋实验室赠;体外转录试剂盒(T7启动子)、无细胞表达试剂盒、质粒中提试剂盒均购自普洛麦格生物技术有限公司,假病毒表达载体Pet 32a-AR(在Pet32a表达载体多克隆位点中插入MS2噬菌体的成熟蛋白基因、衣壳蛋白基因和衣壳蛋白基因下游影响噬菌体包装的19 bp发夹结构)由中国检验检疫科学院赠;传染性胰腺坏死病毒(Infectious pancreatic necrosis virus,IPNV)、传染性造血组织坏死病病毒(Infectious haematopoietic necrosis virus,IHNV)、鲤春病毒血症病毒(Spring viraemia of carp virus,SVCV)由东北农业大学兽医微生物免疫教研室保存。
1.2印物
根据GenBank中SAV基因序列设计扩增E2(1 375 bp)片段上下游引物,分别为含有HindⅢ酶切位点上游引物P1和含有NotⅠ酶切位点下游引物P2,检测引物为上游引物E2F和下游引物E2R,扩增目的片段长为516 bp。引物序列见表1。引物均由吉林省库美生物科技有限公司合成。
1.3SAV假病毒阳性品的制备方法
1.3.1SAV E2基因扩增
Trizol法提取SAV 1(4 640毒株)病毒总RNA,反转录成cDNA,以P1和P2为引物PCR扩增。PCR反应条件:95℃预变性5 min,94℃变性50 s,55℃退火50 s,72℃延伸1.5 min,循环数为35个,终延伸72℃10 min。将SAV E2基因片段回收,构建重组质粒pMD18-TS-SAV E2,进行序列测定比对分析。
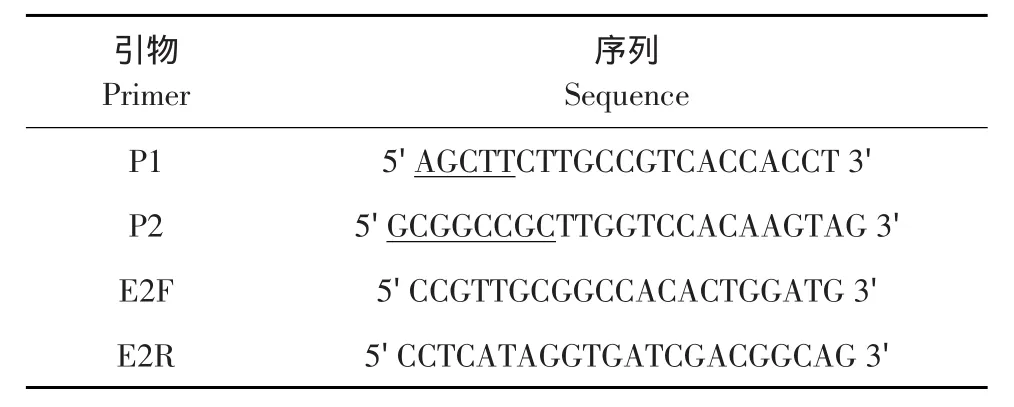
表1 引物序列Table 1Sequnce of primers
1.3.2假病毒重组质粒Pet32a-AR-SAVE2/TG1构建
假病毒表达载体Pet32a-AR用NotⅠ和HindⅢ双酶切后,经1%琼脂糖凝胶电泳分离后,胶回收纯化试剂盒切胶回收两段目的基因,将SAV E2和Pet 32a-AR用T4DNA连接酶连接,16℃连接12 h,转化大肠杆菌TGI感受态。涂布含50 μg·mL-1氨苄抗性的平板,37℃过夜培养,挑取单个菌落提取质粒,进行酶切和PCR鉴定。
1.3.3假病毒mRNA提取及体外转录
用质粒中提试剂盒提取Pet 32a-AR-SAV E2重组质粒,将质粒用限制性内切酶BSSH II线性化后胶回收,PCR鉴定后测定线性化DNA浓度,用体外转录试剂盒转录,反应条件为37℃4 h。转录体系如下:5×Buffer(15 mmol·L-1)20 μL;rNTP(25 mmol·L-1each)30 μL;线性化假病毒DNA 2 μg;转录酶10 μL;ddH2O加至总体积100 μL。在100 μL转录体系内加入10 μL DNase I(RNase free),37℃1 h,去除转录产物中的假病毒DNA。Trizol法提取转录产物中mRNA,紫外分光光度计测量RNA浓度。以提纯RNA溶液为模板,使用引物P1和P2分别作PCR和RT-PCR检测,经鉴定,PCR检测结果为阴性,RT-PCR检测结果为阳性,说明溶液中只有假病毒mRNA无DNA。
1.3.4无细胞表达系统表达SAV假病毒
使用无细胞表达系统表达假病毒,表达体系:Rabbit Reticulocyte Lysate 70 μL;Amino Acid Mixture Minus Leucine(1 mmol·L-1)1 μL;Amino Acid Mixture Minus Methionine(1 mmol·L-1)1 μL;RNasin Ribonuclease Inhibitor 2 μL;RNA 4 μg;ddH2O加至总体积100 μL。反应条件为30℃90 min,反应产物即为SAV假病毒阳性制品,-80℃保存备用。
1.4SAV RT-PCR反应体系建立
Trizol法提假病毒RNA,反转录制备cDNA。以cDNA为模板,E2F和E2R各1 μL,10×PCR buffer 2.5 μL,dNTPs(2.5 mmol·L-1)2.5 μL,rTaq DNA(5 U·μL-1)0.5 μL,ddH2O 15 μL,cDNA模板2.5 μL。反应条件:95℃10 min;94℃30 s、58℃30 s、72℃50 s,35个循环;72℃10 min。PCR产物经电泳分析后作测序比对分析。
1.5PCR反应条件的优化
优化PCR反应退火温度(54.0℃、54.8℃、55.6℃、57.5℃、58.4℃)及上下游引物浓度(0.2、0.4、0.6、0.8和1.0 μmol·L-1),确定最佳退火温度和引物浓度。
1.6特异性试验
Trizol法提取IPNV、IHNV、SVCV和SAV假病毒溶液RNA,分别反转录为cDNA,采用优化后扩增程序PCR反应扩增,灭菌双蒸水作为阴性对照,确定建立的RT-PCR检测方法特异性。
1.7敏感性试验
提取SAV假病毒溶液的RNA,测定其浓度为159.6 ng·μL-1、混合均匀后用DEPC水倍比稀释(101~107),将各稀释度RNA反转录成cDNA,以上述cDNA为模板作PCR检测。
1.8重复性试验
使用本方法重复检测同一样品3次,比较每次结果是否一致,分析建立的RT-PCR方法稳定性。
1.9SAV细胞培养毒检测
采用RT-PCR方法进行SAV1(V4640)、SAV2(V4619),SAV5(V4638)毒株细胞培养物检测,0.8%琼脂糖凝胶电泳观察检测结果。
2 结果与分析
2.1SAV E2基因扩增结果
提取SAV1细胞培养物RNA,反转录成cDNA,以其为模板,扩增到1 375 bp目的片段,目的片段与SAV E2基因片段预测大小一致(见图1),测序后序列比对结果正确。
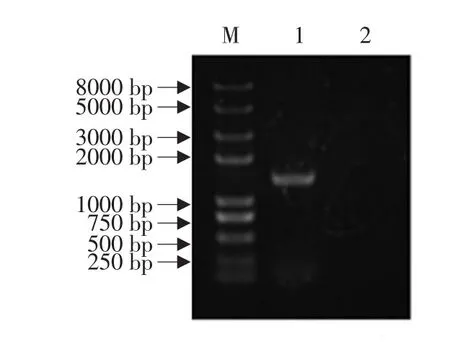
图1 SAV1 E2基因PCR扩增结果Fig.1Amplification result of SAV1 E2 gene
2.2SAV E2假病毒重组质粒Pet32a-AR-SAV E2的鉴定
重组质粒Pet32a-AR-SAV E2用限制性内切酶HindⅢ单酶切得到8 999 bp大小片段,限制性内切酶HindⅢ和NotⅠ双酶切得到7 624和1 375 bp大小片段(见图2),单、双酶切得到的目的片段均与预期大小的核酸片段相同,表明SAV E2基因正确插入Pet 32a-AR假病毒表达载体。

图2 重组质粒Pet 32a-AR-SAV E2酶切鉴定结果Fig.2Restriction map of Pet 32a-AR-SAV E2
2.3假病毒表达鉴定
提取假病毒溶液中的RNA,取适量转录为cDNA,分别作PCR检测鉴定。结果显示以cDNA为模板作RT-PCR检测结果为阳性,以RNA为模板作PCR检测为阴性(见图3)。试验结果表明外源核酸物质(E2的RNA)被假病毒表达蛋白所包裹,获得SAV假病毒溶液可作为SAV RT-PCR检测的阳性质控品。
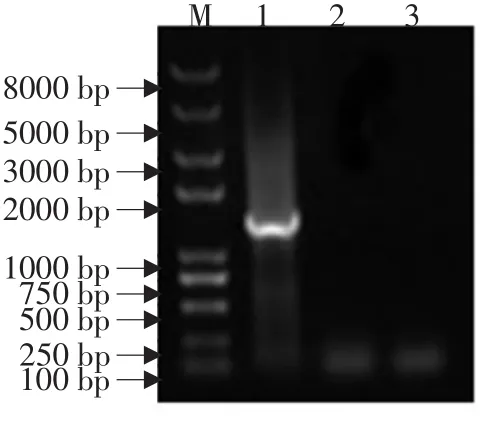
图3 假病毒表达PCR及RT-PCR鉴定结果Fig.3Identification of false virus expression
2.4RT-PCR引物浓度及退火温度优化
取不同浓度引物及退火温度作PCR反应,确定最佳引物浓度和退火温度。结果显示浓度均为1.0 μmol·L-1时目的片段扩增效果最佳(见图4A),退火温度为55.6、57.5和58.4℃时目的条带扩增效果均较好(见图4B),最终将最佳退火温度确定为57.5℃。

图4 RT-PCR引物浓度及退火、温度确定Fig.42Optimization of the RT-PCR reaction conditions
2.5特异性试验
采用本方法对SVCV、IPNV、IHNV及SAV假病毒溶液cDNA作RT-PCR扩增,仅SAV假病毒扩增产物中出现目的片段,而SVCV IPNV、IHNV和ddH O扩增结果均为阴性(见图5),结果表明本RT-PCR检测方法具有较强特异性。
2.6敏感性试验
对SAV假病毒溶液RNA十倍比稀释,分别反转录合成cDNA,以上cDNA为模板分别作PCR反应,结果显示SAV E2最低检测量为1.59 pg(见图6)。
2.7重复性试验结果
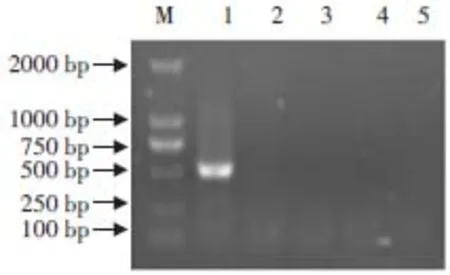
图5 RT-PCR特异性检测Fig.5Specific detection of RT-PCR
分别对同一样品作3次检测,结果完全一致。说明本RT-PCR方法重复性好,结果稳定可靠。
2.8SAV细胞培养毒的检测结果
以SAV1(V4640)、SAV2(V4619)、SAV5(V4638)三种不同亚型毒株的cDNA为模板,利用本RT-PCR方法检测,检测结果均为阳性,而ddH2O扩增结果为阴性,说明本RT-PCR方法检测适用性高(见图7)。
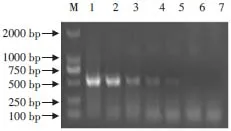
图6 RT-PCR敏感性试验Fig.6Sensitivity assay of RT-PCR
3 讨论与结论
近年来鲑科鱼类养殖业迅猛发展,各养殖区之间贸易频繁,SAV感染鲑科鱼类引起鲑鱼胰腺病和晕睡病已经扩散至欧洲各国[18-20],给鲑鳟养殖业带来巨大经济损失。我国目前尚未发现此病,但建立SAV快速、灵敏、特异检测方法,对预防外来水生动物疫病尤为重要。
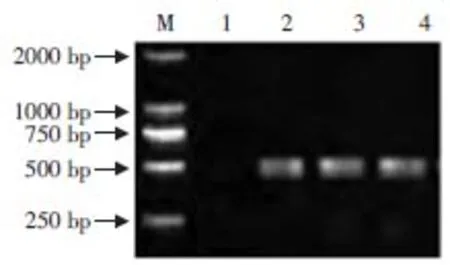
图7 SAV细胞培养毒的检测结果Fig.7Detection of SAV cell culture virus
目前,鲑鱼甲病毒检测方法主要有病毒分离、ELISA和免疫组化等。Karlsen等从患有鲑鱼胰腺病垂死鱼心脏和肾脏中分离得到SAV 3型[21],传代培养并作病理学分析;Taksdal等根据鲑鱼甲病毒糖蛋白E1基因序列成功制备4H1单克隆抗体,对病毒免疫组化检测[22]。但病毒分离、ELISA和免疫组化检测方法耗时较长,不适用口岸检测和临床诊断。Villoing等陆续建立反转录-聚合酶链式反应(RT-PCR)和TaqMan探针PCR检测方法[23-26]。RT-PCR技术在鱼类RNA病毒检测应用广泛,如传染性胰腺坏死病病毒(IPNV)[27-28]和鱼棒状病毒[29]。可见运用RT-PCR技术作鱼类RNA病毒检测应用前景广阔。
本研究通过假病毒制备技术,获得假病毒重组质粒,该质粒在无细胞表达系统中能够将插入的E2基因片段转录成RNA,合成MS2蛋白能够包裹RNA,自发组装成类似病毒粒子,成为SAV假病毒溶液。制备假病毒与MS2噬菌体类似,能耐受核酸酶降解,稳定性较好,不具复制和传染能力,消除其他检测方法中被检测病毒传播风险,避免严重经济损失。以SAV假病毒溶液为阳性核酸质控品,E2F和E2R作为检测引物建立RT-PCR检测方法,阳性对照可得到约516 bp特异性片段,对SAV三种不同亚型检测结果均为阳性,对SVCV、IHNV和IPNV检测结果均为阴性,且SAV最低检出量达达1.59 pg水平,用本RT-PCR方法在不同时间对每份样品作3次重复检测,结果一致。该方法敏感性高、特异性强、稳定性好,可用于SAV临床诊断和流行病毒学研究。
[1]Tz-Chun G,Johansson D X,Øyvind H,et al.A 6K-deletion variant of salmonid alphavirus is non-viable but can be rescued through RNA recombination[J].Plos One,2014,9(7):100184.
[2]Crane M,Hyatt A.Viruses of fish:an overview of significant pathogens[J].Viruses,2011,3(11):2025-2046.
[3]Jansen M D,Taksdal T,Wasmuth M A,et al.Salmonid alphavirus(SAV)and pancreas disease(PD)in Atlantic salmon,Salmo salar L.,in freshwater and seawater sites in Norway from 2006 to 2008[J].J Fish Dis,2010,33(5):391-402.
[4]Graham D A,Fringuelli E,Rowley H M,et al.Geographical distribution of salmonid alphavirus subtypes in marine farmed Atlantic salmon,Salmo salar L,in Scotland and Ireland[J].J Fish Dis,2012,35(10):755-765.
[5]Braceland M,Bickerdike R,Tinsley J,et al.The serum proteome of Atlantic salmon,Salmo salar,during pancreas disease(PD)following infection with salmonid alphavirus subtype 3(SAV3)[J]. J Proteomics,2013,94:423-436.
[6]Ringuelli E,Rowley H M,Wilson J C,et al.Phylogenetic analyses and molecular epidemiology of European salmonid alphaviruses(SAV)based on partial E2 and nsP3 gene nucleotide sequences[J].J Fish Dis,2008,31(11):811-823.
[7]Karlsen M,Villoing S,Rimstad E,et al.Characterization of untranslated regions of the salmonid alphavirus 3(SAV3)genome and construction of a SAV3 based replicon[J].Virol J,2009,6: 173.
[8]Hikke M C,Stine B,Stephane V,et al.Salmonid alphavirus glycoprotein E2 requires low temperature and E1 for virion formation and induction of protective immunity[J].Vaccine,2014,32(47):6206-6212.
[9]Welsh M,Weston J,Borghmans B J,et al.Biochemical characterization of salmon pancreas disease virus[J].J Gen Virol,2000,81(3):813-820.
[10]Xu C,Øystein Evensen,Munang'Andu H M.De Novo assembly and transcriptome analysis of Atlantic salmon macrophage/ dendritic-like TO cells following type I IFN treatment and Salmonid alphavirus subtype-3 infection[J].Bmc Genomics,2015,16(1):1-16.
[11]Lopez-Jimena B,Lyons P,Herath T,et al.The effect of dietary n-3/n-6 polyunsaturated fatty acid ratio on salmonid alphavirus subtype 1(SAV-1)replication in tissues of experimentally infected rainbow trout(Oncorhynchus mykiss)[J].Veterinary Microbiology,2015,178(1-2):19-30.
[12]Cheng X,Mutoloki S,Øystein Evensen.Superior protection conferred by inactivated whole virus vaccine over subunit and DNA vaccines against salmonid alphavirus infection in Atlantic salmon(Salmo salar L.)[J].Vaccine,2012,30(26):3918-3928.
[13]Rheme C,Ehrengruber M U,Grandgirard D.Alphaviral cytotoxicity and its implication in vector development[J].Exp Physiol,2005,90(1):45-52.
[14]Gomez-Casado E,Estepa A,Coll J M.A comparative review on European-farmed finfish RNA viruses and their vaccines[J].Vaccine,2011,29(15):2657-2671.
[15]Aadersen L,Hodneland K,Nylund A.No influence of oxygen levels on pathogenesis and virus shedding in Salmonid alphavirus(SAV)-challenged Atlantic salmon(Salmo salar L.)[J].Virol J,2010,7:198.
[16]Graham D A,Frost P,McLaughlin K,et al.A comparative study of marine salmonid alphavirus subtypes 1-6 using an experimental cohabitation challenge model[J].J Fish Dis,2011,34(4): 273-286.
[17]Villoing S,Beatzotti M,Chilmonczyk S,et al.Rainbow trout sleeping disease virus is an atypical alphavirus[J].J Virol,2000,74(1):173-183.
[18]Mcloughlin M F,Nelson R N,Mccormick J I,et al.Clinical and histopathological features of naturally occurring pancreas disease in farmed Atlantic salmon,Salmo salar L.[J].Journal of Fish Diseases,2002,25(1):33-43.
[19]Graham D A,Taylor C,Rodgers D,et al.Development and evaluation of a one-step real-time reverse transcription polymerase chain reaction assay for the detection of salmonid alphavirusesinserumandtissues[J].DiseasesofAquatic Organisms,2006,70(1-2):47-54.
[20]Suzuki S.Hosono N,Kusuda R.Detection of aquatic birnavirus gene from marine fish using a combination of reverse transcription and nested PCR[J].J Mar Biotechnol,1997,5:205-209.
[21]Karlsen M,Hodnelang K,Endresen C,et al.Genetic stability within the Norwegian subtype of salmonid alphavirus(family Togaviridae)[J].Arch Virol,2006,151(5):861-874.
[22]Taksdal T,Olsen A B,Bjerk S I,et al.Pancreas disease in farmedAtlantic salmon,Salmo salar L.,and rainbow trout,Oncorhynchus mykiss(Walbaum),in Norway[J].Journal of Fish Diseases,2007,30(9):545-58.
[23]Hikke M C,Verest M,Vlak J M,et al.Salmonid alphavirus replication in mosquito cells:towards a novel vaccine production system[J].Microb Biotechnol,2014,7(5):480-484.
[24]Rodger H,Mitchell S.Epidemiological observations of pancreas disease of farmed Atlantic salmon,Salmo salar L.,in Ireland[J].J Fish Dis,2007,30(3):157-167.
[25]Nylund S,Andersen L,Saevareid I,et al.Diseases of farmed Atlantic salmon Salmo salar associated with infections by the microsporidian Paranucleospora theridion[J].Dis Aquat Organ,2011,94(1):41-57.
[26]Tadiso T M,Krasnov A,Skugor S,et al.Gene expression analyses of immune responses in Atlantic salmon during early stages of infection by salmon louse(Lepeophtheirus salmonis)revealed biphasic responses coinciding with the copepod-chalimus transition[J].BMC Genomics,2011,12:141.
[27]Lopez-Lastra M,Gonzalez M,Jashes M,et al.A detection method for infectious pancreatic necrosis virus(IPNV)based on reverse transcription(RT)-polymerase chain reaction(PCR)[J].J Fish Dis 1994,17:269-282.
[28]Boucher P,Le Ven A,Baudin-Laurencin F.Transmission expenmentale de la maladie du sommeil:histologie et caracterisation de l'agent infectieux[J].Nouv Sci Technol,1995,13:115-117.[29]Bruchhof B.Marquardt O,Enzmann P J.Differential diagnosis of fish pathogenic rhabdoviruses by reverse transcri ptase-dependent polymerase chain reaction[J].J Virol Methods 1995,55:111-119.
Positive nucleic acid material preparation and establishment of a RTPCR for salmonid alphavirus
LIU Min1,DU Hang1,SONG Aochen1,GAO Shuai1,TANG Lijie2,JIANG Ye1,LI Yijing2
(1.School of Animal Science and Technology,Northeast Agricultural University,Harbin150030,China;2.SchoolofVeterinaryMedicine,NortheastAgricultural University,Harbin 150030,China)
To rapidly identify the pathogens causing salmonid alphavirus(SAV),a RT-PCR was developed with primers designed according to the sequences of theE2 gene(1 375 bp)of SAV available in GenBank.Amplified theSAV E2 gene,using false virus preparation technology to obtain the SAV fake virus solution wrapped with E2 material nucleic acid(RNA).SAV RT-PCR detection method was established with the SAV false virus as positive nucleic acid material quality control and the designed primers of E2F:5'CCG-TTG-CGG CCA-CAC-TGG-ATG 3',E2R:5'CCT-CAT0AGG TGATCG-ACG-GCA-G 3'as detection primers.Results showed that we could amplify the specific fragment ofSAV E2 DNA gene(516 bp)better when the optimum concentration of primers was 1.0 μmol·L-1and the annealing temperature was 57.5℃at the same time.Detection result of SAV1,SAV3,SAV5 were all positive by the established RT-PCR method,but no specific amplifications from other virus(SVCV, IPNV,IHNV),this assay could detecting the minimum detection of SAV nucleic acids was 1.59 pg.Eachsample had been detectived three times at different times through this RT-PCR,and there was no change between the detection results of same samples.Comprehensive research results showed the established RT-PCR(use the SAV false virus nucleic acid material as positive control)had strong specificity,high sensitivity,good stability and repeatability characteristics,could be used in clinical diagnosisanddetectionofSAV.
salmonid alphavirus;SAV E2;nucleic acid;RT-PCR;detection method
S942.3
A
1005-9369(2016)07-0056-06
2016-04-11
国家科技支撑计划项目(2013BAD12B02)
刘敏(1965-),女,教授,博士,研究方向为水产动物疾病及免疫学。E-mail:liumin-707@163.com
