LiCl溶液中基于固态纳米孔的DNA检测
2016-10-24沙菁石鸿佼
沙菁 石鸿佼 徐 冰
(东南大学江苏省微纳生物医疗器械设计与制造重点实验室, 南京 211189)(东南大学机械工程学院, 南京 211189)
LiCl溶液中基于固态纳米孔的DNA检测
(东南大学江苏省微纳生物医疗器械设计与制造重点实验室, 南京 211189)(东南大学机械工程学院, 南京 211189)
采用阳离子体积较小的LiCl溶液作为缓冲液,研究DNA在不同浓度下通过固态纳米孔时的行为特征,以探究DNA的过孔机理.实验结果表明,当LiCl溶液浓度从0.1mol/L变化到1.0mol/L时,由DNA过孔所引起的相对阻塞离子电流信号比值从0.014 42降低至0.002 79,而DNA完全通过纳米孔所需时间从0.30ms增加至1.87ms.对比1mol/LKCl,NaCl,LiCl三种溶液中DNA的过孔结果发现,在LiCl溶液中DNA的过孔时间分别为KCl,NaCl溶液中的6.2和5.3倍,说明LiCl溶液能够有效降低DNA的过孔速度.此外,实验发现LiCl溶液中DNA主要以线性非折叠和折叠2种形态过孔. 因此,增加LiCl溶液浓度可延长DNA的过孔时间,但会使相对阻塞离子电流信号幅值降低.
纳米孔;LiCl溶液;DNA;阻塞离子电流;过孔时间
纳米孔测序技术是下一代基因测序技术中最可行的技术方案之一,主要是通过物理方法直接对DNA序列进行读取.纳米孔测序技术的原理可以简单描述为: 单个生物分子通过充满离子溶液的纳米孔通道时,会引起通道内电学性质的变化,通过分析离子电流的阻塞信号,可实现单分子检测,甚至是DNA中4种碱基的辨识[1].纳米孔测序技术具有结构简洁、速度快、操作简便等特点,故而得到了广泛的关注[2].
目前,固态纳米孔大都利用硅及其衍生物制造而成,一般使用离子束或电子束在硅或其他材料薄膜表面加工出纳米尺度的孔径,再进一步对孔的形状和大小进行修饰.与生物纳米孔相比,固态纳米孔具有高稳定性、孔径大小和通道长度可控、可调节的表面特性以及可与设备结合的优势[3-5].
纳米孔测序技术的关键在于对DNA穿过纳米孔机理的分析,即对DNA通过纳米孔所引起的阻塞离子电流信号进行辨识检测.对过孔信号起主要影响作用的是缓冲溶液中的正离子,目前的实验研究一般在KCl或NaCl溶液中进行,但K+和Na+的离子半径较大,与DNA表面的吸附力不强,在DNA通过纳米孔时容易脱落[6].为了深入研究纳米孔的过孔机理,本文将LiCl溶液作为缓冲液,研究了不同溶液浓度下DNA通过Si3N4纳米孔的行为特征.
1 实验材料和方法
1.1纳米孔的制备
本实验使用了Si3N4固态纳米孔.其加工步骤如下: ① 利用低压化学气相沉积技术,在2.5mm×2.5mm×0.4mm的硅基底上沉积一层100nm厚的Si3N4薄膜.② 采用光刻技术和反应离子刻蚀的方法,在Si3N4薄膜一侧的中心处加工出一个720μm×720μm的正方形窗口.③ 通过湿法刻蚀技术,在硅基底上刻蚀出一个160μm×160μm的深槽,且刻蚀到Si3N4薄膜时停止.此时,该刻蚀部分薄膜下硅基底已被完全腐蚀,薄膜处于悬空状态.④ 在膜的中心区域1μm×1μm正方形内,利用离子束刻蚀技术将薄膜减薄至10nm左右.⑤ 在减薄区域采用离子束刻蚀技术加工出一个直径约30nm的纳米孔.
为了减弱实验过程中电介质产生的干扰噪声,将纳米孔芯片浸泡在125 ℃的食人鱼溶液(V(H2SO4)∶V(H2O2)=3∶1)中30min,并用去离子水清洗3遍;然后,以高纯氮气吹干表面水渍,同时在孔口附近涂上一层薄PDMS[7].
1.2实验原理与方法
实验原理示意图如图1所示.纳米孔将导电的电解质溶液分成2个区域.通过施加外电场,产生通过纳米孔的基准离子电流.当生物分子通过纳米孔时,离子电流被部分阻塞,根据离子电流的波动便可分析生物分子的信息.在本实验中,被检测的生物分子为λDNA,它是一种长度为48.5kbp的双链DNA,直径为2.2nm,等电位为3.5~4.0.当溶液pH=8.0时,DNA表面带负电.因此,施加相应电场时,DNA在电场力的驱动下可以通过纳米孔.
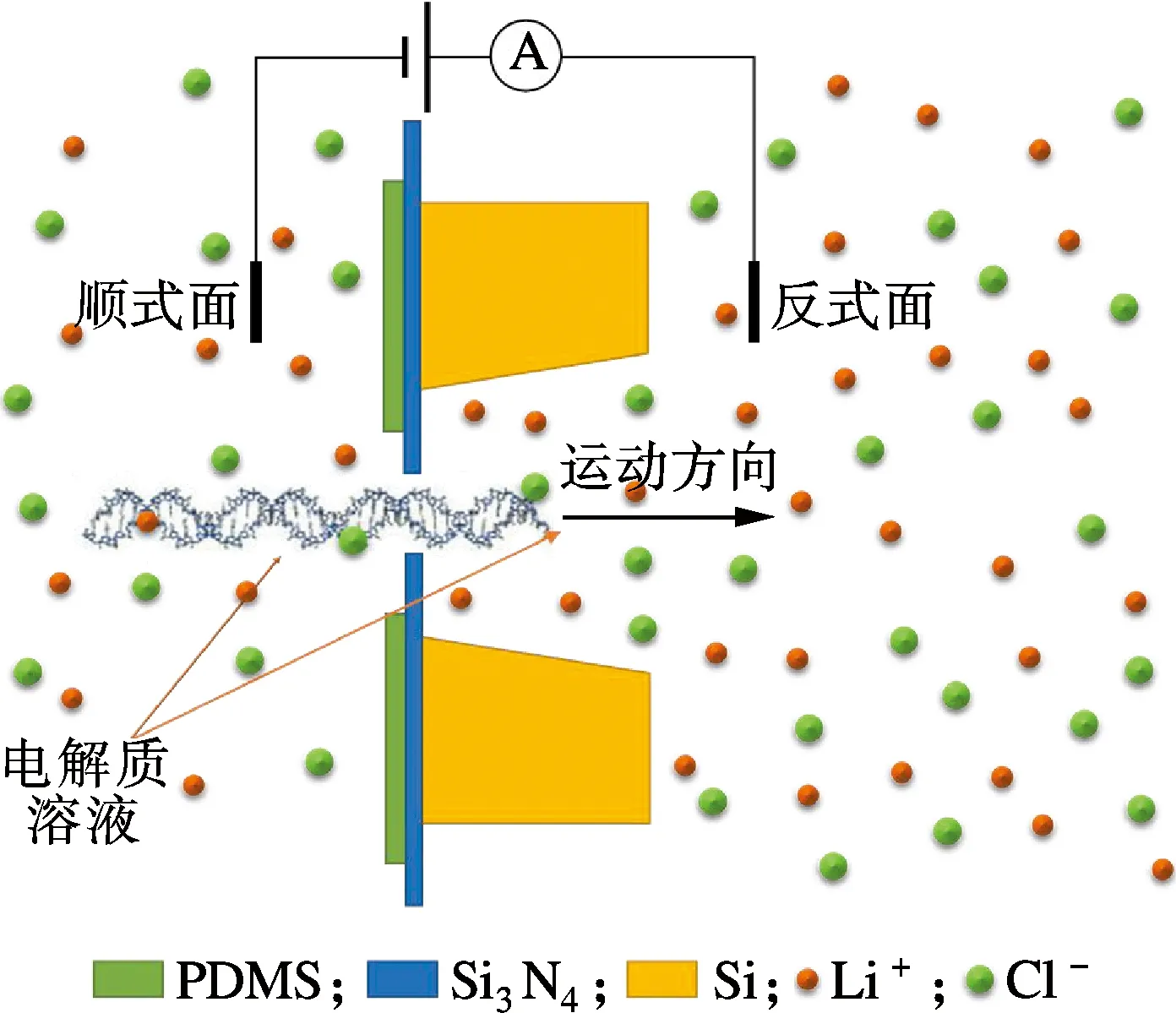
(a) 电压驱动DNA过孔

(b) 氮化硅纳米孔电镜图图1 DNA过孔实验原理示意图
实验在pH=8.0的LiCl溶液中进行,实验装置示意图如图2所示.首先,在2个液池中加入LiCl溶液.为了使2个液池间的离子运动只发生在纳米孔内,将孔的两端垫上橡胶垫圈密封,并将其与液池组装在一起,使各孔中心在一条直线上, 从而保证其密封性. 将λDNA(购于Takara公司) 添加在液池的cis端,2个Ag/AgCl电极插入液池中,并连接到HEKA膜片钳放大器(HEKAEPC10)上,以100kHz采样频率、10kHz低通道滤波频率来检
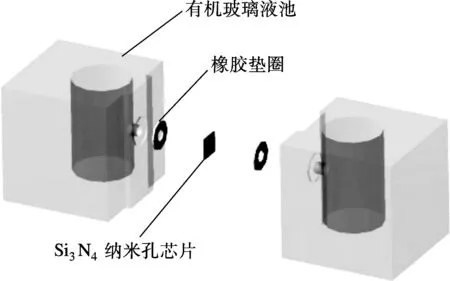
图2 过孔实验装置示意图
测离子电流.为屏蔽外来电噪声的影响,实验在法拉第笼中进行.
2 实验结果和分析
2.1阻塞离子电流信号幅值
DNA过孔时的阻塞离子电流大小主要由DNA占位体积决定,但壁面电荷、DNA带电量、溶液浓度梯度等也会对其产生影响[8-10].下面研究LiCl溶液浓度为1.0,0.5,0.1mol/L时DNA过孔导致的阻塞离子电流信号幅值变化.
如图3(a)所示,当施加电压V0=1 000mV时,不同LiCl溶液浓度下DNA过孔所造成的阻塞离子电流信号幅值接近,峰值均为650pA左右,从事件百分比上看,无法区分不同浓度的LiCl溶液中DNA过孔情况,而利用相对阻塞离子电流信号则不仅可进行有效区分,还能反映出不同浓度对DNA过孔情况的影响.
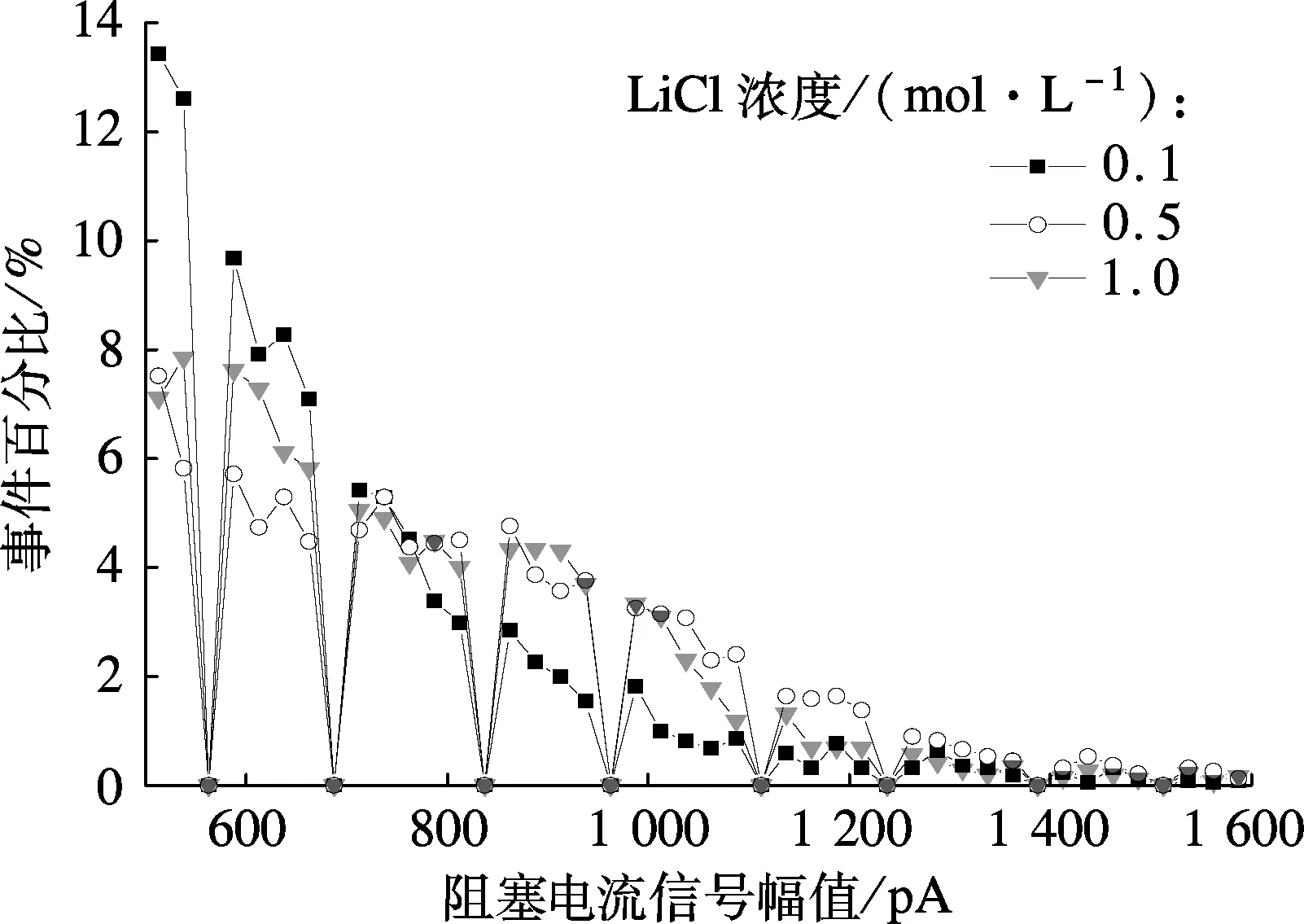
(a) 阻塞离子电流信号

(b) 相对阻塞离子电流信号图3 不同浓度下DNA过孔幅值比较
根据孔内离子电流计算公式[11-12],可以推导出相对阻塞离子电流信号与DNA占位体积的关系,即
I0=GΔV
(1)
(2)
ΔI=ΔGΔV
(3)
ΔI=-[I0Λ/(HeffA)][1+f(dm/Dp,lm/Heff)]≈
-I0Λ/(HeffA)
(4)
式中,I0为基准离子电流;ΔI为阻塞离子电流;ΔV为施加的电压;G为孔的电导;σ为溶液电导率,在1.0mol/LLiCl溶液中σ=8.98S/m;l为氮化硅纳米孔膜厚,此处l=10nm;ΔG为电导的变化量;Λ为DNA在孔内的占位有效体积;Heff为纳米孔膜的厚度;A为孔口面积;HeffA为纳米孔体积;dm为分子直径;Dp为纳米孔直径;lm为分子长度;为了简化计算,一般取 f(dm/Dp,lm/Heff)=0.
由式(3)和(4)可知,阻塞离子电流ΔI的变化量与电导ΔG的变化量、DNA在孔内的占位有效体积Λ呈线性相关.因此,可将ΔI/I0等价于ΔG/G[12].由图3(b)和表1可以看出,当LiCl浓度为1.0,0.5,0.1mol/L时,由DNA过孔所引起的相对阻塞离子电流信号比值ΔI/I0=0.002 79,0.005 08,0.014 42,即随LiCl溶液浓度的下降,ΔG/G呈现上升趋势.
当纳米孔中充满电解质溶液时,孔壁和DNA表面会呈现带电性质,在本文实验中这两者都带负电荷.带电表面在溶液中会存在电性相反的离子聚集现象,从而形成双电层[9].双电层的厚度可用Debye长度κ-1来描述,而Debye长度与溶液浓度c的关系为
(5)
式中,ξ0为电常数;ξr相对静电介质常数;KB为波尔兹曼常数;T为室内温度;e为元电荷量.
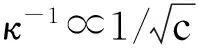
2.2过孔时间
一般来说,长链DNA在电场作用下以线性而非团聚的方式穿过纳米孔[13].采用纳米孔检测DNA上碱基的序列,在理论上是可行的.但是,对于目前大多数DNA检测实验而言,DNA的过孔速度太快,过孔时间太短,无法达到检测单碱基的精度要求.因此,对于DNA过孔时间的研究十分必要.
当施加电压为1 000mV、LiCl溶液浓度为0.1,0.5,1.0mol/L时,DNA的过孔时间见图4(a).由图可知,当溶液浓度从0.1mol/L增加到1.0mol/L时,DNA的过孔时间峰值由0.36ms增加到1.87ms,说明随着LiCl溶液浓度的增加,DNA的过孔时间增大,过孔速度降低.

(a) LiCl溶液
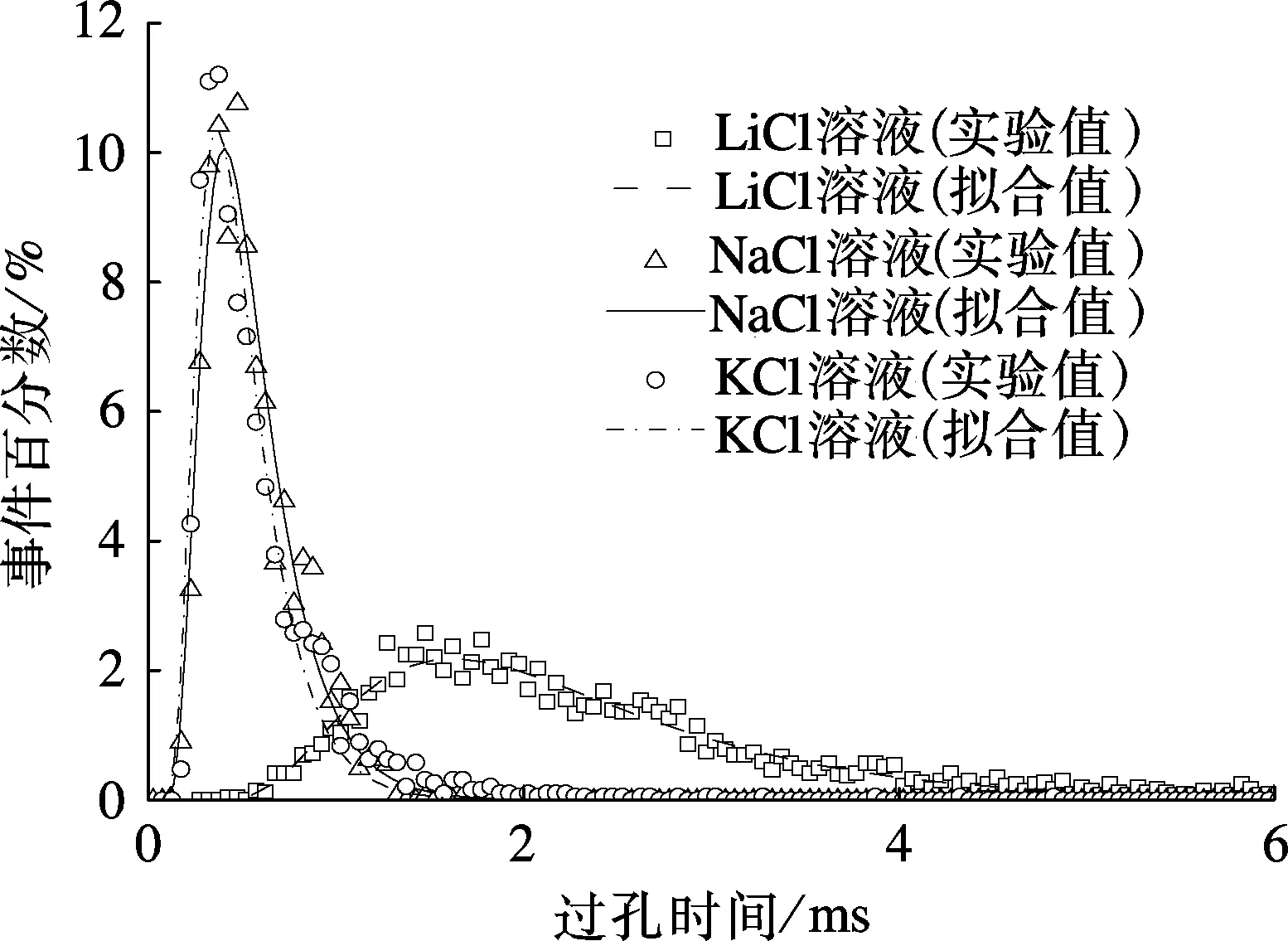
(b) 不同溶液中的DNA过孔时间分布

(c) 不同溶液中的DNA过孔时间峰值图4 DNA过孔时间分析
在溶液中,DNA的过孔速度与溶液黏度、环境温度、离子浓度、溶液pH值等因素有关[14-15].改变溶液浓度,会影响吸附在DNA表面的正离子数量[6].当pH=8.0时,DNA表面带负电荷,LiCl溶液浓度增加,则DNA表面吸附的Li+离子数增加,从而导致DNA的有效净电荷减少.因此,当DNA在电场力驱动下过孔时,其有效净电荷下降,驱动力减小,进而导致过孔速度下降,过孔时间变长[6].
另外,对比1.0mol/LLiCl,NaCl,KCl溶液中DNA的过孔时间发现(见图4(b)和(c)),1.0mol/LLiCl溶液中DNA的过孔时间最长(1.87ms),1.0mol/LNaCl溶液次之(0.35ms),1.0mol/LKCl溶液最短(0.30ms),这与文献[6]的结论一致.LiCl溶液中DNA的过孔时间分别为KCl和NaCl溶液中的6.2和5.3倍.究其原因在于:这3种阳离子与DNA表面的结合强度不一样,Li+与DNA结合能力最强,Na+次之,K+最弱[6].DNA过孔时,Li+紧紧吸附在DNA表面,导致DNA的有效净电荷减少,电场驱动力下降,过孔速度降低; 而K+容易脱落,DNA表面的净电荷数增加,导致驱动力上升,过孔速度增加.
2.3过孔方式
本实验中,纳米孔直径为38nm,DNA直径约2.2nm,即纳米孔的直径明显大于DNA的直径.当DNA在电场驱动下过孔时,可能会直接线性非折叠过孔或者折叠过孔,甚至2条DNA同时过孔.
1.0mol/LLiCl溶液中DNA的过孔形态分析结果见图5.图5(a)中出现了双Gauss峰(峰值P1=581pA,P2=819pA).根据纳米孔内DNA直径公式[12],有
ΔG=Gp-GIN=G(d)-G(dIN)
(6)
(7)
式中,d为孔的有效直径;dIN为DNA在孔内时的等效孔径.
由式(1)~(3)和式(6)~(7),可以计算出峰值P1对应的DNA直径d1=2.3nm,该数值接近双链DNA直径(2.2nm),说明此时DNA以直接线性非折叠线性方式过孔.而峰值P2对应的DNA直径d2=2.74nm,略大于双链DNA直径,可判断为DNA折叠过孔.
由图5(b)可知,实验中还存在2条DNA同时过孔的情况,但是数量较少.
图5(c)为过孔形态示意图.图中,过孔形态1为双链DNA直接线性非折叠过孔,过孔形态2为DNA链折叠过孔,过孔形态3表示折叠和非折叠的DNA链同时过孔.如图5(d)所示,3种过孔形态所对应的阻塞离子电流信号幅值分别约为600,800,1 200pA.
3 结语
本文研究了DNA在不同浓度LiCl溶液中的过孔行为特征.结果表明,在高浓度Li+环境下,DNA表面双电层对阻塞离子电流信号幅值的影响可以忽略,但是在低浓度Li+环境下时,双电层的影响会使得相对阻塞离子电流信号幅值增大.当LiCl溶液浓度由0.1mol/L增加到1.0mol/L时,DNA过孔时间随之增加,这与DNA表面吸附的阳离子数量有关.因此,增加LiCl溶液浓度可延长DNA的过孔时间,但会使相对阻塞离子电流信号幅值降低.另外,通过比较1.0mol/LLiCl,NaCl,KCl溶液的实验结果后发现,DNA的过孔时间与阳离子和DNA表面吸附强度有关.最后,对1.0mol/LLiCl溶液中DNA的过孔方式进行了分析,当纳米孔的直径明显大于DNA直径时,DNA主要以直接线性非折叠和线性折叠2种方式通过纳米孔.
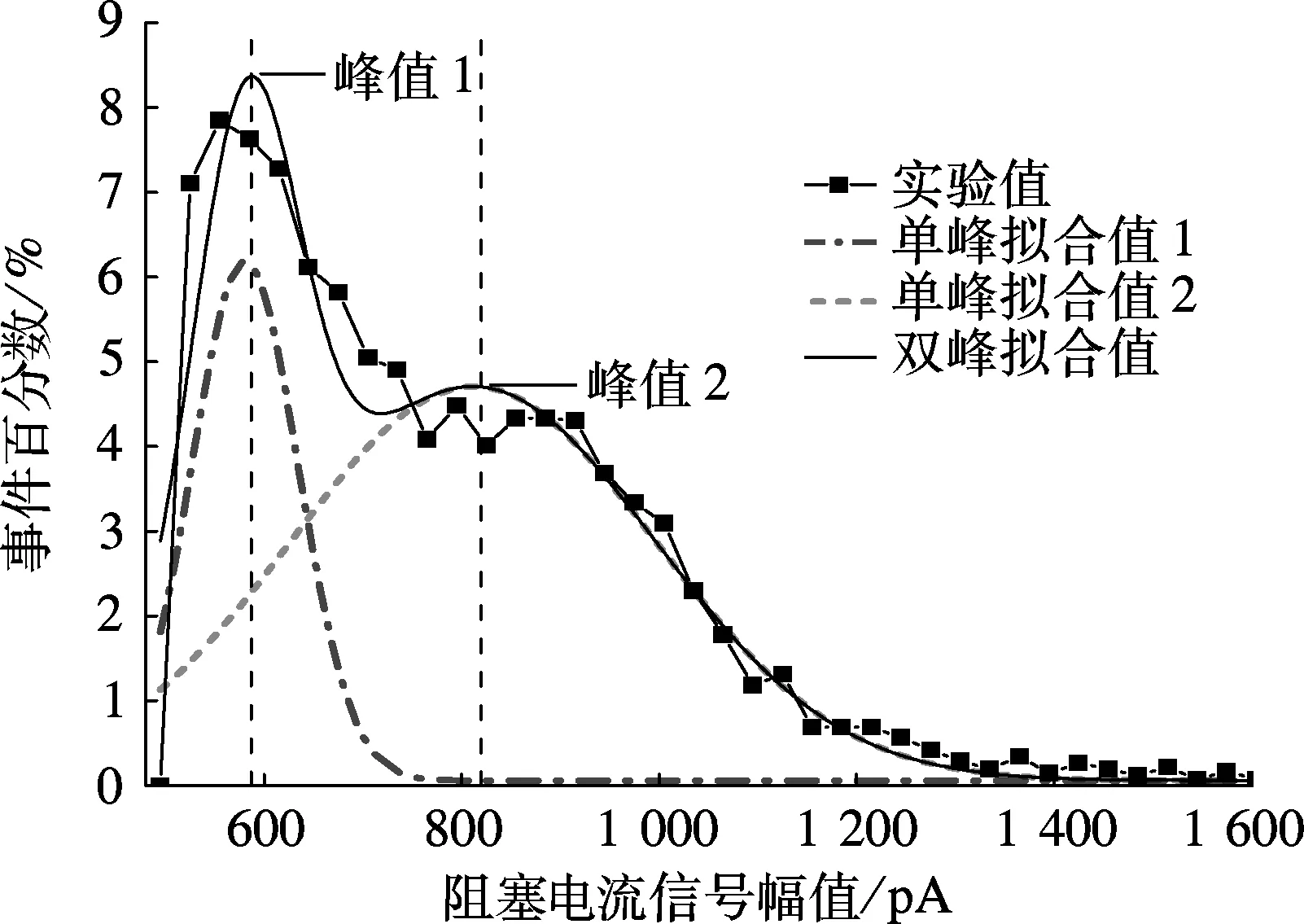
(a) 双Gauss峰值拟合的DNA阻塞离子电流信号幅值曲线
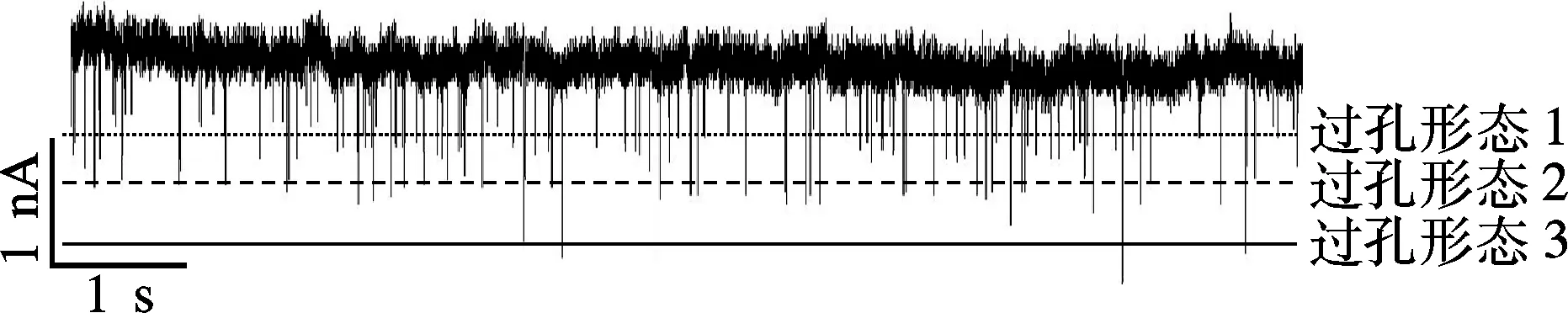
(b) 阻塞离子电流信号

(c) 过孔形态示意图
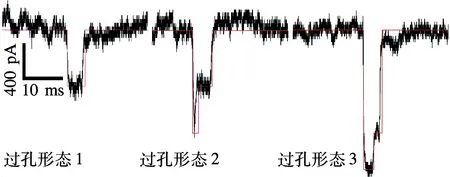
(d) 局部放大阻塞离子电流信号图5 1.0 mol/L LiCl溶液中DNA过孔形态分析
< class="emphasis_italic">References
)
[1]KasianowiczJJ,BrandinE,BrantonD,etal.Characterizationofindividualpolynucleotidemoleculesusingamembranechannel[J]. Proc Natl Acad Sci USA, 1996, 93(24):13770-13773.DOI:10. 1073/pnas. 93. 24. 13770.
[2]StormAJ,StormC,ChenJ,etal.FastDNAtranslocationthroughasolid-statenanopore[J]. Nano Lett, 2005, 5(7):1193-1197.DOI:10. 1021/nl048030d.
[3]KeyserUF,VanD,DekkerC,etal.OpticaltweezersforforcemeasurementsonDNAinnanopores[J]. Review of Scientific Instruments, 2006, 77(10):105105.
[4]MellerA,NivonL,BrantonD.Voltage-drivenDNAtranslocationsthroughananopore[J]. Phys Rev Lett, 2001, 86(15):3435-3438.DOI:10. 1103/PhysRevLett. 86. 3435.
[5]ShaJ,NiZ,LiuL,etal.Anovelmethodoffabricatingananoporebasedonaglasstubeforsingle-moleculedetection[J]. Nanotechnology, 2011, 22(17):175304.DOI:10. 1088/0957-4484/22/17/175304.
[6]KowalczykSW,WellsDB,AksimentievA,etal.SlowingdownDNAtranslocationthroughananoporeinlithiumchloride[J]. Nano Lett, 2012, 12(2): 1038-1044.
[7]ZhangY,WuG,MaJ,etal.Temperatureeffectontranslocationspeedandcapturerateofnanopore-basedDNAdetection[J]. Sci China Technol Sci, 2014, 58(3):519-525.DOI:10. 1007/s11431-014-5674-2.
[8]SmeetsRM,KeyserUF,KrapfD,etal.SaltdependenceofiontransportandDNAtranslocationthroughsolid-statenanopores[J]. Nano Lett, 2006, 6(1):89-95.DOI:10. 1021/nl052107w.
[9]HeY,TsutsuiM,ScheicherRH,etal.Mechanismofhowsalt-gradient-inducedchargesaffectthetranslocationofDNAmoleculesthroughananopore[J]. Biophys J, 2013, 105(3):776-782.DOI:10. 1016/j.bpj. 2013. 05. 065. [10]WanunuM,MorrisonW,RabinY,etal.ElectrostaticfocusingofunlabelledDNAintonanoscaleporesusingasaltgradient[J]. Nat Nanotechnol, 2010, 5(2):160-165.DOI:10. 1038/nnano. 2009. 379.
[11]KowalczykSW,GrosbergAY,RabinY,etal.ModelingtheconductanceandDNAblockadeofsolid-statenanopores[J]. Nanotechnology, 2011, 22(31):315101.DOI:10. 1088/0957-4484/22/31/315101.
[12]LiJ,FologeaD,RollingsR,etal.Characterizationofproteinunfoldingwithsolid-statenanopores[J]. Protein and Peptide Letters, 2014, 21(3): 256-265.
[13]LingDY,LingXS.OnthedistributionofDNAtranslocationtimesinsolid-statenanopores:AnanalysisusingSchrodinger’sfirst-passage-timetheory[J]. Journal of Physics-Condensed Matter, 2013, 25:37510237-1-37510237-6.
[14]MellerA,BrantonD.SinglemoleculemeasurementsofDNAtransportthroughananopore[J]. Electrophoresis, 2002, 23(16): 2583-2591.
[15]FologeaD,UplingerJ,ThomasB,etal.SlowingDNAtranslocationinasolid-statenanopore[J]. Nano Lett, 2005, 5(9): 1734-1737.
DetectionofDNAbasedonsolid-statenanoporeinLiClsolution
ShaJingjieShiHongjiaoXuBing
(JiangsuKeyLaboratoryforDesignandManufactureofMicro-NanoBiomedicalInstruments,SoutheastUniversity,Nanjing211189,China) (SchoolofMechanicalEngineering,SoutheastUniversity,Nanjing211189,China)
ThebehaviorsofDNAtranslocationthroughsolid-statenanoporesinthesolutionswithdifferentconcentrationsarestudiedbyusingtheLiClsolutionwithsmallvolumeofcationsasthebuffersolution,andthemechanismofDNAtranslocationisinvestigated.TheresultsshowthatwhentheconcentrationoftheLiClsolutionincreasesfrom0.1to1.0mol/L,therelativeionicblockagecurrentratiocausedbyDNApassingthoughporesdeclinesfrom0.014 42to0.002 79andthetimeofDNApassingthroughtheporesincreasesfrom0.30to1.87ms.Thecomparisonresultsfor1mol/LKCl,NaClandLiClsolutiondemonstratethatthedwelltimeofDNAintheLiClsolutionis6.2and5.3timesofthoseinKClandNaClsolutions,respectively,indicatingthatthedwellvelocitycanbeeffectivelyreducedbytheLiClsolution.Moreover,DNAisdriventhroughnanoporesinunfoldedandfoldedstatesintheLiClsolution.Therefore,theincreaseoftheLiClconcentrationcanprolongthedwelltimeofDNA,butreducetheamplitudeoftherelativeionicblockagecurrent.
nanopore;LiClsolution;DNA;ionicblockagecurrent;dwelltime
10.3969/j.issn.1001-0505.2016.05.014
2016-02-16.作者简介:沙菁(1980—),女,博士,副教授,major212@seu.edu.cn.
国家自然科学基金资助项目(51375092,51675101)、东南大学优秀青年教师教学科研资助项目(2242015R30002).
:10.3969/j.issn.1001-0505.2016.05.014.
TH113;Q78
A
1001-0505(2016)05-0982-05
