苏淮猪VRTN基因克隆、组织表达特征与多态性分析
2016-10-18吴一尘李平华王钧顺刘红林李齐发
吴一尘,杜 星,李平华,2,吴 艳,王钧顺,刘红林,李齐发
(1南京农业大学动物科技学院,南京 210095;2南京农业大学淮安研究院,江苏淮安 223005;3淮阴种猪场,江苏淮安 223322)
苏淮猪VRTN基因克隆、组织表达特征与多态性分析
吴一尘1,杜星1,李平华1,2,吴艳3,王钧顺3,刘红林1,李齐发1
(1南京农业大学动物科技学院,南京 210095;2南京农业大学淮安研究院,江苏淮安 223005;3淮阴种猪场,江苏淮安 223322)
【目的】获得苏淮猪VRTN基因编码区序列,了解其序列特征和组织表达特征,分析苏淮猪VRTN基因ins291位点的多态性。【方法】以苏淮猪卵巢组织cDNA为模板,采用克隆测序技术分离获得苏淮猪VRTN基因编码区序列。利用BioEdit 7.0软件分析其序列特征和蛋白理化性质。利用Clustal W软件进行核苷酸和蛋白质序列比对分析。利用UCSC基因组浏览器与NCBI基因组数据库进行猪VRTN基因的染色体定位和基因组结构分析。利用SMART软件预测蛋白质功能域,CPHmodels软件预测蛋白质三级结构。随机选取3头成年苏淮猪母猪,屠宰后立即采集心、肝、脾、肺、肾、下丘脑、肌肉和卵巢等组织,提取组织总RNA,采用RT-PCR技术分析苏淮猪VRTN基因的组织表达谱。采集106头成年苏淮猪繁殖母猪耳组织样,提取基因组DNA,采用 PCR-凝胶电泳分析苏淮猪VRTN基因ins291位点的多态性。【结果】苏淮猪VRTN基因编码区序列全长为2 097bp,与其它哺乳动物如人、小鼠、牛和羊的一致性分别为84.98%、74.53%、85.84%和86.45%,而与鸡的一致性只有54.42%。基因组结构分析发现猪VRTN基因由2个外显子和1个内含子构成,定位在猪7号染色体上。苏淮猪VRTN基因编码蛋白含有698个氨基酸残基,与人、小鼠、牛和羊等的一致性分别为85.55%、70.07%、86.20%和86.06%,但与鸡的一致性只有51.17%,可见哺乳动物VRTN基因在进化过程中比较保守。氨基酸组分分析显示苏淮猪VRTN蛋白氨基酸序列存在全部20种氨基酸,其中Leu(亮氨酸)含量最高,达到10.44%,而Asp天冬氨酸含量最低,只有1.43%。蛋白结构分析发现苏淮猪VRTN蛋白含有 HTH结构域等典型结构域,由6个α螺旋、5个β折叠以及若干无规则卷曲组成。RT-PCR分析表明VRTN基因在苏淮猪心、肝、脾、肺、肾、下丘脑、卵巢和肌肉组织等组织中均有表达,说明VRTN基因是一个广泛表达的基因。在苏淮猪群体中检测到VRTN基因ins291位点的3种基因型,其中仅有93bp条带的为野生纯合型(wt/wt),仅有384bp条带的为突变纯合型(Q/Q),含有384和93bp条带的为杂合型(wt/Q)。在苏淮猪群体中wt/wt型为优势基因型,基因型频率为0.717;wt为优势等位基因,基因频率是0.816;高脊椎数等位基因Q的频率为0.184。遗传多态性分析发现苏淮猪VRTN基因ins291位点的多态信息含量为0.331,为中度多态位点,杂合度为0.40,变异程度相对较低。【结论】 获得了苏淮猪VRTN基因序列,其表达无组织特异性;苏淮猪群体中存在高脊椎数等位基因Q。
苏淮猪;VRTN基因;克隆;组织表达谱;多态性
0 引言
【研究意义】脊椎数是影响猪胴体长和产肉量的重要经济性状,也是一个高遗传力性状(遗传力可达0.7以上)[1]。家猪的祖先野猪的胸腰椎数共有19根[2],而西方商业猪种在长期选育的过程中对体型的大小特别是体长进行了高强度的人工选择,胸腰椎数可达20—23根[3-4]。研究发现每增加一根脊椎数可使成年猪的胴体增长80mm,体重可增加4—8 kg,单位产肉效率明显提高[5-6]。因此,揭示猪脊椎数性状形成的分子机制对提高猪的产肉性能和养殖效益等均具有重要的实践意义。【前人研究进展】由于与生产性能关系密切,猪脊椎数性状在生产实践和科学研究领域均引起了广泛关注,科学家一直在寻找影响猪脊椎数的QTL、主效基因和遗传标记。2000年WADA等[7]首先利用318个遗传标记(主要为微卫星)对梅山猪×哥廷根猪资源家系进行全基因组扫描,发现在1号和2号染色体上存在影响猪脊椎数的QTL。2003年SATO等[8]利用180个微卫星标记和梅山猪×杜洛克猪家系,发现7号染色体上也存在影响猪脊椎数的QTL。2011年MIKAWA等[9]将7号染色体上影响猪脊椎数的QTL进行了精细定位,认为精细QTL内唯一的编码蛋白基因VRTN(Vertebrae Development Homolog,是人类C14orf115同源基因)是候选基因,且Q/Q型个体的脊椎数显著高于wt/wt型。随后多个课题组利用Illumina PorcineSNP60 Beadchip在不同的资源家系和群体中均发现VRTN是影响猪脊椎数的主效基因,如REN等[10]和FAN等[11]在白色杜洛克猪×二花脸猪家系,FAN等[11]在二花脸猪×通城猪家系、苏太猪群体和杜长大三元杂交猪群体,DUIJVESTEIJN等[12]在大白猪群体。欧阳子璇[13]在白色杜洛克猪×二花脸猪家系中发现VRTN基因ins291和NV027 C>A等2个位点符合因果突变的条件,其中ins291位点为291 bp的大片段插入突变(PRE1元件),为因果突变的可能性更大。FAN等[11]进一步证实ins291突变可能就是影响猪脊椎数的致因突变。【本研究切入点】近年来,在国内猪新品种(品系)培育过程中脊椎数的选育正逐渐引起关注[13]。苏淮猪是2011年国家审定的新品种,是以新淮猪为母本,与大白猪杂交培育而成的。但目前关于苏淮猪脊椎数的研究还未见报道。【拟解决的关键问题】拟以苏淮猪为研究对象,以VRTN为候选基因,克隆苏淮猪VRTN基因编码区全序列,了解其序列特征和组织表达特征,分析苏淮猪VRTN基因ins291位点的多态性,以期为揭示苏淮猪脊椎数性状形成机制和苏淮猪选育提高提供理论依据。
1 材料与方法
1.1试验时间、地点
本试验于2014年12月至2015年10月在南京农业大学动物遗传育种实验室进行。
1.2试验动物
在淮阴种猪场随机选取3头成年苏淮猪,屠宰后立即采集心、肝、脾、肺、肾、下丘脑、肌肉和卵巢等组织,置于液氮中冻藏,用于提取组织总RNA。随机选取健康无病、体况良好的成年苏淮猪繁殖母猪106头,采集耳组织样,置于装有75%酒精的离心管中,用冰盒带回实验室,用于提取基因组DNA。
1.3DNA和RNA提取
采用常规的酚/氯仿法提取苏淮猪耳基因组DNA。采用TRlzol Reagent(Invitrogen公司)提取苏淮猪各组织总RNA,采用PrimeScriptTMRT Master Mix试剂盒(Takara公司)进行反转录,具体方法详见说明书。反转录产物置于-20℃冰箱中保存。
1.4RT-PCR反应与克隆测序
根据猪VRTN基因序列(NM_001195113.1),利用Premier 5.0软件设计2对引物(P1和P2),扩增苏淮猪VRTN基因编码区序列,引物序列见表1。RT-PCR反应程序:98℃预变性30s;98℃ 5 s、退火30 s、72℃ 1.5 min 35个循环;最后72℃延伸7 min。PCR 产物用 1.5%琼脂糖凝胶电泳分离,用Gel Purification Kit凝胶回收试剂盒(Omega公司)进行PCR产物纯化。纯化产物克隆入pMD19-T vector(TaKaRa公司),并转化到DH5α感受态细胞中;采用质粒提取试剂盒(Axygen公司)提取质粒,送由苏州金唯智公司进行测序。

表1 PCR引物及反应条件Table 1 Primers and conditions of PCR
1.5序列分析
采用DNAMAN、BioEdit 7.0等生物信息学软件对苏淮猪VRTN基因编码区序列进行拼接、氨基酸序列预测和蛋白性质分析。用Clustal W(http://www.ebi. ac.uk)进行核苷酸和蛋白质序列比对分析。利用UCSC基因组浏览器(http://genome.ucsc. edu/cgi-bin/hgBlat)与NCBI基因组数据库(http://www.ncbi.nlm.nih.gov/ mapview/)进行猪VRTN基因的染色体定位和基因组结构分析。利用SMART软件(http://smart.emblheidelberg.de)预测蛋白质功能域,CPHmodels软件(http://www.cbs.dtu.dk/services/CPHmodels)预测蛋白质三级结构。哺乳动物VRTN基因序列均从GenBank数据库下载,包括人(NM_018228.2)、倭黑猩猩(XM_003824163)、小鼠(NM_001168588.1)、大鼠(XM_003750199.3)、绵羊(XM_012111602.1)、牛(NM_001206630.1)和鸡(XM_421265.4)。
1.6苏淮猪VRTN基因组织表达谱分析
以GAPDH基因为内参,根据猪VRTN基因(NM_001195113.1)和GAPDH基因mRNA序列(NM_001206359.1)设计引物P3、P4(表1),以苏淮猪各组织cDNA为模板进行RT-PCR扩增。PCR产物进行1.5% 琼脂糖凝胶电泳,观察结果并拍照记录。
1.7苏淮猪VRTN基因ins291位点的多态性分析
根据文献[11]中报道的影响猪脊椎数性状的VRTN基因的g.20311_20312 ins291位点,设计引物P5(表1),以苏淮猪基因组DNA为模板进行PCR扩增。扩增产物用 1.5%琼脂糖凝胶电泳分离,根据条带大小对苏淮猪不同个体VRTN基因ins291位点进行分型。多态信息含量和杂合度等的计算方法见文献[14]。
2 结果
2.1苏淮猪VRTN基因PCR扩增
以苏淮猪肌肉组织cDNA为模板,利用引物P1与P2进行PCR扩增,PCR扩增产物经过琼脂糖凝胶电泳检测,得到特异性良好且与目的片段大小一致的扩增片段(图1)。测序后发现扩增片段长度分别为1 328和898 bp,与引物设计时预期长度一致。
2.2苏淮猪VRTN基因编码区序列分析
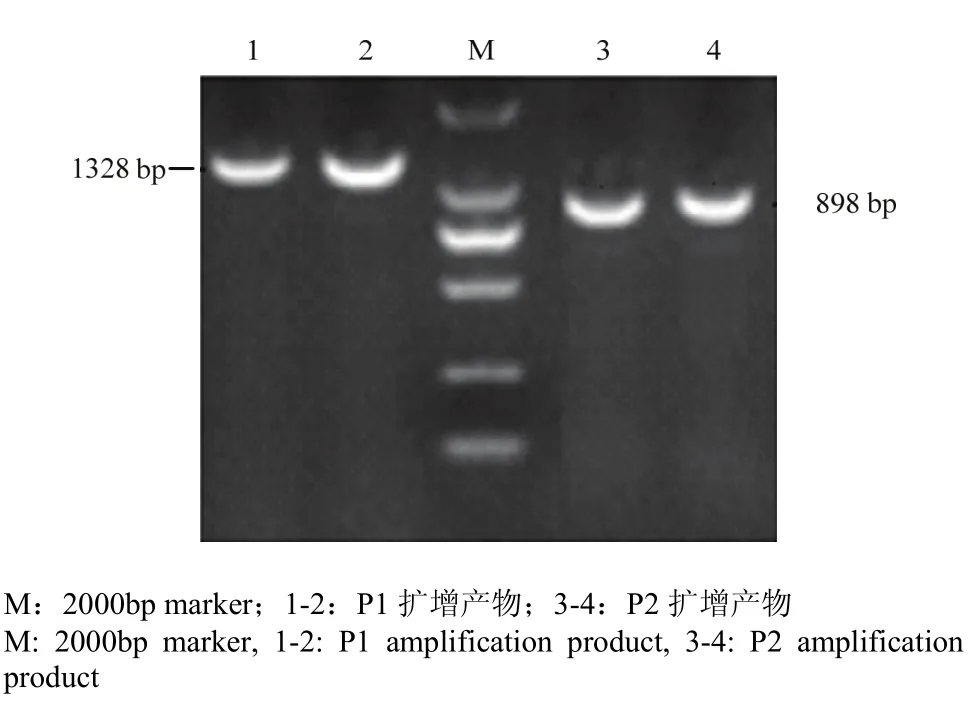
图1 苏淮猪VRTN基因PCR扩增产物电泳图谱Fig. 1 Agarose gel photograph of VRTN gene in Suhuai pig

图2 苏淮猪VRTN基因核苷酸序列与预测的氨基酸序列Fig. 2 Nucleotide and amino acid sequence of VRTN gene in Suhuai pig
通过克隆测序和序列拼接获得了苏淮猪VRTN基因完整的编码区序列,序列长度为2 097 bp(图2)。采用BioEdit 7.0软件对苏淮猪VRTN基因编码区核苷酸序列的碱基组成进行分析,结果发现CG含量(64.27%)明显高于AT含量(35.73%)。BLAST比对发现苏淮猪VRTN基因编码区核苷酸序列与引物源序列(NM_001195113.1)的一致性为99.95%。苏淮猪VRTN基因编码区核苷酸序列与其它哺乳动物如人、倭黑猩猩、小鼠、大鼠、牛和羊的一致性分别为84.98%、85.12%、74.53%、73.68%、85.84%和86.45%,而与鸡的一致性只有54.42%。哺乳动物相近物种间VRTN基因编码区核苷酸序列的一致性更高,如人和倭黑猩猩(99.38%)、牛和羊(96.48%)。电子染色体定位发现VRTN基因定位在猪7号染色体上,介于AbcD4基因(XM_013989158.1)与SYNDIG1L基因(XM_013989161.1)之间。基因组结构分析发现猪VRTN基因由2个外显子和1个内含子构成。
2.3苏淮猪VRTN蛋白氨基酸序列分析

图3 苏淮猪 VRTN基因氨基酸序列与其他哺乳动物的同源性比较Fig. 3 Identical alignments of VRTN amino acid sequence in Suhuai pig with other mammals
苏淮猪VRTN基因编码蛋白含有698个氨基酸残基(图3)。氨基酸组分分析显示苏淮猪VRTN蛋白氨基酸序列存在全部20种氨基酸,其中Leu(亮氨酸)含量最高,达到10.44%,而Asp天冬氨酸含量最低,只有1.43%。氨基酸残基中酸性氨基酸(Asn、Asp、Gln、Glu)有98个,碱性氨基酸(Arg、His、Lys)有89个;正电荷残基(Asp、Glu)有52个,负电荷残基(Arg、Lys)有75个。同源性分析发现苏淮猪VRTN蛋白氨基酸序列与人、倭黑猩猩、小鼠、大鼠、牛和羊等的一致性分别为85.55%、84.80%、70.07%、70.33%、86.20%和86.06%,但与鸡的一致性只有51.17%。同样地,哺乳动物相近物种间VRTN蛋白氨基酸序列一致性更高,如人和倭黑猩猩(99.00%)、牛和羊(96.71%)。
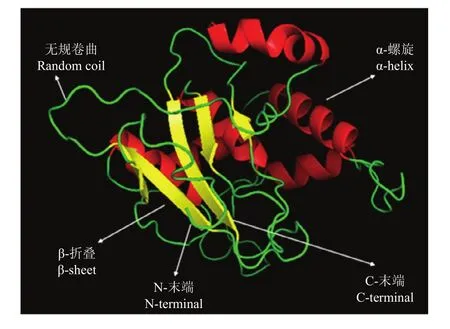
图4 苏淮猪VRTN蛋白三级结构预测Fig. 4 Space structure prediction analysis of VRTN protein in Suhuai pig
2.4苏淮猪VRTN基因编码蛋白特征分析
苏淮猪VRTN蛋白的分子量为77.8467kD,理论等电点pI为 9.37,体外半衰期为30 h,不稳定系数63.41,疏水性均值(GRAVY)为-0.274。ProtScale和BioEdit软件分析发现在苏淮猪VRTN蛋白270—320、400—450、550—600氨基酸之间有3个较强的亲水性区域,说明苏淮猪VRTN蛋白具有较强的亲水性。NCBI与SMART在线软件对VRTN蛋白功能结构域进行分析,结果并未在苏淮猪VRTN蛋白上发现跨膜结构,但发现苏淮猪VRTN蛋白氨基酸序列存在2个低复杂区域(LCR)(5Glu—20Glu;247Glu—275Ser)和2个内部重复序列(RPT)(291Val—436Arg;530Ser—687Ala),另外检测到一个HTH结构域(276Gln—306Glu)(图3)。利用CPHmodels在线工具进行苏淮猪VRTN蛋白质三级结构预测分析,结果见图4。从图4可以看出VRTN蛋白包含6个α螺旋、5个β折叠以及若干无规则卷曲结构。
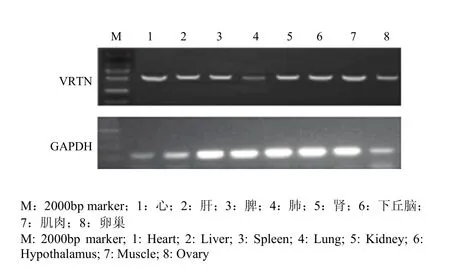
图5 苏淮猪VRTN基因组织表达谱Fig. 5 Normal expression profile of VRTN gene in Suhuai pig
2.5苏淮猪中VRTN基因组织表达谱分析
以苏淮猪各个组织cDNA为模板,利用引物P3与P4进行RT-PCR分析,PCR产物的琼脂糖凝胶电泳结果见图5。可以看出,在苏淮猪心、肝、脾、肺、肾、下丘脑、卵巢和肌肉等组织中均检测到VRTN基因的表达,说明VRTN基因是一个广泛表达的基因。
2.6苏淮猪VRTN基因ins291位点多态性分析
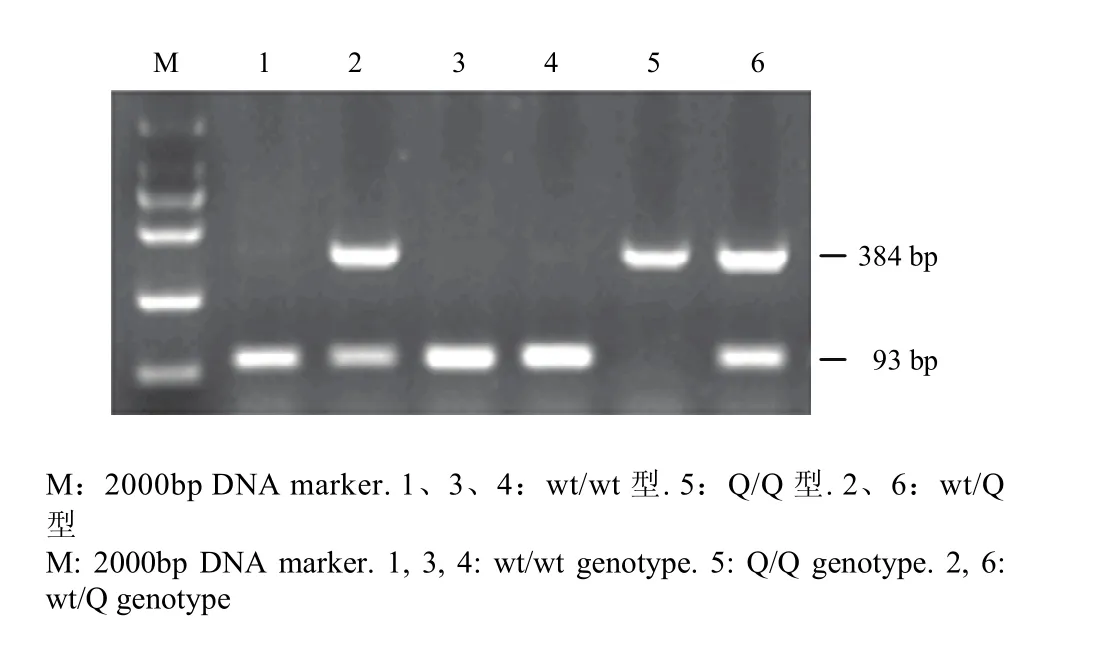
图6 苏淮猪VRTN基因ins291位点基因分型Fig. 6 The genotype patterns of the VRTN ins 291 mutation in Suhuai pig individuals
在苏淮猪群体中检测到VRTN基因ins291位点的3种基因型(图6),其中仅有93bp条带的为野生纯合型(wt/wt),仅有384 bp条带的为突变纯合型(Q/Q),含有384和93 bp条带的为杂合型(wt/Q)。苏淮猪VRTN基因ins291位点的基因型频率、基因频率分析结果见表2。在苏淮猪群体中wt/wt型为优势基因型,基因型频率为0.717;wt为优势等位基因,基因频率是0.816。另外,遗传多态性分析发现苏淮猪VRTN基因ins291位点的多态信息含量为0.331,为中度多态位点,杂合度为0.40,变异程度相对较低。
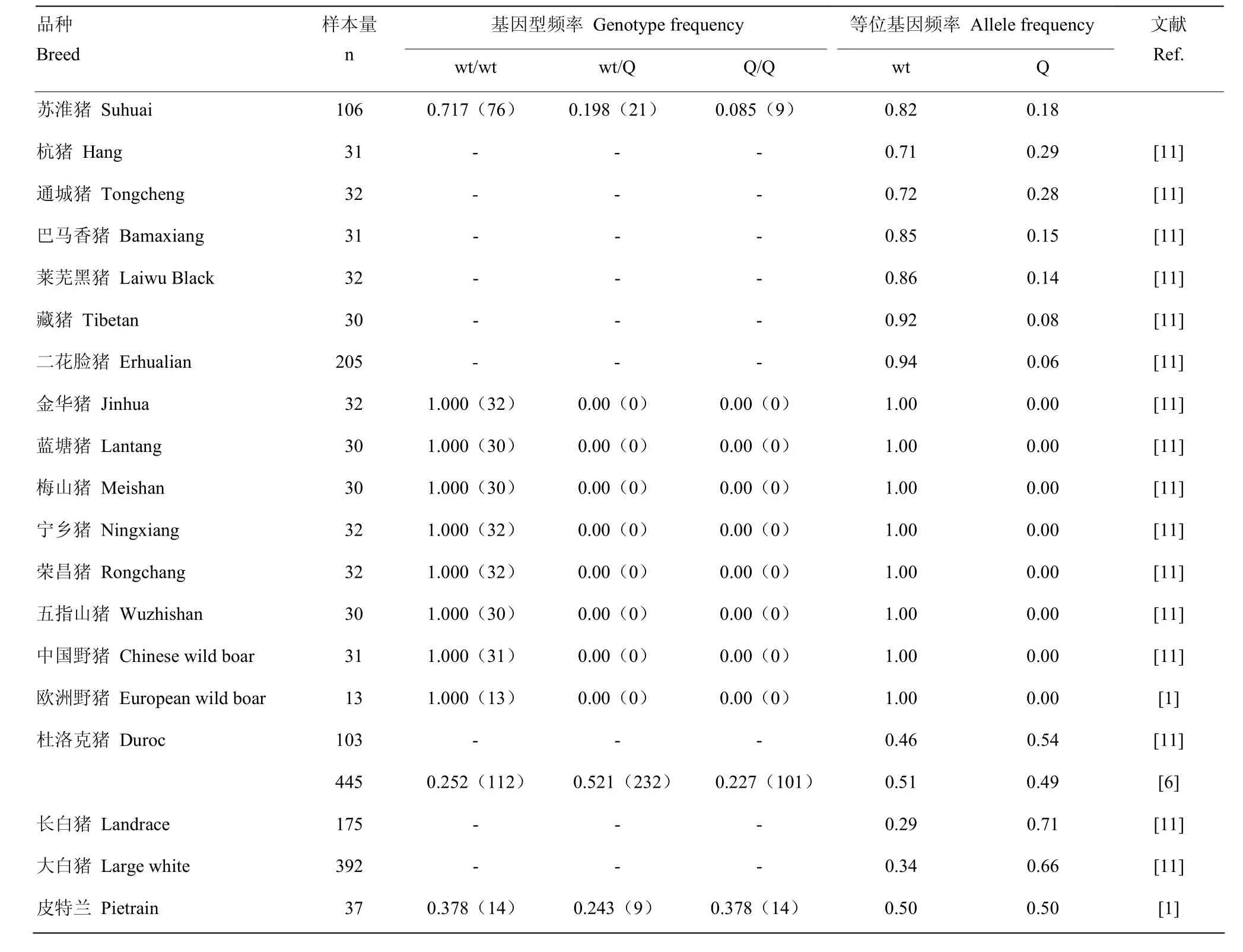
表2 苏淮猪和其它猪种VRTN基因ins291位点的基因型频率和基因频率Table 2 Polymorphism of VRTN ins291 in Suhuai pig and other pig breeds
3 讨论
研究表明在猪基因组上存在着大量的QTL,而在这些QTL内一些候选基因的多态性与猪性状之间存在着潜在的联系,例如IGF2基因内含子3上g.3072G>A位点多态性与猪肌肉生长高度关联[15],MC4R基因上 p.D298N(c.892G>A)位点错义突变导致猪采食量和日增重都显著减少[16],VRTN基因ins291突变是影响猪脊椎数的致因突变[11],PPARD基因G32E 位点多态性与猪耳朵面积显著关联[17],PHKG1基因g.8283C>A突变引起猪肌糖原含量升高和肉质下降[18],等等。VRTN基因位于猪7号染色体上,介于控制脂肪分解和乳头数性状的数量性状基因座之间[9,12]。猪VRTN基因全长9 570 bp,有2个外显子,其中一个是编码蛋白[19]。本文通过克隆测序获得了苏淮猪VRTN基因编码区序列,发现苏淮猪核甘酸(氨基酸)序列与其它哺乳动物比较的一致性均高于73%(70%),明显低于猪某些基因和哺乳动物其它物种的同源性,如Smad4基因(高于86%和98%)[20]、ECE基因(高于87%和96%)[21]、Rb1基因(高于89%和91%)[22]和MSRB3基因(高于84%和88%)[23],而与猪NR5A2基因[24]相近,说明哺乳动物VRTN基因在进化过程中相对保守,这与前人研究发现VRTN基因位于突变位点较为常见的区域是一致的[9,12]。苏淮猪VRTN蛋白特征预测发现VRTN蛋白不具备跨膜功能,包含2个LCR和2个RPT区域,以及1个HTH结构域。研究发现HTH结构域包含螺旋-反转-螺旋基础结构,为一个序列特异性结构单元,常见于转录调节因子蛋白,用以识别靶基因的转录调控序列[25],提示VRTN蛋白可能具有调控转录的功能,但目前尚未见到相关研究报道。
VRTN基因是影响猪脊椎数性状的主效基因,于2011年首先在大白猪群体中被鉴定[9]。VRTN基因多态性被认为是影响猪脊椎数变异的主要因素,MIKAWA等[9]在猪VRTN基因中检测到9个突变位点,其中单倍型Q比单倍型wt的脊椎数多1.02根,且这些突变影响VRTN基因在胚胎发育早期的转录,其中单倍型Q型高于单倍型wt。进一步分析发现,NV123位点中Q等位基因是插入了一个291bp的PRE1元件(一种猪SINE元件)形成的,而SINE元件可以作为增强子元件调控相应基因的表达[9,26]。欧阳子璇[13]利用白色杜洛克猪×二花脸猪家系研究认为NV123ins291位点可能是影响猪脊椎数性状的因果突变位点(QTN),随后FAN等[11]进一步证实了这个观点。本研究以ins291位点为对象,分析了苏淮猪群体VRTN基因ins291位点的多态性,结果在苏淮猪群体中发现了高脊椎数等位基因的存在,Q等位基因的频率为0.18,明显低于另一个等位基因wt的频率(0.82)。研究发现在西方猪商业化猪种中VRTN基因ins291位点Q等位基因频率均较高,例如杜洛克猪、长白猪、大白猪和皮特兰猪中Q等位基因频率分别高达0.54、0.71、0.66和0.50(表2),这与西方猪种脊椎数较多和体长较长是一致的[1,11]。国内地方猪种中一般检测不到Q等位基因或其频率较低,如梅山猪、金华猪等Q等位基因频率均为0[11]。中外猪种脊椎数和VRTN基因ins291位点Q等位基因频率的差异,是因为西方猪种在近、现代选育过程中注重对猪体长的选育,高强度地选育了具有较多脊椎数的品种,从而人为的提高了西方猪种Q等位基因频率[27]。在苏淮猪群体中检测到VRTN基因ins291位点Q等位基因,可能与苏淮猪血统有关,即在苏淮猪的培育过程中引入了西方猪种大白猪的血统[28]。
4 结论
本研究分离了苏淮猪脊椎数主效基因VRTN基因编码区全序列,了解了其基因特征和蛋白性质。RT-PCR发现VRTN基因在苏淮猪各种组织中广泛表达。在苏淮猪群体中鉴定出VRTN 基因ins291位点高脊椎数等位基因Q的存在,为苏淮猪胴体长和产肉性状的进一步选育提高提供了理论基础和分子标记。
References
[1] URGOS C, LATORRE P, ALTARRIBA J, CARRODEGUAS J A,VARONA L, LÓPEZ-BUESA P. Allelic frequencies of NR6A1 and VRTN, two genes that affect vertebrae number in diverse pig breeds: a study of the effects of the VRTN insertion on phenotypic traits of a Duroc × Landrace-Large White cross. Meat Science, 2015, 100: 150-155.
[2] MIKAWA S, MOROZUMI T, SHIMANUKI S, HAYASHI T,UENISHI H, DOMUKAI M, OKUMURA N, AWATA T. Fine mapping of a swine quantitative trait locus for number of vertebrae and analysis of an orphan nuclear receptor, germ cell nuclear factor(NR6A1). Genome Research, 2007. 17(5): 586-593.
[3] KING J, ROBERTS R. Carcass length in the bacon pig: its association with vertebrae numbers and prediction from radiographs of the young pig. Animal Production Science, 1960, 2: 59-65.
[4] BERGE S. Genetical researches on the number of vertebrae in the pig. Animal Science, 1948, 7: 233-238.
[5] MIKAWA S, HAYASHI T, NII M, SHIMANUKI S, MOROZUMI T,AWATA T. Two quantitative trait loci on Sus scrofa chromosomes 1 and 7 affecting the number of vertebrae. Journal of Animal Science,2005, 83(10): 2247-2254.
[6] NAKANO H, SATO S, UEMOTO Y, KIKUCHI T, SHIBATA T, KADOWAKI H, KOBAYASHI E, SUZUKI K. Effect of VRTN gene polymorphisms on Duroc pig production and carcass traits, and their genetic relationships. Animal Science Journal, 2015, 86(2): 125-131.
[7] WADA Y, AKITA T, AWATA T, FURUKAWA T, SUGAI N, INAGE Y,ISHII K, ITO Y, KOBAYASHI E, KUSUMOTO H, MATSUMOTO T,MIKAWA S, MIYAKE M, MURASE A, SHIMANUKI S, SUGIYAMA T, UCHIDA Y, YANAI S, YASUE H. Quantitative trait loci (QTL)analysis in a Meishan x Gottingen cross population. Animal Genetics,2000, 31(6): 376-384.
[8] SATO S, OYAMADA Y, ATSUJI K, NADE T, KOBAYASHI E,MITSUHASHI T, NIRASAWA K, KOMATSUDA A, SAITO Y,TERAI S, HAYASHI T, SUGIMOTO Y. Quantitative trait loci analysis for growth and carcass traits in a Meishan x Duroc F2resource population. Journal of Animal Science, 2003, 81(12):2938-2949.
[9] MIKAWA S, SATO S, NII M, MOROZUMI T, YOSHIOKA G,IMAEDA N, YAMAGUCHI T, HAYASHI T, AWATA T. Identification of a second gene associated with variation in vertebral number in domestic pigs. BMC Genetics, 2011, 12: 5.
[10] REN D R, REN J, RUAN G F, GUO Y M, WU L H, YANG G C,ZHOU L H, LI L, ZHANG Z Y, HUANG L S. Mapping and fine mapping of quantitative trait loci for the number of vertebrae in a White Duroc × Chinese Erhualian intercross resource population. Animal Genetics, 2012, 43(5): 545-551.
[11] FAN Y, XING Y, ZHANG Z, AI H, OUYANG Z, OUYANG J, YANG M, LI P, CHEN Y, GAO J, LI L, HUANG L, REN J. A further look at porcine chromosome 7 reveals VRTN variants associated with vertebral number in Chinese and Western pigs. PloS One, 2013, 8(4): e62534.
[12] DUIJVESTEIJN N, VELTMAAT J M, KNOL E F, HARLIZIUS B. High-resolution association mapping of number of teats in pigs reveals regions controlling vertebral development. BMC Genomics,2014, 15: 542.
[13] 欧阳子璇. 猪7号染色体上影响脊椎变异的VRTN因果突变的筛选和验证[D]. 南昌: 江西农业大学, 2012.
OUYANG Z X. Identification of VRTN causal variations for vertebral numbers on pig chromosome 7[D]. Nanchang: Jiangxi Agricultural University, 2012. (in Chinese)
[14] 吴井生, 王金玉. FSHR基因第10外显子多态性及其与小梅山猪产仔数的相关性. 中国农业科学, 2012, 45(13): 2728-2736.
WU J S, WANG J Y. Polymorphism of exon10 of FSHR gene and its relationship with litter size in Xiaomeishan pigs. Scentia Agricultura Sinica, 2012, 45(13): 2728-2736. (in Chinese)
[15] VAN LAERE A S, NGUYEN M, BRAUNSCHWEIG M, NEZER C,COLLETTE C, MOREAU L, ARCHIBALD A L, HALEY C S,BUYS N, TALLY M, ANDERSSON G, GEORGES M, ANDERSSON L. A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig. Nature, 2003, 425(6960): 832-836.
[16] KIM K S, LARSEN N, SHORT T, PLASTOW G, ROTHSCHILD M F. A missense variant of the porcine melanocortin-4 receptor (MC4R)gene is associated with fatness, growth, and feed intake traits. Mammalian Genome, 2000, 11: 131-135.
[17] REN J, DUAN Y, QIAO R, YAO F, ZHANG Z, YANG B, GUO Y,XIAO S, WEI R, OUYANG Z, DING N, AI H, HUANG L. A missense mutation in PPARD causes a major QTL effect on ear size in pigs. PLoS Genetics, 2011, 7(5): e1002043.
[18] MA J, YANG J, ZHOU L, REN J, LIU X, ZHANG H, YANG B, ZHANG Z, MA H, XIE X, XING Y, GUO Y, HUANG L. A splice mutation in the PHKG1 gene causes high glycogen content and low meat quality in pig skeletal muscle. PLoS Genetics, 2014, 10(10): e1004710.
[19] HIROSE K, MIKAWA S, OKUMURA N, NOGUCHI G, FUKAWA K, KANAYA N, MIKAWA A, ARAKAWA A, ITO T, HAYASHI Y,TACHIBANA F, AWATA T. Association of swine vertnin (VRTN)gene with production traits in Duroc pigs improved using a closed nucleus breeding system. Animal Science Journal, 2013, 84(3): 213-221.
[20] 赵永祥, 刘吉英, 潘增祥, 张久峰, 姚勇, 周吉隆, 谢庄, 徐银学, 刘红林, 李齐发. 二花脸猪Smad4基因的克隆与卵巢组织mRNA的表达水平. 中国农业科学, 2012, 45(23): 4883-4890.
ZHAO Y X, LIU J Y, PAN Z X, ZHANG J F, YAO Y, ZHOU J, XIE Z,XU Y X, LIU H L, LI Q F. Cloning and mRNA expression of Smad4 gene in ovaries of Erhualian pig. Scentia Agricultura Sinica, 2012,45(23): 4883-4890. (in Chinese)
[21] WANG Y D, ZHANG J, LI C H, XU H P, CHEN W, ZENG Y Q,WANG H. Molecular cloning, sequence characteristics, and tissue expression analysis of ECE1 gene in Tibetan pig. Gene, 2015, 571(2): 237-244.
[22] HU X, LUO P, PENG X, SONG T, ZHOU Y, WEI H, PENG J, JIANG S. Molecular cloning, expression pattern analysis of porcine Rb1 gene and its regulatory roles during primary dedifferentiated fat cells adipogenic differentiation. General and Comparative Endocrinology,2015, 214: 77-86.
[23] ZHANG Y, LIANG J, ZHANG L, WANG L, LIU X, YAN H, ZHAO K, SHI H, ZHANG T, LI N, PU L, WANG L. Porcine methionine sulfoxide reductase B3: molecular cloning, tissue-specific expression profiles, and polymorphisms associated with ear size in Sus scrofa. Journal of Animal Science and Biotechnology, 2015, 6: 60.
[24] 姚勇, 潘增祥, 张久峰, 赵永祥, 谢庄, 刘红林, 徐银学, 李齐发.二花脸猪NR5A2基因克隆与卵巢组织转录水平分析. 南京农业大学学报, 2013, 36(3): 133-138.
YAO Y, PAN Z X, ZHANG J F, ZHAO Y X, XIE Z, LIU H L, XU Y X, LI Q F. Cloning and trascriptional level of NR5A2 gene in ovaries of Erhualian pig. Journal of Nanjing Agricultural University, 2013,36(3): 133-138. (in Chinese)
[25] ZHANG C, ZHOU Z, ZHANG W, CHEN Z, SONG Y, LU W, LIN M,CHEN M. The site-directed A184S mutation in the HTH domain of the global regulator IrrE enhances Deinococcus radiodurans R1 tolerance to UV radiation and MMC shock. Journal of Microbiology and Biotechnology, 2015, 25(12): 2125-2134.
[26] SASAKI T, NISHIHARA H, HIRAKAWA M, FUJIMURA K,TANAKA M, KOKUBO N, KIMURA-YOSHIDA C, MATSUO I,SUMIYAMA K, SAITOU N, SHIMOGORI T, OKADA N. Possible involvement of SINEs in mammalian-specific brain formation. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 4220-4225.
[27] FONTANESI L, SCHIAVO G, SCOTTI E, GALIMBERTI G,CALÒ D G, SAMORÈ A B, GALLO M, RUSSO V, BUTTAZZONI L. A retrospective analysis of allele frequency changes of major genes during 20 years of selection in the Italian Large White pig breed. Journal of Animal Breeding and Genetics, 2015, 132(3): 239-246.
[28] 李隐侠, 徐银学, 陈杰, 于传军. 苏淮猪7个功能基因的多态性及其与生产性能的相关性. 南京农业大学学报, 2008, 31(3): 102-106.
LI Y X, XU Y X, CHEN J, YU C J. Polymorphsims of seven functional genes and their corrlation to production performance in Suhuai pigs. Journal of Nanjing Agricultural University, 2008, 31(3): 102-106. (in Chinese)
(责任编辑林鉴非)
Sequence Cloning, Tissue Expression Profile and Polymorphism of VRTN Gene in Suhuai Pig
WU Yi-chen1, DU Xing1, LI Ping-hua1,2, WU Yan3, WANG Jun-shun3, LIU Hong-lin1, LI Qi-fa1
(1College of Animal Science and Technology, Nanjing Agriculture University, Nanjing 210095;2Huaian Academy of Nanjing Agricultural University, Huaian 223005, Jiangsu;3Huaiyin Pig Breeding Farm, Huaian 223322, Jiangsu)
【Objective】The aim of this study is to obtain the coding region sequence of Suhuai VRTN gene, identify its characteristics and expression patterns, analyze the polymorphism of VRTN ins291 in Suhuai pig population. 【Method】The codingsequence of Suhuai pig VRTN gene was isolated by cloning and sequencing. The BioEdit 7.0 software was used to analyze the molecular characteristics and physicochemical properties of Suhuai pig VRTN. Nucleotide and amino acid sequences alignment was performed using Clustal W. Genomic organization and chromosomal locations were investigated by comparing the cDNA and corresponding genomic sequence (UCSC server and NCBI database). Motif analysis was performed using the online programs SMART, and protein tertiary structure was predicted by CPHmodels server. Tissue samples of heart, liver, spleen, lung, kidney,hypothalamus, ovary and skeleton muscle were obtained from adult female Suhuai pigs (n=3). Total RNA was extracted using a Trizol kit, and the expression patterns of VRTN gene of Suhuai pig were analyzed by RT-PCR. The ear samples were obtained from adult female Suhuai pigs (n=106), and genomic DNA was extracted by a conventional phenol-chloroform extraction method. PCR-Gel electrophoresis was performed to analyze the polymorphism of VRTN ins291. 【Result】The full length of the coding sequences of Suhuai pig VRTN gene is 2097 bp, which shared similarities of 84.98%, 74.53%, 85.84% and 86.45% to human, mouse,cattle and sheep, respectively, but only 54.42% to chicken. Genomic structure analysis showed that pig VRTN gene includes 2 exons and 1 intron, and is located on chromosome 7. The Suhuai pig VRTN gene encodes 698 amino acid residues, which shared similarities of 85.55%, 70.07%, 86.20% and 86.06% to human, mouse, cattle and sheep, respectively, but only 51.17% to chicken,indicated that VRTN is conserved in the evolution process of mammalian. Protein structure analysis found that VRTN protein of Suhuai pig contains several typical domains including HTH domain, and comprised 6 α-helices, 5 β-sheets and many random coils. Real-time PCR analysis revealed that the VRTN gene is expressed in heart, liver, spleen, lung, kidney, hypothalamus, ovary and skeleton muscle of Suhuai pig, suggesting that VRTN is widely expressed in various tissues of pig. Genotyping ins291 of VRTN gene in 106 Suhuai pigswas done, and three genotypes were detected. In Suhuai pigs, the frequencies of wt/wt, wt/Q and Q/Q genotypes were 0.717, 0.198 and 0.085. The frequencies of high vertebrate number of Q allele was 0.184. The polymorphic information content of VRTN ins291 in Suhuai pigs was 0.331. The heterozygosity of VRTN ins291 in Suhuai pigs was 0.40.【Conclusion】 The coding region of VRTN gene was isolated, and its expression showed no tissue-specificity. The presence of high number of vertebrate Q allele in Suhuai pig population was confirmed.
Suhuai pig; VRTN gene; cloning; tissue expression profile; polymorphism
2015-12-26;接受日期:2016-07-08
国家科技支撑计划(2015BAD03B01)、江苏省农业科技自主创新资金(SCX(12)2097)、南京农业大学SRT计划(1515A08)
联系方式:吴一尘,E-mail:wuyichen@njau.edu.cn。杜星,E-mail:2014205003@njau.edu.cn。吴一尘和杜星为同等贡献作者。通信作者李齐发,E-mail:liqifa@njau.edu.cn
