陆地棉GhPYR1基因的克隆和功能分析
2016-10-13刘妍孟志刚孙国清王远周焘郭三堆张锐
刘妍 孟志刚 孙国清 王远 周焘 郭三堆 张锐
(中国农业科学院生物技术研究所,北京 100081)
陆地棉GhPYR1基因的克隆和功能分析
刘妍 孟志刚 孙国清 王远 周焘 郭三堆张锐
(中国农业科学院生物技术研究所,北京 100081)
植物激素脱落酸(Abscisic acid,ABA)在植物应对干旱、盐碱等逆境胁迫以及植物种子萌发、根伸长、芽休眠等阶段发挥重要作用。PYR/PYL/RCAR蛋白家族是ABA受体,与ABA结合后能够启动ABA信号传导通路,诱导ABA应答基因的表达。利用电子克隆和RT-PCR技术从陆地棉中克隆了GhPYR1基因,其编码的GhPYR1蛋白与拟南芥中AtPYR1蛋白相似度为73%。将GhPYR1蛋白序列与拟南芥14个PYR/PYL/RCAR家族成员蛋白序列进行比对并构建进化树,发现它与拟南芥PYR/PYL/RCAR蛋白亚家族III亲缘关系最近。过表达GhPYR1基因的T3代拟南芥在外源ABA处理下,其种子萌发和初期根生长均滞后于野生型,表现出对ABA更加敏感;高盐和干旱胁迫对转基因种子的萌发抑制更强烈,但苗期胁迫处理下转基因拟南芥的长势却明显优于野生型;同时在外源ABA诱导条件下ABA应答基因RD29A、RAB18的表达量较野生型有明显提高。以上结果说明GhPYR1基因编码的蛋白是ABA的受体,过表达该基因能够提高植物对ABA的敏感性和增强应对逆境胁迫的能力。
陆地棉;GhPYR1基因;生物信息学分析;生理功能分析
植物激素脱落酸(Abscisic acid,ABA),在植物应对干旱、盐碱等非生物胁迫及植物种子成熟、休眠等发育过程中发挥着重要作用[1,2]。2009年,Ma和Park[3,4]两个实验团队分别通过遗传筛选和酵母双杂交的方法,在拟南芥中证实了PYR/ PYL/RCAR蛋白是ABA的受体。拟南芥中该家族有14个成员,分别命名为PYR1和PYL1-13,它们都是可溶性蛋白质,含有START(STAR-RELATED LIPID-TRANSFER)特征区域的蛋白质,分布于细胞质和细胞核内[5]。PYR/PYL/RCAR家族蛋白是ABA信号通路的起点,而ABA信号通路是一个双重负调控系统[6,7],核心组分除PYR/PYL/RCAR外,还包括PP2Cs、SnRK2s。在通常情况下,未结合ABA的PYR/PYL/RCAR蛋白不与PP2C互作,PP2C通过解磷酸化抑制SnRK2的活性。当应答来自环境或发育的信号时,植物体内ABA的含量增加,并与PYR/PYL/RCAR结合使其发生变构[8-10],然后与PP2C互作,抑制PP2C的磷酸酶活性[3,4],从而使SnRK2被激活,活化的SnRK2磷酸化下游的转录因子[11,12],启动ABA应答基因RD29A、RAB-18[13,14]等的表达。继拟南芥之后,其他高等植物,如玉米、大豆、水稻、草莓、青蒿等[15-18]的ABA受体研究也逐步展开,结果表明,在这些作物中过表达PYR/PYL/RCAR家族基因成员,能够提高转基因作物对ABA的敏感性、抗旱性和促进果实成熟等。
本研究克隆了陆地棉中GhPYR1基因全长,构建了过表达载体,获得了过表达GhPYR1基因的T3代拟南芥,并初步鉴定转GhPYR1基因的拟南芥在种子萌发、根生长期间对ABA、盐碱和干旱的敏感性以及苗期耐逆性和ABA应答基因的表达情况,旨在为解析ABA通路与植物逆境胁迫应答的关系提供依据。
1 材料与方法
1.1 材料
1.1.1 植物材料 陆地棉Gossypium hirsutum L. Y18、拟南芥Columbia Col-0由本实验室保存。
1.1.2 载体和菌株 Gateway入门载体Hbskb1-HI-380、Gateway目的载体pEarelyGate330、农杆菌GV3101感受态均由本实验室保存;大肠杆菌Trans-T1感受态和克隆载体pEASYTM-Blunt Zero购于北京全式金生物技术有限公司。
1.1.3 仪器与试剂 荧光定量PCR仪(Bio-Rad公司chromo4);原平皓公司RNA提取试剂盒;东洋纺公司ReverTra Ace反转录试剂盒;天根公司Fast HiFidelity PCR Kit;Promega公司GoTag qPCR Master Mix;Invitrogen公司 Gateway LR Clonase ⅡEnzyme Mix;各化学试剂均购于Sigma公司。
1.2 方法
1.2.1 DNA的提取和cDNA和合成 以陆地棉Y18叶片为材料,使用改良的CTAB法提取棉花的基因组DNA;RNA的提取过程参照EASYspin RNA提取试剂盒,提取的RNA用ReverTra Ace反转录试剂盒反转录成cDNA。反应体系20 μL:先将RNA 11 μL、Oligo(dT)201 μL混合后,65℃ 5 min;再加入5×RT Buffer 4 μL、RNase inhibitor(10 U/μL)1 μL、ReverTra Ace 1 μL、dNTP Mix(10 mmol/L)2 μL。反应条件:42℃ 30 min,99℃ 5 min,4℃ 5 min。

表1 引物序列表
1.2.2 陆地棉GhPYR1基因编码区的克隆 因为雷蒙德氏棉的祖先被公认为是陆地棉(Gossypium hirsutum)D亚基因组的供体,所以在陆地棉基因组尚未公布的前提下,本研究以雷蒙德氏棉(Gossypium raimondii)的基因组序列为参考,以AtPYR1基因序列Blast雷蒙德氏棉基因组数据库,并结合开放阅读框预测工具ORF Finder 预测得到可能的GrPYR1基因的编码序列。在基因编码区两侧设计引物GhPYR1F/R(表1),以陆地棉DNA和cDNA为模板进行PCR扩增。扩增体系为50 μL:Fast HiFidelity Polymerase 1 μL、5×Fast HiFidelity PCR Buffer 10 μL、10 mmol/L上下游引物各2 μL、模板DNA/cDNA 2 μL、ddH2O 33 μL。PCR反应程序为:94℃预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸30 s,30个循环;最后72℃延伸10 min。回收纯化PCR产物与克隆载体pEASYTM-Blunt Zero连接并转入大肠杆菌感受态Trans-T1,挑取克隆子用GhPYR1F/R引物进行菌落PCR检测,将阳性克隆子送北京擎科新业生物技术有限公司进行测序。
1.2.3 GhRYR1基因的序列分析 将GhPYR1基因编码的蛋白序列Blast Swiss-Prot蛋白数据库;利用MEGA 5.0将GhPYR1和AtPYR1、AtPYL1-13蛋白序列进行比对,构建NJ系统进化树;利用Protparam tool(http://web.expasy.org/protparam/)在线程序对GhPYR1的基本性质进行分析。利用在线程序PSORT(http://psort.hgc.jp/form.html)进行亚细胞定位预测。
1.2.4 过表达载体的构建 在GhPYR1基因编码序列的5'和3'端分别加上NotⅠ和SbfⅠ两个酶切位点,PCR扩增获得目的条带,回收后连接到到pEASYTM-Blunt Zero克隆载体上,测序正确后与Gateway入门载体Hbskb1-HI-380同时进行NotⅠ/ SbfⅠ双酶切,回收目的片段经T4连接酶连接后构建GhPYR1-380入门载体。然后通过Gateway LR反应构建过表达载体,反应体系5 μL:入门载体GhPYR1-380 1 μL、目的载体pEarlyGate330 3 μL、Gateway LR Clonase Ⅱ Enzyme Mix 1 μL,轻轻混匀后,25℃孵育6 h后加1 μL Proteinase K Solution 1 μL结束反应。产物转化大肠杆菌Trans-T1感受态,进行菌落PCR和测序鉴定,获得阳性克隆子,成功构建植物表达载体pOEGhPYR1。
1.2.5 拟南芥转化和鉴定 将表达载体pOEGhPYR1通过热激法转化农杆菌GV3101感受态细胞,将鉴定为阳性的农杆菌通过蘸花法转化野生型拟南芥植株。将T1代拟南芥种子在含50 mg/L 卡那霉素的MS固体培养基上进行筛选,提取 T1代抗性植株的DNA进行PCR鉴定,为保证检测的正确性,每个抗性植株分别用两对引物进行鉴定,一对特异扩增GhPYR1基因,另一对特异扩增部分CaMV 35S 启动子和GhPYR1基因,引物序列见表1。收获PCR鉴定为阳性的T1代植株种子,经T2代繁殖及进一步抗性筛选最终获得纯合T3代株系,并对T2、T3代转基因株系进行PCR鉴定。
1.2.6 转基因植株的生理实验分析 为了分析种子萌发时转基因拟南芥对ABA、高盐及干旱的反应情况,将野生型和转基因植株的种子分别播种到MS及分别含0.5 μmol/L ABA、200 mmol/L甘露醇、100 mmol/L NaCl的MS固体培养基上。每种培养基上,野生型和转基因植株的3个株系分别点40-50粒种子,每种处理做3个重复。然后4℃冰箱黑暗春化3 d,于气候培养箱23℃/20℃,16 h光照/8 h黑暗条件下萌发5 d后进行拍照和种子萌发率的统计分析。
为了分析外源ABA、高盐及干旱条件下转基因拟南芥根生长初期的生长情况,首先将WT、OEGhPYR1-1、OEGhPYR1-2在MS上萌发7 d,7 d后将植株转移至含5 μmol/L ABA、200 mmol/L甘露醇、100 mmol/L NaCl的MS固体培养基上继续培养,7 d后进行拍照并统计根长。
1.2.7 ABA应答基因的表达分析 将在MS培养基上生长10 d的野生型和转基因型拟南芥幼苗用10 μmol/L ABA 处理3 h。收集ABA处理前和ABA处理后的拟南芥材料后液氮速冻。提取拟南芥的RNA并反转录为cDNA作为模板。通过实时定量PCR进行ABA应答基因RD29A、RAB18的表达量分析,内参对照选择拟南芥GAPDH基因。本实验所用引物见表1。反应体系20 μL:GoTag qPCR Master Mix(2×)10.0 μL、cDNA 2 μL、上下游引物(10 μmol/L)各0.4 μL、ddH2O 7.2 μL,每次实验设定3个重复。反应程序为:98℃ 2 min;98℃ 15 s,60℃ 15 s,72℃ 30 s,40个循环。统计每个反应的荧光强度达到阈值时对应的循环数(Ct)。目的基因相对于内参基因的相对表达量用2-△△Ct法计算。
2 结果
2.1 GhPYR1基因克隆、序列分析及载体构建
分别以陆地棉DNA和cDNA为模板进行PCR扩增,均获得大小约600 bp左右、单一、清晰的条带(图1-A和B)。测序结果显示,扩增产物长618 bp,编码205个氨基酸残基,ORF区无内含子。将GhPYR1基因序列Blast陆地棉基因组,结果显示GhPYR1基因在陆地棉A基因组和D基因组的第3号染色体上各有一个拷贝,说明克隆得到GhPYR1基因。将GhPYR1序列Blast Swiss-Prot数据库,显示GhPYR1具有PYR/PYL/RCAR蛋白家族的特征结构域(图2-A),且与拟南芥AtPYR1有最高相似度,为73%(图2-B)。
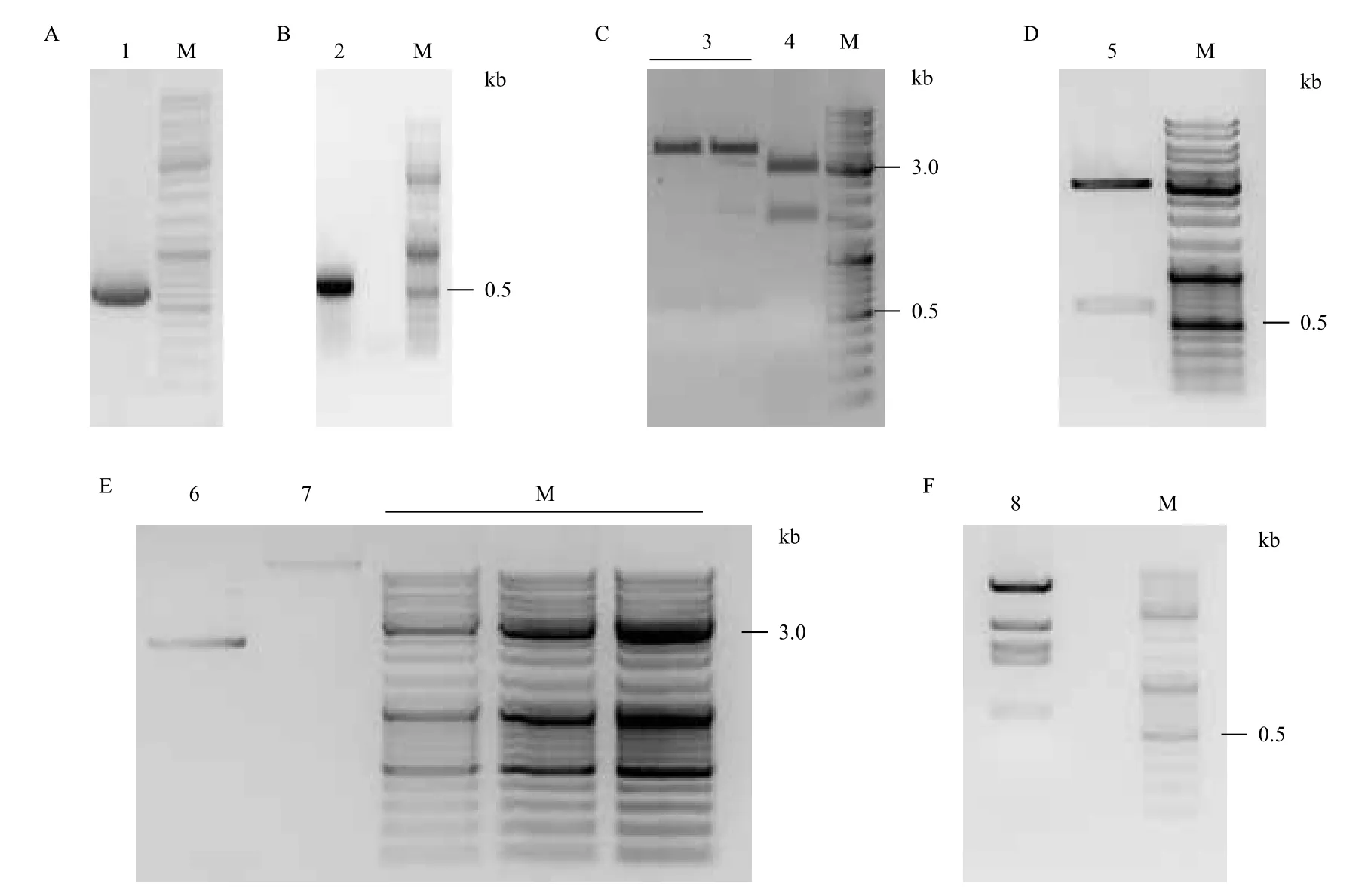
图1 GhPYR1基因的克隆及载体
利用MEGA 5.0软件分析GhPYR1和拟南芥中14个PYR/PYL/RCAR家族成员,并进行NJ进化树分析,结果(图3)表明,拟南芥的PYR/PYL/ RCAR家族被分成了3个亚家族,而GhPYR1与拟南芥第Ⅲ亚家族的亲缘关系最近。利用Protparam tool在线程序分析GhPYR1的基本性质,结果表明,其由205个氨基酸残基组成,蛋白分子式为C1001H1584N280O318S7,分子量为22853.6 Da,等电点pI为5.47,亲水性平均数为-0.364,属于水溶性蛋白。利用在线程序PSORT预测GhPYR1的亚细胞定位特性,结果表明GhPYR1定位于细胞质的分值为0.65,而定位于叶绿体基质、叶绿体类囊体膜、叶绿体类囊体间隙的分值均为0.2,因此推测GhPYR1主要分布于细胞质中。
将GhPYR1基因首先克隆到pEASYTM-Blunt Zero载体上,随后经NotⅠ和SbfⅠ双酶切与载体Hbskb1-HI-380连接,构建成功GhPYR1-380入门载体(图1-C和D)。最后入门载体GhPYR1-380与目的载体pEarelyGate330在定量后进行LR反应,构建成功表达载体pOEGhPYR1(图1-E和F)。
2.2 转GhPYR1基因拟南芥获得
将pOEGhPYR1载体转化农杆菌,PCR鉴定(图4-A)为阳性后,利用蘸花法转化野生型拟南芥,T1代转基因拟南芥PCR鉴定结果如图4-B所示。每个抗性植株分别用两对引物进行鉴定,产物大小分别为615 bp和826 bp,野生型受体拟南芥无特异扩增。经鉴定最终获得T1代转株GhPYR1基因拟南芥阳性植株39株。收获T2代拟南芥种子进行筛选后得到各个株系的拟南芥单株,经PCR鉴定(图4-C)后进行繁代,收获得到T3代种子后进行抗性筛选,选择在筛选平板上不分离的株系进行PCR鉴定后(图4-D),最终获得3个纯合转基因株系,分别命名为OEGhPYR1-1、OEGhPYR1-2、OEGhPYR1-3。
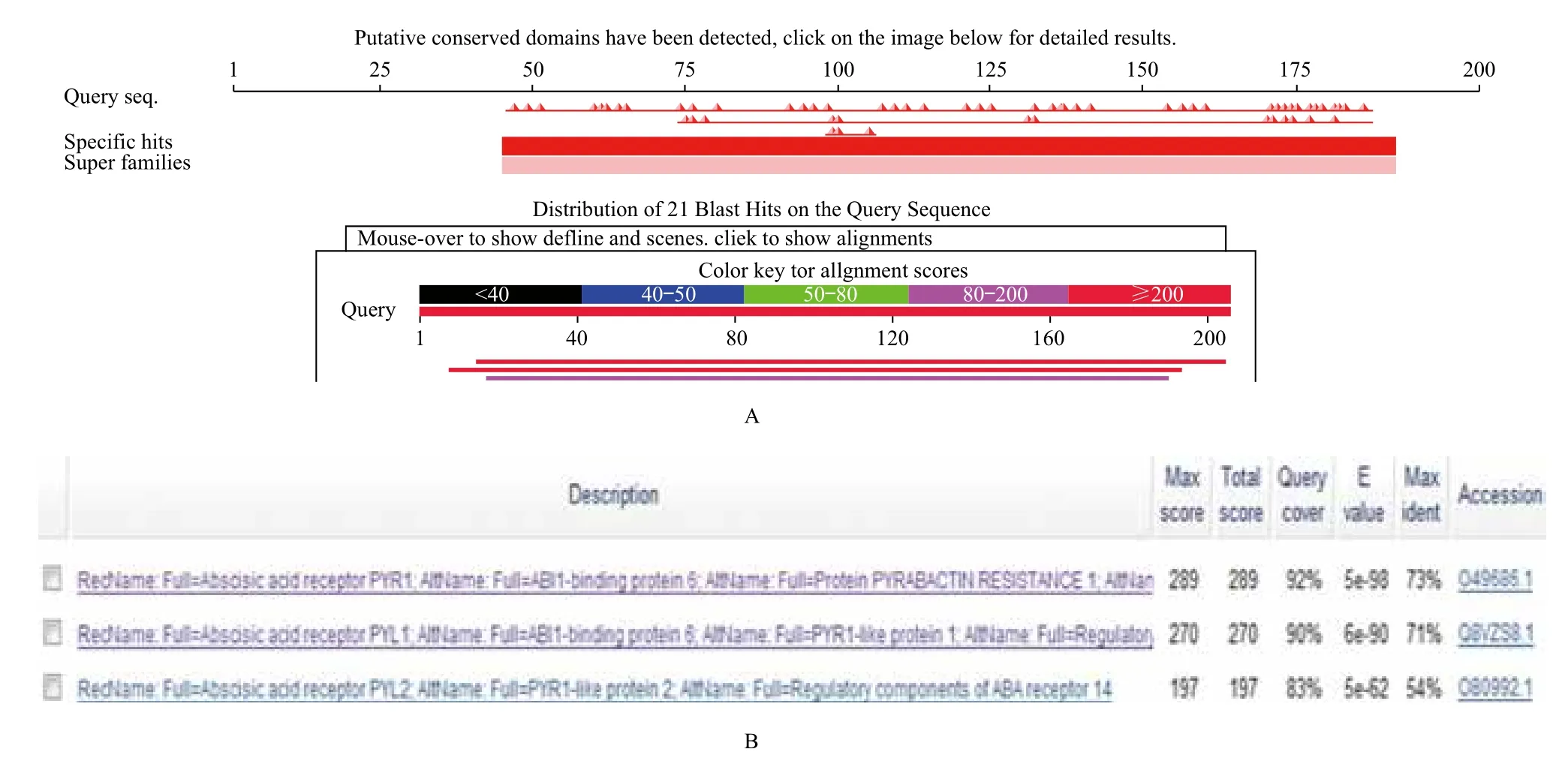
图2 GhPYR1蛋白Blast NCBI Swiss-Prot结果
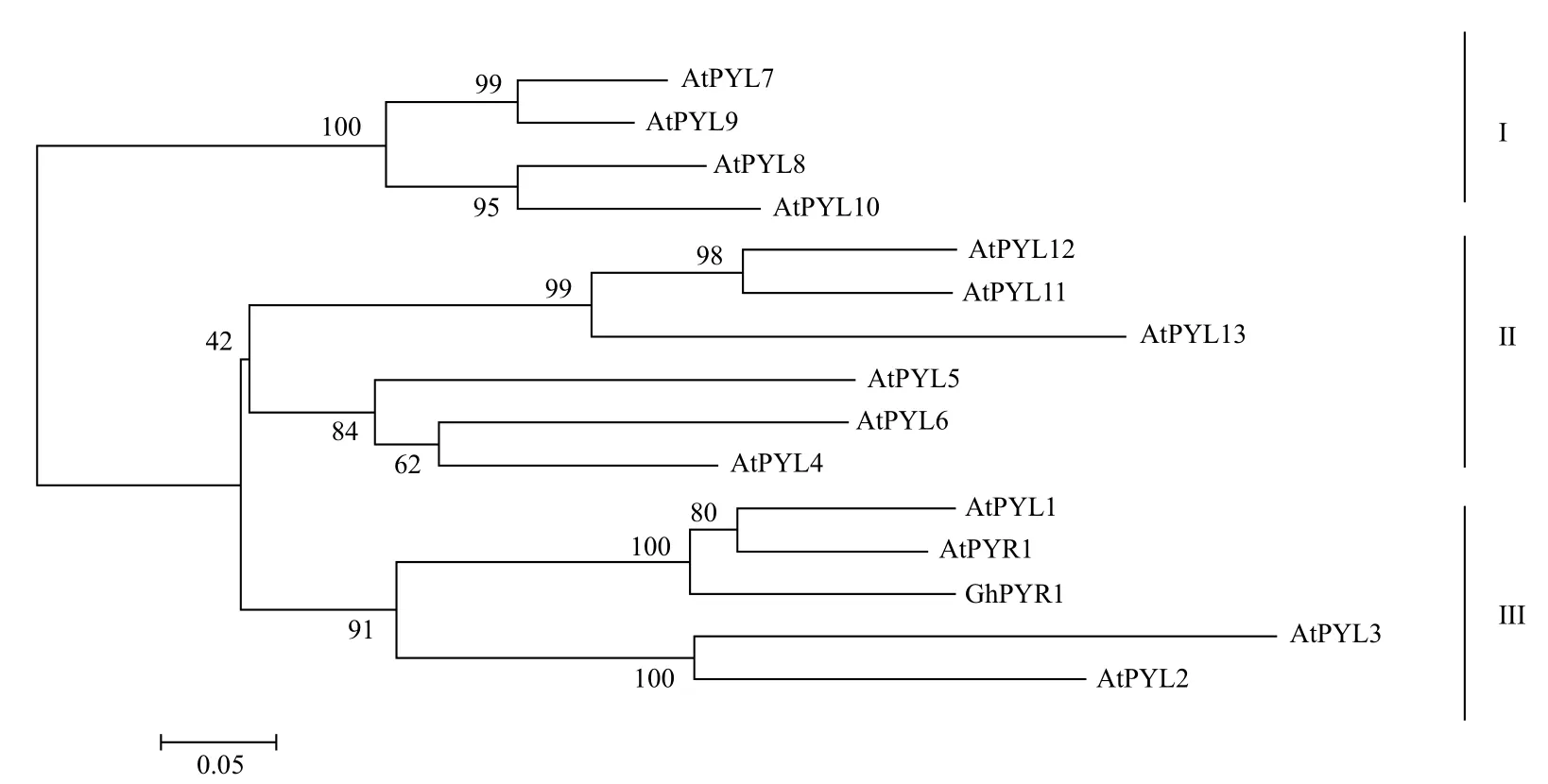
图3 GhPYR1与拟南芥14个PYR/PYL/RCAR蛋白的进化树分析
2.3 转GhPYR1基因拟南芥的生理鉴定
2.3.1 T3代转GhPYR1基因拟南芥的种子萌发实验 以野生型拟南芥为对照,在MS和分别含0.5 μmol/L ABA、100 mmol/L NaCl、200 mmol/L 甘露醇的MS固体培养基上进行转基因拟南芥种子萌发实验,结果如图5所示。图5-A显示,在MS培养基上,野生型和转基因型拟南芥表现一致,均能正常萌发,长出绿色的子叶,萌发率在95%-98%之间(表2);而在含0.5 μmol/L ABA MS的培养基上,转基因拟南芥和野生型之间表型表现出显著差异,野生型种子大多数能正常萌发,并长出绿色的子叶,5 d萌发率在90%以上,3个转基因株系则表现为萌发滞后,同期观察长出绿色子叶的种子数量很少,萌发率在75%-87%之间(图5-B,表2),野生型和转基因株系间存在极显著差异(图6)。在含100 mmol/L NaCl的培养基上,所有拟南芥的萌发率都下降,但野生型相对下降幅度较小,5 d萌发率仍维持在80%以上,3个转基因株系则大幅度下降,5 d萌发率仅有5%-8%左右(图5-C,表2),野生型和转基因株系间存在极显著差异(图6)。类似地,在含200 mmol/L 甘露醇培养基上,3个转基因株系的5 d萌发率下降至6%-14%(图5-D,表2),与野生型之间同样表现出极显著差异(图6)。干旱、高盐等生理逆境会引起细胞内脱水[19]进而植物通过增加ABA的合成和ABA代谢的调整使得植物体内ABA的含量上升[20],上述结果表明,转GhPYR1基因拟b .5 b .5南芥植株比野生型对ABA更敏感,而ABA含量的增加,无论是外源的,还是内源的,都会影响拟南芥种子的萌发,使其萌发率下降,萌发期滞后。
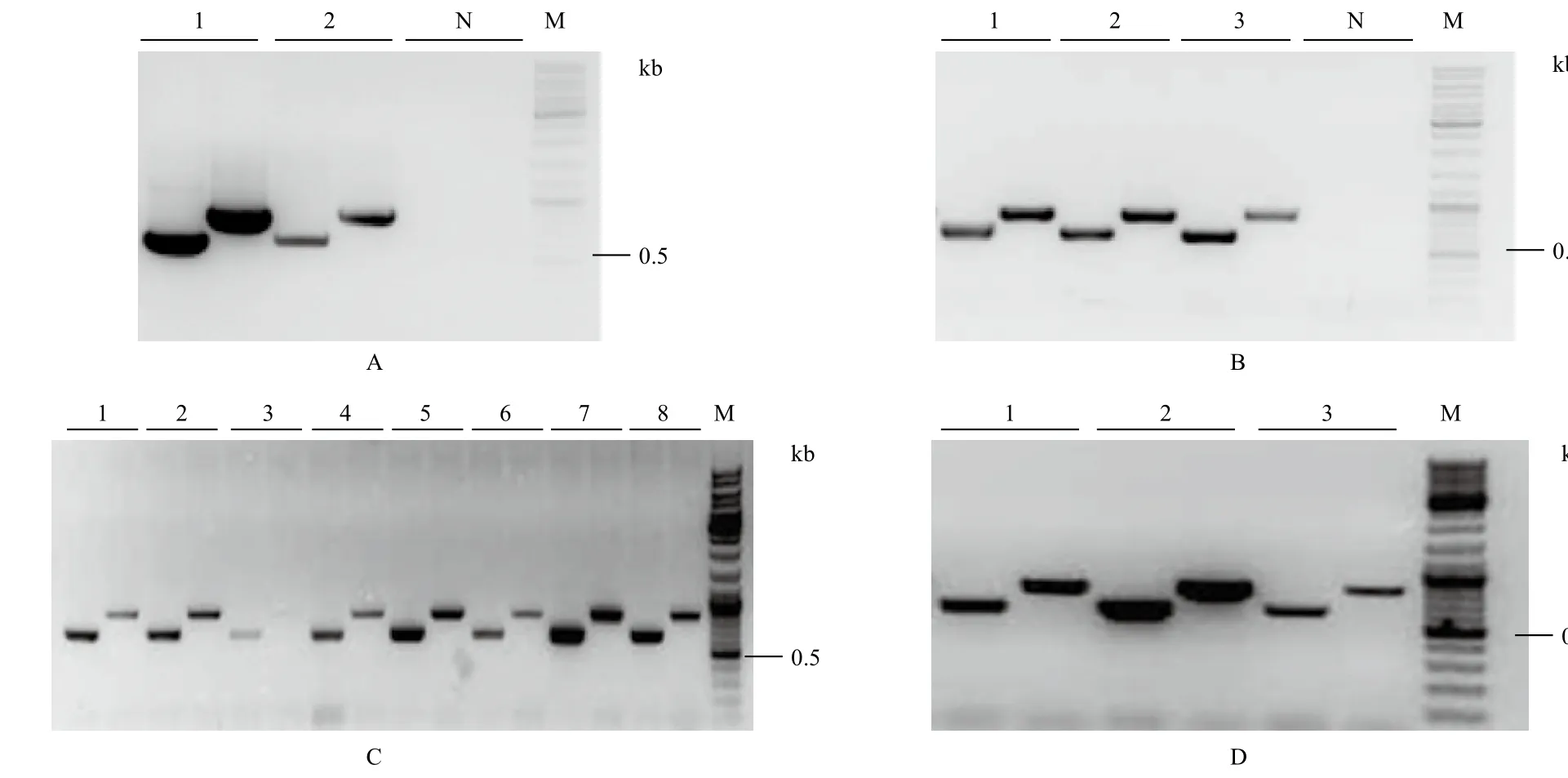
图4 农杆菌及转基因拟南芥的鉴定
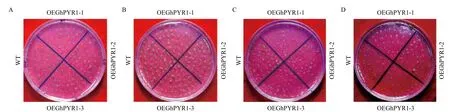
图5 T3代转GhPYR1基因拟南芥的种子萌发实验
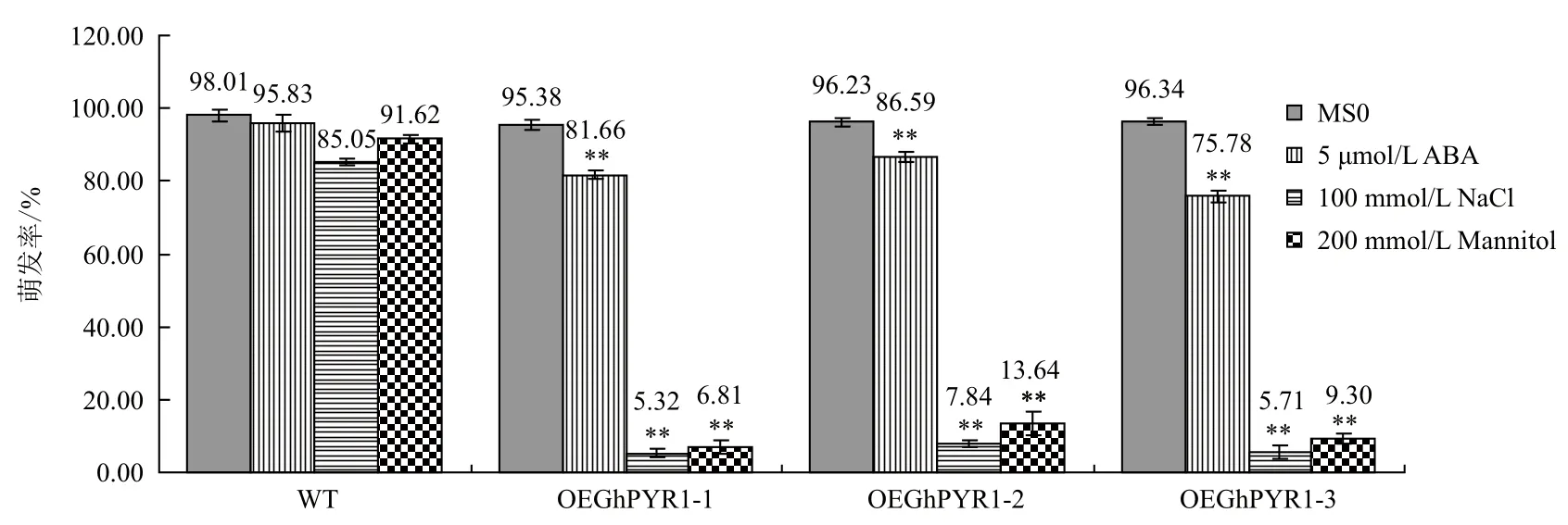
图6 不同处理下野生型和转基因型拟南芥的萌发率
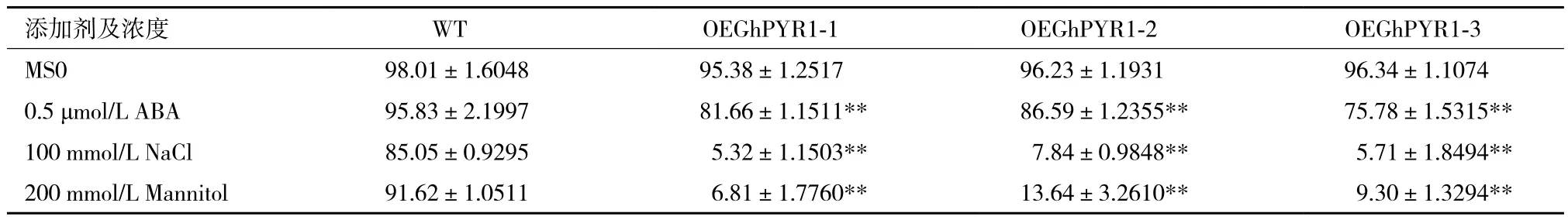
表2 不同处理下野生型和转基因型拟南芥萌发率/%
2.3.2 转GhPYR1基因拟南芥苗期耐逆实验 拟南芥在MS平板上萌发生长7 d后,转移至分别含5 μmol/L ABA、100 mmol/L NaCl、200 mmol/L甘露醇的MS固体培养基上继续培养7 d,生长状况如图7-9图所示。在5 μmol/L 外源ABA处理下(图7-A),转GhPYR1基因的拟南芥植株的平均根长不足5 cm,而野生型拟南芥的平均根长在6.5 cm以上,极显著于转基因拟南芥(图7-B)。这一结果表明ABA具有抑制拟南芥根生长的特性,而且,转基因拟南芥的根生长被抑制程度更深,说明转基因拟南芥比野生型对ABA更加敏感。
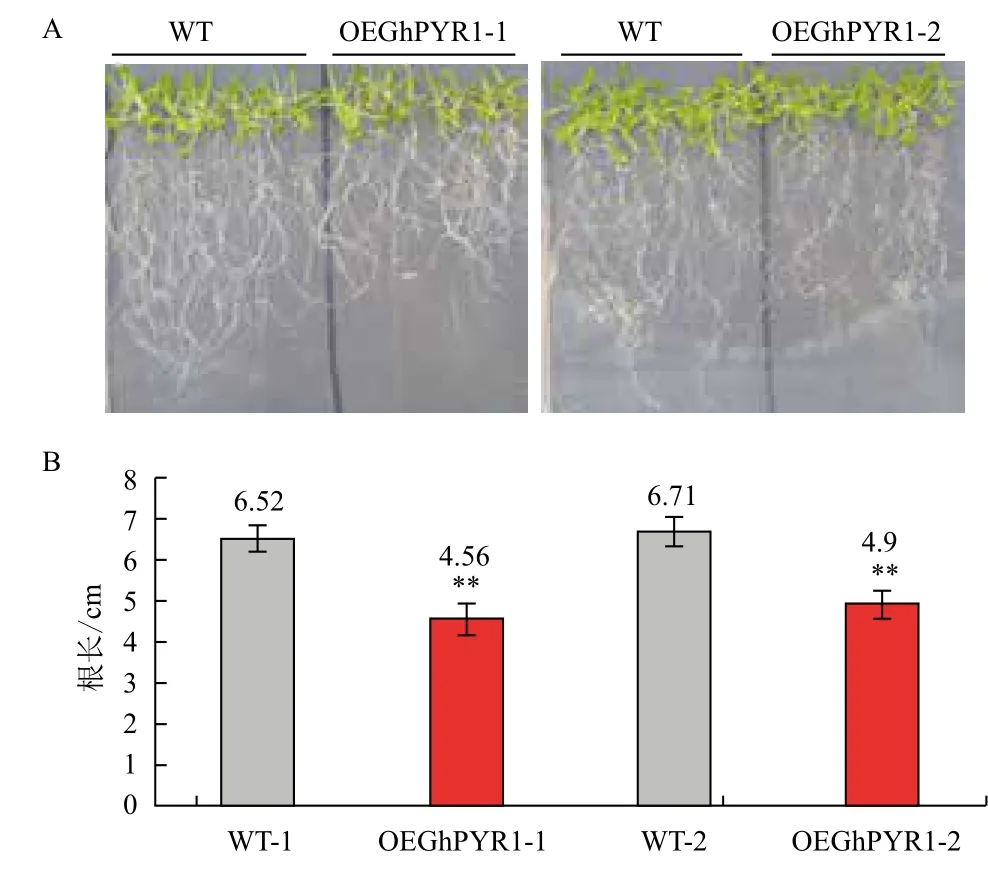
图7 苗期野生型和转基因拟南芥在外源ABA条件下生长情况
在100 mmol/L NaCl处理下,转GhPYR1基因的拟南芥植株生长状况优于野生型(图8-A),转基因拟南芥的平均根长在4.4 cm以上,而野生型的在3.7 cm左右,显著短于转基因拟南芥(图8-B)。结果表明,在种子萌发后期根的生长过程中,过表达GhPYR1基因的拟南芥较野生型更能适应高盐环境。同样地,在模拟干旱条件下,GhPYR1基因过表达植株根的生长优于野生型(图9-A),转基因拟南芥的平均根长在6.8 cm以上,而野生型的根长在6.0 cm左右,差异极显著于转基因拟南芥(图9-B)。上述结果表明,100 mmol/L NaCl和200 mmol/L甘露醇均为拟南芥苗期生长的逆境,会抑制拟南芥的生长,但转基因拟南芥相对生长状况要优于野生型,说明转基因拟南芥具有较强的耐逆性。
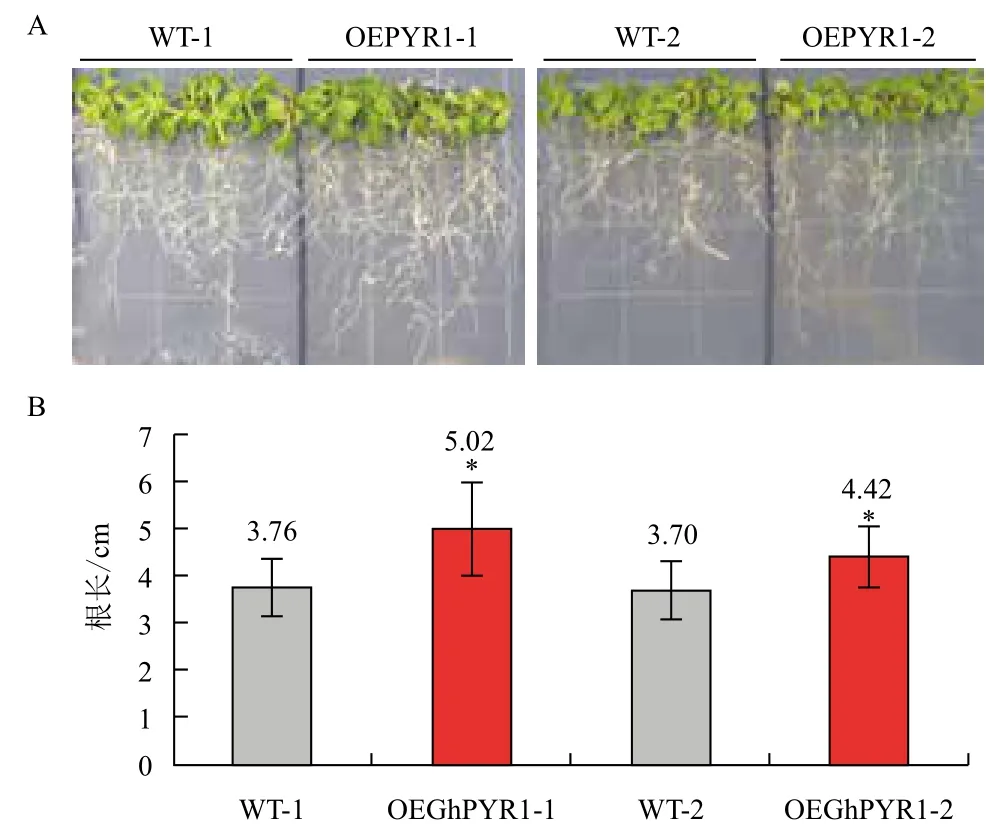
图8 苗期野生型和转基因拟南芥在高盐条件下生长情况
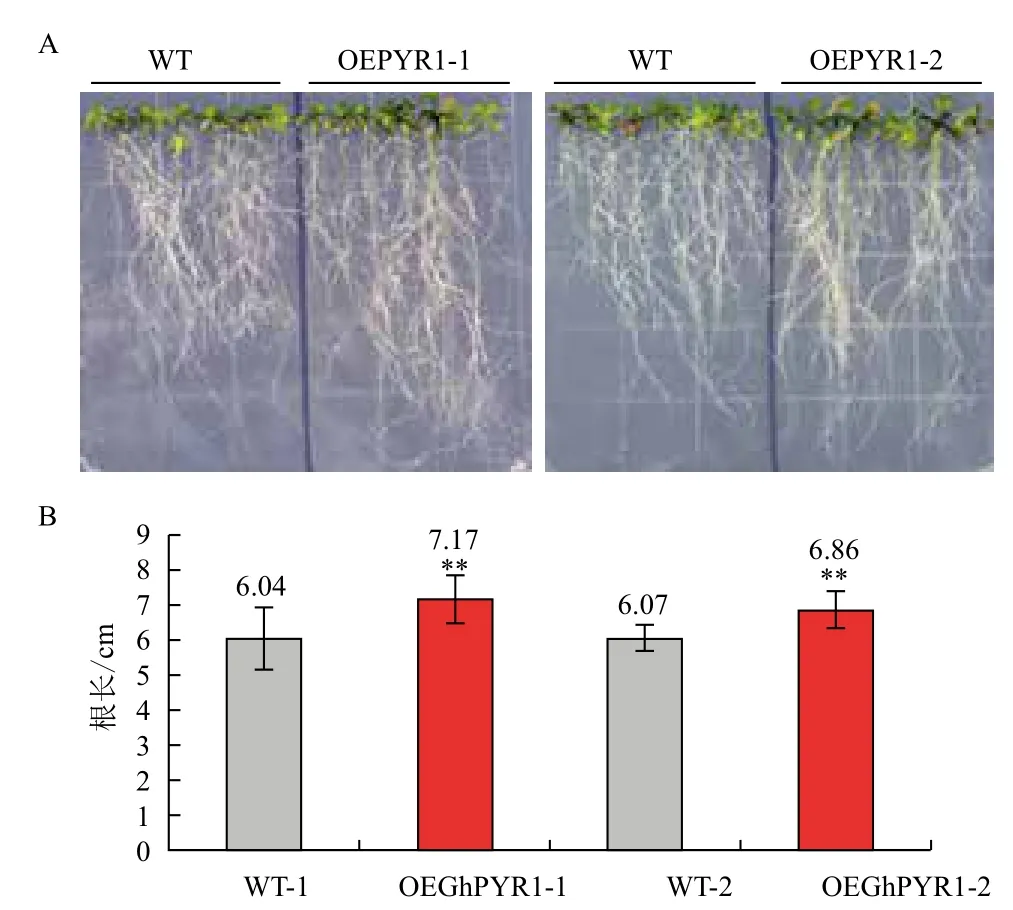
图9 苗期野生型和转基因拟南芥在模拟干旱条件下的生长情况
2.4 ABA应答基因的表达分析
为了探明ABA与受体结合后,是否能启动下游应答基因的表达,利用qRT-PCR的方法,分析了在外源ABA处理前和处理后,野生型和转GhPYR1基因拟南芥中ABA应答基因RD29A和RAB18B的表达量变化,结果如图10所示。在ABA处理前,转GhPYR1基因的拟南芥中RA29A基因的表达量略高于野生型,是野生型的1.81倍,RAB18基因的表达量略低于野生型,无明显差异;在ABA处理后,转GhPYR1基因的拟南芥和野生型中,RD29A基因的表达量都有所增加,野生型中增加了20%,而转基因中增加了622%,达到处理前野生型的8.08倍;类似地,RAB18基因的表达量在处理后的野生型和转GhPYR1基因拟南芥中均有所增加,在野生型中增加了56%,在转基因中增加了457%,达到处理前野生型的5.38倍。这些结果表明,在转GhPYR1基因的拟南芥中,ABA通路的活性要高于野生型,特别是经ABA处理后,活性大幅提升,应答基因大量表达,以使植物应对干旱、高盐等逆境。
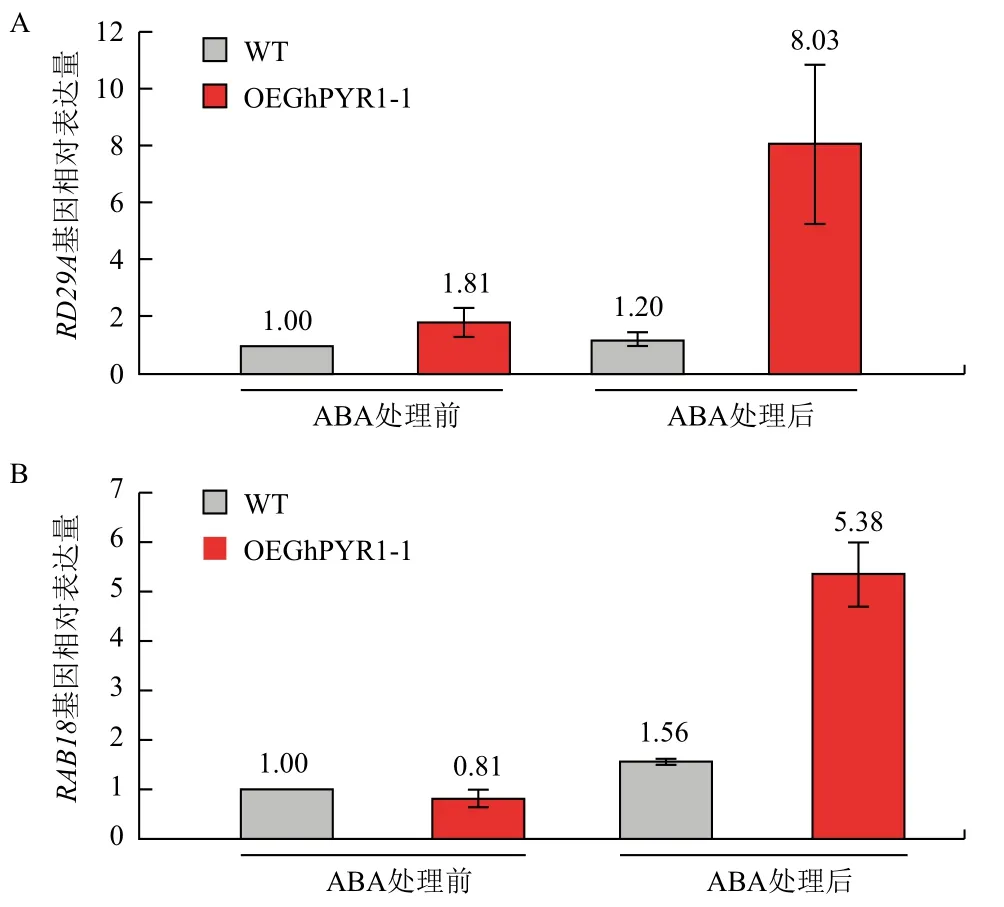
图10 野生型和转基因型拟南芥中ABA应答基因表达量比较
3 讨论
ABA通路在植物应对干旱、盐碱等非生物胁迫及植物种子成熟、休眠等发育过程中发挥着重要的作用。植物遭遇干旱、盐碱等逆境胁迫时,体内ABA含量增加,ABA受体通过与ABA结合感知这一信号,从而激活ABA通路,提高植物体内耐逆相关基因表达,从而使植物度过逆境胁迫期。因此,提高植物对ABA信号的敏感性,有助于植物对逆境胁迫作出快速及时的反应,提高植物的抗逆性。
本研究通过同源克隆的方法,从陆地棉中克隆了GhPYR1基因。该基因在陆地棉中只有一个外显子,ORF长618 bp,编码205个氨基酸残基,编码序列含有PYR/PYL/RCAR蛋白家族的特征结构域,与拟南芥中AtPYR1蛋白相似度为73%,与拟南芥中的AtPYR1、AtPYL1、AtPYL2及AtPYL3关系最近,同属于第Ⅰ亚家族,亚细胞定位预测其最可能分布于细胞质。过表达GhPYR1基因的拟南芥在添加外源ABA的情况下表现出种子休眠期延长,萌发滞后,苗期生长发育迟缓的现象,说明其对ABA的敏感性增强,这一结果和在拟南芥中过表达青蒿的PYR/PYL/RCAR家族基因AaPYL9,提高了转基因拟南芥种子在萌发时对ABA的敏感性[17]相一致。
种子萌发包括吸胀、萌动和萌发等连续的生理过程,是一个需水较多的生理时期。干旱、盐碱等逆境,由于缺水会抑制种子萌发,导致萌发滞后等现象发生,这是植物的一种自我保护手段,而ABA通路应该是参与这一自我保护调控过程的主要因子。干旱、盐碱等逆境会诱导植物体内ABA含量增加,从而激活和增强ABA通路,引起下游耐逆基因的大量表达,同时抑制萌动、萌发等相关基因的表达。本研究中拟南芥种子萌发时添加外源ABA的实验,证实了植物在种子萌发期对ABA信号的应答,拟南芥将环境中ABA含量升高这一现象,解读为存在干旱、盐碱等逆境,从而启动植物体内的逆境应答反应,而不是正常状态下的萌发,因而出现了和干旱、盐碱状态下相似的表型。过表达GhPYR1基因的拟南芥,因为对ABA的信号更加敏感,所以在相同浓度的外源ABA或逆境条件下,其种子的萌发被抑制得更为强烈。
在植物生长期,ABA通路是植物应对逆境的主要信号通路。本研究对苗期的拟南芥进行干旱和高盐处理,转基因拟南芥表现出更好的生长势即说明了这一点,因为在转基因拟南芥中,ABA通路被激活的程度,要数倍强于野生型在同等条件下被激活的程度。RD29A基因的表达量,在逆境下野生型只增加20%,而在转基因拟南芥中却增加了622%,达到处理前野生型的8.08倍;RAB18B基因的表达量,在逆境下野生型中增加56%,而在转基因中则增加了457%,是处理前野生型的5.38倍。也正因为转基因拟南芥对ABA更敏感,所以在只添加ABA的模拟逆境环境下,转基因拟南芥的生长势要弱于野生型,因为拟南芥将环境中的ABA信号,解读为逆境信号,而同等信号强度下,转基因拟南芥激活的ABA通路的基因表达要数倍于野生型,而对应的它抑制的与促进植物生长有关的通路基因的表达,也应数倍于野生型。而恰恰因为这只是一个模拟逆境的状态,并不存在真实逆境对植物生长的伤害,所以,ABA通路表达弱的野生型拟南芥反而长势优于通路表达强的转基因拟南芥。
本研究结果证明了GhPYR1基因编码的蛋白是ABA的受体,同时证明了过表达GhPYR1基因的拟南芥植株提高了对ABA的敏感性和增强了应对逆境胁迫的能力,但同时也会抑制种子的萌发,所以将该基因用于植物基因工程,用以提高植物耐旱、耐盐碱性的时候,最好避免使用CaMV 35S这种组成型表达的启动子,而代之以组织特异性或诱导表达型启动子,以期在提高目的基因在靶标组织表达的同时,降低其对种子萌发的抑制。
4 结论
GhPYR1基因是拟南芥中AtPYR1基因的同源基因,在拟南芥中过表达GhPYR1基因能够提高转基因植株对ABA的敏感性。表现为在外源ABA存在、干旱和高盐胁迫条件下,其种子的休眠期延长,萌发率下降。同时,在苗期,转基因拟南芥对外源ABA敏感,更能适应高盐及干旱环境。在外源ABA条件下,ABA应答基因的表达量提高,使得植物能够对ABA信号做出及时反应,以启动植物体内的信号传导机制,使植物安全度过逆境胁迫期。
[1]Cutler SR, Rodriguez PL, Finkelstein RR, et al. Abscisic acid:emergence of a core signaling network[J]. Annu Rev Plant Biol,2010, 61:651-679.
[2]Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses[J]. Annu Rev Plant Biol, 2006, 57:781-803.
[3]Ma Y, Szostkiewicz I, Korte A, et al. Regulators of PP2C phosphatase activity function as abscisic acid sensors[J]. Science, 2009, 324(5930):1064-1068.
[4]Park SY, Fung P, Nishimura N, et al. Abscisic Acid Inhibits Type 2C Protein Phosphatases via the PYR/PYL Family of START Proteins[J]. Science, 2009, 324(5930):1068-1071.
[5]余小林, 胡帅, 王芳展, 等. PYR/PYL/RCAR蛋白介导植物ABA的信号转导[J]. 遗传, 2012, 34(5):560-572.
[6]Umezawa T, Sugiyama N, Mizoguchi M, et al. Type 2C protein phosphatases directly regulate abscisic acid-activated protein kinases in Arabidopsis[Z]. 2009:106, 17588-17593.
[7]Vlad F, Rubio S, Rodrigues A, et al. Protein phosphatases 2C regulate the activation of the snf1-related kinase OST1 by abscisic acid in Arabidopsis[J]. The Plant Cell, 2009, 21(10):3170-3184.
[8]Santiago J, Dupeux F, Round A, et al. The abscisic acid receptor PYR1 in complex with abscisic acid[J]. Nature, 2009, 462(7273):665-668.
[9]Nishimura N, Hitomi K, Arvai AS, et al. Structural mechanism of abscisic acid binding and signaling by dimeric PYR1[J]. Science,2009, 326(5958):1373-1379.
[10] Santiago J, Dupeux F, Round A, et al. The abscisic acid receptor PYR1 in complex with abscisic acid[J]. Nature, 2009, 462(7273):665-668.
[11]Fujii H, Verslues PE, Zhu J. Identification of two protein kinases required for abscisic acid regulation of seed germination, root growth, and gene expression in Arabidopsis[J]. The Plant Cell Online, 2007, 19(2):485-494.
[12]Furihata T, Maruyama K, Fujita Y, et al. Abscisic acid-dependent multisite phosphorylation regulates the activity of a transcription activator AREB1[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(6):1988-1993.
[13]Jeannette E, Rona JP, Bardat F, et al. Induction of RAB18 gene expression and activation of K+outward rectifying channels depend on an extracellular perception of ABA in Arabidopsis thaliana suspension cells[J]. Plant J, 1999, 18(1):13-22.
[14]Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses[J]. Annu Rev Plant Biol, 2006, 57:781-803.
[15]Kim H, Hwang H, Hong JW, et al. A rice orthologue of the ABA receptor, OsPYL/RCAR5, is a positive regulator of the ABA signal transduction pathway in seed germination and early seedling growth[J]. Journal of Experimental Botany, 2012, 63(2):1013-1024.
[16]Bai G, Yang D, Zhao Y, et al. Interactions between soybean ABA receptors and type 2C protein phosphatases[J]. Plant Molecular Biology, 2013, 83(6):651-664.
[17]Zhang F, Lu X, Lv Z, et al. Overexpression of the Artemisia orthologue of ABA receptor, AaPYL9, enhances ABA sensitivity and improves artemisinin content in Artemisia annua L.[J]. PLoS One, 2013, 8(2):e56697.
[18] Chai YM, Jia HF, Li CL, et al. FaPYR1 is involved in strawberry fruit ripening[J]. Journal of Experimental Botany, 2011, 62(14):5079-5089.
[19] Fujita Y, Fujita M, Shinozaki K, et al. ABA-mediated transcriptional regulation in response to osmotic stress in plants[J]. Journal of Plant Research, 2011, 124(4):509-525.
[20] Nambara E, Marion-Poll A. Abscisic acid biosynthesis and catabolism[J]. Annual Review of Plant Biology, 2005, 56(1):165-185.
(责任编辑 李楠)
Cloning and Function Analysis of Gene GhPYR1 in Gossypium hirsutum L.
LIU Yan MENG Zhi-gang SUN Guo-qing WANG Yuan ZHOU Tao GUO San-dui ZHANG Rui
(Biotechnology Research Institute,Chinese Academy of Agricultural Sciences,Beijing 100081)
The phytohormone abscisic acid(ABA)plays an important role in response to drought, salinity and other abiotic stress. It is also a key regulator involved in different plant developmental stages such as seed germination, root elongation, and bud dormancy. The PYR/ PYL/RCAR protein family is the ABA receptor, which can initiate the ABA signaling pathway and induce the expression of ABA response genes. In this study, gene GhPYR1 was cloned from Gossypium hirsutum L. with electronic cloning and RT-PCR technology. GhPYR1 protein exhibited 73% sequence identity with Arabidopsis AtPYR1 protein. Protein sequence alignment among GhPYR1 and 14 family members of PYR/PYL/ RCAR in Arabidopsis as well as the construction of phylogenetic tree revealed that gene GhPYR1 was most closely related to subfamily III of PYR/PYL/RCAR protein in Arabidopsis. T3generation Arabidopsis over-expressing gene GhPYR1were more sensitive to exogenous ABA, as its seed germination and early root growth lagged behind than wild type. The germination of transgenic seeds was more strongly inhibited by high salt and drought stress, however, the transgenic lines were significantly better than the wild ones in the seeding stage. Moreover, the ABA-responsive gene RD29A and RAB18 in transgenic Arabidopsis were significantly induced by ABA compared to the wild type. The above results show that the protein encoded by gene GhPYR1 is the receptor of ABA and over-expression of GhPYR1 can improve the sensitivity of plant to ABA and enhance the ability to respond to stress in plants.
Gossypium hirsutum L.;GhPYR1 gene;bioinformatics analysis;physiological function analysis
10.13560/j.cnki.biotech.bull.1985.2016.02.012
2015-05-13
“863”项目(2013AA102601)
刘妍,硕士研究生,研究方向:植物基因工程;E-mail:liuyan4368@sina.com
张锐,研究员,博士生导师,研究方向:棉花基因工程;E-mail:zhangrui71@126.com
