miRNA qPCR检测方法研究进展及其应用
2016-10-13冯世鹏
冯世鹏
(海南大学农学院 海南省热带生物资源可持续利用重点实验室,海口 570228)
miRNA qPCR检测方法研究进展及其应用
冯世鹏
(海南大学农学院 海南省热带生物资源可持续利用重点实验室,海口 570228)
miRNA是目前国际研究的热点领域之一,而miRNA表达检测是miRNA研究的基础内容。针对miRNA序列短小的特点,开发出了多种检测方法,其中miRNA qPCR定量检测是应用最广泛的方法。就miRNA抽提,miRNA qPCR定量检测原理及miRNA qPCR定量检测流程包括引物设计、反应体系设置、内参基因筛选等方面进行了深入探讨,并对miRNA定量检测研究方法的发展趋势进行了展望,以期为研究者进行miRNA qPCR定量检测提供参考。
miRNA;stem-loop RT-PCR;poly(A)加尾法RT-PCR;延伸法RT-PCR;数字PCR
miRNA是真核生物体内普遍存在的一类非编码序列长度约22个碱基的单链小RNA分子,由特定发卡结构(Stem-loop)的前体剪切加工而成。成熟的miRNA与相关蛋白组成沉默诱导复合体(RNA-induced silencing complex,miRISC)与靶基因mRNA结合,降解mRNA或者抑制mRNA翻译成蛋白质,从而调控靶基因的表达[1,2]。目前在英国Sanger中心miRBase数据库(最新版本21,2014年6月释放)(http://www.mirbase.org/)中共有28 645条成熟miRNA记录,涵盖物种包括囊泡藻界(Chromalveolata)、后生动物亚界(Metazoa)、黏菌门(Mycetozoa)、绿色植物亚界(Viridiplantae)及病毒(Viruses)5大类,其中记录的成熟miRNA人类(Homo sapiens)2 588个,小鼠(Mus musculus)1 915个,秀丽线虫(Caenorhabditis elegans)434个,水稻(Oryza sativa)713个,拟南芥(Arabidopsis thaliana)413个[3,4]。
MiRNA参与生物体生长、发育、衰老和凋亡等多个进程,其发挥的作用越来越受到研究者的重视。根据miRNA序列短小的特点,开发了多种检测方法,如Northern blot杂交法[5],Primer extension(引物延伸法)[6],Signal-amplifying ribozymes(核酶法)[7],Invader assay(侵入探针法)[8],Bead-based assay(磁珠法)[9],qPCR法[10],miRAGE(miRNA表达序列分析法)[11],深度测序法[12]等。
以上这些方法的优劣及使用情况已经有相关文献[13]进行了报道,其中qPCR方法使用最多,本文将对近年miRNA qPCR方法的研究进展进行论述,以期对今后miRNA的qPCR定量检测提供参考意见。
1 miRNA抽提方法
目前对于miRNA的抽提主要有两类方法。一类是沉淀法,通过强有机试剂裂解细胞释放RNA,再用乙醇、异丙醇、乙酸钠、LiCl等试剂进行RNA沉淀,常见的裂解液如不同公司的Trizol试剂(含有强有机试剂异硫氰酸胍等);另一类是柱吸附法,通过裂解液裂解细胞释放RNA后,用吸附柱分离纯化小RNA分子(表1)。
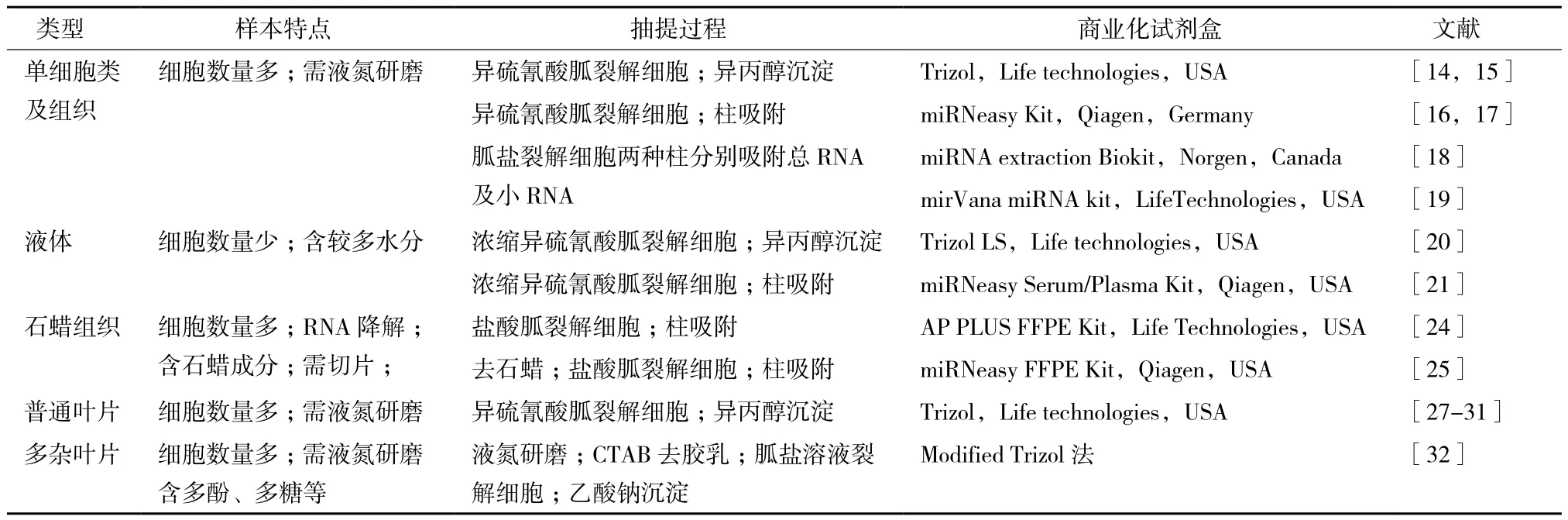
表1 常见样本miRNA抽提方法汇总表
1.1 细胞类样本及动物组织样本miRNA抽提
对于人、动物、植物细胞,微生物细胞个体及人与动物组织样本,常用沉淀法法抽提总RNA,该方法通过Trizol试剂裂解细胞释放RNA,再进行总RNA的沉淀,此方法易于操作,所抽提的RNA里面包含miRNA等小RNA分子,并可满足大多数情况下miRNA检测之用[14,15];有些吸附柱可同时吸附所有类型的RNA,利用这种吸附柱也可抽提含miRNA的总RNA分子[16,17],也有部分吸附柱只能吸附大片段的RNA或者只能吸附小片段的RNA,基于这类吸附柱开发的试剂盒对于RNA的抽提则需要将大片段的RNA(>200 nt)与小片段(<200 nt)RNA分别提取[18,19]。有部分实验需要较高纯度的miRNA(如miRNA测序、miRNA的表达谱研究等),可在抽提的总RNA基础上进一步采用吸附柱或凝胶电泳纯化分离纯化miRNA分子。
对于血样、唾液、精液等液体样本,因其所含细胞少、水分多等特点需用浓缩型的miRNA抽提试剂[20,21]。Ho等[22]比较了不同公司6款针对液体样本的miRNA抽提方法,发现其抽提效果稍有差异,该结果可为研究者进行商业化试剂盒选择时提供参考。Tzimagiorgis等[23]对于血浆、血清、唾液、脑髓液等体液样本游离核酸的来源、样本前处理及miRNA抽提方法进行了综合讨论,可为液体样本miRNA抽提提供参考。
对于石蜡样本,因其RNA有不同程度降解,所含石蜡会影响后续实验反应,样本miRNA抽提前需进行去石蜡操作[24,25],不同公司开发了针对石蜡样本的miRNA抽提试剂盒,其实际使用使用效果有一定差异,选择时需谨慎[26]。
1.2 植物组织样本miRNA抽提
由于植物的根、茎、叶、花、果不同组织或者不同植物的同一组织中所含的次生代谢产物的种类及含量不一样,导致植物组织的miRNA抽提方法更加多样化。对于模式植物拟南芥、大部分禾本科植物、部分其他科植物的叶片来说,用Trizol进行总RNA抽提,其中所包含的miRNA分子可用于大多数情况下的miRNA表达检测[27-31]。对于一些特殊植物,如橡胶树叶片含有较多胶乳成分,香蕉和荔枝等水果树叶片含多糖等成分,其总RNA及miRNA的抽提需要进行去除这些成分的前期处理[32]。
1.3 miRNA质控
包含miRNA的总RNA抽提完毕之后需进行质量检测,包括RNA完整性、浓度、纯度的检测。其中RNA的完整性主要用两种方法检测:一是RNA凝胶电泳(PAGE或琼脂糖凝胶电泳均可),28S条带的亮度约为18S条带的2倍,则证明所抽提总RNA完整性较好;二是进行RIN值(RNA integrity number)检测(如用Agilent 2100进行芯片电泳),RIN值大于7则证明样本所抽提RNA完整性较好。RNA的浓度及纯度可用测吸光度的方法进行测定检测260 nm、280 nm和230 nm三个波长的吸光值A260、A280和A230,常用A260吸光值与浓度换算关系是OD值相当于RNA浓度44 ng/mL;A260与A280的比值可评估是否有DNA或者蛋白质污染,该比值大于2.0则证明RNA纯度较高;A260与A230的比值是看有机杂质残留,该值越大则认为RNA样本含有机杂质越少[33]。
2 miRNA qRT-PCR检测
2.1 miRNA前处理
miRNA由于其自身特点,对其进行特异性检测面临一些挑战。如序列长度太短,只有22 bp;GC含量不均一;缺少类似polyA的公共序列;存在初级转录本(pri-miRNA)、前体(pre-miRNA)、成熟miRNA三种形式;miRNA家族成员间序列相差几个碱基,甚至只差一个碱基[14]。因此对于miRNA的检测方法设计,从得到的RNA样本即开始综合考虑。
获得含有miRNA的高质量RNA样本后,在第一链cDNA合成之前,按照对该样本RNA的处理方式分3种类型:一是不做任何处理(图1-A);二是使用poly(A)聚合酶(poly(A)polymerase,PAP)在miRNA的3'端连接poly(A)尾(表2,图1-B);三是在miRNA的3'端或两端均添加linker(图1-C,图1-D)。

图1 成熟miRNA的前处理
2.1.1 Poly(A)加尾法 其最显著的好处在于为所有待检测miRNA引入类似mRNA的poly(A)尾,便于后续反转录时通过带poly(T)的反转录引物一次将所有的miRNA反转录为cDNA,对于用qPCR法进行miRNA表达谱研究来说非常实用。其缺点是加尾完毕需进行RNA纯化,无论是用乙醇沉淀[34]还是通过吸附柱[35]回收来纯化,均会损失一部分miRNA表达信息。另外,PAP酶的反应效率也很难达到100%,这也会导致一部分miRNA表达信息的损失,从而导致低表达的miRNA无法检测,其他可检测的miRNA表达量下降;另外由于不同公司的PAP酶及不同的反应条件会导致poly(A)尾的长短产生一定的差异,导致qPCR检测时出现杂峰,在反转录引物设计时必须考虑这种情况,并尽力避免。Poly(A)加尾法所需的总RNA量目前文献报道在0.5 μg以上,因该方法后续处理纯化的损失,因此建议起始量在条件许可的情况下可适当增加(如2 μg),这样有利于检测低表达的miRNA,同时加尾处理后的RNA可用于其他miRNA检测实验,也不会浪费。
2.1.2 linker连接法 在成熟的miRNA一端或者两端同时使用T4 RNA连接酶进行linker连接,其优点是:直接增加了miRNA的长度,使引物设计时可选择引物结合范围增大;在3'端引入linker,可用通用引物进行反转录;若同时在一端或者两端引入公共接头,如需要可设计通用引物。其缺点是:受T4 RNA连接酶反应效率影响,易导致低丰都表达的miRNA难以检测[36]。
2.2 第一链cDNA合成
2.2.1 Poly(A)加尾法 对于通过poly(A)加尾处理过的RNA样本来说,根据待检测的miRNA数量,取适量RNA加入带poly(T)的反转录引物进行反应即可(表2)。该反转录引物常用这种形式表示adapter(T)nVN,从5'端开始依次可分为3部分(图2-C):(1)adapter部分,用于引入通用反向引物位点,文献报道其长度范围集中在32-75 nt,其结构为线性或者发卡状。为了进一步引入通用taqman探针结合位点,或者后续实验有其他应用如构建文库,该部分可设计为较长的片段,并设计成发卡结构也增加其稳定性[37,38]。(2)poly(T)n部分,于miRNA的poly(A)尾互补配对,长度在12-25 nt之间(表2)。由于PCR反应过程中需扩增该片段,为避免扩增过程中出现聚合酶打滑现象,同时较长的引物片段会增加引物合成的困难,建议这部分的序列设计尽量不要太长。(3)VN部分,V代表A、G和C三者之一,N代表A、T、G和 C四者之一(表2),这一部分的作用是与miRNA互补配对,锚定反转录时的起始位置,避免由于poly(A)尾长短的差异导致qPCR时出现杂峰(图2-C)。这部分的长度有1nt[38],2nt[24,35],5-7 nt[39],其中2 nt使用最多,该部分长度小于或等于2 nt的属于通用引物,包含由3种(1 nt情况)或者12种(2 nt情况)引物的混合物,其反转录产物理论上可以检测所有的miRNA;大于2 nt的属于特异引物,主要用于进行特定miRNA的检测,提高qPCR检测的特异性[39]。
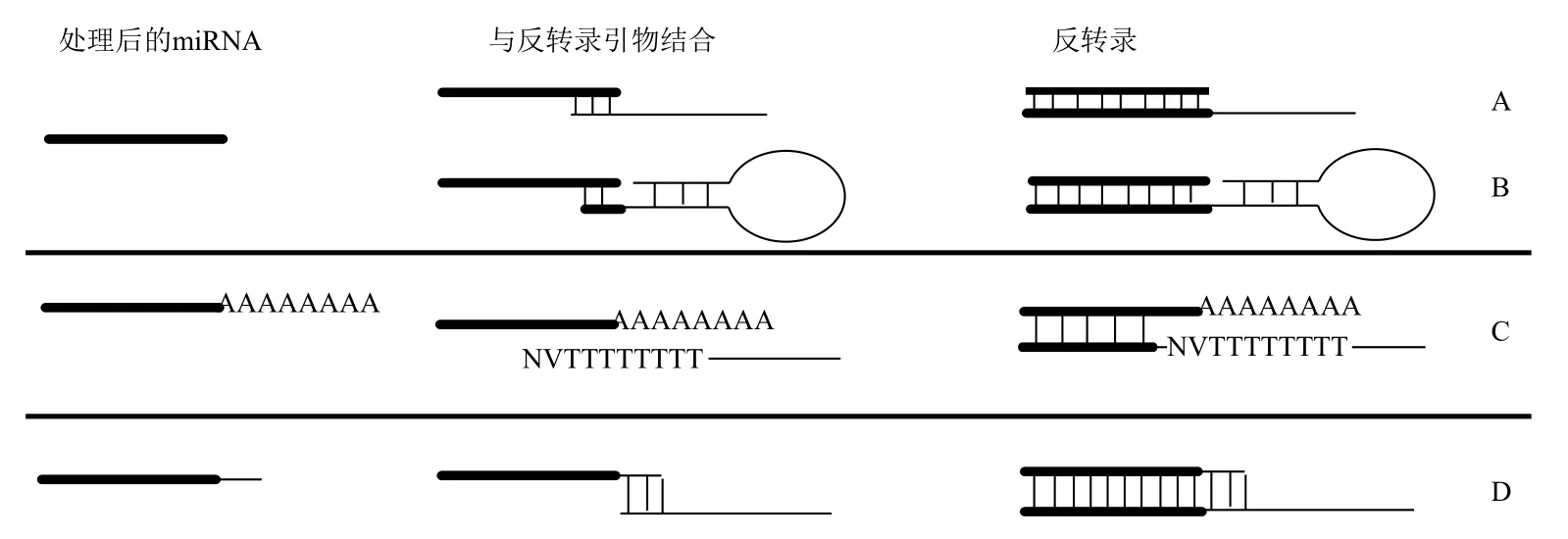
图2 反转录反应流程图
2.2.2 Stem-loop 引物反转录或线性引物反转录 对于未经加尾处理的RNA样本来说,需加入miRNA的特异引物进行miRNA的反转录(图2-A,2-B),若需同时检测多个miRNA,则必须将这些miRNA的特异引物混合后进行反转录反应。miRNA特异引物一般包括2部分,从5'端开始依次是:(1)延长部分,该部分的作用是引入反向引物结合位点,其结构可分为线性和stem-loop发卡结构两种。线性结构反转录引物可通过引物的联合设计取得较理想的检测效果,如qPCR正向引物LNA修饰[40];或通过RT反转录引物与反向引物的协同设计,使miRNA有特异的RT引物及qPCR反向引物等[41]。发卡结构可看成是在线性结构基础上的改进,发卡结构对于线性结构来说,在反转录时可提高反转录引物与miRNA结合的稳定性,并由于发卡结构的位阻效应可避免反转录引物与前体miRNA的结合,从而提高miRNA检测的灵敏度和特异性[10]。在发卡结构引物中还可引入dU碱基,在反转录完毕后加入鸟嘧啶DNA糖基化酶(uracil-DNA glycosylase,UDG),在dU碱基处将DNA断裂为两段,这样可去除反转录过程中剩余的RT引物,而已经转录完毕的cDNA在dU处断裂后余下的片段也可作为qPCR反应的模板,这样进一步提高qPCR检测的特异性[42]。(2)与miRNA互补配对部分,这部分与miRNA检测特异性密切相关。这部分的长度集中在6-8 nt,Lao等[43]测试这部分长度对于RT引物与miRNA结合效率的影响,从5-10 nt,在8 nt之前,随着长度的增加,RT引物结合效率显著增加;在8 nt之后则增加不明显。Jung等[44]测试获得了类似的结果,同时发现发卡结构的序列与miRNA结合位点间的gap越大,RT引物与miRNA结合的效率越差。
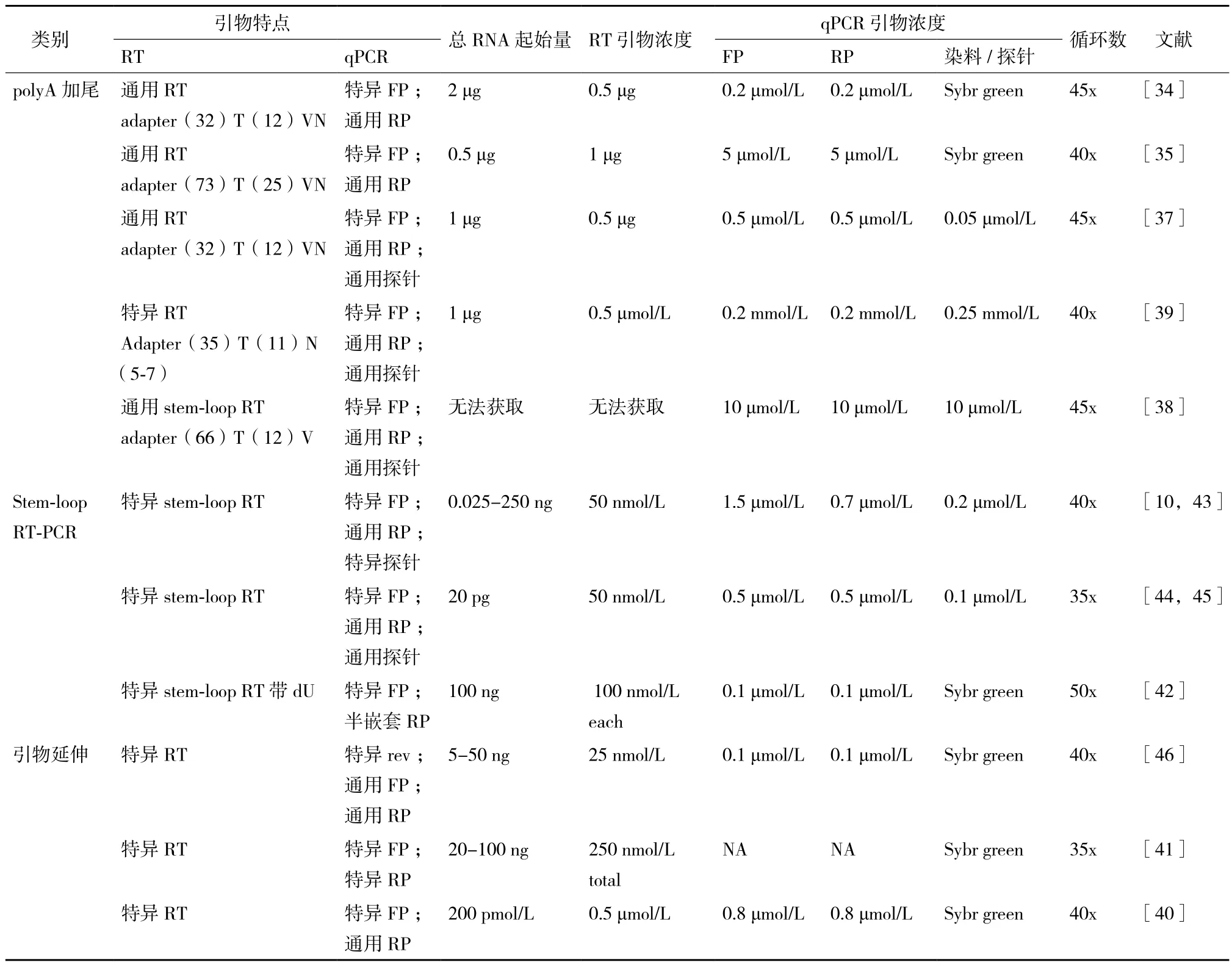
表2 miRNA qRT-PCR方法汇总表
2.2.3 Linker引物反转录 在miRNA的3'端连上linker后,由于引入了通用的反转录引物接合位点,可直接使用通用反转录引物进行反转录反应(图2-D);也可设计成miRNA特异的反转录引物。若设计为通用反转录引物,一般分为2部分,从5'端依次是延长部分,进一步引入通用qPCR反向引物位点;与linker配对部分,实现加尾后的miRNA反转录。若设计为miRNA特异的反转录引物,则设计时会进一步延伸若干碱基与miRNA配对,从而实现特定miRNA的反转录[36]。
2.2.4 反转录引物浓度设定 在引物浓度方面,进行特定miRNA检测时的RT引物浓度集中在50 nmol/L范围;如果要同时检测多个miRNA,则将这些miRNA相应的RT引物混合,每条引物均为50 nmol/L;带poly(T)的引物浓度序列明显高于其他非poly(T)的引物浓度(500 nmol/L以上)(表2)。
2.3 qPCR反应
miRNA反转录成cDNA后,均经过适当延长,已经可以进行qPCR扩增,其检测方法有染料法(如Sybr green染料)(图3-A)和探针法(如Taqman探针)(图3-B)。qPCR的引物根据其特点可分为以下几种类型。
2.3.1 染料法 (1)特异正向引物(forward primer,FP)和通用反向引物(reverse primer,RP)。特异正向引物从5'端开始依次分为延长部分以及与miRNA序列一致部分两部分:延长部分,序列为人为设计,可进行LNA修饰,主要用于调节引物的GC含量、Tm值、长度等,使其与下游引物相匹配;与miRNA序列一致部分是提供miRNA检测特异性的最重要部分,对于部分polyA加尾法来说,miRNA检测特异性直接决定于这一个部分的特异结合情况。通用反向引物来自与反转录引物的延长部分,所有的miRNA反转录引物这部分均设计相同的序列,导致可以采用通用反向引物[34,35]。(2)特异正向引物及特异反向引物。其中特异正向引物与前一种情况相同。特异反向引物来源于针对特定的miRNA设计的特异反转录引物,这些特异反转录引物除了与miRNA互补配对部分提供特异性外,针对不同的miRNA,其反转录引物延伸部分也不完全一样,而qPCR反向引物必须根据反转录引物的序列进行设计,最终导致各个miRNA有不同的特异反向引物[41];或者是使用半嵌套引物,反向引物有部分序列与miRNA互补配对,也导致不同的miRNA有特异的反向引物[42]。(3)通用正向引物、通用反向引物。这是由于在PCR体系中除了正反向引物外,还加入了另一条引物,该引物从5'端开始依次分两部分:延伸部分,提供了通用正向引物序列;与miRNA序列一致部分,提供了引物检测的特异性;通用反向引物由反转录引物序列提供[46]。
2.3.2 探针法 (1)特异正向引物、通用反向引物、特异探针。其中特异探针是由于探针设计的位置有一部分与miRNA序列匹配,导致不同的miRNA需用特异探针,进一步提高了检测的特异性[10]。(2)特异正向引物、通用反向引物、通用探针。其中探针的位置设计在反转录引物的延伸部分,由于不同miRNA其延伸部分相同或者至少探针结合位置序列相同,因此可使用通用探针[37-39,44,45]。
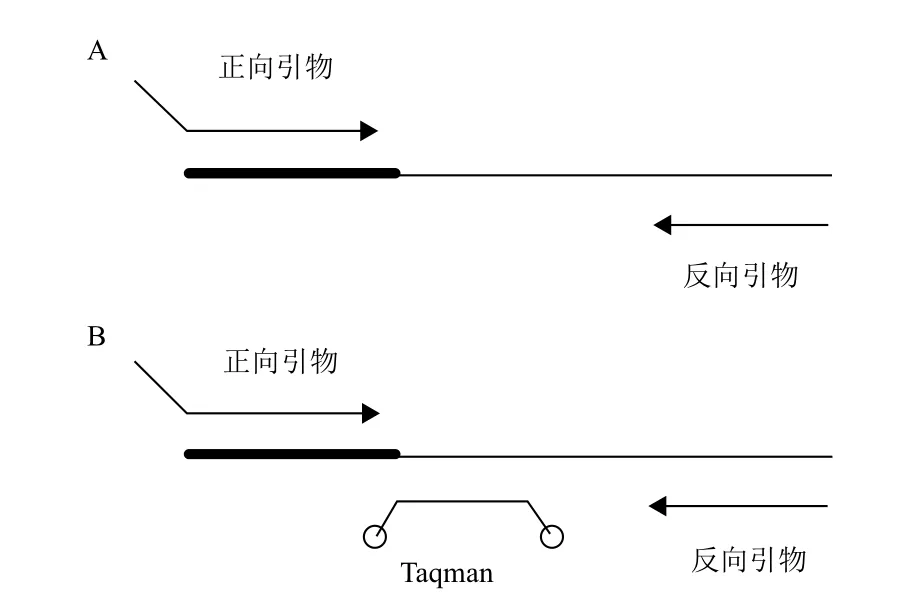
图3 miRNA qPCR反应
2.3.3 qPCR反应体系 (1)引物浓度确定。qPCR正反向引物的使用浓度集中在0.1-1 μmol/L之间(表2),其中尤以0.1-0.2 μmol/L浓度使用较多,增加正反向引物的浓度,可提高检测灵敏度,但同时也可导致非特异扩增现象的产生,因此引物浓度需经预实验确定,一般起始浓度建议0.2 μmol/L。探针的浓度集中在0.1-0.5 μmol/L之间,其浓度也需预实验确定,起始浓度建议0.1 μmol/L。(2)循环数设定。qPCR循环数集中在35 x-45 x之间(表2),循环数的增加也会导致非特异扩增现象,一般进行qPCR定量检测结果统计时循环数在18-25之间,则数据分析结果比较可信,因此建议miRNA qPCR时的循环数设置在35x即可。(3)qPCR方法选择。在miRNA进行qPCR检测的众多方法之中,何种是最好的尚无定论,从检测特异性及实验花费来说有相同的趋势:特异探针法≥通用探针法≥Sybr green染料法,在实际操作中应结合实际情况综合考虑。Adhikari等[47]比较poly(A)加尾法和stem-loop RT-PCR方法对于植物的miRNA检测效果,认为由于植物miRNA的甲基化,导致stem-loop RT-PCR检测方法比polyA加尾法更准确可信。Jung等[44]比较stem-loop RT-PCR时用通用探针获得的qPCR检测特异性和灵敏度与特异探针一致,但是通用探针可大幅降低实验消耗费用。Varkonyi-Gasic等[45]比较认为stem-loop RT-PCR方法使用通用探针法检测的特异性要优于Sybr green染料法。整体来说,poly(A)加尾法,操作过程较繁琐,但是由于引入通用poly(A)尾,可使用通用反转录引物进行反转录,适合进行miRNA qPCR表达谱组学研究;stem-loop RT-PCR方法检测灵敏度高,比较适合进行少数miRNA的特异检测。如需同时检测多个miRNA,可将这些miRNA的反转录引物混合反转录,但混合后可能会导致非特异扩增[43],因此在miRNA的反转录引物混合前最好通过预实验确定这些引物混合的适用性。
2.3.4 其他qPCR检测方法 (1)一步法qRT-PCR。Yan等[48]尝试用一步法进行miRNA的qPCR检测,该方法的原理是用一条反转录引物RT1将miRNA反转录为cDNA,用另一条引物RT2与cDNA模板结合并通过聚合酶延伸相互补齐,最后用P1与P2引物进行qPCR扩增,其中P1是RT1引入的结合位点,P2是RT2引入的结合位点。对于不同的miRNA其RT1与RT2引物均有部分与miRNA对应,因此这2条引物是特异的;P1与P2可根据需求使其变为通用引物或者miRNA特异引物。该方法的优点是一步完成qPCR检测,减少操作步骤;其最大的缺点是4条引物在一起,可能导致非特异扩增。(2)钳形探针法。Huang等[49]开发了一种钳形探针方法(Prince probe)进行miRNA二步法qPCR检测,其原理是用长链钳形探针与miRNA配对,由于钳形探针的5'端与3'端部分与miRNA完全互补配对,并且第一个碱基与最后一个碱基重叠同时利用反转录酶的活性特定,在40℃时5'端与miRNA完全互补配对,捕获miRNA;温度降低至25℃时,3'端与miRNA部分互补配对,实现miRNA的反转录;反转录产物即可用于qPCR扩增。该方法的灵敏度与特异性与stem-loop方法类似,但是其最大的优点在于可区分成熟miRNA及其前体。(3)数字PCR技术。数字PCR技术(digital PCR)是一项新的技术,目前已应用于进行miRNA的绝对定量检测[50-52],该方法分4步完成,第一步将定量PCR技术的所使用的引物、探针或者染料(如Taqman探针或者EvaGreen染料)、特殊的数字PCR反应液混合均匀;第二步使用特殊的仪器及反应板制备微滴,10 000微滴以上,每一个微滴相当于一个单独PCR反应;第三步将制备好含微滴的样本转入普通96孔PCR板并进行普通PCR扩增;第四步将PCR扩增产物使用特殊仪器进行荧光信号的检测实现对基因的绝对定量。该技术最大的特点是仪器和试剂的改进导致可进行微量样本的绝对定量检测;其最大的缺点是须使用特殊的仪器、试剂、耗材,运行成本昂贵。
3 内参基因选择
目前公认的理想内参基因需要符合以下几个条件:在不同组织及不同生理条件下表达稳定;表达量较高,易于检测;与目标基因有相似的性质,如目标基因与内参基因均为蛋白编码基因或均为非编码RNA等。其中第一条最重要也最难达到,不同物种不同生理条件下使用的内参基因各不相同(表3),即使被广泛使用的内参基因Actin和GAPDH,在一定条件下,其表达会发生改变或受到其他基因的调控[69]。因此,对特定条件下的内参基因进行实验筛选是一种严谨的做法,但要求每一位研究者对所使用的内参基因进行实验测定也不现实。理论上不存在完全理想的内参基因,只能寻找相对表达稳定的基因,对于新鲜的组织及细胞样本,用于miRNA检测的常见内参基因是U6及5S;对于血样,常见的内参基因是miR-16,借助外源加入其他物种的miRNA作内参,或者联合使用多种内参基因(表3)。
4 结语
本文对目前所使用的miRNA qPCR定量检测方法进行了总结,从RNA抽提,miRNA qPCR方法选择,miRNA qPCR流程中的引物设计、模板浓度、引物浓度、循环数等反应条件设置,内参基因选择等各方面进行了探讨,并指出了在一般条件下进行实验设计时的相应参数。miRNA qPCR技术今后的发展方向将是准确、易操作、所涉及到的试剂性价比高,如在不损失检测灵敏度与特异性的条件下尽可能采用通用引物或者通用探针,在样本量珍贵稀少时仍可准确检测等。未来对于miRNA定量检测的要求将会越发严格,包括样本制备、抽提RNA质控、内参选择、实验重复性方面将会有越来越高的要求,这样一方面可以保证实验的可重复性,并提高不同研究者的实验数据可比性;另一方面对研究者的实验设计及实验操作等方面提出了较高的要求,需要尽快适应。

表3 实验所筛选的内参基因汇总
[1] Rana TM. Illuminating the silence:understanding the structure and function of small RNAs[J]. Nat Rev Mol Cell Biol, 2007, 8 (1):23-36.
[2] Schwarz DS, Zamore PD. Why do miRNAs live in the miRNP?[J]. Genes Dev, 2002, 16 (9):1025-1031.
[3] Kozomara A, Griffiths-Jones S. miRBase:annotating high confidence microRNAs using deep sequencing data[J]. Nucleic Acids Res, 2014, 42:D68-D73.
[4] Griffiths-Jones S, Saini HK, Van Dongen S, Enright AJ. miRBase:tools for microRNA genomics[J]. Nucleic Acids Res, 2008, 36:D154-D158.
[5] Lee RC, Feinbaum R, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5):843-854.
[6] Zeng Y, Cullen BR. Sequence requirements for micro RNA processing and function in human cells[J]. RNA, 2003, 9(1):112-123.
[7] Hartig JS, Grune I, Najafi-Shoushtari SH, et al. Sequence-specific detection of MicroRNAs by signal-amplifying ribozymes[J]. J Am Chem Soc, 2004, 126(3):722-723.
[8] Allawi HT, Dahlberg JE, Olson S, et al. Quantitation of microRNAs using a modified Invader assay[J]. RNA, 2004, 10(7):1153-1161.
[9] Babak T, Zhang W, Morris Q, et al. Probing microRNAs with microarrays:Tissue specificity and functional inference[J]. RNA, 2004, 10(11):1813-1819.
[10] Chen C, Ridzon DA, Broomer J, et al. Real-time quantification of microRNAs by stem-loop RT-PCR[J]. Nucleic Acids Res, 2005,33(20):e179.
[11] Cummins JM, He Y, Leary RJ, et al. The colorectal microRNAome[J]. Proc Natl Acad Sci USA, 2006, 103(10):3687-3692.
[12] Kozomara A, Griffiths-Jones S. miRBase:annotating high confidence microRNAs using deep sequencing data[J]. Nucleic Acids Res, 2014, 42:D68-D73.
[13] Benes V, Castoldi M. Expression profiling of microRNA using realtime quantitative PCR, how to use it and what is available[J]. Methods, 2010, 50(4):244-249.
[14] Ding LF, Jiang Z, Chen QY, et al. A functional variant at miR-520a binding site in PIK3CA alters susceptibility to colorectal cancer in a Chinese Han population[J]. Biomed Res Int, 2015, clx.doi. org/10.1155/2015/373252.
[15] Chen R, Jiang N, Jiang Q, et al. Exploring microRNA-like small RNAs in the filamentous fungus Fusarium oxysporum[J]. PLoS One, 2014, 9(8):e104956.
[16] Halper B, Hofmann M, Oesen S, et al. Influence of age and physical fitness on miRNA-21, tgF-β and its receptors in leukocytes of healthy women[J]. Exec Intell Rev, 2015, 21:154-163.
[17] Tominaga N, Kosaka N, Ono M, et al. Brain metastatic cancer cells release microRNA-181c-containing extracellular vesicles capable of destructing blood-brain barrier[J]. Nat Commun, 2015, 6:6716. doi:10. 1038/ncomms7716. .
[18] Poudel S, Aryal N, Lu CF. Identification of microRNAs and transcript targets in Camelina sativa by deep sequencing and computational methods[J]. PLoS One, 2015, 10(3):e0121542.
[19] Cerdá-Olmedo G, Mena-Durán AV, Monsalve V, et al. Identification of a microRNA signature for the diagnosis of Fibromyalgia[J]. PLoS One, 2015, 10(3):e0121903.
[20] Lo YMD, Tsui NBY, Chiu RWK, et al. Plasma placental RNA allelic ratio permits noninvasive prenatal chromosomal aneuploidy detection[J]. Nat Med, 2007, 13(2):218-223.
[21] Sarrion I, Milian L, Juan G, et al. Role of Circulating miRNAs as biomarkers in idiopathic pulmonary arterial hypertension:possible relevance of miR-23a[J]. Oxid Med Cell Longev, 2015, 2015:792846.
[22] Ho YK, Xu WT, Too HP. Direct quantification of mRNA and miRNA from cell lysates using reverse transcription real time PCR:a multidimensional analysis of the performance of reagents and workflows[J]. PLoS One, 2013, 8(9):e72463.
[23] Tzimagiorgis G, Michailidou EZ, Kritis A, et al. Recovering circulating extracellular or cell-free RNA from bodily fluids[J]. Cancer Epidemiol, 2011, 35(6):580-589.
[24] Wakabayashi K, Mori F, Kakita A, et al. Analysis of microRNA from archived formalin-fixed paraffin-embedded specimens of amyotrophic lateral sclerosis[J]. Acta Neuropathol Commun,2014, 2(1):173.
[25] Pan LJ, Huang SN, He RQ, et al. Decreased expression and clinical significance of miR-148a in hepatocellular carcinoma tissues[J]. Eur J Med Res, 2014, 19(1):68.
[26] Doleshal M, Magotra AA, Choudhury B, et al. Evaluation and validation of total RNA extraction methods for microRNA expression analyses in formalin-fixed, paraffin-embedded tissues[J]. J Mol Diagn, 2008, 10(3):203-211.
[27] Reiche M, Li YJ, Li JY et al. Inhibiting plant microRNA activity:molecular SPONGEs, target MIMICs and STTMs all display variable efficacies against target microRNAs[J]. Plant Biotechnol J,2015, 13(7):915-926.
[28] Yin FQ, Qin C, Gao J, et al. Genome-wide identification and analysis of drought-responsive genes and microRNAs in tobacco[J]. Int J Mol Sci, 2015, 16(3):5714-5740.
[29] Wang YL, Zhao ZL, Deng MJ, et al. Identification and functional analysis of microRNAs and their targets in Platanus acerifolia under lead(Pb)stress[J]. Int J Mol Sci, 2015, 16(4):7098-7111.
[30] Zhou J, Cheng Y, Yin M, et al. Identification of Novel miRNAs and miRNA expression profiling in wheat hybrid necrosis[J]. PLoS One, 2015, 10(2):e0117507.
[31] Yu R, Wang Y, Xu L et al. Transcriptome profiling of root micro-RNAs reveals novel insights into taproot thickening in radish(Raphanus sativus L. )[J], BMC Plant Biol, 2015, 15(1):30.
[32] Peng J, Xia ZH, Chen L, et al. Rapid and efficient isolation of high-quality small RNAs from recalcitrant plant species rich in polyphenols and polysaccharides[J]. PLoS One, 2014, 9(5):e95687.
[33] Becker C, Hammerle-Fickinger A, Riedmaier I, et al. mRNA and microRNA quality control for RT-qPCR analysis[J]. Methods,2010, 50(4):237-243.
[34] Shi R, Chiang VL. Facile means for quantifying microRNA expression by real-time PCR[J]. Biotechniques, 2005, 39(4):519-524.
[35] Ro S, Park C, Jin JL, et al. A PCR-based method for detection and quantification of small RNAs[J]. Biochem Biophys Res Commun, 2006, 351(3):756-763.
[36] Mishim T, Mizuguchi Y, Kawahigashi Y, et al. RT-PCR-based analysis of microRNA(miR-1 and -124)expression in mouse CNS[J]. Brain Res, 2007, 1131(1):37-43.
[37] Reichenstein I, Aizenberg N, Goshen M, et al. A novel qPCR assay for viral encoded microRNAs[J]. J Virol Methods, 2010, 163(2):323-328.
[38] Luo X, Zhang J, Wang HJ, et al. PolyA RT-PCR-based quantification of microRNA by using universal TaqMan probe[J]. BiotechnolLett, 2012, 34(4):627-633.
[39] Kang K, Zhang XY, Liu HT, et al. A novel real-time PCR assay of microRNAs using SPoly(T), a specific oligo(dT)reverse transcription primer with excellent sensitivity and specificity[J]. PLoS One, 2012, 7(11):e48536.
[40] Raymond CK, Roberts BS, Garrett-engele P, et al. Simple,quantitative primer-extension PCR assay for direct monitoring or microRNAs and short-interfering RNAs[J]. RNA, 2005, 11(11):1737-1744.
[41] Wang XW. A PCR-based platform for microRNA expression profiling studies[J]. RNA, 2009, 15(4):716-723.
[42] Wan GQ, Lim QE, Too HP. High-performance quantification of mature microRNAs by real-time RT-PCR using deoxyuridineincorporated oligonucleotides and hemi-nested primers[J]. RNA, 2010, 16(7):1436-1445.
[43] Lao KQ, Xu NL, Yeung V, et al. Multiplexing RT-PCR for the detectio n of multiple miRNA species in small samples[J]. Biochem Biophys Res Commun, 2006, 343(1):85-89.
[44] Jung U, Jiang XO, Kaufmann SHE, et al. A universal TaqManbased RT-PCR protocol for cost-efficient detection of small noncoding RNA[J]. RNA, 2013, 19(12):1864-1873.
[45] Varkonyi-Gasic E, Wu RM, Wood M, et al. Protocol:a highly sensitive RT-PCR method for detection and quantification of microRNAs[J]. Plant Methods, 2007, 3:12.
[46] Sharbati-Tehrani S, Kutz-Lohroff B, Bergbauer R, et al. miR-Q:a novel quantitative RT-PCR approach for the expression profiling of small RNA molecules such as miRNAs in a complex sample[J]. BMC Mol Biol, 2008, 9:34.
[47] Adhikari S, Turner M, Subramanian S. Hairpin priming is better suited than in vitro Polyadenylation to generate cDNA for plant miRNA qPCR[J]. Mol Plant, 2013, 6(1):229-231.
[48] Yan JL, Zhang N, Qi C, et al. One-step real time RT-PCR for detection of microRNAs[J]. Talanta, 2013, 110:190-195.
[49] Huang TH, Yang J, Liu GP, et al. Quantification of mature micro-RNAs using pincer probes and real-time PCR amplification[J]. PLoS One, 2015, 10(3):e0120160.
[50] Faragó N, Kocsis ÁK, Lovas S, et al. Digital PCR to determine the number of transcripts from single neurons after patch-clamp recording[J]. Biotechniques, 2013, 54(6):327-336.
[51] Miotto E, Saccenti E, Lupini L, et al. Quantification of circulating miRNAs by droplet digital PCR:comparison of EvaGreen- and TaqMan-based chemistries[J]. Cancer Epidemiol Biomarkers Prev, 2014, 23(12):2638-2642.
[52] Li N, Ma J, Guarnera MA, et al. Digital PCR quantification of miRNAs in sputum for diagnosis of lung cancer[J]. J Cancer Res Clin Oncol, 2014, 140(1):145-150.
[53] Davoren PA, McNeill RE, Lowery AJ, et al. Identification of suitable endogenous control genes for microRNA gene expression analysis in human breast cancer[J]. BMC Mol Biol, 2008, 9:76.
[54] Ratert N, Meyer HA, Jung M, et al. Reference miRNAs for miRNAom e Analysis of urothelial carcinomas[J]. PLoS One,2012, 7(6):e39309.
[55] Wotschofsky Z, Meyer HA, Jung M, et al. Reference genes for the relative quantification of microRNAs in renal cell carcinomas and their metastases[J]. Anal Biochem, 2011, 417(2):233-241.
[56] Sanders I, Holdenrieder S, Walgenbach-Brünagel G, et al. Evaluation of reference genes for the analysis of serumn miRNA in patients with prostate cancer, bladder cancer and renal cell carcinoma[J]. Int J Urol, 2012, 19(11):1017-1025.
[57] Song JN, Bai ZG, Han W, et al. Identification of suitable reference genes for qPCR analysis of serum microRNA in gastric cancer patients[J]. Dig Dis Sci, 2012, 57(4):897-904.
[58] Zheng G, Wang H, Zhang X, et al. Identification and validation of reference genes for qPCR detection of serum microRNAs in colorectal adenocarcinoma patients[J]. PLoS One, 2013, 8(12):e83025.
[59] Li Y, Zhang LQ, Liu F et al. Identification of endogenous controls for analyzing serum exosomal miRNA in patients with hepatitis B or hepatocellular carcinoma[J]. Dis Markers, 2015, 893594 doi:10.1155/20151893594.
[60] Zhu HT, Dong QZ, Wang G, et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients[J]. Mol Biotechnol, 2012, 50(1):49-56.
[61] Jurcevic S, Olsson B, Klinga-Levan K. Validation of suitable endogenous control genes for quantitative PCR analysis of microRNA gene expression in a rat model of endometrial cancer[J]. Cancer Cell Int, 2013, 13(1):45.
[62] Tea M, Michael MZ, Brereton HM, et al. Stability of small non-coding RNA reference gene expression in the rat retina during exposure to cyclic hyperoxia[J]. Mol Vis, 2013, 19:501-508.
[63] Wang Y, Tang NP, Hui TT, et al. Identifi cation of endogenous reference genes for RT-qPCR analysis of plasma microRNAs levels in rats with acetaminophen-induced hepatotoxicity[J]. J Appl Toxicol, 2013, 33(11):1330-1336.
[64] Giu Y, Li M, Zhang K, et al. Identification of suitable endogenous control microRNA genes in normal pig tissues[J]. Anim Sci J,2011, 82(6):722-728.
[65] Wessels JM, Edwards AK, Zettler C, et al. Selection and validation of reference genes for miRNA expression studies during porcine pregnancy[J]. PLoS One, 2011, 6(12):e28940.
[66] Li J, Wei HX, Li Y, et al. Identification of a suitable endogenous control gene in porcine blastocysts for use in quantitative PCR analysis of microRNAs[J]. Sci China Life Sci, 2012, 55(2):126-131.
[67] Li D, Liu H, Li Y, et al. Identification of suitable endogenous control genes for quantitative RT-PCRanalysis of miRNA in bovine solid tissues[J]. Mol Biol Rep, 2014, 41(10):6475-6480.
[68] Johansen I, Andreassen R. Validation of miRNA genes suitable as reference genes in qPCR analyses of miRNA gene expression in Atlantic salmon(Salmo salar)[J]. BMC Res Notes, 2014, 7:945.
[69] Kou SJ, Wu XM, Liu Z, et al. Selection and validation of suitable reference genes for miRNA expression normalization by quantitative RT-PCR in citrus somatic embryogenic and adult tissues[J]. Plant Cell Rep, 2012, 31(12):2151-2163.
[70] Lin YL, Lai ZX. Evaluation of suitable reference genes for normaliza tion of microRNA expression by real time reverse transcription PCR analysis during longan somatic embryogenesis[J]. Plant Physiol Biochem, 2013, 66:20-25.
[71] Ferdous J, Li Y, Reid N, et al. Identification of reference genes for quantitative expression analysis of microRNAs and mRNAs in barley under various stress conditions[J]. PLoS One, 2015, 10(3):e0118503.
[72] Sikand K, Singh J, Ebron JS, et al. Housekeeping gene selection advisory:glyceraldehyde-3-phosphate dehydrogenase(GAPDH)and b -Actin are targets of miR-644a[J]. PLoS One, 2012, 7(10):e47510.
(责任编辑 狄艳红)
Research Advance on miRNA qPCR Methods and Its Application
FENG Shi-peng
(Hainan Key Laboratory for Sustainable Utilization of Tropical Bioresource,College of Agriculture,Hainan University,Haikou 570228)
miRNA is one of the hottest research fields in the world. The detection of miRNA expression is the fundamental content in miRNA research. Due to the feature of miRNAs being short and small, varied methods were developed, and quantitative miRNA qPCR is the most widely used one. This paper reviews the developments of miRNA extraction method, quantitative detection principle of miRNA qPCR,primer design, the reaction condition, and reference gene selection. The developments of miRNA qPCR methods in the future are also discussed. All of these may provide a reference for researchers in the field when they detect miRNA by qPCR method.
miRNA;stem-loop RT-PCR;poly(A)tailing RT-PCR;extension RT-PCR;digital PCR
10.13560/j.cnki.biotech.bull.1985.2016.02.008
2015-04-30
国家自然科学基金项目(31460178),海南省自然科学基金项目(314042),海南省教育厅项目(HNKY2014-16),海南大学科研启动项目(kyqd1306)
冯世鹏,男,博士,讲师,研究方向:miRNA功能与作用机制;E-mail:feng_shipeng@hainu.edu.cn
