枝干树皮宏基因组DNA的提取
2016-10-11赵玲云范东颖李燕芳颜霞黄丽丽
赵玲云范东颖李燕芳颜霞黄丽丽
(1. 西北农林科技大学生命科学学院,杨凌 712100;2. 西北农林科技大学植物保护学院,杨凌 712100;3. 旱区作物逆境生物学国家重点实验室,杨凌 712100)
枝干树皮宏基因组DNA的提取
赵玲云1,3范东颖1,3李燕芳1,3颜霞1,3黄丽丽2,3
(1. 西北农林科技大学生命科学学院,杨凌 712100;2. 西北农林科技大学植物保护学院,杨凌 712100;3. 旱区作物逆境生物学国家重点实验室,杨凌 712100)
旨在得到一种适用于提取多种枝干树皮的高质量DNA的方法。参考前人提取植物DNA的经验,综合了研磨时加PVP、缓冲液洗涤、SDS与CTAB结合使用、高浓度KAc低温冻融、高浓度NaCl条件下异丙醇沉淀、DNA完全溶解后加微量RNase A处理等操作,所得5种基因组DNA浓度介于190-970 ng/μL,纯度都很高,皆能被限制性内切酶切割,都可用作模板扩增到细菌16S rRNA基因片段,以苹果树皮DNA为模板扩增到苹果BIP和PDI基因且苹果树皮基因组DNA经测序公司检测,质量满足高通量测序法研究微生物多样性对环境宏基因组的要求。
枝干树皮;DNA提取;KAc;RNase A;高通量测序
苹果树腐烂病、梨枝枯病、杨树溃疡病等枝干皮层病害严重影响树木的生长,甚至导致植株的死亡。目前对此类病害的防治以施用化学药剂为主,但该方法易引发农药残留、环境污染、病原物产生抗药性等问题。20世纪80年代,微生态防治在植物病害防治中兴起[1]。植物微生态学是研究植物内微生物的组成、功能、演替、微生物互作及其与寄主间相互关系的生命分支。以往人们采用纯培养技术研究微生物的种群结构,但环境中大多数微生物处于存活但不能培养的状态[2]。Illumina公司的MiSeq、Roche公司的454等第二代测序技术直接提取微生物环境宏基因组DNA,扩增微生物保守区域内的序列可变区进行高通量测序、比对、分析,为研究环境微生物组成提供了新的手段[3]。例如Sun等[4]利用16S rDNA V1-V3区高通量测序技术研究施用生防菌Phlebiopsis gigantea后挪威云杉树桩内细菌群落的变化。若要使用高通量测序技术研究树皮内微生物组成,进而借助微生态理论防治枝干皮层病害,提取高质量的树皮宏基因组DNA是首要任务。另外,以植物树皮为材料提取遗传物质,可以降低空间和季节等因素对取样的限制,为植物分子生物学研究提供便利条件。
目前人们已成功地从植物叶片、果实等材料中提取出DNA[5,6]。植物DNA的提取方法有很多,传统的方法有CTAB法和SDS法,国内外生物公司还开发了多种商品化的植物DNA提取试剂盒,但由于不同植物中次生代谢产物的种类和含量差异很大,有时同种植物不同器官或组织的次生代谢产物的种类和含量也不一样,使得针对某种材料优化的DNA提取方法不一定适用于其它材料[7]。枝干树皮组织厚且硬,富含多糖、多酚、单宁、色素及其他次生代谢物质,用简易CTAB法分离出的DNA因多酚氧化而呈褐色,多糖、单宁等物质与DNA结合成黏稠的胶状物,用改良的CTAB法[8,9]、常用的SDSCTAB结合法[10-12]以及天根植物DNA提取试剂盒也不能避免多糖与核酸共沉淀,提取的基因组DNA纯度低,不能进行稳定的PCR扩增和限制性酶切反应。因此本研究继续尝试整合多步除杂步骤,旨在得到一种适合提取多种枝干树皮高质量DNA的方法,为应用高通量测序技术研究树皮内微生物组成和其他植物分子生物学研究提供参考。
1 材料与方法
1.1 材料
试验样品:苹果树皮、杨树树皮、梨树树皮、柳树树皮和枣树树皮采自西北农林科技大学经济树木园和周边村落。样品采集后4 h内经液氮冷冻后贮存于-80℃冰箱。
仪器:Thermo 702超低温冰箱,海门其林贝尔QL-901漩涡混合器,精宏XMTE 8112水浴锅,Eppendorf 5810R高速冷冻离心机,Thermo NanoDrop 2000分光光度计,Bio-Rad S1000TMThermalcycler PCR仪,JUNYI JY600c电泳仪,Gene Genius Bioimaging System凝胶成像系统。
试剂:PCR buffer、MgCl2和dNTPs等PCR反应试剂购自TaKaRa,Taq DNA聚合酶、限制性内切酶Hind Ⅲ、限制性内切酶EcoRⅠ等购自Thermo scientific,2×Es Taq MasterMix、RNase A购自康为世纪,16S引物由北京华大基因合成,引物MdPDIF、MdPDIR、MdBipF、MdBipR由Invitrogen公司合成,DNA Marker购自Vazyme,其他化学试剂为国产分析纯。
缓冲液BufferⅠ:200 mmol/L Tris-HCl(pH8.0),50 mmol/L EDTA(pH8.0),250 mmol/L NaCl,0.5% β-巯基乙醇。
2% SDS缓冲液:2% SDS,2% PVP,1 mol/L NaCl,100 mmol/L Tris-HCl(pH8.0),50 mmol/L EDTA(pH8.0)。
2% CTAB缓冲液:2% CTA,2% PVP,1.4 mol/L NaCl,100 mmol/L Tris-HCl(pH8.0),20 mmol/L EDTA(pH8.0)。
1.2 方法
1.2.1 树皮宏基因组DNA的提取 实验首先以苹果树皮为材料,主要参考文献[7-12]探索适合苹果枝干树皮DNA的提取方法,之后用得到的方法提取杨树、梨树、柳树、枣树树皮宏基因组DNA,观察该法通用性。具体步骤为:(1)取约1 g于-80℃保存的苹果树皮和0.1 g PVP放入预冷的研钵中,加液氮用研杵碾压样品,待剩余少量液氮时快速用力研磨,重复研磨3-4次,将适量彻底研碎的细粉迅速转移至2 mL离心管(管中体积约1 mL),加入1.5mL BufferⅠ涡旋混匀后冰浴10 min,期间不时摇匀。(2)4℃,5 000×g离心10 min,弃上清,沉淀中加入700 μL 65℃预热的2% SDS缓冲液和100 μL β-巯基乙醇混匀,65℃水浴1 h。(3)加入300 μL 5 mol/L KAC(pH6.0),混匀后-20℃放置1 h。(4)4℃,12 000×g离心10 min,取上清,加入1/5体积65℃预热的2% CTAB缓冲液,混匀,65℃水浴40 min。(5)冷却至室温,加入1/2体积酚∶氯仿∶异戊醇抽提2次,每次5 min。(6)4℃,12 000×g离心10 min,取上清,加入1/2体积5 mol/L NaCl 和等体积异丙醇,混匀后-20℃放置4 h。(7)4℃,12 000×g离心10 min,弃上清,70%乙醇洗涤沉淀两次,每次10 min,过夜风干沉淀,加100 μL灭菌水,37℃水浴2 h溶解DNA后加入0.5 μL RNase A(10 mg/mL),37℃过夜,最后置4℃冰箱备用,若长期保存应放于-80℃。
1.2.2 DNA质量检测
1.2.2.1 DNA产量和质量分析 分别取1 μL苹果树、杨树、梨树、柳树和枣树树皮DNA,用Thermo NanoDrop 2000分光光度计测定各样本DNA的浓度及A260/A280和A260/A230值。
1.2.2.2 琼脂糖凝胶电泳检测各样本DNA质量 取8 μL DNA样品与1 μL 10×loading buffer混合,90 V电压下1%琼脂糖凝胶中电泳50 min,溴化乙锭染色,Gene Genius Bio-imaging System凝胶成像系统中观察并拍照。
1.2.2.3 DNA限制性内切酶酶切 选择限制性内切酶HindⅢ和EcoRⅠ分别酶切5种树皮DNA。采用10 μL反应体系,其中DNA样品2.5 μL,限制性内切酶1 μL,10×buffer 1 μL,灭菌水5.5 μL。37℃放置2 h,90 V电压下1%琼脂糖凝胶中电泳检测酶切情况。
1.2.2.4 PCR扩增树皮内细菌16S rRNA基因序列 分别以5种树皮DNA为模板,用16S通用引物27F:5'-AGAGTTTGATCCTGGCTCAG-3';1492R:5'-GGTTACCTTGTTACGACTT-3'扩增苹果树、杨树、梨树、柳树、枣树树皮环境中细菌的16S rRNA基因。扩增体系为25 μL,其中10×PCR Buffer 2.5 μL,25 mmol/L MgCl22 μL,10 μmol/L 引物各0.5 μL,2.5mmol/L dNTP 0.4 μL,5 U/μL Taq酶0.2 μL,灭菌水18.4 μL,模板DNA 0.5 μL。所用PCR扩增程序为:95℃预变性10 min;95℃变性30 s,55℃退火30 s,72℃延伸90 s,34个循环;最后72℃延伸10 min。取8 μL PCR产物与1 μL 10×loading buffer混合,90V电压下1%琼脂糖凝胶中电泳检测扩增情况。
1.2.2.5 PCR扩增苹果基因 以苹果树皮DNA为模板,用两对引物MdBipF:5'-ACACCATGGCTGGCTCTTG-3';MdBipR:5'-TTAAAGCTCGTCATGCGACTC-3'和MdPDIF:5'-TATGGCGTCGTCTTCTAGGGT -3';MdPDIR:5'-CGTTAGAGGCTCCATCCTTTCCG-3'分别扩增苹果Bip和PDI基因,扩增体系为25 μL,其中2×Es Taq MasterMix 12.5 μL,灭菌水12 μL,模板DNA 0.5 μL。所用扩增程序同1.2.2.4,取5 μL PCR产物,90 V电压下1%琼脂糖凝胶中电泳检测扩增情况。
1.2.2.6 测序公司检验苹果树皮宏基因组DNA质量将6份苹果树皮宏基因组DNA送北京诺禾致源公司,经电泳检测合格后,以该DNA为模板扩增树皮环境中细菌16S高变区,判断 DNA质量是否满足后续建库测序需求。
2 结果
2.1 DNA浓度及纯度测定
用分光光度计测定DNA的浓度和纯度,高纯度DNA的A260/A280比值应在1.8-2.0之间,A260/A230的数值处于2.0左右时,表明小分子盐离子、色素、多糖或醇等杂质含量较少。表1显示,5种植物树皮的宏基因组DNA产量满足常规实验需求,且纯度很高。

表1 五种树皮DNA的浓度及纯度值
2.2 DNA电泳检测
琼脂糖凝胶电泳(图1)显示,5种树皮DNA各显示出一条高分子量条带,点样孔较干净,均无明显的拖尾现象,说明5种DNA都相对完整,多糖等杂质去除干净,DNA稳定无降解。
2.3 限制性内切酶酶切结果
图2显示,5种DNA都能被HindⅢ和EcoRⅠ识别并在2 h内切割完全。说明所提DNA纯度高,抑制内切酶活性的多糖等杂质含量很少。
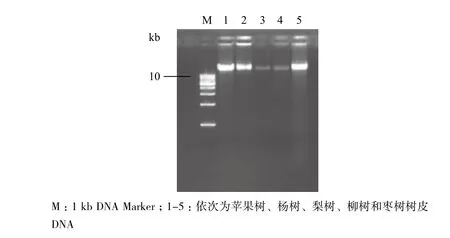
图1 五种树皮基因组DNA电泳图

图2 五种树皮基因组DNA限制性内切酶酶切图谱
2.4 树皮内细菌16S rRNA基因和苹果基因PCR扩增产物检测结果
以5种树皮DNA为模板,用16S引物扩增细菌16S rRNA基因PCR产物电泳及以苹果树皮DNA为模板扩增Bip和PDI基因,结果(图3)显示,所有扩增产物主条带明亮清晰,16S rRNA基因大小为1 500 bp左右,Bip和PDI基因3 000 bp以上,符合相应片段特征。此项结果进一步证明该方法提取到高质量的树皮宏基因组DNA。
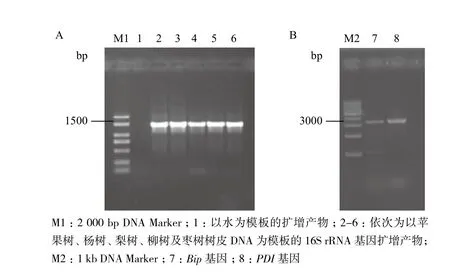
图3 五种树皮内细菌16S rRNA基因(A)及苹果Bip和PDI基因扩增产物(B)电泳图
2.5 测序公司检验苹果树皮DNA质量
经检测,本研究方法提取到的苹果树皮宏基因组DNA电泳条带整齐明亮,送样过程中略有降解,以此DNA为模板扩增到的16S rRNA基因V4+V5高变区序列为A级,即PCR产物目的条带单一(图4),大小正确,总量满足两次或者两次以上建库需要。综合各项指标确定该宏基因组DNA质量合格,可不经PCR扩增而直接用于后续建库测序。
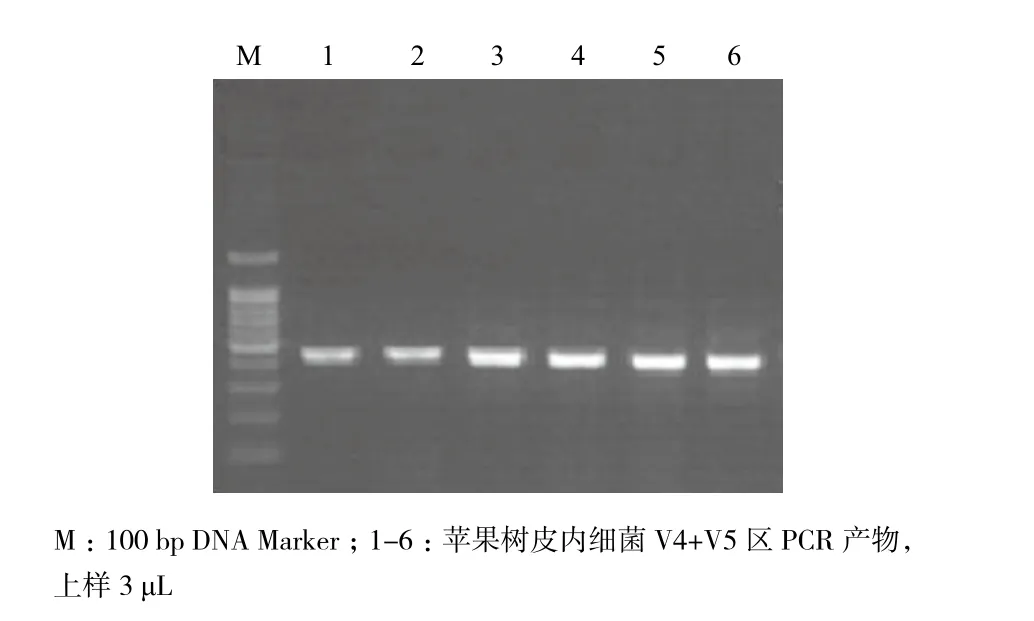
图4 苹果树皮内细菌16S rRNA基因V4+V5区PCR产物电泳图
3 讨论
提取高质量的枝干树皮宏基因组DNA是应用高通量测序技术研究树皮内微生物组成,进而借助微生态理论防治枝干皮层病害的前提,以树皮为材料提取遗传物质还可以使取样免受空间和季节等因素的限制,为限制性酶切、PCR扩增等植物分子生物学研究提供便利。从当前发表的植物DNA 提取文献看,各种方法的建立大多基于CTAB和SDS两种提取技术之上,但由于提取对象、缓冲液成分及提取步骤的差异,使得 DNA 提取效果不尽相同[13]。顽拗型枝干树皮材料厚且硬,富含干扰DNA提取的次级代谢产物,需要更完善的提取操作才能得到质量合格的DNA。
研磨好的材料中加入洗涤液BufferⅠ,洗去了大量色素等杂质,减轻后续除杂过程的负担。为了去除植物中的酚类物质,防止其氧化变褐后与核酸不可逆结合,普遍是在研磨时或提取液中加入适量的高分子螯合剂PVP和抗氧化剂β-巯基乙醇,也可以用TCEP、抗坏血酸、硼砂等试剂抑制氧化反应,避免DNA褐化[14]。适当地提高β-巯基乙醇的量还能有效去除多糖等次生物质[15]。SDS在高温(55-65℃)条件下能裂解细胞,同时与蛋白质和多糖结合成复合物,释放出核酸,之后加入高浓度KAc或NH4Ac低温放置,使形成的复合物转变为溶解度更小的钾盐形式,沉淀更加完全,离心后除去[16]。CTAB能与核酸形成复合物,此复合物在高盐(>0.7 mol/L)浓度下可溶,并稳定存在,经过酚∶氯仿∶异戊醇(25∶24∶1)抽提去除蛋白质、多糖、色素等杂质,最后用异丙醇或乙醇将DNA沉淀出来,离心弃上清后,CTAB-核酸复合物再用70%酒精浸泡可洗脱掉CTAB[17,18]。
植物组织往往富含多糖,多糖污染是提取植物DNA时遇到的最棘手的问题。多糖的许多理化性质与DNA很相似,所以很难将它们分开。Dellaporta等[19]认为加入高浓度的KAc有利于除去多糖;Fang等[20,21]认为在1.0-2.5 mol/L NaCl的高盐TE中,用无水乙醇沉淀DNA能除去多糖。
以往在提取植物DNA时,大多选用植物叶片等材料,选取以上操作的几个方面即可满足植物分子生物学的要求[5,7,9,18,21]。本研究的目的是得到适用于高通量测序的枝干树皮宏基因组DNA,对DNA质量要求较高,又因枝干树皮属顽拗型植物材料,实验综合多步抑制多酚氧化、除蛋白、除多糖等处理,才得到质量合格的基因组DNA且该DNA适用于普通植物分子生物学研究。研究中发现,略去DNA提取过程中洗涤、加KAc -20℃放置、加CTAB 65℃水浴、沉淀时加NaCl、DNA完全溶解后加RNase A中的任何一步操作,都会影响DNA的质量和稳定性,其中KAc和RNase A处理是至关重要的两个步骤,分别对所提DNA的纯度和稳定性起着决定性的作用。使用本实验方法提取5种树皮DNA,产量差异较大,可能与植物本身特性有关。
4 结论
本研究得到一种适合多种枝干树皮基因组DNA的提取方法,该法操作简单,所用试剂廉价易得,仪器常见,所得5种树皮DNA完整性好,纯度高,苹果树皮DNA满足高通量测序法研究微生物多样性对环境宏基因组DNA的要求。经PCR扩增苹果基因和5种树皮内细菌16S rRNA基因及酶切实验验证,此方法得到的DNA也适用于植物分子生物学研究。
[1] 李宝聚, 王莉, 陈捷. 植物病害微生态防治研究[J]. 北方园艺,2006(6):89-91.
[2] 段曌, 肖炜, 王永霞, 等. 454测序技术在微生物生态学研究中的应用[J]. 微生物学杂志, 2012, 31(5):76-81.
[3] 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报, 2011, 51(4):445-457.
[4]Sun H, Terhonen E, Koskinen K, et al. The impacts of treatment with biocontrol fungus(Phlebiopsis gigantea)on bacterial diversity in Norway spruce stumps[J]. Biological Control, 2013, 64(3):238-246.
[5]Sharma P, Purohit SD. An improved method of DNA isolation from polysaccharide rich leaves of Boswellia serrata Roxb[J]. Indian Journal of Biotechnology, 2012, 11(1):67-71.
[6]Sharma K, Mishra AK, Misra RS. A simple and efficient method for extraction of genomic DNA from tropical tuber crops[J]. African Journal of Biotechnology, 2008, 7(8):1018-1022.
[7]李金璐, 王硕, 于婧, 等. 一种改良的植物DNA提取方法[J].植物学报, 2013, 48(1):72-78.
[8]高洁, 李巧明. 羽叶金合欢的DNA提取和SSR引物筛选[J].云南植物研究, 2008, 30(1):64-68.
[9]Japelaghi RH, Haddad R, Garoosi GA. Rapid and efficient isolation of high quality nucleic acids from plant tissues rich in polyphenols and polysaccharides[J]. Molecular Biotechnology, 2011, 49(2):129-137.
[10]何卫龙, 杨立恒, 王晓锋, 等. 马尾松胚乳DNA高效提取及SSR-PCR体系优化[J]. 林业科技开发, 2013, 27(1):15-18.
[11]于华会, 杨志玲, 杨旭, 等. 不同提取方法对厚朴叶片总DNA提取效果的影响[J]. 中南林业科技大学学报, 2010, 30(4):45-49.
[12]Niu C, Kebede H, Auld DL, et al. A safe inexpensive method to isolate high quality plant and fungal DNA in an open laboratory environment[J]. African Journal of Biotechnology, 2008, 7(16):2818-2822.
[13]权俊萍, 贾晓鹰, 戴丽娜, 等. 薰衣草高质量DNA提取及ISSR-PCR多重化体系的建立[J]. 生物技术通报, 2012(5):151-157.
[14]陈林杨, 宋敏舒, 查红光, 等. 一种改良的植物基因组DNA通用提取方法[J]. 植物分类与资源学报, 2014, 36(3):375-380.
[15]黄绍辉, 方炎明. 改进的SDS-CTAB法提取濒危植物连香树总DNA[J]. 武汉植物学研究, 2007, 25(1):98-101.
[16]易庆平, 罗正荣, 张青林. 植物总基因组DNA提取纯化方法综述[J]. 安徽农业科学, 2007, 35(25):7789-7791.
[17] 旭芬. 基因工程实验指导[M]. 第2版. 北京:高等教育出版社,2006.
[18]Zhang L, Wang B, Pan L, et al. Recycling isolation of plant DNA,a novel method[J]. Journal of Genetics and Genomics, 2013, 40(1):45-54.
[19]Dellaporta SL, Wood J, Hicks JB. A plant DNA minipreparation:versionII[J]. Plant Molecular Biology Reporter, 1983, 1(4):19-21.
[20]Fang G, Hammar S, Grumet R. A quick and inexpensive method for removing polysaccharides from plant genomic DNA[J]. Biotechniques, 1992(13):52, 54, 56.
[21]Plant M. Optimization of DNA isolation and PCR protocol for ISSR analysis of Nothapodytes nimmoniana:A threatened anticancerous medicinal plant[J]. Asian Journal of Biotechnology,2012, 4(2):100-107.
(责任编辑马鑫)
The Extraction of Metagenom DNA in Branch Bark
ZHAO Ling-yun1,3FAN Dong-ying1,3LI Yan-fang1,3YAN Xia1,3HUANG Li-li2,3
(1. College of Life Sciences,Northwest A&F University,Yangling 712100;2. College of Plant Protection,Northwest A&F University,Yangling 712100;3. State Key Laboratory of Crop Stress Biology for Arid Areas,Yangling 712100)
A suitable method capable of extracting high-quality metagenom DNA from a variety of branch barks was developed based on previous experiences of plant DNA extraction. The method consisted of several steps:adding PVP while grinding materials, washing bark powder with bufferⅠ, simultaneously using SDS and CTAB, low-temperature frozen and then melting samples after adding high concentration of potassium acetate(KAc), precipitating DNA using isopropanol under high concentration of NaCl, and adding trace RNase A after DNA completely dissolved. Finally, the concentrations of genomic DNA obtained by this method ranged from 190 to 970 ng/μL with all in high purity. All DNA can be digested by restriction endonuclease and used as templates for bacterial 16S rRNA gene amplification. The DNA of apple bark as the template was amplified into gene BIP and PDI, then the genome DNA of apple bark was detected by a sequencing firm, and the quality satisfied the requirements of environmental metagenome for studying microbial diversity by high-throughput sequencing.
branch bark;DNA extraction;KAc;RNase A;high-throughput sequencing
10.13560/j.cnki.biotech.bull.1985.2016.01.018
2015-03-24
国家自然科学基金项目(31101476,31171796),陕西省科学技术研究发展计划项目(2013K01-45),杨凌示范区科技计划项目(2014NY-41),果树腐烂病防控技术研究与示范项目(201203034)
赵玲云,女,硕士研究生,研究方向:微生物资源与利用;E-mail:hebeizhaolingyun@163.com
颜霞,女,博士,副教授,研究方向:微生物资源与利用;E-mail:yanxia@nwsuaf.edu.cn黄丽丽,女,教授,研究方向:果树病害综合治理;E-mail:huanglili@nwsuaf.edu.cn
