芥菜型油菜A9染色体BAC物理图谱构建及比较基因组分析
2016-10-10刘芳瑛刘显军胡学芳刘旭东徐海鹏刘忠松
刘芳瑛,陆 赢,王 卓,刘显军,胡学芳,刘旭东,徐海鹏,刘忠松
(湖南农业大学油料作物研究所,长沙 410128)
芥菜型油菜A9染色体BAC物理图谱构建及比较基因组分析
刘芳瑛,陆赢,王卓,刘显军,胡学芳,刘旭东,徐海鹏,刘忠松*
(湖南农业大学油料作物研究所,长沙 410128)
为获得高精确度的芥菜型油菜A9染色体物理图,本研究利用定位在A9染色体上的标记对芥菜型油菜BAC文库进行三维混合PCR筛选,共得到阳性BAC 3200个;构建了由16个重叠群、5个单拷贝组成的芥菜型油菜A9染色体物理图谱。根据最小路径原则挑选代表整个物理图的种子BAC 435个,并进行双末端测序,得到质量良好的序列862条;按照BAC平均插入片段为126 kb,可知所有重叠群累积达52.08 M,覆盖A9染色体的1.37倍。利用BLAST软件(按参数值E<1e-10)将种子BAC末端序列比对到白菜和甘蓝型油菜A9染色体假分子上,结果显示该物理图谱分别覆盖了36.4、31.0 Mb的物理距离,相当于全基因组组装长度的93.9%、91.8%;在862条BESs中,分别有702条(81.40%)定位在白菜A9染色体上,646条(74.9%)定位在甘蓝型油菜A9染色体上,表明芥菜型油菜A9染色体同白菜A9染色体同源性高于甘蓝型油菜A9染色体;芥菜型油菜A9染色体同甘蓝型油菜A9染色体,相对白菜A9染色体6413031~7827177、14856710~18458808的位置发生了两处相同的分别长为1.4、3.6 Mb的倒位,白菜和芥菜型油菜A9染色体相对于甘蓝型油菜15676847~16064302的区域存在长为0.98 Mb的插入片段,该区域位于前述的倒位区域,而且与预测但未组装出的甘蓝型油菜着丝粒位置一致。
芥菜型油菜;A9染色体;物理图谱;比较基因组
芥菜型油菜属于十字花科芸薹属中芥菜的油用类型,是由白菜和黑芥通过杂交和染色体自然加倍形成的异源四倍体(AABB,n=2x=18,1068 Mb)[1]。在其进化过程中经历了多次全基因组倍增事件(α,β,γ,bU复制事件)[2,3],产生了大量重复序列。多倍性和重复序列使基因组增大且更复杂,从而增加了测序成本,降低了测序的准确性[4]。目前,获得基因组序列主要有两种方法[5]:全基因组鸟枪法测序策略(WGS)和基于物理图的克隆连克隆测序策略(CBCS)。新一代测序技术以高通量、时间少、低成本[6]受到科学家们的关注和青睐,但其产生的大量短序列不仅加大组装过程中计算机的运行量,而且对测序错误率敏感,在基因组重复区甚至出现组装错误或序列丢失的现象[7]。CBCS选择能覆盖整个基因组的最低数目BAC进行测序,一个克隆长约126 kb,可以跨越重复区,从而减少空隙(GAP)数,提高了组装质量。完整、高质量的基因组参考序列是进行基因预测、基因组功能注释、进化及比较基因组学研究的基础,而克隆连克隆测序策略是获得精细基因组序列的前提[8,9]。 BAC重叠群的构建主要有酶切指纹分析、分子标记筛选、BAC末端测序[10]等方法。酶切指纹分析利用HICF[11](高信息量酶切指纹技术)根据相同的指纹图谱确定BAC之间的重叠关系,再通过指纹重叠群(FPC)或线性拓扑重叠群(LTC)两种软件修饰[12]组装成重叠群。该方法可用于未知基因组测序。分子标记法包括核酸杂交[13]和PCR筛选[14]。两者都需要参考已知序列设计探针或引物对BAC文库进行筛选确定阳性克隆之间的重叠关系。该方法可以实现遗传图谱和物理图谱的整合。BAC末端测序则通过把末端序列与已知近缘生物基因组比对,从而把BAC锚定到染色体上[15],重叠群侧翼的BAC末端序列还可以用来设计探针或引物进行染色体步移筛选,填补重叠群之间的空隙。完整的物理图谱往往需要几种方法综合使用才能完成。 在芸薹属植物A、B、C三个基因组中,A、C基因组高度同源[16],其中A9染色体与C8、C9染色体部分同源[17]。根据已测序的白菜[18]和甘蓝型油菜[19]基因组数据显示,在A基因组的10条染色体中,A9染色体最长,白菜A9染色体组装长度为38.8 Mb,而甘蓝型油菜A9染色体组装长度为33.8 Mb,另有4.1 Mb序列组装方向未知、不均匀地分布在甘蓝型油菜A9染色体上。A9染色体是易于发生重排的染色体,着丝粒区域是易于发生重排的地方[20]。Cheng等[21]根据着丝粒特异序列认为白菜A9染色体的着丝粒位于10.4~11.3 Mb的位置,而在19.0~19.1 Mb也存在失活的着丝粒痕迹。Mason等[22]采用半四分体(half-tetrad)分析方法,认为甘蓝型油菜A9染色体的着丝粒位于15.6~15.9 Mb的区域,对应白菜A9染色体14.8~15.4 Mb的区域。A9染色体携带许多控制重要性状的基因如种子大小、种皮颜色、油脂合成、硫苷合成[23~26]等相关的基因,因而构建A9染色体物理图不仅具有理论意义,更具实用价值。
本研究利用定位在A9染色体的标记对构建的ZBjuH BAC文库,进行三维混合PCR 步移筛选,构建A9染色体BAC物理图谱,依据最小瓦片路径(Minimal Tiling Path,MTP)原则,挑取尽可能覆盖全基因组的BAC作为种子BAC,进行双末端测序,将测序成功的BES与已测序的白菜和甘蓝型油菜A9染色体序列进行BLAST,分析芸薹属植物A9染色体的结构变异。
1 材料与方法
1.1芥菜型油菜BAC文库的构建
提取芥菜型油菜作图群体亲本紫叶芥S8自交系叶片总DNA,构建芥菜型油菜紫叶芥BAC文库(187个384孔板)。构建步骤参照文献[27]操作,由华中农业大学罗美中教授实验室协助完成。
1.2BAC质粒池的混池方法
BAC质粒DNA的提取参照《分子克隆实验指南》(萨姆布鲁克等,2005)进行。为简化PCR筛选过程,本实验室构建了3个水平的BAC质粒DNA池(一级池、二级池和三级池)。一级池:由每相邻10块384孔板的BAC克隆质粒DNA混和成一管,共19管。二级池:由每块384孔板的BAC克隆质粒DNA混和成一管,共187管。三级池分为横向三级池和纵向三级池。横向三级池由一个384孔板中的每相邻2行共48个BAC克隆质粒DNA(如002号平板的002-A/B、002-C/D、……、002-O/P)混和而成,共187×8管;纵向三级池由384孔板中的连续2列共32个BAC克隆质粒DNA,(如004号平板的002-1/2、002-3/4、……、002-23/24)混合而成,共187×12管。在所构建的一级池、二级池、横向三级池、纵向三级池中都包含了整个BAC文库。
1.3引物的设计
共设计了引物1728对,其中参考白菜基因组[18]序列设计STS引物311对,参考芥菜型油菜四川黄籽种皮的非冗余转录本(unigene)序列设计STS引物107对,参考芥菜型油菜Survey序列设计STS引物126对,利用BAC末端序列设计引物1184对。引物设计采用Primer premier 3或Primer premier 5软件。设计引物时设定的标准为:产物大小200~500 bp,引物长度控制在20~23个碱基,正反引物Tm值60℃左右,GC含量尽量大于50%。
1.4BAC单克隆的筛选
(1)用程序降落PCR筛选BAC文库。95℃预变性5 min;94℃预变性30 s,62℃退火30 s,72℃延伸30 s,3个循环;94℃预变性30 s,59℃退火30 s,72℃延伸30 s,3个循环;94℃预变性30 s,56℃退火30 s,72℃延伸30 s,28个循环;72℃延伸10 min,22℃保存。退火温度根据引物进行适当调整。
(2)BAC文库筛选步骤。对19个一级池进行筛选,确定阳性克隆位于哪组相邻的10块384孔板中;筛选阳性一级池包含的所有二级池,确定阳性克隆位于哪块384孔板中;筛选阳性二级池对应的横向和纵向三级池,把阳性克隆定位于384孔板中横向和纵向交叉所包含的4个单克隆;对4个单克隆进行筛选,得到单个阳性克隆。PCR产物采用1.5%琼脂糖凝胶电泳检测。
1.5BAC末端测序和序列分析
BAC末端序列由上海生物工程技术公司采用Sanger方法进行测序,测序引物为M13R和S2。利用chromas软件分析BES测序峰图以判断测序质量,利用BLASTN程序与BRAD、NCBI上白菜、甘蓝型油菜基因组数据等进行序列比对分析,利用比对联配长度和E值等数据初步排除假阳性BAC,推断BAC在染色体序列假分子上的大概位置,并设计末端引物用于验证和延伸。 挑选被最多标记筛选到、代表整个物理图谱的BAC作为种子BAC,利用BLAST软件将种子BAC的两个BES分别比对到甘蓝型油菜和白菜A9染色体假分子上(E<1e-10),对芥菜型油菜A9染色体BAC物理图谱与白菜、甘蓝型油菜A9染色体进行共线性分析。
2 结果与分析
2.1芥菜型油菜A9染色体物理图
利用PCR方法进行染色体步移筛选,得到阳性BAC共3200个,将其中2070个BAC进行末端测序,获得BAC末端序列(BES)3410条,构建了由16个重叠群、5个单拷贝(BAC数低于5个)组成的芥菜型油菜A9染色体物理图谱。按照最小瓦片路径原则挑选了代表整个物理图的种子BAC 435个,并送往生工进行末端测序,共获得862条BES用于序列比对分析(表1)。按照每个BAC的平均插入片段为126 kb估算,所有重叠群累积共52.08 M,芥菜型油菜A9约为38 Mb,可知该物理图谱覆盖A9染色体的1.37倍,因此估计每个BAC延伸长度约为87.6 kb。其中最长的重叠群包含62个BACs,长约5.4 Mb,覆盖A9染色体的14.3%。N50值(达到总长度一半时,最小的contig长度)为2.8 Mb,包含32个BACs。Contig包含的BACs越多表明组装越准确。排除BAC数小于5的5个单拷贝,重叠群的平均覆盖长度约为1.68 Mb。
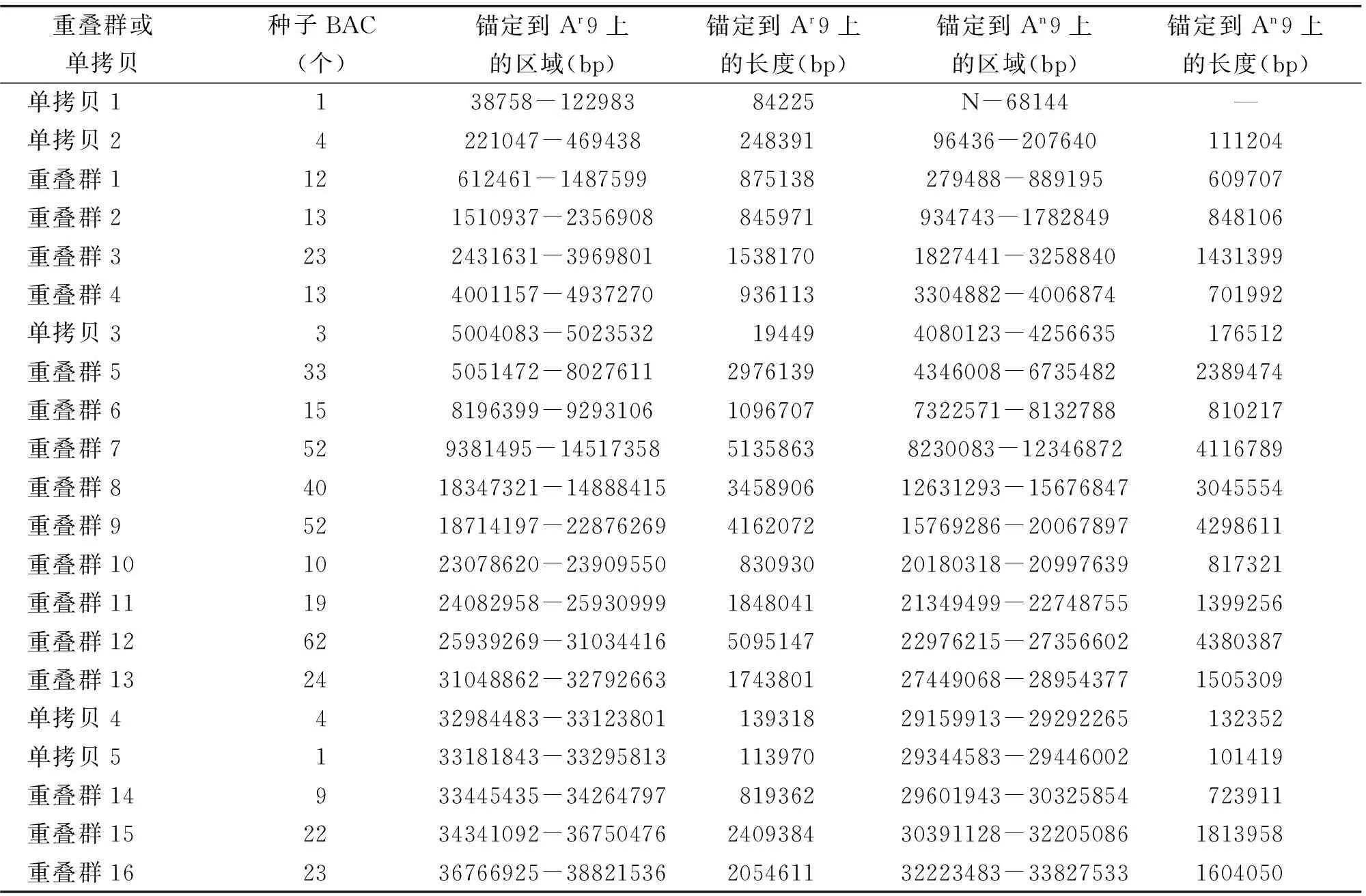
表1 芥菜型油菜A9染色体物理图及比对到白菜、甘蓝型A9染色体的区域Table 1 BAC physical map of Aj9 and positions mapped to Ar9、An9
注:Ar9表示白菜A9染色体,Aj9表示芥菜型油菜A9染色体,An9表示甘蓝型油菜A9染色体。BAC数少于5个的被定义为单拷贝。N表示未比对到A9上,—表示锚定到A9上的区域长度未知。
2.2芸薹属植物A9染色体结构比较
甘蓝型油菜和芥菜型油菜的A基因组都来源于白菜[1],但它们的基因组组成不同,前者为AACC,后者为AABB。A、C基因组亲缘关系近,但与B基因组亲缘关系远[16]。有研究表明,种间杂种的基因型会影响减数分裂时的染色体配对联会及以后的交换重组[28]。不同的基因组遗传背景下,芥菜型油菜和甘蓝型油菜是否发生同样的结构变化有待研究。本研究利用芥菜型油菜A9染色体的种子BAC末端序列,与白菜和甘蓝型油菜的假分子序列进行比较,研究A9染色体在进化过程中的结构变异。
2.2.1A9染色体宏观共线性比较
将种子BES与白菜scaffold序列[18]进行比对,芥菜型油菜A9物理图对应白菜基因组上50个scaffold。Li等[29]利用芸薹属60 K Infinium SNP芯片对472份甘蓝型油菜种质进行关联分析,对甘蓝型油菜各染色体序列支架的排列和方向也进行了整理。将3种油菜A9染色体结构进行比较(图1,封三)可知:相较于白菜,同是异源四倍体的芥菜型油菜和甘蓝型油菜A9染色体上锚定了之前白菜没有进行定位的多个序列支架,如BraScf178、BraScf321、BraScf17_front、BraScf344、BraScf185;芥菜型油菜和甘蓝型油菜又有其特异的支架,芥菜型油菜包含特异的BraScf960、BraScf70、BraScf282、BraScf460、BraScf496及插入BraScf006内的BraScf174,甘蓝型油菜包含特异的BraScf311、BraScf103、BraSc120、BraSc106,白菜包含特异的BraScf393;BraScf481在甘蓝型油菜中未找到,在芥菜型油菜中发生转座;芥菜型油菜中BraScf159和BraScf113的位置与白菜一致,与甘蓝型油菜发生颠倒;BraScf66~BraScf134与甘蓝型油菜一致,与白菜发生颠倒。
2.2.2A9染色体微观共线性比较
用BLAST将862条种子BAC末端序列(BES)定位到白菜和甘蓝型油菜A9染色体假分子上,分别覆盖了36.4、31.0 Mb的区域,分别占假分子组装长度的93.9%、91.8%。在这862条BESs中,分别有702条(81.40%)定位在白菜A9染色体上、646条(74.9%)定位在甘蓝型油菜A9染色体上,表明芥菜型油菜A9染色体与白菜A9染色体的同源性高于甘蓝型油菜A9染色体。甘蓝型油菜基因组测序时,75个总长33.8 Mb的scaffolds通过遗传标记定向锚定在A9染色体上,另有163个总长4.1 Mb的scaffolds不能通过遗传标记定向锚定到A9染色体因而被归为“chrA9_random”;“chrA9_random”上的scaffolds不均匀地分布在甘蓝型油菜A9染色体上[19]。上述646条定位到甘蓝型油菜A9染色体的BES中,591条比对在A9染色体上,55条定位在 “chrA9_random”上(图2红色区域,封三);在未定位到甘蓝型油菜A9上的224条BESs中,有47条BESs定位在“chrAnn_random”上(图2中绿色区域,封三)。与白菜A9染色体假分子[18]相比,BAC重叠群中,075M22R ~159A17L对应白菜A9染色体6413031~7827177区域有约1.4 Mb大小的倒位,对应白菜scaffold57的末端。185J13L ~176B07R之间对应白菜A9染色体14888415 ~18347321区域有约3.6 Mb倒位,对应白菜scaffold134~scaffold66。而与甘蓝型油菜A9染色体假分子[19]相比,BAC重叠群中,094O07R~108C15L之间的BES均比对到chrAnn_random上,对应甘蓝型油菜15676847~15769286的区域,该区域与预测的甘蓝型油菜着丝粒位置一致。因而,推测该缺失区域很可能是由于着丝粒区域序列高度重复或缺乏可利用的遗传标记而无法组装到A9染色体上造成的。
3 讨论
3.1BAC物理图谱构建中的问题
本研究使用A9特异性标记及BAC末端序列标记步移筛选法,构建了芥菜型油菜A9染色体BAC物理图谱。在物理图谱的构建过程中,BAC文库的筛选是最关键也是最耗时的步骤,三级DNA池的逐步筛选法大量减少了文库筛选过程,而且每个阳性克隆都经过了4次验证,结果可靠。但是,文库筛选过程中由于基因组中的重复序列仍存在假阳性现象,因而在引物设计过程中可以与白菜或甘蓝型油菜A9假分子序列比对,使引物唯一比对在预测目的区域。另外可以增加引物长度、提高退火温度、采用PCR产物验证等方法减少假阳性出现的概率。目前所构建的芥菜型油菜A9染色体物理图仍然存在一些空隙,且多对应白菜scaffold的末端。scaffold的末端一般是因为位于重复序列而无法将相邻scaffold连接起来。 因此空隙存在的地方很可能是重复区域,但也可能是由于HindIII内切酶的偏好型,导致某些区域难以形成126 kb左右的片段,而导致BAC文库中没有代表该区域的阳性克隆。因此,在BAC文库构建过程中通常需要采用多酶切的方法。
3.2芸薹属植物A9染色体比较分析
通过芥菜型油菜种子BAC末端序列与白菜、甘蓝型油菜基因组序列比对,发现芥菜型油菜和甘蓝型油菜A9染色体在异源四倍体的形成过程中发生了两处相同的染色体倒位。预测的甘蓝型油菜A9染色体着丝粒跨越其中一处倒位,同Parkin等[20]认为的A9染色体是易于发生重排的染色体,着丝粒是易于发生重排的区域结论一致。芥菜型油菜A9染色体物理图和白菜基因组包含了预测的甘蓝型油菜着丝粒区域,表明针对大而复杂基因组测序时,基于物理图的克隆连克隆测序策略优于全基因组鸟枪法测序策略。芥菜型油菜A9染色体与白菜A9染色体的同源性高于甘蓝型油菜A9染色体。甘蓝型油菜全基因组序列显示A、C亚基因组之间,特别是A1与C1,A2与C2,A9与C9之间的同源交换非常频繁[19],因此推测这有可能是因为甘蓝型油菜在进化过程中,A基因组与高度同源的C基因组之间的同源重组使甘蓝型油菜A9产生了异于白菜和芥菜型油菜的变异,也可能是甘蓝型油菜组装时,遗传标记数目有限导致scaffolds无法特异定位在A9上。
4 结论
本研究构建了由16个重叠群、5个单拷贝组成的芥菜型油菜A9染色体物理图,选择其中435个BAC作为A9染色体测序的候选克隆,通过BAC末端序列与Ar9、An9假分子比对,发现Aj9与Ar9同源性高于An9;Aj9同An9,相对Ar9发生了两处相同的分别长为1.4、3.6 Mb的倒位;Aj9物理图覆盖了An9未组装出的着丝粒区域。
[1]UN.Genome analysis inBrassicawith special reference to the experimental formation ofB.napusand peculiar mode of fertilization[J].Japanese Journal of Botany,1935,7:389-452.
[2]Bowers JE,Chapman BA,Rong J,et al.Unravelling angiosperm genome evolution by phylogenetic analysis of chromosomal duplication events[J].Nature,2003,422:433-438.
[3]Bancroft I,Bancroft I,Botanik HC.Genetics and Genomics of theBrassicaceae[M].New York:Springer,2011.
[4]Jackson SA,Iwata A,Lee SH,et al.Sequencing crop genomes:approaches and applications[J].New Phytologist,2011,191:915-925.
[5]Catherine F,Leach JE,Jane R,et al.Crop genome sequencing:lessons and rationales[J].Trends in Plant Science,2011,16(2):77-88.
[6]Shendure J,Ji H.Next-generation DNA sequencing[J].Nature Biotechnology,2008,26(10):1135-1145.
[7]Paszkiewicz K,Studholme DJ.De novo assembly of short sequence reads[J].Briefings in Bioinformatics,2010,11(5):457-472.
[8]Vivien M.Next-generation sequencing:The genome jigsaw[J].Nature,2013,501:263-268.
[9]Pan Y,Deng Y,Lin H,et al.Comparative BAC-based physical mapping ofOryzasativassp.indicavar.93-11 and evaluation of the two rice reference sequence assemblies[J].Plant Journal,2014,77(5):795-805.
[10]汤飞宇,张天真.重叠群物理图谱的构建及其应用[J].基因组学与应用生物学,2009,28(1):195-201.
[11]Luo MC,Thomas C,You FM,et al.High-throughput fingerprinting of bacterial artificial chromosomes using the snapshot labeling kit and sizing of restriction fragments by capillary electrophoresis[J].Genomics,2003,82(3):378-389.
[12]Barabaschi D,Magni F,Volante A,et al.Physical mapping of bread wheat chromosome 5A:An integrated approach[J].Plant Genome,2015,8(3):1-24.
[13]Mozo T,Dewar K,Dunn P,et al.A complete BAC-based physical map of theArabidopsisthalianagenome[J].Nature Genetics,1999,22(3):271-275.
[14]Vu GT,Caligari PD,Wilkinson MJ.A simple,high throughput method to locate single copy sequences from Bacterial Artificial Chromosome (BAC) libraries using High Resolution Melt analysis[J].BMC Genomics,2010,11(10):301.
[15]Mun J,Kwon S,Seol Y,et al.Sequence and structure ofBrassicarapachromosome A3[J].Genome Biology,2010,11(9):79-82.
[16]栗茂腾,张椿雨,刘列钊,等.芸薹属A,B和C基因组之间关系研究进展[J].遗传,2005,27(4):671-676.
[17]Panjabi P,Jagannath A,Bisht NC,et al.Comparative mapping ofBrassicajunceaandArabidopsisthalianausing Intron Polymorphism (IP) markers:homoeologous relationships,diversification and evolution of the A,B and CBrassicagenomes[J].BMC Genomics,2008,9(4):113.
[18]Wang X,Wang H,Wang J,et al.The genome of the mesopolyploid crop speciesBrassicarapa[J].Nature Genetics,2011,43(10):1035-1039.
[19]Boulos C,France D,Shengyi L,et al.Plant genetics.Early allopolyploid evolution in the post-NeolithicBrassicanapusoilseed genome[J].Science,2014,345:950-953.
[20]Parkin I.Chasing ghosts:comparative mapping in theBrassicaceae[J].Plant Genetics & Genomics Crops & Models,2010,9:153-170.
[21]Cheng F,Mandáková T,Wu J,et al.Deciphering the diploid ancestral genome of the mesohexaploidBrassicarapa[J].Plant Cell,2013,25(5):1541-1554.
[22]Mason AS,Rousseau-Gueutin M,Morice J,et al.Centromere locations inBrassicaA and C genomes revealed through half-tetrad analysis[J].Genetics,2015,115:183-210.
[23]刘显军,袁谋志,官春云,等.芥菜型油菜黄籽性状的遗传、基因定位和起源探讨[J].作物学报,2009,35(5):839-847.
[24]Ji F,Yan L,Lei S,et al.Characterization of metabolite quantitative trait loci and metabolic networks that control glucosinolate concentration in the seeds and leaves ofBrassicanapus[J].New Phytologist,2012,193(1):96-108.
[25]Hui W,Jian W,Silong S,et al.Glucosinolate biosynthetic genes inBrassicarapa[J].Gene,2011,487(2):135-142.
[26]Zhao J,Huang J,Chen F,et al.Molecular mapping ofArabidopsisthalianalipid-related orthologous genes inBrassicanapus[J].Theoretical & Applied Genetics,2012,124(2):407-421.
[27]Luo M,Wing RA.An improved method for plant BAC library construction[J].Methods in Molecular Biology,2003,236:3-20.
[28]Mason AS,Huteau V,Eber F,et al.Genome structure affects the rate of autosyndesis and allosyndesis in AABC,BBAC,CCAB[J].Chromosome Research,2010,18(6):655-666.
[29]Li F,Chen B,Xu K,et al.Genome-wide association study dissects the genetic architecture of seed weight and seed quality in rapeseed (BrassicanapusL.)[J].DNA Research,2014,21(4):1-13.
Construction of BAC Physical Map on Chromosome A9 in Brassica juncea and Comparative Genome Analysis
LIU Fangying,LU Ying,WANG Zhuo,LIU Xianjun,HU Xuefang,LIU Xudong,XU Haipeng,LIU Zhongsong*
(Oil Crops Research Institute,Hunan Agricultural University,Changsha,Hunan 410128,China)
To abtain a precise physical map ofBrassicajunceachromosome A9,the markers located on the A9 chromosome were used in this study to screenBrassicajunceaBAC library by three-dimensional mixed PCR screening.A total of 3200 positive BAC were screened out;constructing a physical map composed of 16 contigs and 4 singletons.According to the principle of the minimum path,435 BACs were chosen as seed BACs to represent the whole physical map and were subject to end sequencing,at last,862 qualified sequences were received.As the average insert size of BAC is 126 kb,the contigs accounted up to 52.08 M,covering 1.37 times of chromosome A9.By BLAST,the seed BAC end sequences were mapped to chromosome A9 pseudomolecule ofBrassicarapaandBrassicanapusrespectively,results showed that the physical map covered 36.4 Mb and 31.0 Mb,equivalent to 93.9%、91.8% of the whole genome assemble length.Among the 862 BAC end sequences,702 sequences (81.4%) were located onBrassicarapaA9 chromosome and 646 sequences(74.9%)toBrassicanapusA9 chromosome,which meansBrassicajunceaA9 chromosome have higher homology withBrassicarapaA9 chromosome thanBrassicanapusA9 chromosome.Compared withBrassicanapusA9 pseudomolecule and BAC end sequence ofBrassicajunceaA9 chromosome,there are two chromosome inversions about 1.4 Mb、3.6 Mb in length at the position of 6413031-7827177 and 14856710-18458808 inBrassicarapaA9.While compared with BAC end sequence ofBrassicajunceaA9,there is a inserted mutation about 0.98 Mb in length at the position of 15676847-16064302 ofBrassicanapusA9 pseudomolecule,which lie in one of the two chromosome inversions and was consist with the predicted position of centromere ofBrassicanapusA9 chromosome remained to assembled.
Brassicajuncea;chromosome A9;physical map;compare genomics
2016-03-07
刘芳瑛(1991-),女,硕士研究生,Email:365698105@qq.com。*通信作者:刘忠松,博士,教授,Email:zsliu48@sohu.com。
国家自然科学基金(30971799)。
S565.403.2
A
1001-5280(2016)04-0347-07
10.16848/j.cnki.issn.1001-5280.2016.04.01
