茶树炭疽病抗性的QTL分析
2016-09-21徐礼羿谭礼强王丽鸳齐桂年成浩韦康
徐礼羿,谭礼强,王丽鸳,齐桂年,成浩,韦康
1.四川农业大学园艺学院,四川 成都 611130;2.中国农业科学院茶叶研究所/国家茶树改良中心,浙江 杭州 310008
茶树炭疽病抗性的QTL分析
徐礼羿1,2,谭礼强1,2,王丽鸳2,齐桂年1*,成浩2,韦康2
1.四川农业大学园艺学院,四川 成都 611130;2.中国农业科学院茶叶研究所/国家茶树改良中心,浙江 杭州 310008
以茶树SSR遗传连锁图谱为基础,选取龙井43为母本,白毫早为父本的170株F1遗传群体为试验材料,于2014年对该群体分别进行了茶树炭疽病抗性性状的田间观测和室内侵染试验,并采用复合区间作图法对该性状进行 QTL定位与分析。结果显示:从 F1群体病叶上分离纯化出一种茶树炭疽病病菌 HZ-1,经NCBI BLAST比对,其ITS基因序列与炭疽菌(Colletotrichum sp.)的亲缘关系最近,序列相似度为99%。对F1群体的炭疽病抗性表型分析发现,田间环境下的感病单株的占比(41%)高于室内环境(24%)。QTL分析显示,在6个不同的遗传连锁群(Linkage group,LG)上共检测到8个QTLs,单个QTL的LOD阈值变幅为2.53~6.80,单个QTL的表型变异贡献率的变幅为5.6%~13.8%。LG10存在1个控制茶树炭疽病抗性性状的主效QTL,LOD值6.80,表型变异贡献率13.8%。
炭疽病抗性;QTL定位;茶树;SSR遗传图谱
炭疽病是广泛存在于木本、草本植物中,由炭疽菌(Colletotrichum)引起的真菌病害,主要分布在热带与亚热带地区[1]。在园艺植物中炭疽病原菌会攻击草莓[2-3]、芒果[4]、咖啡豆[5]、梨[6]、茶树[7]等经济作物的果实、叶片等部位,极大地降低作物的产量和品质,严重时会导致植株枯死。因此,炭疽病菌被认为是世界上排名第八的植物病原真菌[8]。目前有研究显示[9],能侵染茶树的炭疽病菌包括 C.karstii、C.fioriniae、C.fructicola、C.siamense、C.sp.1和C.sp.2等6个种。
茶树[Camellia sinensis(L.)kuntze]是多年生木本植物,其叶片是加工茶叶的原料。2015年中国茶叶产量 227.8万 t,为世界第一,因此茶树已成为我国重要的经济作物[10-12]。我国四大茶区主要分布在亚热带和热带地区,气候很适宜炭疽菌的生长和繁殖,每年的春夏之际是炭疽病害爆发的时期,全国各茶区受害严重。因此,选育具有优良炭疽病抗性茶树品种成为茶树育种的一个新方向。随着分子标记技术的发展,利用分子标记构建遗传连锁图谱,进而通过QTL(Quantitative trait locus,QTL)解析能揭示由多基因或寡基因控制的自然抗性等数量性状,找到与目标性状紧密连锁或共分离的标记,借助分子标记辅助选择能显著提高育种效率[13-14]。目前,国内外研究已发现多个控制辣椒炭疽病抗性相关的主效QTLs[15-17],茶树方面却尚未发现与炭疽病抗性相关的QTLs,故而本研究旨在利用实验室前期构建完成的SSR(Simple sequences repeats)连锁遗传图谱,对作图群体的炭疽病抗性进行田间观测和室内侵染试验,比较不同环境下群体的抗性差异,并分别进行QTL分析,发掘相关的QTL位点,为进一步精细定位和克隆相关功能基因,以及分子标记辅助育种提供理论基础。
1 材料与方法
1.1试验材料
选用龙井43(LJ43,母本)与白毫早(BHZ,父本)为亲本杂交得到的367株F1代中随机选取170株用于QTL定位的材料。
1.2炭疽病菌的鉴定
从群体中挑取病害叶片,用刀片将病菌孢子(HZ-1)从病叶病斑上刮下置于显微镜下观察后涂于PDA培养基中培养3 d。3 d后经筛选纯化培养于新的PDA培养基中。培养7 d后用得到纯化的病菌菌落提取 DNA(DP305 Plant Genomic DNA Kit,TIANGEN),采用真菌通用引物ITS1/ITS4(ITS1:TCCGTAGGTGAACCTGCGG;ITS4:TCCTCCGCTTATTGATATGC)进行PCR扩增。PCR扩增体系:2×Taq Master Mix for PAGE(Vazyme biotech,Nanjing,China)3.5μL,ddH2O4.1μL,上下游引物各0.2 μL和DNA模板20 ng。PCR反应程序:95℃预变性4 min;95℃变性30 s,55℃退火30 s,72℃延伸30 s,重复35个循环;72℃最终延伸10 min。PCR扩增产物经DNA测序,结果在NCBI上比对确认菌种。
1.3田间观测
于2014年8~9月在中国农业科学院茶叶研究所资源圃分别调查了群体亲本和 170株单株的叶片罹病率并进行分级。评价标准参考文献[18](表1)。
1.4室内侵染试验
参照 Yoshiyuki和 Katsuyuki[19-20]的病菌接种方法,取带有2片成熟叶的嫩梢5个,在叶片表面用昆虫针刺伤形成伤口,用小喷雾器喷施纯化后的茶树炭疽病菌的孢子悬浮液(事先将孢子悬浮液浓度配制为1×106个·mL-1)。侵染后将嫩梢插在装有水的容器中并套袋保湿在密闭环境中(26℃,10 000 lx,12 h light/12 h dark),7 d后观察并记录病斑直径。重复试验1次,以计算算术平均数作为结果进行抗性分级(表1)。
1.5数据分析,图谱构建与QTL定位
使用DNASTAR SeqMan(http://www.dnastar.com)对菌株测序结果进行拼接,拼接序列在NCBI上比对。采用Excel2016(https://products.office.com)进行表型数据分析并作图。利用已构建完成的具有237个标记的SSR遗传连锁图谱(LJ43♀×BHZ♂)作为定位图谱[21],该图谱总长1156.0 cM,平均图距为5.21 cM,共有15个连锁群。在MapQTL5.0(https://www.kyazma.nl/index.php/mc.MapQTL)软件中对茶树炭疽病抗性进行分析,采用复合区间作图法(MQM Mapping,MQM),先根据区间作图法(Interval Mapping,IM)的结果选择离连锁群上LOD峰值最近的标记作为辅因子(Cofactor),再进行 MQM Mapping。本试验中若连锁群区间的 LOD 值>2.5,则认为该连锁群区间中存在1个QTL位点,并计算QTLs对应的贡献率。QTL的命名遵循McCouch[22]的命名原则。QTL定位结果通过Mapchart(http://www.microm-online.de)软件作图。

表1 茶树炭疽病抗性分级Table 1 The classification of anthracnose resistance in tea plants
2 结果与分析
2.1茶树炭疽病菌的鉴定
拼接序列(表2)经NCBI BLAST比对结果表明(图1),本试验中所观测、分离、纯化的病菌菌株 HZ-1与 Colletotrichum sp.和Colletotrichum gloeosporioides的亲缘关系最近,且序列相似度均为 99%。因此,可以推断本试验中所使用的病菌 HZ-1确为炭疽菌。
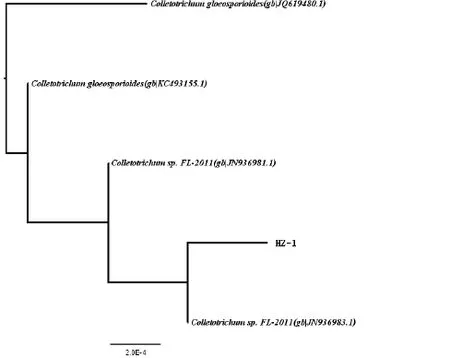
图1 真菌ITS基因序列的进化树Fig.1 The evolutionary tree of ITS gene in fungi

表2 炭疽菌(HZ-1)ITS基因序列Table 2 ITS gene sequence of HZ-1

图2 室内试验前后病菌孢子形态对比Fig.2 The shape contrast of spores before and after the indoor test
室内试验中发病叶上的病菌孢子与用于侵染的病菌孢子在显微镜下观察形态一致(图2),表明发病叶的病斑确为病菌HZ-1的侵染而造成的。
2.2亲本与F1群体的表型抗性
室内试验表明(图 3),病菌感染造成的病斑直径在0.95~7.15 mm之间,并呈现出正态分布的趋势。其中,病斑直径在2~3 mm范围的群体单株数最多,达到85株。亲本BHZ和LJ43的病斑直径分别为0.95 mm和3.10 mm。说明该群体及亲本对炭疽菌的抗性存在明显的差异。
对室内试验数据进行抗性分级后与田间调查进行比较,结果表明(图4):双亲的抗性表型保持一致,母本 BHZ抗性等级均为Lv.3,父本 LJ43抗性等级均为 Lv.7,而 F1群体的抗性表型分布存在一定差异。在田间调查的数据中,除抗性等级Lv.9的单株仅占9%外,Lv.3、Lv.5、Lv.7各抗性等级分布较为均衡,分别为28%、31%、32%。在室内试验中,抗性等级分布较为集中,超过70%的单株抗性等级为Lv.5,而Lv.3、Lv.9仅各占2%和4%。
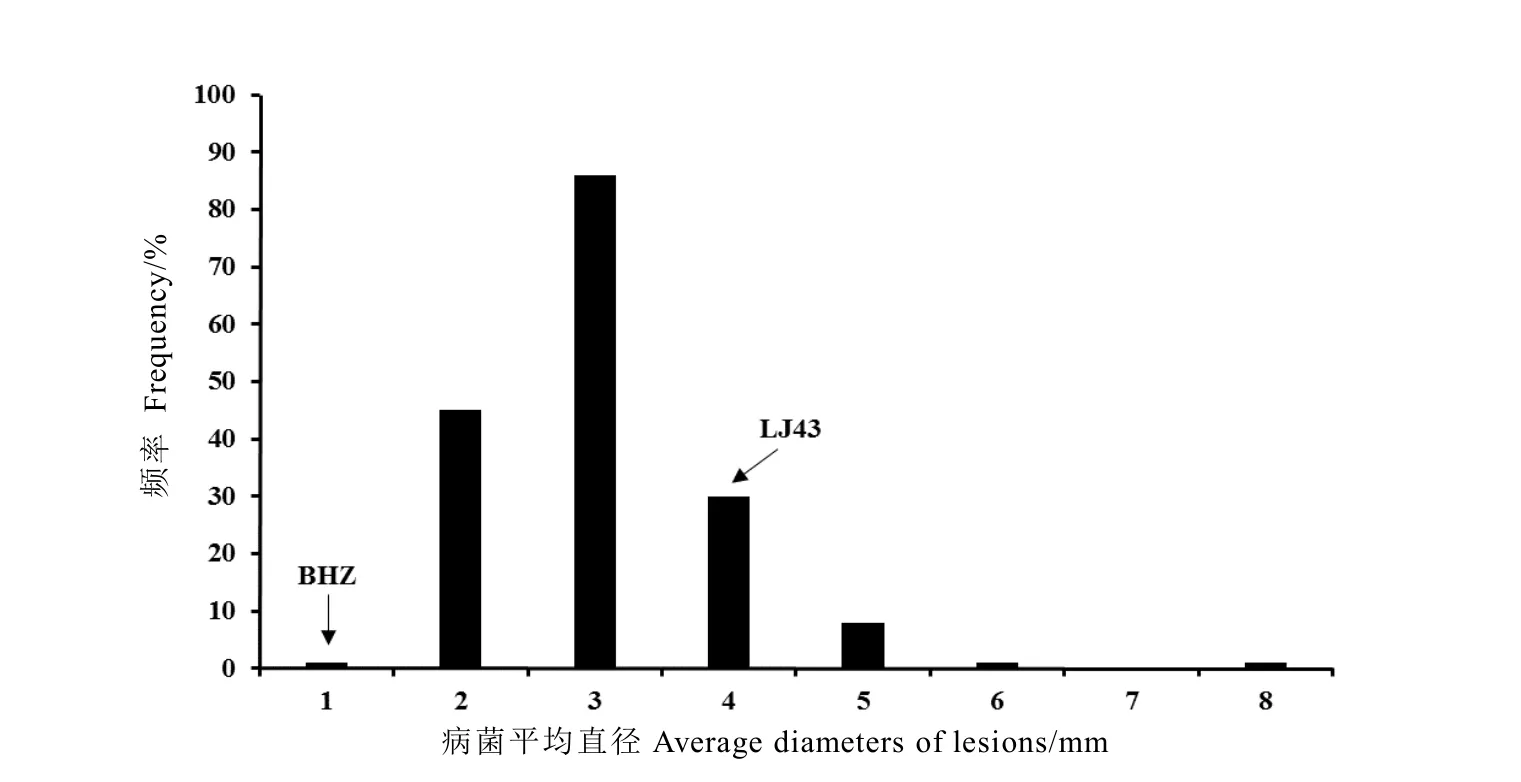
图3 室内试验中病斑平均直径的分布情况Fig.3 The distribution of average diameters of lesions in the indoor test
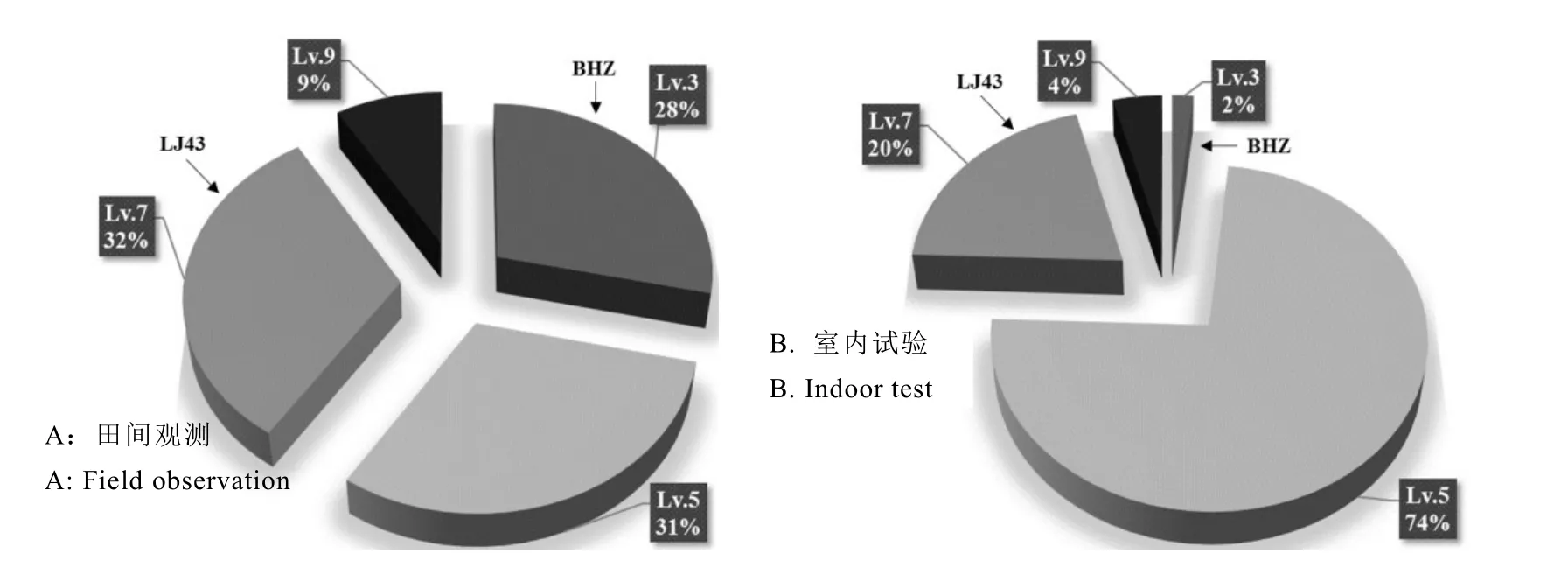
图4 亲本与F1群体的茶树炭疽病抗性等级分布Fig.4 The distribution of resistance levels of tea plant in the parent and F1 group
2.3炭疽病抗性的QTL分析
田间观测的数据分析结果显示(表3,图5),共检测到3个炭疽病抗性的QTL位点,分别分布在第6、第8、第10号连锁群上,命名为 qAR-6.1、qAR-8.1、qAR-10.1,贡献率的变异幅度为8.3%~13.8%。与qAR-6.1相关联的标记为CsFM1719,与qAR-8.1相关联的标记为 CsFM1865,与 qAR-10.1相关联的标记为CsFM1889,上述QTL位点的LOD值分别为4.31、4.31、6.80。
室内试验的数据分析结果显示,共检测到5个炭疽病抗性的QTL位点,分别分布在第3、第6、第10、第13、第15连锁群上,命名为qAR-3.1、qAR-6.2、qAR-10.2、qAR-13.1、qAR-15.1,贡献率的变异幅度为5.6%~7.9%。与 qAR-3.1相关联的标记为 CsSSR110,与qAR-6.2相关联的标记为 CsFM1769,与qAR-10.2相关联的标记为 CsFM1106,与qAR-13.1相关联的标记为TM425,与qAR-15.1相关联的标记为CsFM1931,上述QTL位点的LOD值分别为6.0、5.6、6.9、6.7、7.9。
综上所述,在田间观测和室内试验中发现的抗炭疽病的QTL位点并不完全一致,并且表型数据显示各抗性等级占比存在差异,说明该性状受环境因素影响较大。

表3 炭疽病抗性QTL定位结果Table 3 The consequence of QTL mapping for anthracnose resistance

图5 茶树炭疽病QTLs在连锁群上的位置Fig.5 Location of QTLs for tea plant anthracnose on the linkage map
3 讨论
3.1不同环境条件下茶树炭疽病抗性的差异
茶树的炭疽病抗性是受环境因素影响较大的性状,因此本研究主要从田间观测数据与室内试验数据对该性状进行QTL定位,并相互验证。在表型数据统计分析和QTL分析中,通过比较可以发现田间观察数据和室内试验数据均不完全一致。本研究中,在田间环境感病植株(Lv.7、Lv.9)所占的比例(41%)要高于室内环境(24%)。因此我们推测:相较于室内试验的密闭、恒定光照和温度的环境,田间环境开放,所具有的空气流动、气温变化,降雨量的改变等因素有利于病菌孢子在群体间相互传播,对植株造成多次侵染。该推测与Mouen[23]的研究结果相一致,其研究认为气温的波动和降水的变化是炭疽病蔓延的重要因素。
3.2茶树炭疽病QTL分析
目前,国内外对茶树数量性状的相关QTL研究较少。田中淳一等[24]采用 RAPD标记对“薮北”ד静-印杂131”F1群体的46个单株构建遗传连锁图谱后进行QTL定位,检测到 1个与茶氨酸含量相关的 QTL。Kamunya等[25]采用 100个标记对阿萨姆茶种内杂交的43个F1单株构建遗传连锁图谱,共检测到18个与茶树产量相关的QTLs。马建强[26]、王雪敏[27]采用SSR标记以“迎霜”ד北跃单株”F1群体的 183个单株构建遗传连锁图谱,共检测到3个与咖啡碱含量相关的主效QTLs、3个可可碱含量相关的主效QTLs、12个芽叶形态相关的主效 QTLs、9个与儿茶素组分相关的主效QTLs。
本研究首次对茶树炭疽病抗性性状进行QTL定位,在QTL定位过程中,共有8个与茶树炭疽病抗性相关的QTLs被发现。其中第10号遗传连锁群上28.7~30.7 cM处的QTL位点 qAR-10.1,LOD阈值 6.80,表型变异率13.8%为主效 QTL位点,受环境因素影响较小,而且在室内试验中其邻近区域 36.9~38.7 cM处也检测到一个QTL位点qAR-10.2,LOD阈值2.92,表型变异率6.9%。其他QTL位点的表型变异率在5.6%~8.8%之间。李慧慧等[28]研究认为,对200个样本的群体而言,PVE=5% 的 QTL定位到 10 cM置信区间内的概率为77%,PVE=10%的QTL定位到10 cM置信区间内的概率为 97%。因此我们初步判断 QTL位点qAR-10.1的邻近区间内应存在与茶树炭疽病抗性的相关基因。第 3、6、8、13、15号连锁群上检测到的QTLs未能在田间环境和室内环境条件下于连锁群的邻近区间上重复检测到,推测其为受环境影响较大的微效QTLs。下一步可以结合QTL位点紧密连锁的SSR标记对QTL进行精细定位,发掘出相关基因。
[1]Cannon PF,Damm U,Johnston PR,et al.Colletotrichum-current status and future directions [J].Studies in Mycology,2012,73:181-213.
[2]Fang XP,Chen WY,Xin Y,et al.Proteomic analysis of strawberry leaves infected with colletotrichum fragariae [J].Journal of Proteomics,2012,75(13):4074-4090.
[3]Weber N,Veberic R.Mikulic-Petkovsek M,et al.Metabolite accumulation in strawberry(Fragaria × ananassa Duch.)fruits and runners in response to Colletotrichum nymphaeae infection [J].Physiological and Molecular Plant Pathology,2015,92:119-129.
[4]Hong KQ,Gong DQ,Zhang LB,et al.Transcriptome characterization and expression profiles of the related defense genes in postharvest mango fruit against Colletotrichum gloeosporioides [J].Gene,2016,576(1):275-283
[5]Loureiro A,Nicole MR,Várzea V,et al.Coffee resistance to Colletotrichum kahawae is associated with lignification,accumulation of phenols and cell death at infection sites [J].Physiological and Molecular Plant Pathology,2012,77(1):23-32.
[6]Sanders GM,Korsten L.Comparison of cross inoculation potential of south african avocado and mango isolates of colletotrichum gloeosporioides [J].Microbiological Research,2003,158(2):143-150.
[7]Katsuyuki Y,Akiko O,Kengo Y,et al.Induction of disease resistance in tea(Camellia sinensis L.)by plant activators [J].JARQ,2010,44(4):391-398.
[8]Dean R,JAL Van Kan,Pretorius ZA,et al.The top 10 fungal pathogens in molecular plant pathology [J].Molecular Plant Pathology,2012,13:414-430.
[9]刘威.茶树炭疽病的病原鉴定及其遗传多样性分析[D].福州:福建农林大学,2013.
[10]Tapan K.Tropical Species,Tea Breeding[M].Springer New York:Breeding Plantation Tree Crops,2009:545-587.
[11]封槐松.2015年全国茶叶生产稳定发展结构优化产值增加[J].茶世界,2016(1):31-32.
[12]Lee CJ,Chen LW,Chen LG,et al.Correlations of the components of tea tree oil with its antibacterial effects andskin irritation [J].Journal of Food and Drug Analysis,2013,21(2):169-176.
[13]Xu YB,Crouch JH.Marker-assisted selection in plant breeding:from publications to practice [J].Crop Science,2008,48(2):391-407.
[14]Sun CY,Mao SL,Zhang ZH,et al.Resistances to anthracnose(colletotrichum acutatum)of Capsicummature green and ripe fruit are controlled by a major dominantcluster of QTLs on chromosome P5 [J].Scientia Horticulturae,2015,181:81-88.
[15]Lee J,Hong J,Do J,et al.Iden-tification of QTLs for resistance to anthracnose to two colletotrichum species inpepper [J].Crop Sci Biotechnol,2010,13:227-233.
[16]Kim S,Kim KT,Kim DH,et al.Identification of quantitative trait loci associated withanthracnose resistance in chili pepper(Capsicum spp.)Korean [J].Hortic SciTechnol,2010,28:1014-1024.
[17]Voorrips RE,Finkers R,Sanjaya L,et al.QTL mapping of anthrac-nose(Colletotrichum spp.)resistance in a cross between Capsicum annuum and C.Chinense [J].Theor Appl Genet,2004,109:1275-1282.
[18]陈亮,杨亚军,虞富莲,等.茶树种质资源描述规范和数据标准[M].北京:中国农业出版社,2005.
[19]Yoshiyuki.Phenotypes and genotypes related to tea gray blight disease resistance in the genetic resources of tea in Japan [J].JARQ,2003,37(1):31-35.
[20]Katsuyuki,Yoshiyuki.Evaluation of anthracnose resistance among tea genetic resources by wound-inoculation assay [J].JARQ,2006,40(4):379-386.
[21]Tan LQ,Wang LY,Qi GN,et al.Floral transcriptome sequencing for ssr marker development and linkage map construction in the tea plant(Camellia sinensis)[J].Plos one,2013,8(11):e81611.
[22]McCouch SR,Cho Y G,Yano M,et al.Report on QTL nomenclature [J].New slett,1997,14:11-13.
[23]Mouen B,Bieysse D,Njiayouom I,et al.Effect of cultural practices on the development of arabica coffee berry disease,caused by colletotrichum kahawae [J].Crop Protection,2007,119:391-400.
[24]田中淳一,渡部育夫.チセの成叶のテアニンの含量のQTL解析[J].茶業研究報告,1996,84(别册):46-47.
[25]Kamunya SM,Wachira FN,Pathak RS,et al.Genomic mapping and testing for quantitative trait loci in tea [Camellia sinensis(L.)O.Kuntze][J].Tree Genetics & Genomes,2010,6:915-929.
[26]马建强.茶树高密度遗传图谱构建及重要性状 QTL定位[D].杭州:中国农业科学院茶叶研究所,2013.
[27]王雪敏.茶树儿茶素含量的遗传与 QTL分析[D].杭州:中国农业科学院茶叶研究所,2013.
[28]李慧慧,张鲁燕,王建康.数量性状基因定位研究中若干常见问题的分析与解答[J].作物学报,2010,36(6):918-931.
QTL Analysis for Anthracnose Resistance in Tea Plant(Camellia sinensis)
XU Liyi1,2,TAN Liqiang1,2,WANG Liyuan2,QI Guinian1*,CHENG Hao2,WEI Kang2
1.College of Horticulture,Sichuan Agricultural University,Chengdu 611130,China; 2.National Center for Tea Improvement,Tea Research Institute of the Chinese Academy of Agricultural Sciences(TRICAAS),Hangzhou 310008,China
In order to provide a basis for breeding tea plants with anthracnose resistance,170 F1 plants,derived from LJ43♀×BHZ♂,were used to constructed a linkage map by SSR markers.Field observation and indoor test were carried out in 2014.The data of phenotypic characters were used for QTL mapping and analysis by the method of MQM mapping.A pathogen was isolated from a diseased leaf of the F1 plants,and its gene sequence of ITS had 99% similarity with Colletotrichum sp.based on NCBI BLAST.The plants grown in open field were more easy to be infected by the pathogen than those grown in rooms.Eight QTLs were detected in six different linkage groups by QTL analysis.The LOD and PVE of individual QTLs ranged from 2.53 to 6.80 and 5.6% to 13.8%,respectively.A main QTL with LOD 6.80 and PVE 13.8% was detected in LG10.
anthracnose resistance,QTL mapping,tea plant,SSR linkage map
S571.1;S435.711
A
1000-369X(2016)04-432-08
2016-01-19
2016-02-15
四川省茶树育种科技专项(2012-12CGZHZX0579)、浙江省茶树农业新品种选育重大科技专项(2012C12905)、国家茶叶产业技术体系(nycytx-23)。
徐礼羿,男,硕士研究生,主要从事茶树分子生物学研究。*通讯作者:guinian5612@163.com
