猪EGFL6基因生物信息学分析
2016-09-21孙彩霞乔瑞敏韩雪蕾李新建河南农业大学牧医工程学院郑州450002
孙彩霞,李 明,乔瑞敏,韩雪蕾,李新建(河南农业大学牧医工程学院,郑州 450002)
猪EGFL6基因生物信息学分析
孙彩霞,李明,乔瑞敏,韩雪蕾,李新建*
(河南农业大学牧医工程学院,郑州450002)
为了解猪EGFL6基因基本特性,克隆该基因CDS区,运用生物信息学方法预测分析其氨基酸理化性质、同源性、蛋白功能及二三级结构等。结果表明,猪EGFL6基因CDS区全长1 665 bp,编码554个氨基酸,与人、马、小鼠、绵羊等氨基酸相似性均在75%以上,亲缘关系较近,该蛋白具有亲水性和不稳定性,是分泌蛋白,有12个潜在磷酸化位点,5个保守结构域,无跨膜结构区,无规则卷曲是二三级结构中最主要结构元件。该研究为进一步揭示猪EGFL6基因功能奠定基础。
EGFL6;生物信息学;猪
孙彩霞,李明,乔瑞敏,等.猪EGFL6基因生物信息学分析[J].东北农业大学学报,2016,47(4):16-23.
Sun Caixia,Liming,Qiao Ruimin,et al.Bioinformatics analysis ofEGFL6 gene in pig[J].Journal of Northeast Agricultural University,2016,47(4):16-23.(in Chinese with English abstract)
网络出版时间2016-4-22 10:01:11[URL]http://www.cnki.net/kcms/detail/23.1391.S.20160422.1001.010.html
表皮生长因子样结构域蛋白6(Epidermal growth factor-like domain 6,EGFL6)是类表皮生长因子超家族蛋白成员之一,Yeung等首次发现,证实其位于人的Xp22染色体,因具有MAM结构域,故又命名为MAM[1-2]。人EGFL6蛋白含有553个氨基酸,相对分子质量为6.1×104,包含5个类表皮生长因子结构域和一个羧基端MAM结构域。小鼠、狗、猪的EGFL6基因均有12个外显子组成,位于X染色体;鸡EGFL6基因有12个外显子组成,位于1号染色体,而斑马鱼EGFL6基因有13个外显子组成,位于9号染色体。研究表明,EGFL6基因在一些肿瘤组织如乳腺癌、肺癌、脑脊膜瘤、黑色素瘤和卵巢癌等肿瘤组织呈高表达趋势,尤其是卵巢癌TEC中表达量较高[3-4]。
亢中奎等首次开展人EGFL6基因对肿瘤细胞株A375和SKOV3增殖试验,发现人EGFL6促进A375和SKOV3增殖,但增殖作用机制有待研究[5];吕成定等表明,沉默EGFL6基因表达抑制人黑素瘤A375细胞增殖和侵袭能力,有可能成为黑素瘤治疗潜在靶标[6];EGFL6基因在癌症发生和发展中发挥重要作用,可作为潜在肿瘤分子标记物,为诊断和治疗提供依据。EGFL6基因表达于小鼠毛囊和颅面区间质[7],通过旁分泌途径对肥胖人群脂肪组织具有上调作用,成熟脂肪组织是EGFL6主要来源,促进脂肪组织衍生的基质血管细胞(ADSVC)增殖[8]。EGFL6基因在成骨样细胞上表达,促进内皮细胞血管生成和移植[9],表明EGFL6参与系列生理过程对血管生成和细胞移植具有重要作用。杨晶等制备较高效价EGFL6多克隆抗体,不仅可与原核表达EGFL6全长及片段蛋白特异性结合,也能识别细胞株中天然EGFL6蛋白,为进一步研究EGFL6相关性质与功能提供基础[10]。
目前,猪EGFL6基因相关研究相对较少。本试验通过前期高通量测序筛选,发现EGFL6基因在不同品种猪群中表达差异显著,可为预测其生物信息学功能,深入了解EGFL6基因及后续研究奠定基础。
1 材料与方法
1.1EGFL6基因序列获取
1.1.1试验样品
采集猪肾脏组织,液氮保存,以备后用。
1.1.2主要试验步骤
① TRlzon提取总RNA,PrimeScriptTMRT re⁃agent Kit with gDNA Eraser(Taraka)试剂盒反转录,获得全基因组cDNA;
②以NCBI上XM_005673431.2为模板设计引物,PCR扩增;
③将扩增产物送上海生工测序,获得EGFL6基因编码区序列。
1.2同源性分析
从NCBI-Blastp对比结果中选择几种具有代表性物种,如人、马、犬、猫、大鼠、小鼠、绵羊等,并下载其氨基酸序列,利用MEGA6.0进行序列比对并构建进化树。
1.3EGFL6生物信息学分析
利用NCBI、ExPASy、ProtFun等公共数据库及在线软件包分析EGFL6基因生物信息学,预测其理化性质、蛋白质功能、信号肽、保守结构域、跨膜结构域、以及疏水性/亲水性、亚细胞定位、激酶磷酸化修饰位点、二级和三级结构。主要软件或在线网站相关信息见表1。

表1 预测基因结构和功能应用的网站和软件Table 1 Webs and software for predicting the gene structure and function
2 结果与分析
2.1EGFL6基因序列信息
通过PCR扩增,并测序,结果表明,EGFL6基因CDS(Coding sequence)区长 1 665 bp,对 ORF (Open reading frame)阅读框预测,编码554个氨基酸(见图1)。
2.2同源性分析
通过NCBI数据库中Blastp程序,将EGFL6氨基酸序列进行同源性比对,数据显示,猪与人、马、犬、猫、牛、小鼠、大鼠、绵羊、鸡、鸭、斑马鱼氨基酸序列相似性分别为79%、83%、81%、80%、76%、76%、75%、81%、59%、60% 和48%;利用Mega 6.0软件对12个物种EGFL6基因氨基酸序列多重比对,构建系统进化树(见图2)
2.3EGFL6氨基酸一级结构及理化性质预测
通过ExPASy ProtParam在线预测EGFL6基因编码氨基酸序列组成成分和理化性质。结果显示,554个氨基酸原子组成式为C2645H4205N779O798S45,共8 472个原子,相对分子质量为61 128.9。带正电荷氨基酸数目(77)大于带负电荷氨基酸数目(64),等电点(PI)为8.56,偏碱性。EGFL6在280 nm波长下消光系数为63 225m-1·cm-1(假设所有半胱氨酸均来自胱氨酸)和60 850m-1·cm-1(假设无半胱氨酸残基存在),半衰期为30 h。不稳定系数<40表示蛋白质稳定,>40表示蛋白质不稳定[11],预测不稳定系数分值为44.02,表示EGFL6蛋白质不稳定。脂肪系数为70.04,预测总平均亲水系数为-0.462。蛋白质疏水性预测是根据GRAVY预测,GRAVY值范围2~-2,正值表明此蛋白为疏水性蛋白,负值表明为亲水蛋白。因此,该蛋白为亲水蛋白。

图1 EGFL6基因CDS序列及预测的氨基酸序列Fig.1 CDS and predicted amino acid sequence of EGFL6
2.4EGFL6蛋白质功能预测
Protfun 2.2预测EGFL6蛋白细胞水平功能、酶类别及基因本体论范畴,结果见表2。该蛋白细胞水平功能是细胞膜组成(0.790),可能参与氨基酸生物合成、运输和绑定;属于酶类可能性为0.456,最可能是水解酶;在基因本体范畴预测中应激反应可能性为0.136,机率最大为1.550,其对转录调节有重要作用。

图2 不同物种系统进化树Fig.2 Phylogenetic tree of different species
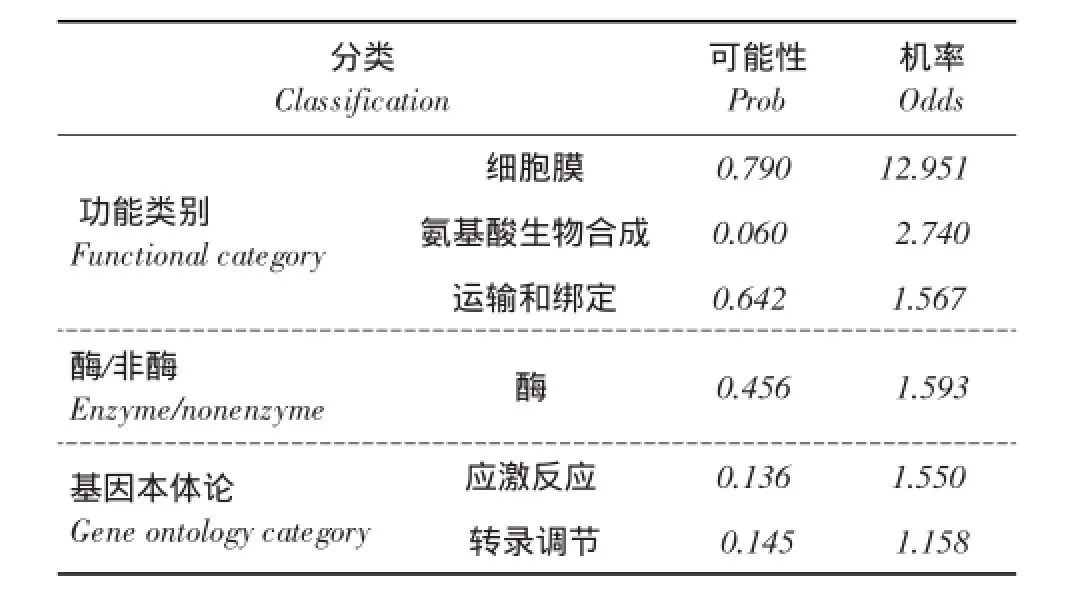
表2 EGFL6蛋白功能预测Table 2 Function prediction of EGFL6 protein
2.5EGFL6信号肽预测
Signal P-4.1 Server软件预测EGFL6信号肽,结果见表3。C(C-score)代表原始信号肽裂解位点记分,C值越大表示该点出现裂解位点可能性越大;S(S-score)代表信号肽评分,评分越大表示该氨基酸位于信号肽区域概率越大,评分越低表示相应氨基酸不含信号肽或位于成熟蛋白部分;Y(Y-score)是最有可能的信号肽裂解位点,基于C值和S值的斜率得出几何平均数,在Y值最大处具有最大C值和陡峭S斜率。D(D-score)是S均值和最大Y值加权平均值,用于区分信号肽和非信号肽。D=0.450是区分信号肽和非信号肽临界值,EGFL6蛋白预测D=0.813,因此根据信号肽假说推测EGFL6蛋白含有信号肽,属于分泌蛋白,且剪切位点位于18~19个碱基VAG-GG(见图3)。

表3 EGFL6保守结构域位点Table 3 Site of EGFL6 conserved domain
2.6EGFL6保守结构域预测
通过NCBI保守结构域数据库(Conserved Do⁃main Database,CDD)分析EGFL6序列保守结构域,结果见图4。
EGFL6基因氨基酸序列有5个保守结构域,属于EGF_CA和MAM超家族两个超家族,5个结构域依次是EGF_C、cE、EGF_CA、EGF_CA、MAM。EGF_CA超家族具有钙离子结合功能,主要存在于膜结合蛋白和动物胞外蛋白上;MAM超家族是胞外结构域,主要介导蛋白间相互关系,具有细胞粘附功能。EGFL6基因与两个家族具有共同结构域,说明其具有类似功能。预测5个保守结构域相应具体位点如表3所示。
2.7EGFL6跨膜结构域预测
使用TMHMM Server version 2.0预测EGFL6蛋白跨膜结构,结果见图5。
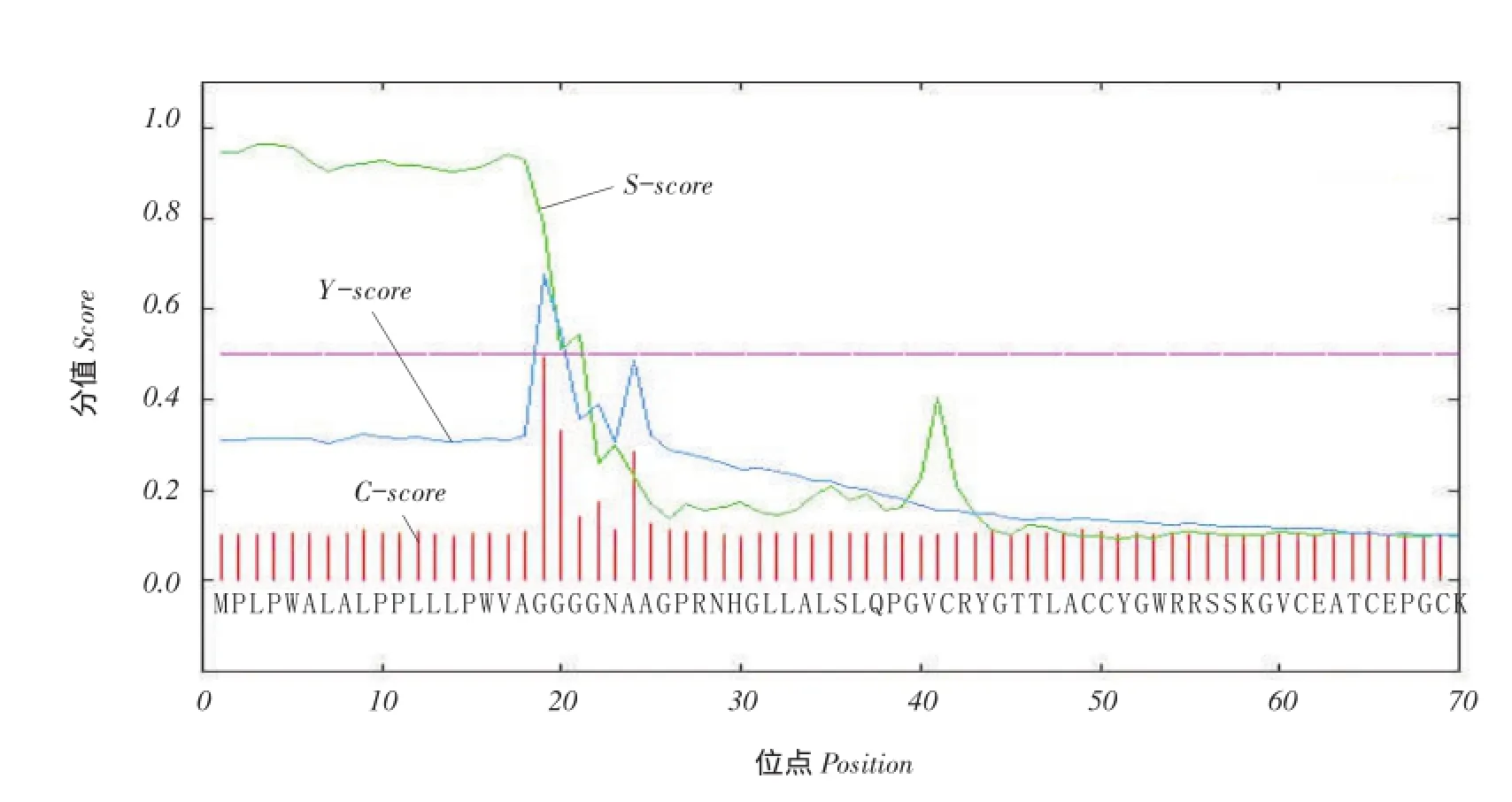
图3 EGFL6信号肽预测Fig.3 Signal peptide prediction of EGFL6
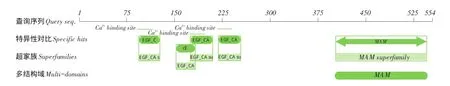
图4 EGFL6保守结构域预测Fig.4 Conserved domain of EGFL6

图5 EGFL6跨膜结构域预测Fig.5 Transmembrance prediction of EGFL6
由图5可知,该氨基酸无跨膜螺旋(TMHs),即无跨膜区;非跨膜螺旋区(ExpAA)预测值为0.74291,该值>18时说明预测蛋白为跨膜蛋白,蛋白N端位于膜内一侧可能性是0.03527,总体该序列位于膜外,EGFL6基因所编码蛋白为非跨膜蛋白。
2.8EGFL6亚细胞定位预测
运用PSORTⅡ工具预测EGFL6亚细胞定位,根据k-NN算法预测结果见表4。EGFL6位于细胞核概率最大,占整体的52.2%,其次是线粒体,预测概率为26.1%,位于细胞外基质(包括细胞壁)和细胞质概率分别是13.0%和8.7%。
2.9EGFL6亲水性/疏水性
利用ExPASY ProtScale在线软件预测EGFL6氨基酸序列亲水性/疏水性,分值代表其疏水性大小。负值越大(绝对值)表示亲水性越强,正值越大表示疏水性越强。通过分析发现,整条链在第537处甘氨酸,具有最大疏水值(2.422),在第344处谷氨酸,具有最小疏水值(-3.667)。EGFL6蛋白N末端疏水,C末端亲水,整条链中亲水性氨基酸残基多于疏水性氨基酸残基。因此,可推测EGFL6是可溶性蛋白(见图6)。

表4 EGFL6亚细胞定位Table 4 Subcellular location of EGFL6

图6 EGFL6疏水性分析Fig.6 Hydrophilia/hydrophobicity analysis of EGFL6
2.10EGFL6激酶磷酸化修饰位点分析
蛋白质磷酸化是基因序列翻译后修饰一种重要机制,对代谢、细胞信号传导、分化、跨膜转运等细胞进程具有重要影响。磷酸化修饰与多种因素有关,通常发生在丝氨酸(S)、苏氨酸(T)、或酪氨酸(Y)3个残基上,被各种蛋白激酶催化。KinasePhos网站预测结果显示,EGFL6有7个丝氨酸激酶(氨基酸位点:57,92,180,323,335,486,497)、4个苏氨酸激酶(290,306,388, 389)和1个酪氨酸激酶(145)潜在磷酸化位点。其中3个磷酸化位点较为活跃,结果见表5。
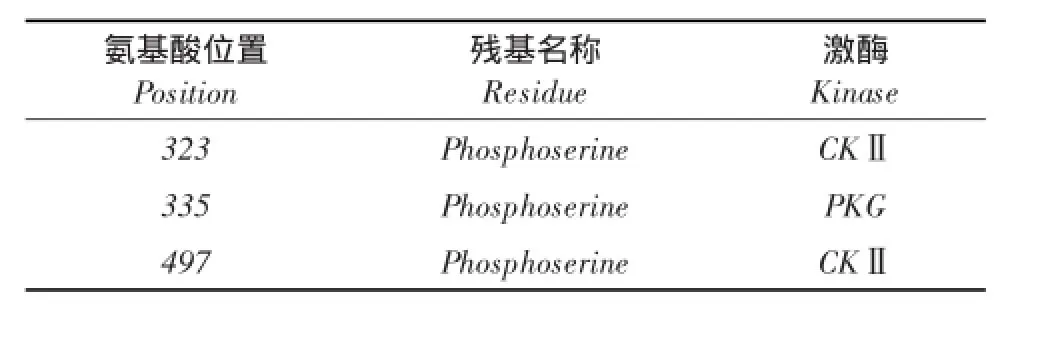
表5 EGFL6活跃磷酸化位点Table 5 Active phosphorylation sites of EGFL6
2.11EGFL6二级结构预测
通过SOPMA程序预测EGFL6蛋白二级结构,发现由259个氨基酸残基组成的无规则卷曲,占整个二级结构的46.75%,是EGFL6二级结构主要构成;β-转角由65个氨基酸构成,占整体11.73%;α-螺旋、延伸链分别由102、128个氨基酸残基构成,在整体中所占比例分别为18.41%和23.10%。如图7所示。
2.12EGFL6三级结构预测
EGFL6蛋白质三维结构预测结果见图8。将猪三维结构和小鼠、大鼠、猫、人空间结构比较发现,猫和人、小鼠及大鼠三维结构差异较大。
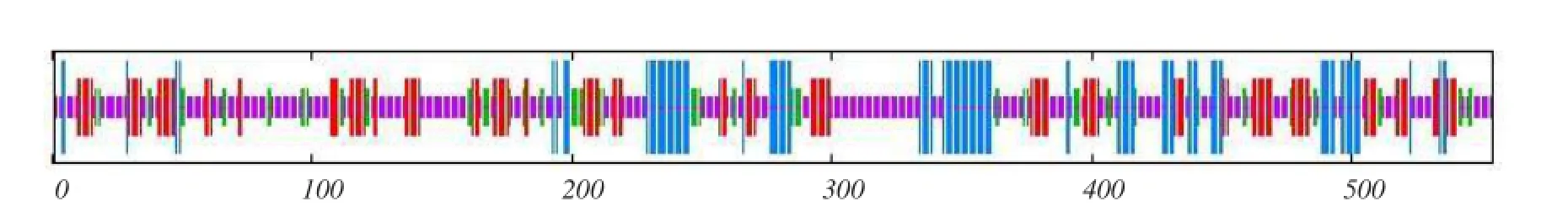
图7 EGFL6蛋白二级结构Fig.7 Secondary structure of EGFL6
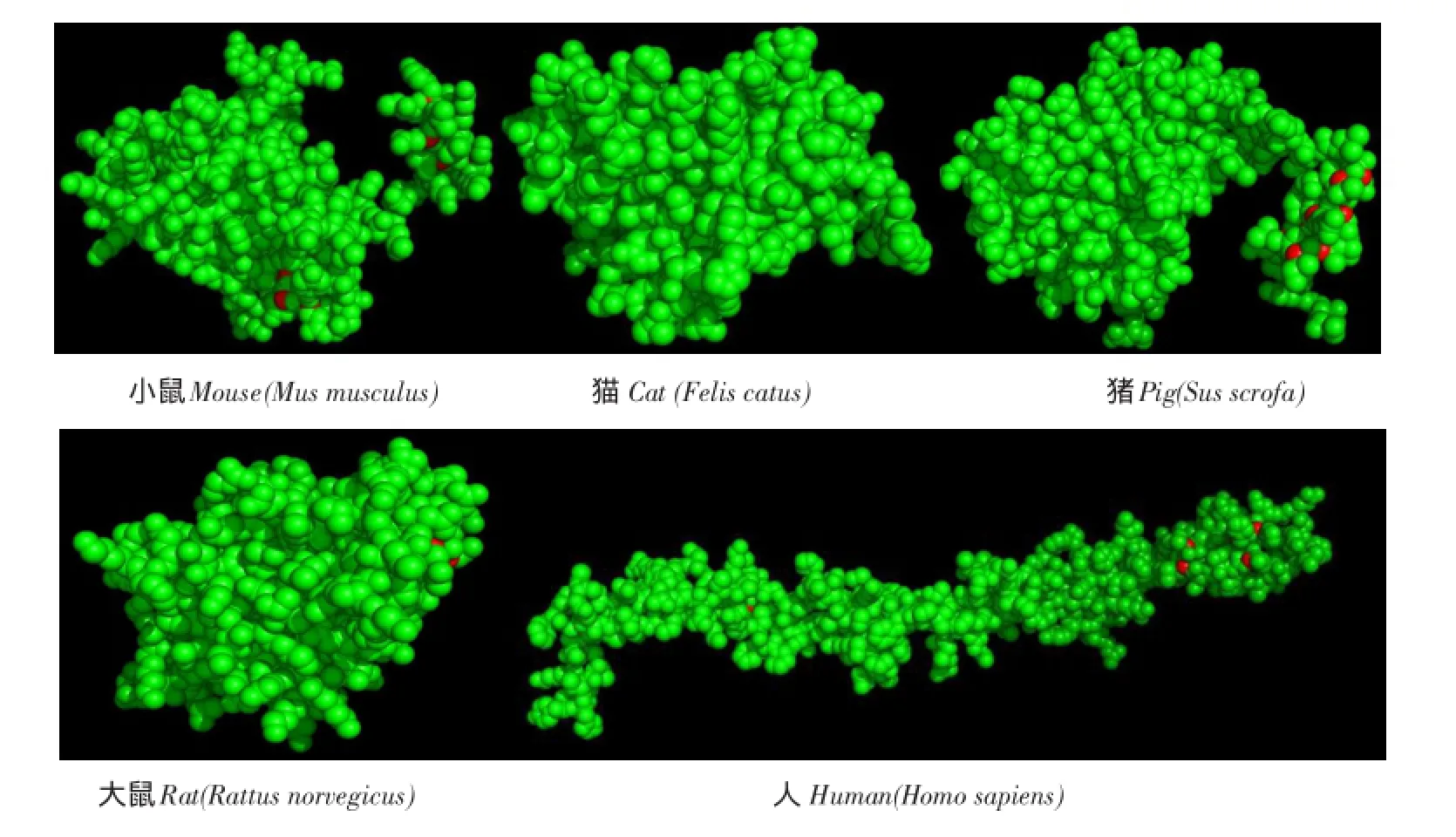
图8 三级分子结构Fig.8 3Dmolecular structures
3 讨论
3.1猪EGFL6基因基本生物信息学分析
猪EGFL6基因位于X染色体上,12个外显子,基因序列全长165 641 bp,编码区长1 665 bp,ORF预测最长编码554个氨基酸;相对分子质量为61 128.9,结构不稳定(不稳定系数分值为44.02),带正电荷,偏碱性,为亲水性蛋白。氨基酸同源性分析表明,猪与马、人、小鼠、牛氨基酸序列相似性较高,分别为83%、79%、76%和76%,与鸡、鸭同源性仅59%和60%,与运用MEGA6.0软件进化树分析结果一致,猪EGFL6基因和马亲缘关系更近,与鸡、鸭亲缘关系较远。
3.2猪EGFL6基因功能预测
运用NCBI的CDD对保守结构域预测发现该基因有5个保守结构域,分别属于MAM和EGF_CA两个超家族。KinasePhos预测12个潜在磷酸化位点,其中3个较活跃磷酸化位点分别位于第323、325和497个氨基酸,第323和497个丝氨基酸磷酸化位点相对应激酶为CⅡ,第325个丝氨酸磷酸化位点对应激酶为PKG,说明EGFL6蛋白活性被改变可能性变大。蛋白功能预测和亚细胞定位主要运用ProtFun和PSORT在线软件,结果表明EGFL6蛋白主要功能是细胞膜组成、氨基酸生物合成,位于细胞核可能性为52.2%[12-13]。Signal P信号肽预测和TMHMM跨膜结构预测结果显示,EGFL6为非跨膜分泌蛋白,与周宇等研究方法[14]一致。
3.3猪EGFL6基因结构预测
SOPMA预测EGFL6蛋白二级结构中无规则卷曲是主要构件,占整个结构46.75%。无规则卷曲(Coil),又称为环区(Loop),柔性大,经常出现在活跃点和对接点,可用于分子识别,在蛋白质特征和功能中起关键作用[15]。对于EGFL6蛋白质三维结构,SWISS-MODEL网络建模服务器使用广泛[16-17]。一般同源性超过30%蛋白质序列均可建立精确结构模型,序列同源性越高则建立模型结构准确性越高[18-20];比较三级结构可知,猪与人EGFL6基因空间构型差别较大,说明尽管氨基酸序列极其相似,但蛋白质翻译加工过程复杂。
4 结论
通过猪EGFL6基因基本生物信息学分析,预测编码氨基酸序列功能,预测软件经大量模型和算法验证,可信性高,可为研究猪EGFL6基因功能提供参考。
[1]Yeung G,Mulero J J,Berntsen R P,et al.Cloning of a novel epi⁃dermal growth factor repeat containing gene EGFL6:Expressed in tumor and fetal tissues[J].Genomics,1999,62(2):304-307.
[2]Buchner G,Orfanelli U,Quaderi N,et al.Identification of a new EGF-repeat-containing gene from human Xp22:A candidate for developmental disorders[J].Genomics,2000,65(1):16-23.
[3]Wang X,Gong Y,Wang D,et al.Analysis of gene expression pro⁃filing inmeningioma:Deregulated signaling pathways associated withmeningioma and EGFL6 overexpression in benignmeningio⁃matissue and serum[J].PloS One,2012,7(12):483-496.
[4]Buckanovich R J,Sasaroli D,Anne O J,et al.Tumor vascular pro⁃teins as biomarkers in ovarian cancer[J].J Clin Oncol,2007,25 (7):852—861.
[5]亢中奎,张晋霞,姜浩武,等.重组人EGFL6在HEK293细胞中的瞬时表达[J].生物技术通讯,2013(9):170-176.
[6]吕成定,吴斌华,翁锐强,等.EGFL6基因沉默对人黑素瘤细胞增殖及侵袭的影响[J].肿瘤,2015,35(4):370-376.
[7]Buchner G,Broccoli V,Bulfone A,et al.Maeg,an EGF-repeat containing gene,is a newmarker associated with dermatome spec⁃ification andmorphogenesis of its derivatives[J].Mech Dev,2000. 98(1-2):179-182.
[8]Oberauer R,Rist W,Lenterm C,et al.EGFL6 is increasingly ex⁃pressed in human obesity and promotes proliferation of adipose tissue—derived stromal vascular cells[J].Mol Cell Biochem, 2010,343(1-2):257-269.
[9]Chim Sm,Qin A,Tickner J,et al.EGFL6 promotes endothelial cellmigration and angiogenesis through the activation of extracel⁃lular signal-regulated kinase[J].J Biol Chem,2011,286(25): 22035-22046.
[10]杨晶,王金胜,张辉挺,等.人源性EGFL6的重组表达及多克隆抗体制备研究[J].免疫学杂志,2013,29(6):517-526.
[11]陈珊珊,郭晋隆,李国印,等.甘蔗过氧化氢酶基因的电子克隆及生物信息学分析[J].生物信息学,2012,10(1):65-70.
[12]李丽莎,李祥龙,毛艳朋,等.山羊TRPM1基因CDS区生物信息学分析[J].河南农业科学,2015,44(12):121-125.
[13]潘蕾,刘燕,韦强,等.家兔MYLl2B基因的克隆及生物信息学分析[J].浙江农业学报,2015,27(6):927-932.
[14]周宇,谢辉,孟庆峰,等.猪博卡病毒GD6株的全基因序列及生物信息学分析[J].畜牧与兽医,2016,48(2):85-89.
[15]刘洪超,胡澍,涂心明.果蝇Tap蛋白结构与功能的生物信息学分析[J].重庆医学,2015,44(17):2311-2314.
[16]Biasinim,Bienert S,Waterhouse A,et al.SWISS-MODEL:mod⁃eling protein tertiary and quanternary structure using evolutionary information[J].Nucleic Acids Research,2014,12:252-258.
[17]谌容,陈敏,杨春贤,等.基于SWISS-MODEL的蛋白质三维结构建模[J].生命的化学,2006,26(1):54-56.
[18]曾炳佳,曹以诚,杜正平,等.同源建模关键步骤的研究动态[J].生物学杂志,2008,25(2):7-10.
[19]汤海旭,丁达夫.用于蛋白质同源模建及三维结构预测的结构比较方法[J].生物物理学报,1995,11(1):60-66.
[20]Fiser A,Dork G,Sali A.Modeling of loops in protein structures[J]. Protein Science,2000,9(9):1753-1773.
Bioinformatics analysis ofEGFL6 gene in pig
SUN Caixia,LIming,QIAO Ruimin, HAN Xuelei,LI Xinjian(School of Animal Science and Veterinarymedicine,Henan Agricultural University,Zhengzhou 450002,China)
To explore the specific character ofEFGL6 gene,CDS region was cloned and some characters ofEGFL6 gene were analyzed by bioinformaticsmethod,including the physical and chemical properties,the homology of encoding amino acid,protein function,secondary and tertiary structure and so on.The results showed that the CDS region ofEGFL6 gene was 1 665 bp,and encoded 554 amino acids. The similarity of amino acid sequence was over 75%with human,horse,rat,sheep.EGFL6 was hydrophilic and unstable.It was secretory protein and it had 12 potential phosphorylation sites and five conserved domains,but had no transmembrane domain,and coil was the primarily component of the corresponding secondary structure and tertiary structure.These results would provide theoretical foundations for the future study.
EGFL6;bioinformatics;pig
S858.28;Q78
A
1005-9369(2016)04-0016-08
2016-02-03
河南省生猪产业技术体系创新团队项目(S2012-06);河南省科技攻关项目(142102110047);国家自然科学基金项目(U150 4322)
孙彩霞(1991-),女,硕士研究生,研究方向为动物遗传育种。E-mail:836353527@qq.com
李新建,副教授,研究方向为动物遗传育种与繁殖。E-mail:lxjlongfei@163.com
