酿酒酵母基因组位置效应对外源基因表达的影响
2016-09-19张文政唐继军李炳志元英进
张文政,唐继军,李炳志,元英进
酿酒酵母基因组位置效应对外源基因表达的影响
张文政1,2,唐继军2,3,李炳志1,2,元英进1,2
1 天津大学 系统生物学教育部重点实验室,天津 300072 2 天津大学 化工学院 天津化学化工协同创新中心,天津 300072 3 天津大学 计算机与科学技术学院,天津 300072
外源基因元件和模块在底盘细胞中发挥特定功能是合成生物学研究的基本过程,而外源元件和模块在基因组中的位置对其功能的实现具有显著影响。为了系统、全面地表征酿酒酵母基因组位置效应对外源基因的表达影响,以绿色荧光蛋白为报告基因,通过双交换同源重组方法,对酿酒酵母单基因敲除库进行高通量转化,构建酿酒酵母基因组单位点荧光标记菌株库。结合流式细胞术和高通量测序技术对单位点荧光标记库菌株进行分析,构建高表达位点库和低表达位点库,共发现促进绿色荧光蛋白表达的位点428个,抑制绿色荧光蛋白表达的位点444个。通过分析高、低表达位点在酵母染色体上的分布,从全基因组尺度上对酿酒酵母基因组整合位置对基因表达的影响进行表征。本研究可为酿酒酵母基因组位置效应的分布规律和产生机理研究提供重要参考,对外源蛋白工业生产和合成生物学中的基因表达精细调控也具有重要的指导意义。
合成生物学,酿酒酵母,染色体位置效应,外源基因表达
酿酒酵母具有蛋白翻译后修饰功能,适合复杂结构蛋白的表达和功能发挥,被广泛应用于复杂化合物的生产[1-3]。合成生物学领域的最新研究进展表明,外源基因元件和模块能够在酿酒酵母细胞中发挥功能,实现复杂天然化合物的生 产[4-6]。外源基因元件或模块通常以游离型质粒为载体存在于酿酒酵母细胞中,或插入酿酒酵母基因组。与游离型质粒相比,将外源基因元件或模块整合至酵母基因组具有可精确控制拷贝数、稳定强、无需抗生素或其他化合物的添加以维持筛选压力等优点。然而研究表明,酿酒酵母基因组对外源基因的表达存在明显的位置效应[7],与插入位点相关的表观遗传效应能够影响外源基因的表达,插入本身可能会激活或者抑制某些基因,造成内源基因的异常表达和有毒代谢产物的积累。因此酿酒酵母基因组位置效应对外源基因表达的影响的研究具有重要意义。
目前,研究人员已在多种生物体系中发现基因组位置效应的存在,如大肠杆菌[8]、酿酒酵 母[9-10]、乳酸乳球菌[11]、果蝇[12]和人类细胞[13]等。Yamane等[9]以为报告基因,将其整合至酵母Ⅲ号染色体的不同位点,通过测定β-半乳糖苷酶的活性表征的转录水平,对酵母Ⅲ号染色体的位置效应进行了研究;Bai等[10]采用类似的方法,在酿酒酵母基因组上选取了20个可能适合外源基因整合的位点,对插入这些位点的基因的表达情况进行了测定,发现不同位点间的表达水平存在明显差异,最高可达20倍;Thomoson等[14]的研究则采用了一种随机插入的策略,并未对报告基因的插入位置进行确定。上述研究仅对酿酒酵母的位置效应对外源基因表达的影响进行了初步探索,研究局限于有限位点或单条染色体,缺乏对整个酿酒酵母基因组的位置效应系统、全面的探究。
酿酒酵母单基因敲除库 (Yeast konck-out collection,简写为YKO)[15]含有菌株4 817株,每个菌株均有1个非必需基因的开放阅读框 (ORF) 被表达盒替代,具有G418抗性。表达盒上下游各含有一个长度为20 bp的“条码区”识别区,用于特异性识别被替换的非必需基因。本研究通过同源重组将表达盒替换为报告基因,结合流式细胞分选技术和高通量测序技术,构建酿酒酵母单位点荧光标记库、高表达位点库和低表达位点库,实现对酿酒酵母基因组多位点上外源基因表达水平差异的表征。本研究方法具有高通量、能够精准定位插入位置的特点,不仅能够筛选出增强和抑制外源基因表达的位点,而且能够从基因组尺度上揭示酵母位置效应的分布规律和产生机理。
1 材料与方法
1.1 菌株与质粒
实验背景菌株均为S288c 菌株 BY4741 ()[16]。以BY4741为背景菌株的酵母单基因敲除库由美国Jef D. Boeke教授实验室保存馈赠。大肠杆菌质粒pUC19[17],酵母复制型质粒pRS416[18],均为本实验室保存。带有基因的质粒pGRB2.3由美国Brendan Cormack教授实验室保存馈赠。用于质粒扩增的菌株为大肠杆菌DH5α,购自北京博迈德生物技术有限公司。
1.2 培养基与试剂
YPD培养基组成成分为10 g/L酵母浸粉,20 g/L蛋白胨,20 g/L葡萄糖。SD培养基组成成分为6.7 g/L不含氨基酸的酵母氮源,2 g/L氨基酸混合物,20 g/L葡萄糖,20 mg/L组氨酸,20 mg/L色氨酸,SD-URA缺陷型培养基添加100 mg/L亮氨酸,SD-LEU缺陷型培养基添加20 mg/L尿嘧啶,用10 mol/L NaOH调节pH至6.5。LB培养基组成成分为10 g/L 氯化钠,5 g/L酵母浸粉, 10 g/L蛋白胨,添加0.1 mg/mL氨苄霉素用于筛选。固体培养基添加20 g/L琼脂粉。培养含有基因的菌株细胞时,培养基中额外添加0.2 mg/mL G418。
培养基所用试剂购自北京鼎国生物技术有限公司;质粒提取试剂盒、DNA纯化试剂盒购自北京天根生化科技有限公司;™ 高保真DNA聚合酶购自美国 Stratagene公司,限制性内切酶、T4 DNA连接酶购自美国Fermentas公司;转化所用PEG3350、DMSO购自美国Sigma公司,鲑鱼精DNA购自北京鼎国生物技术有限公司。
1.3 表达盒构建
突变pUC19质粒去除I酶切位点,作为基因表达载体。采用OE-PCR法[19]构建整合型表达盒,总长3.9 kb,由5个DNA片段组成:与基因同源的上游同源臂和下游同源臂、酿酒酵母内源弱启动子、开放阅读框与其相应的终止子和营养筛选标记,PCR引物见表1。最终得到带有表达盒的整合型质粒pUC19M-1 (图1)。
表1 构建整合型表达质粒引物列表
Table 1 Primer for constructing the designer cassette for integrative transformation
Restriction enzyme sites are indicated in bold.
1.4 酿酒酵母单位点荧光标记库的构建
利用大肠杆菌对整合型质粒pUC19M-1进行扩增,酶切回收目的DNA片段20 μg,对酿酒酵母单基因敲除库进行转化。筛选出具有LEU+G418-表型的转化子,构建单位点荧光标记菌株库。将该菌株库在液体SD-LEU营养缺陷型培养基中进行培养以抑制假阳性细胞的生长。取部分培养后的细胞稀释涂布于YPD固体平板并翻印至SD-LEU营养缺陷型平板以统计酿酒酵母单位点荧光标记库的细胞阴性率。
1.5 酿酒酵母单位点荧光标记库的分选
将酿酒酵母单位点荧光标记库过夜培养,去除培养基后悬于PBS缓冲液中,细胞密度调整至1×107个细胞/mL。用流式细胞仪对细胞进行分选,根据细胞荧光蛋白表达强度的分布图谱 (图2),选取荧光蛋白表达量较高和较低的细胞,分选出的细胞数分别占细胞总量10%。将得到的细胞再次分选,去除干扰细胞,纯化细胞库,形成高表达位点库和低表达位点库。
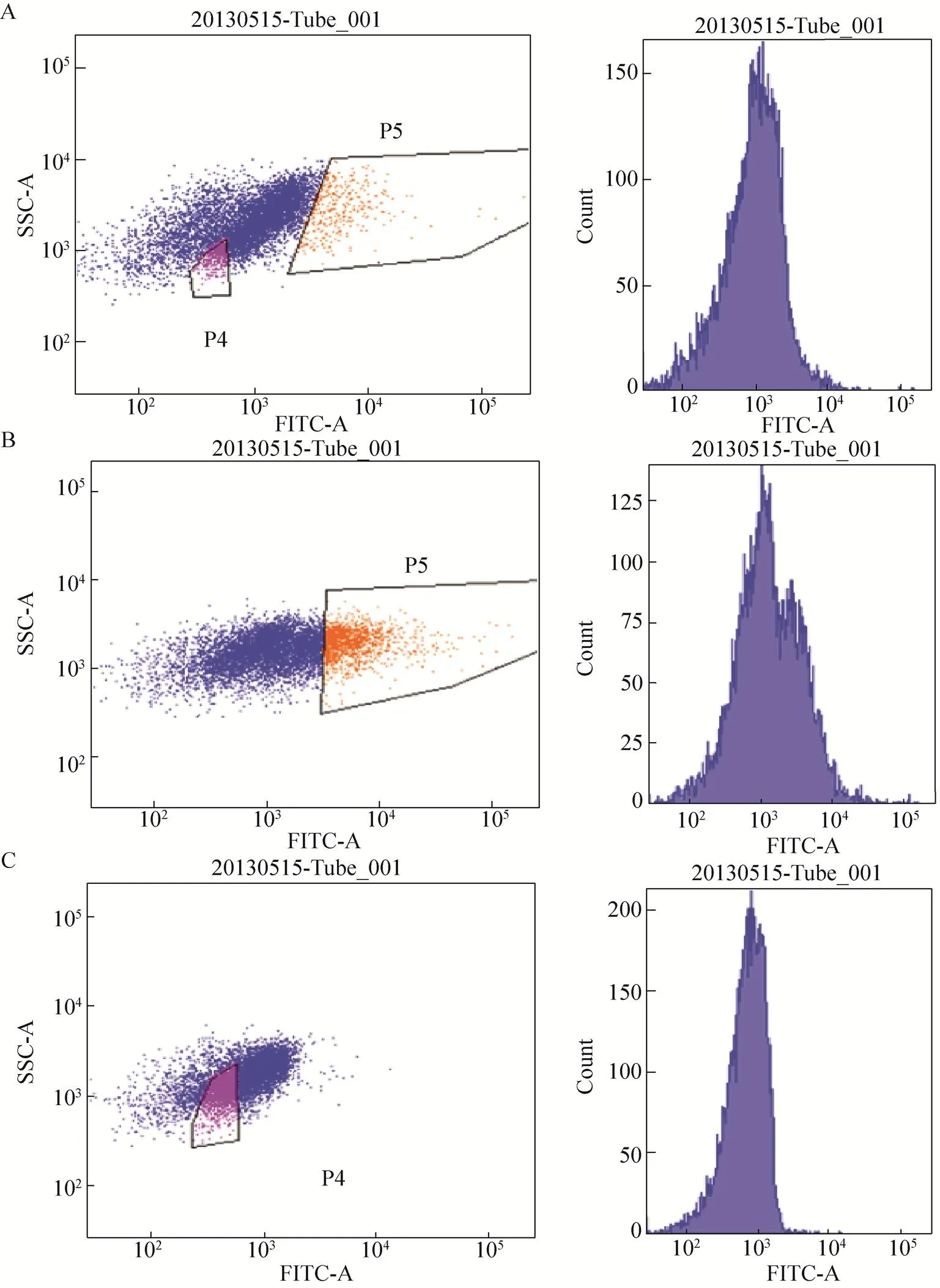
图2 酿酒酵母单位点荧光标记库菌株分选 (A:以酿酒酵母单位点荧光标记库为起始的第一轮分选,P4框中为gfp高表达细胞,P5 框中的gfp低表达细胞;B:以第一轮分选出的gfp高表达细胞为起始进行的第二轮分选;C:以第一轮分选出的gfp低表达细胞为起始进行的第二轮分选)
1.6 高/低表达位点库的测定与分析
提取酿酒酵母单基因敲除库、酿酒酵母单位点荧光标记库、高表达位点库和低表达位点库的基因组DNA,采用Tag-array hybridization法[20]测定表达盒上下游“条码区”的DNA序列,确定库中所含菌株和基因插入位置。
2 结果与讨论
为了系统研究酿酒酵母基因组位置效应对外源基因表达的影响,本实验中对酿酒酵母单基因敲除库进行了高通量转化,以报告基因替换单基因敲除菌株的序列。通过对转化子的表型筛选,得到1.3×104个具有LEU+G418-表型的转化子,构建酿酒酵母基因组单位点荧光标记菌株库。酿酒酵母单基因敲除库中共有菌株4 817株,根据计算,单位点荧光标记库对单基因敲除库的覆盖率为2.6倍,单个单基因敲除菌株被转化的概率为92.8%。单基因敲除菌株库中存在部分生长缓慢菌株,在培养、转化过程中可能遗失;某些基因的缺失会对整合型转化产生影响[21];此外,长片段外源DNA的整合型转化效率远低于游离型质粒转化。考虑上述因素,我们认为实验中所构建的酿酒酵母单点荧光标记库基本实现了对原单基因敲除库的替换。
与抗性筛选不同,营养缺陷型筛选过程中会产生假阳性转化子,为避免这些转化子影响后续的分选和测序,实验中对酵母单位点荧光标记库、高表达位点库和低表达位点库中菌株的表型进行了验证。在统计的4 200个单位点荧光标记库菌株菌落中,表型为LEU-的菌落84个,阴性率为2% (表2)。采用同样的方法对高、低表达位点库的细胞阴性率进行检测,结果显示高表达位点库的阴性率为0%,低表达位点的阴性率为1.1% (表2)。由于经过营养缺陷型培养基中的传代培养和流式细胞仪的分选,两者的阴性率与单位点荧光标记库菌株阴性率相比均有明显降低。但是低位点表达库仍存在1.1%的假阳性细胞,这是由于流式细胞仪的分选受精度所限,未能将部分无荧光强度的细胞与荧光强度较低的细胞区分开来。由于后续的测序实验能够排除假阳性菌株,因此当库中细胞阴性率低于2%时,不会对后续实验结果造成显著影响。
表2 酿酒酵母单位点荧光标记库、高表达位点库和低表达位点库中菌株表型验证
Table 2 Phenotype verification for strains in the starting pool (SP), high-expressed pool (HP) and low-expressed pool (LP)
利用高通量测序技术测定表达盒上下游“条码区”的DNA序列,确定酿酒酵母单位点荧光标记库中所含菌株和基因插入位置。数据分析显示,酿酒酵母单位点荧光标记库中共含菌株3 455个,占原酵母单基因敲除库的72%。分析测定结果少于酵母单基因敲除库菌株数的原因如下:1) 在原始的4 817个菌株中,有部分菌株具有生长缓慢表型。这些菌株无论是在最开始的转化操作,还是后续的培养、分选和测序过程中均可能因为生长过于缓慢丢失。2)基因的缺失和插入位置的不同会对转化效率造成影响[21],使得部分菌株未能成功插入报告基因。3) 分析和测序过程中会造成一部分菌株的缺失。
由酵母单位点荧光标记库的流式细胞分选图 (图2) 可以看出,部分细胞的表达量明显高于或低于其他细胞,说明处于这些位点上的基因表达受位置影响较大。
实验中将位置效应明显的细胞分选出来,构建高表达位点库和低表达位点库。测序分析结果显示,高表达位点库中含有菌株428个,占单位点标记库的12%;低表达位点库中含有菌株444个,占单位点标记库的13% (菌株列表见补充材料)。在荧光显微镜下观察酵母单位点荧光标记库、高表达位点库菌株细胞的表达情况 (图3),图3B为酵母单位点荧光标记库细胞的观察结果,可以看出细胞之间荧光强度存在明显差异;图3C为高表达库菌株的细胞,其荧光强度明显强于低表达库菌株的细胞 (图3D)。对酵母单位点荧光标记库、高表达位点库和低位点表达库荧光表达强度的定量测定与镜检结果相吻合 (图4)。图4B为未分选的酵母单位点荧光标记库细胞的荧光蛋白表达量分布图,沿横坐标轴分布较为分散,说明库中细胞荧光蛋白表达量差异较大;图4C为高表达位点库细胞的荧光蛋白表达量分布,与图4B相比明显沿坐标轴向右偏移,说明细胞中荧光蛋白的表达量明显增强;而从代表低表达位点库的图4D中可以看出,细胞分布沿坐标轴右移,说明细胞中荧光蛋白的表达量明显降低。通过分布图的量化数据可知,低表达位点库菌株的平均表达量为18.02,高表达位点库菌株的平均表达量为137.84,为低表达位点库平均表达量的7.6倍。
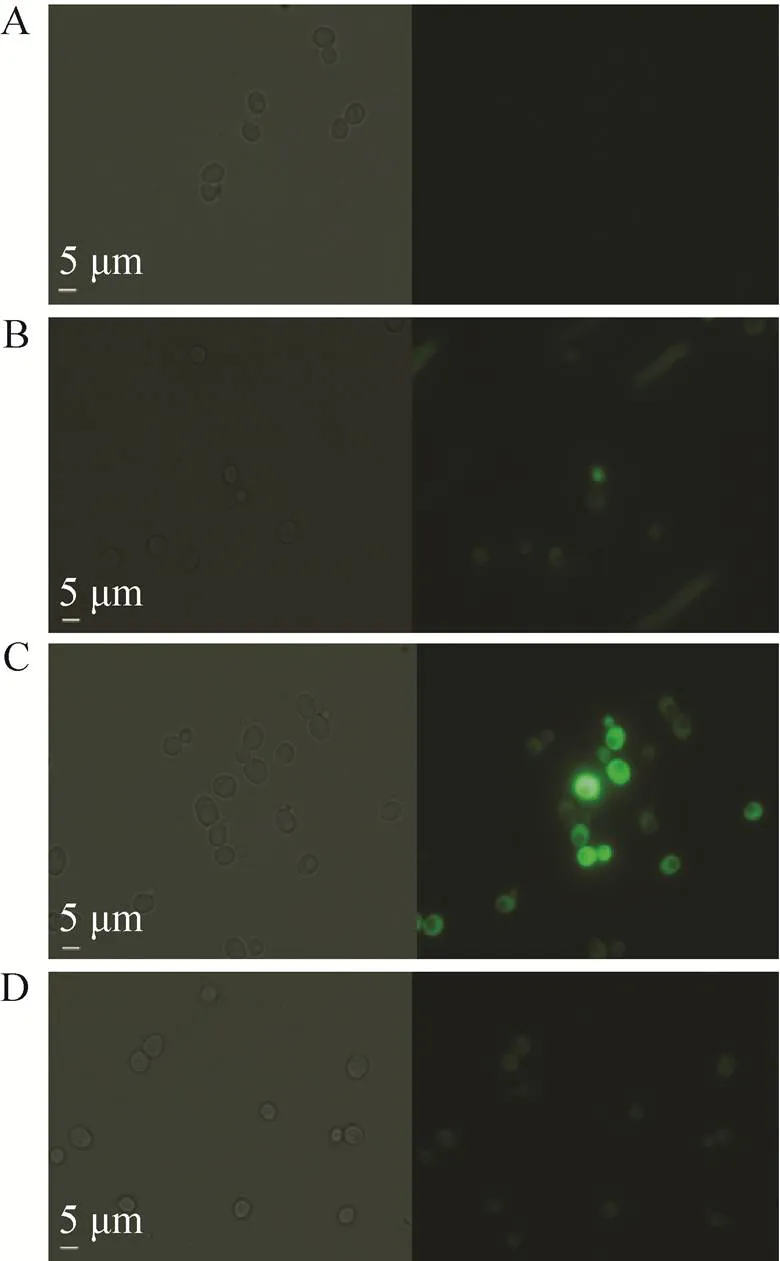
图3 荧光显微镜下酿酒酵母单基因敲除库、高表达位点库和低表达位点库的绿色荧光蛋白表达情况(A:未转化的酿酒酵母单基因敲除库细胞;B:未经过分选的酿酒酵母单位点荧光标记库细胞;C:高表达位点库细胞;D:低表达位点库细胞。左侧图片为白光观测,曝光时间100 ms;右侧图片为荧光观测,曝光时间2 000 ms)
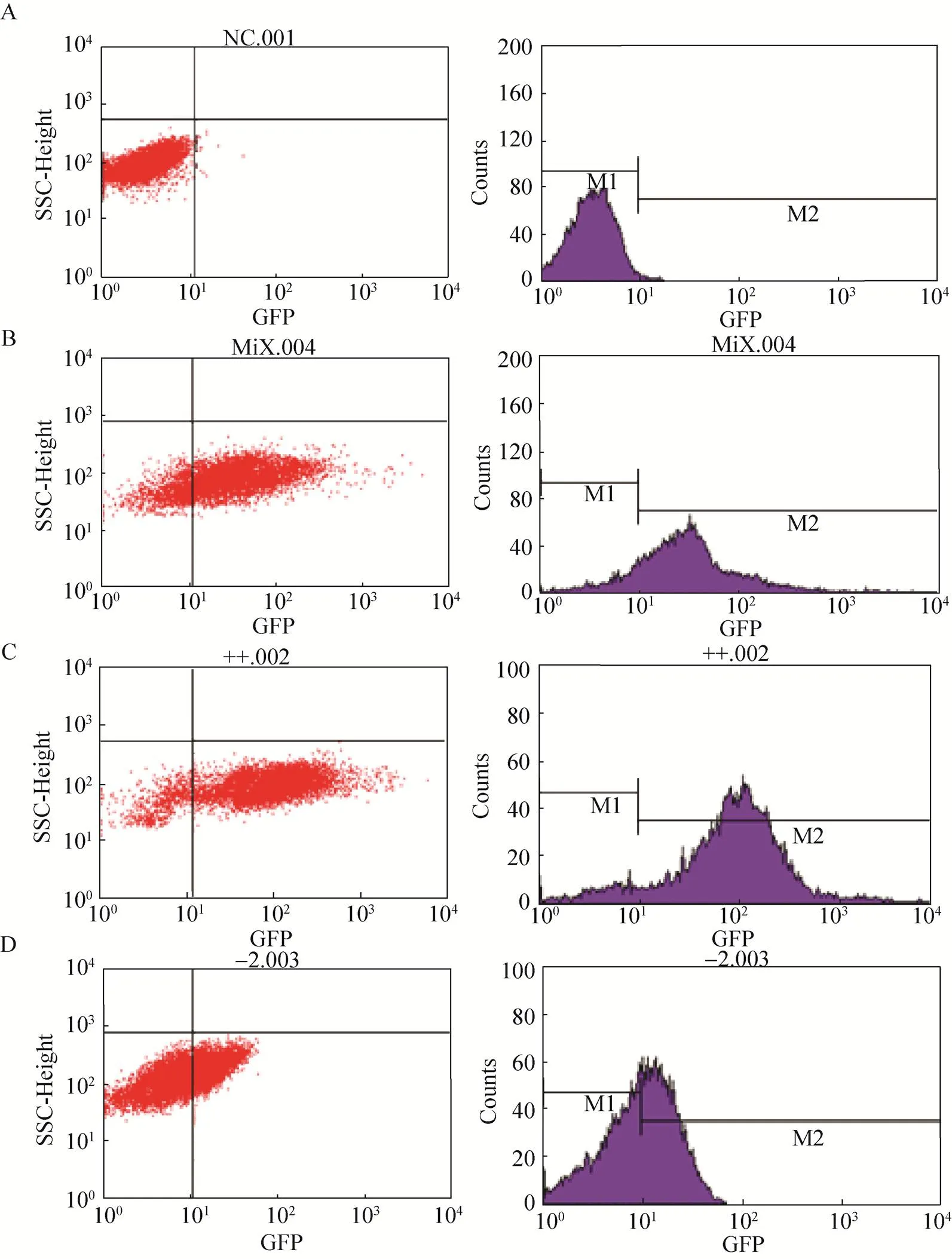
图4 gfp表达量测定图 (A:未转化的酿酒酵母单基因敲除库;B:未经过分选的酿酒酵母单位点荧光标记库;C:经过两轮分选得到的高表达位点库;D:经过两轮分选得到的低表达位点库)
为了进一步探寻酿酒酵母位置效应的分布规律,将高表达位点库和低表达位点库中报告基因所在位点绘制成图谱 (图5)。高/低表达位点分布图显示,对于外源基因表达存在显著影响的位点在酵母基因组的16条染色体上均有分布。一些高表达和低表达位点集中分布在染色体的特定位置,呈现出一定的区域性。存在下调基因表达的“抑制区”和上调外源基因表达的“热点区”。一个典型的例子就是端粒位置效应,表现为处于端粒序列附近的基因的表达受到抑制[22]。从图5中可以看出,低表达位点在端粒位置的分布较为集中,如Ⅶ号、ⅪⅤ号染色体,端粒沉默效应抑制了其附近位点基因的表达,这一现象与染色质的组织结构有关[23-24]。此外,低表达位点还多集中于着丝粒位置和位点,产生这一现象的原因为着丝粒和位点具有特殊的DNA序列,容易与蛋白质结合形成稳定的聚合体,不利于外源蛋白的表达[25-26]。除上述位点之外,染色体上还存在一些外源基因表达抑制区,如Ⅱ号染色体的100-119 kb位置,Ⅷ号染色体的136-159位置,ⅩⅥ号染色体的226-204 kb位置等,其抑制机理有待进一步研究。此外,在染色体上存在一些高表达位点相对较为集中的热点区域,Yamane等[8]和Flagfeldt等[10]在其针对酿酒酵母基因组位置效应的研究中发现热点区域附近存在自主复制序列 (ARS),促进外源基因的表达,本实验证实了这一现象并发现了更多类似热点区,如Ⅳ号染色体755-768 kb (ARS423),Ⅶ号染色体828-849 kb (ARS731),ⅩⅥ号染色体455-465 kb (ARS1633)等。研究过程中我们还发现一些热点区域能够造成外源基因表达量增加,如Ⅷ染色体320-336 kb,ⅩⅢ染色体571-589 kb等。但其作用机理尚不明确。
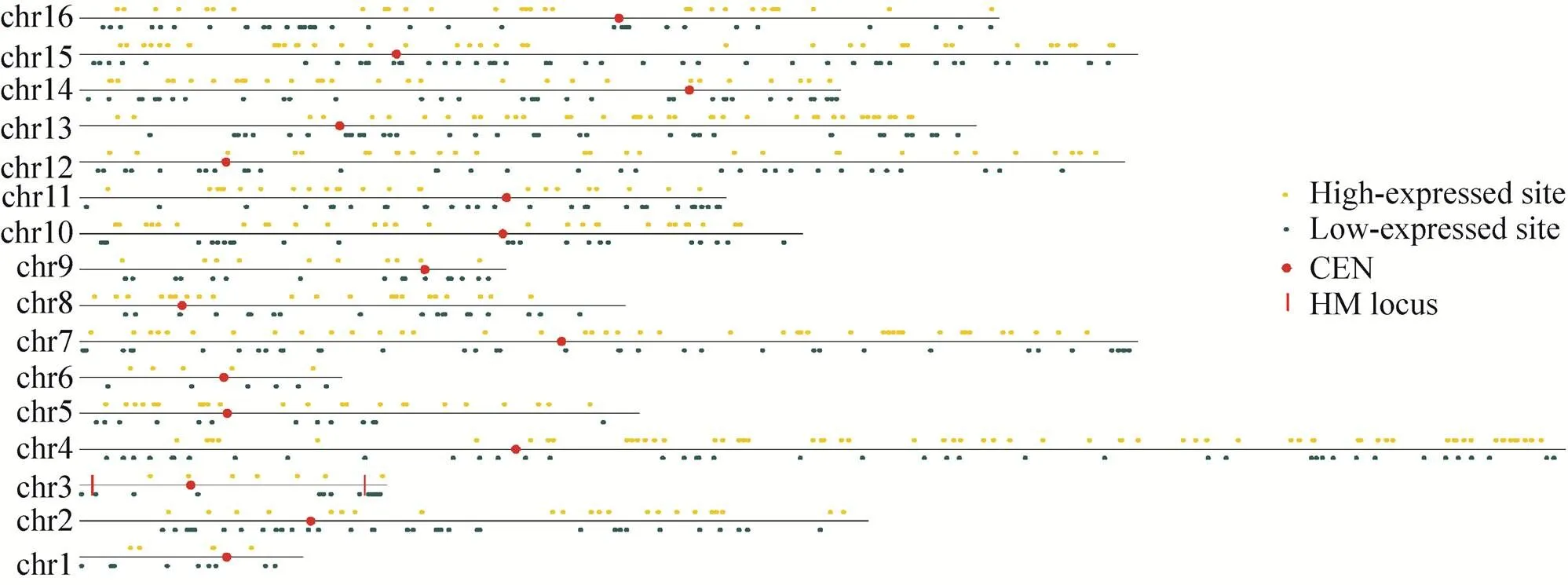
图5 高/低表达位点在酿酒酵母染色体上的分布示意图 (线上部黄色点代表高表达位点;线下部蓝色点代表低表达位点;线中红色点代表染色体着丝粒;Ⅲ号染色体上的红线代表交配型位点 (HML和HMR))
本研究以酿酒酵母单基因敲除库为基础,构建酿酒酵母基因组单位点荧光标记库、高表达位点库和低表达位点库,从全基因组尺度上对酿酒酵母基因组的位置效应进行了表征。获得高表达位点428个,低表达位点444个。通过对这些位点分布的分析揭示了位置效应在染色体上的分布规律。研究结果对合成生物学研究中精确调控外源基因表达和重要蛋白生产工业都具有重要的指导意义。
致谢:感谢美国纽约大学的Jef D. Boeke教授对本研究的悉心指导和支持;本文的测序工作和位点图谱绘制得到了Boeke课题组Anna Sliva和Zheng Kuang的协助,在此对他们表示感谢。
REFERENCES
[1] Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440(7086): 940–943.
[2] Luo YZ, Li BZ, Liu D, et al. Engineered biosynthesis of natural products in heterologous hosts. Chem Soc Rev, 2015, 44(15): 5265–5290.
[3] Froissard M, D'andréa S, Boulard C, et al. Heterologous expression of AtClo1, a plant oil body protein, induces lipid accumulation in yeast. FEMS Yeast Res, 2009, 9(3): 428–438.
[4] Engles B, Dahm P, Jennewein S. Metabolic engineering of taxadiene biosynthesis in yeast as a first step towards Taxol (Paclitaxel) production. Metab Eng, 2008, 10(3/4): 201–206.
[5] Fossati E, Ekins A, Narcross L, et al. Reconstitution of a 10-gene pathway for synthesis of the plant alkaloid dihydrosanguinarine inNat Commun, 2014, 5: 3283.
[6] Siddiqui MS, Thodey K, Trenchard I, et al. Advancing secondary metabolite biosynthesis in yeast with synthetic biology tools. FEMS Yeast Res, 2012, 12(2): 144–170.
[7] Velculescu VE, Zhang L, Zhou W, et al. Characterization of the yeast transcriptome. Cell, 1997, 88(2): 243–251.
[8] Sousa C, de Lorenzo V, Cebolla A. Modulation of gene expression through chromosomal positioning in. Microbiology, 1997, 143(6): 2071–2078.
[9] Yamane S, Yamaoka M, Yamamoto M, et al. Region specificity of chromosome Ⅲ on gene expression in the yeast. J Gen Appl Microbiol, 1998, 44(4): 275–281.
[10] Bai Flagfeldt D, Siewers V, Huang L, et al. Characterization of chromosomal integration sites for heterologous gene expression in. Yeast, 2009, 26(10): 545–551.
[11] Siezen RJ, Bayjanov JR, Felis GE, et al. Genome-scale diversity and niche adaptation analysis ofby comparative genome hybridization using multi-strain arrays. Microb Biotechnol, 2011, 4(3): 383–402.
[12] Markstein M, Pitsouli C, Villalta C, et al. Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nat Genet, 2008, 40(4): 476–483.
[13] Gierman HJ, Indemans MHG, Koster J, et al. Domain-wide regulation of gene expression in the human genome. Genome Res, 2007, 17(9): 1286–1295.
[14] Thompson A, Gasson MJ. Location effects of a reporter gene on expression levels and on native protein synthesis inand. Appl Environ Microbiol, 2001, 67(8): 3434–3439.
[15] Winzeler EA, Shoemaker DD, Astromoff A, et al. Functional characterization of thegenome by gene deletion and parallel analysis. Science, 1999, 285(5429): 901–906.
[16] Brachmann CB, Davies A, Cost GJ, et al. Designer deletion strains derived fromS288C: a useful set of strains and plasmids for PCR-mediated gene disruption and other applications. Yeast, 1998, 14(2): 115–132.
[17] Yanisch-Perron C, Vieira J, Messing J, et al. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. Gene, 1985, 33(1): 103–119.
[18] Sikorski RS, Hieter P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in. Genetics, 1989, 122(1): 19–27.
[19] Horton RM, Hunt HD, Ho SN, et al. Engineering hybrid genes without the use of restriction enzymes: gene splicing by overlap extension. Gene, 1989, 77(1): 61–68.
[20] Pan XW, Yuan DS, Ooi SL, et al. dSLAM analysis of genome-wide genetic interactions in. Methods, 2007, 41(2): 206–221.
[21] Aslankoohi E, Voordeckers K, Sun H, et al. Nucleosomes affect local transformation efficiency. Nucl Acids Res, 2012, 40(19): 9506–9512.
[22] Ottaviani A, Gilson E, Magdinier F. Telomeric position effect: from the yeast paradigm to human pathologies?. Biochimie, 2008, 90(1): 93–107.
[23] Grewal SIS, Moazed D. Heterochromatin and epigenetic control of gene expression. Science, 2003 301(5634): 798–802.
[24] Fox CA, McConnell KH. Toward biochemical understanding of a transcriptionally silenced chromosomal domain in. J Biol Chem, 2005, 280(10): 8629–8632.
[25] Smith JS, Boeke JD. An unusual form of transcriptional silencing in yeast ribosomal DNA. Genes Dev, 1997, 11(2): 241–254.
[26] Aparicio OM, Billington BL, Gottschling DE. Modifiers of position effect are shared between telomeric and silent mating-type loci in. Cell, 1991, 66(6): 1279–1287.
Effect of integration loci of genome on heterologous gene expression in
Wenzheng Zhang1,2, Jijun Tang2,3, Bingzhi Li1,2, and Yingjin Yuan1,2
1 Key Laboratory of Systems Bioengineering (Ministry of Education), Tianjin University, Tianjin 300072, China 2 SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering (Tianjin), School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China 3 School of Computer Science and Technology, Tianjin University, Tianjin 300072, China
Chromosomal integration of heterologous genes or pathways is preferred over the use of episomal plasmids for its inherently stability and thus more desirable in the industrial setting. However, the position of integration of heterologous genes in the genome influences the expression levels. In combination of high throughput transformation of the Yeast Knock-out Collection (YKO) and FACS analysis, the position effect on heterologous reporter genewas identified across the whole genome in yeast. In total 428 high-expressed sites and 444 low-expressed sites were spotted, providing massive data to analyze patterns and reasons for region dependency of gene expression on the genome-wide scale.
synthetic biology,, chromosomal region, heterologous gene expression
October 21, 2015; Accepted: December 4, 2015
工业生物技术
张文政, 唐继军, 李炳志, 等. 酿酒酵母基因组位置效应对外源基因表达的影响. 生物工程学报, 2016, 32(7): 901–911.
Zhang WZ, Tang JJ, Li BZ, et al. Effect of integration loci of genome on heterologous gene expression in. Chin J Biotech, 2016, 32(7): 901–911.
Supported by: International Science and Technology Cooperation Program of China (No. 2015DFA00960), National Natural Science Foundation of China (No. 21390203), Tianjin Municipal Science and Technology Program (No. 13RCGFSY19800).
Corresponding author: Bingzhi Li. Tel: +86-22-27402503; Fax: +86-22-27403888; E-mail: bzli@tju.edu.cn
国家国际科技合作项目 (No. 2015DFA00960),国家自然科学基金 (No. 21390203),天津市科技计划项目 (No. 13RCGFSY19800) 资助。
(本文责编 郝丽芳)
