小麦花发育MADS-box基因的表达模式分析
2016-09-18李海峰刘梦佳王冰华苏亚丽孙其信
李海峰 韩 英 刘梦佳 王冰华 苏亚丽 孙其信,*
1西北农林科技大学农学院 / 旱区作物逆境生物学国家重点实验室, 陕西杨凌 712100;2新疆农业职业技术学院, 新疆昌吉 831100
小麦花发育MADS-box基因的表达模式分析
李海峰1,2,*韩英1刘梦佳1王冰华1苏亚丽1孙其信1,*
1西北农林科技大学农学院 / 旱区作物逆境生物学国家重点实验室, 陕西杨凌 712100;2新疆农业职业技术学院, 新疆昌吉 831100
为解析小麦花发育的分子机制, 构建了MADS-box基因系统进化树, 发现小麦中存在花发育ABCDE模型的各类基因。半定量RT-PCR和定量RT-PCR分析结果显示, A类基因AP1/FUL小麦中的同源基因TaFUL在所有花器官中都表达, 在外稃和內稃中表达水平最高。B类基因TaAP3和C类基因TaAG的表达模式保守, TaAP3在浆片和雄蕊中表达, TaAG在雄蕊和雌蕊中表达。D类基因OsMADS13的同源基因除了在雌蕊表达外, 在浆片也有较高的表达,暗示该基因可能同时影响胚珠和浆片的发育。E类基因TaSEP主要在内稃和内三轮器官表达, 在外稃和护颖中不表达。LHS1是禾本科特有的MADS-box基因亚家族, 发挥E类基因的功能。TaLHS1除了在內稃和外稃表达, 在护颖中也有表达。水稻中控制心皮发育的DROOPING LEAF(DL)基因的同源基因TaDL除了在外稃和心皮表达外, 在护颖中也有表达。根据这些结果, 我们认为控制小麦花发育的分子机制比较保守, 但部分基因功能在进化过程中可能发生了分化。另外, 结合小麦外稃和护颖形态结构分析, TaLHS1以及TaDL的表达模式暗示小麦的护颖和外稃这2个器官可能有共同的起源。
小麦; 花发育; MADS-box基因; 表达模式
双子叶植物拟南芥(Arabidopsis)的花从外向内由4轮花器官组成, 呈同心圆排列, 依次为萼片、花瓣、雄蕊、心皮。20世纪90年代初, 学者根据双子叶植物拟南芥和金鱼草(Antirrhinum)花器官突变体的研究, 提出了花器官发育的ABC模型[1], 随后扩展为ABCDE模型[2-3], 将调控花发育的基因分为A、B、C、D、E共5类。其中, A类和E类基因决定花萼特征, A、B和E类基因一起调控花瓣发育, B、C和E类基因共同决定雄蕊发育, C类和E类基因决定心皮发育, D类和E类决定胚珠发育[2-3]。
单子叶模式植物水稻(Oryza sativa)的小花从外到内同样包括4轮器官, 分别是外(內)稃、浆片、雄蕊和雌蕊。近年来, 随着基因组测序的完成, 调控水稻花器官发育的多个ABCDE类基因功能被解析, 如AP1/FUL类基因OsMADS14、OsMADS15和OsMADS18[4-5], B类基因OsMADS16/SPW[6], C类基因OsMADS3和OsMADS58[7], D类基因OsMADS13和OsMADS21[8-10], E类基因OsMADS1、OsMADS5、OsMADS7、OsMADS8和OsMADS34[11-13], 以及控制心皮发育的基因DROOPING LEAF (DL)[14-15]。除A类基因外, 基于双子叶植物建立的花器官发育模型基本上适用于单子叶植物。
穗粒数是决定小麦(Triticum aestivum)产量三要素之一,提高穗粒数是高产、超高产小麦栽培和品种选育的主攻目标, 而小麦穗粒数是小花分化发育和结实的最终体现[16]。小麦花为复穗状花序, 组成花序的基本单位是小穗。小穗包含2枚颖片和若干朵小花[17]。作为异源六倍体物种, 小麦中有3个FUL (FRUITFULL)基因[18]。在小麦中, 也检测到与拟南芥B类基因PI和AP3、C类基因AG和D类基因STK同源的基因[18], 发现了决定水稻心皮特征DL基因的同源基因, 而且小麦DL和水稻DL的时空表达模式非常相似, 暗示该基因可能同样调控心皮的发育[19]。对小麦中的E类基因研究较多, Anna等[18]和Shitsukawa等[20]分别克隆了WSEP的序列, 发现小麦7A、7B和7D染色体上各有一个部分同源(homoeologous)基因, 均与拟南芥SEP3及水稻MADS7同源; 通过拟南芥异源过量表达以及蛋白质互作分析, 发现这3个基因具有功能上的冗余性[20]。LHS1基因亚家族是禾本科特有的一个MADS-box基因家族[11]。在小麦基因组中检测到3个WLHS1部分同源基因, 分别位于小麦A、B和D基因组的第4染色体上[20]。因为一个大片段的插入, 导致WLHS1-A失去了生物学功能; WLHS1-B和WLHS-D序列相似, 其中WLHS-B因表观遗传学修饰而表达沉默, 推测小麦中主要由WLHS1-D执行LHS1基因的功能[20]。国内学者在小麦花育基因领域的研究也取得一定进展, 如克隆了42个小麦MADS-box基因的序列[21]; 分析了TaMADS1的表达模式, 并发现其在拟南芥中过量表达引起早花和花器官发育异常[22]。
尽管对小麦MADS-box基因的研究逐渐增多, 但还未有系统研究小麦ABCDE类基因表达模式的报道。本研究在构建系统进化树的基础上, 选取部分代表性基因, 利用半定量和定量RT-PCR方法分析了小麦花发育相关的代表性基因在不同花器官中的表达, 并比较了拟南芥及水稻同源基因的表达。研究结果为揭示相关基因的功能和小麦花发育的分子机制奠定了基础。
1 材料与方法
1.1植物材料及花器官形态结构观察
2013年和2014年10月, 连续两年将中国春小麦种植于西北农林科技大学试验田(陕西杨凌), 次年在5月1日前后, 采集各发育时期的花序、小穗和小花材料用于RNA提取。参照文献[17]照相、制石蜡切片和用扫描电子显微镜观察分析。
1.2系统进化树构建方法
根据文献报道, 分别用16个拟南芥MADS-box蛋白和21个水稻MADS-box蛋白序列, 通过BLASTP在NCBI数据库(http://www.ncbi.nlm.nih.gov/)上进行搜索, 寻找小麦、短柄草(Brachypodium distachyon)、玉米(Zea mays)中序列同源的MADS-box转录因子。利用MEGA 5.10软件[23]构建小麦MADS-box基因家族系统发育树, 首先用Muscle程序比对序列, 再根据比对结果选出最优模型, 最后用JJT+G模型、邻近法(neighbor-joining, NJ)构建系统发育进化树, 显示各分支长度。
1.3半定量RT-PCR和荧光定量RT-PCR
根据进化树, 从A、B、C、D、E类中各选一个代表性基因, 即OsMADS14同源基因VRN-1 (AM502869)、OsMADS16同源基因TaAP3 (AM502879)、OsMADS3同源基因TaAG-2A (AM502898)、OsMADS13同源基因TaAGL9(AM502865)、OsMADS7同源基因TaSEP-D (AB295661),根据NCBI数据库序列, 运用Primer Primier 5.0软件设计引物, 由上海生工生物工程有限责任公司合成。禾本科特异的E类基因LHS1和水稻心皮决定基因DROOPING LEAF (DL)在小麦中的同源基因TaLHS1-D (AB295664)和TaDL (AB470269)的半定量和定量引物设计同上。内参基因为ACTIN[20]。引物序列见表1。
用TRIzol总RNA提取试剂盒(Sangon, 上海), 从小麦护颖和外稃、內稃、浆片、雄蕊以及雌蕊中提取总RNA,用反转录试剂盒(PrimeScript RT reagent Kit, Fermentas,Canada)合成第1链cDNA[24], 按窦艳华等[24]描述的方法操作进行半定量RT-PCR。
用CFX96 Real-time PCR detection Systems, 按照SYBR Premix Ex Taq (Tli RNaseH Plus)试剂盒(TaKaRa,大连)操作说明进行荧光定量RT-PCR。PCR反应体系15 μL, 含0.5 μL cDNA (5.0 ng μL–1), 7.5 μL SYBR Premix Ex Taq (2× Sangon, 上海), 各0.75 μL上、下游引物(10 pmol μL–1), ddH2O 5.5 μL。扩增条件为: 95℃预变性30 s; 运行61个循环, 包括95℃变性5 s, 55℃退火30 s, 40个循环;65℃延伸10 s。设3次生物学重复和3次实验重复。按窦艳华等[24]描述的方法分析数据。
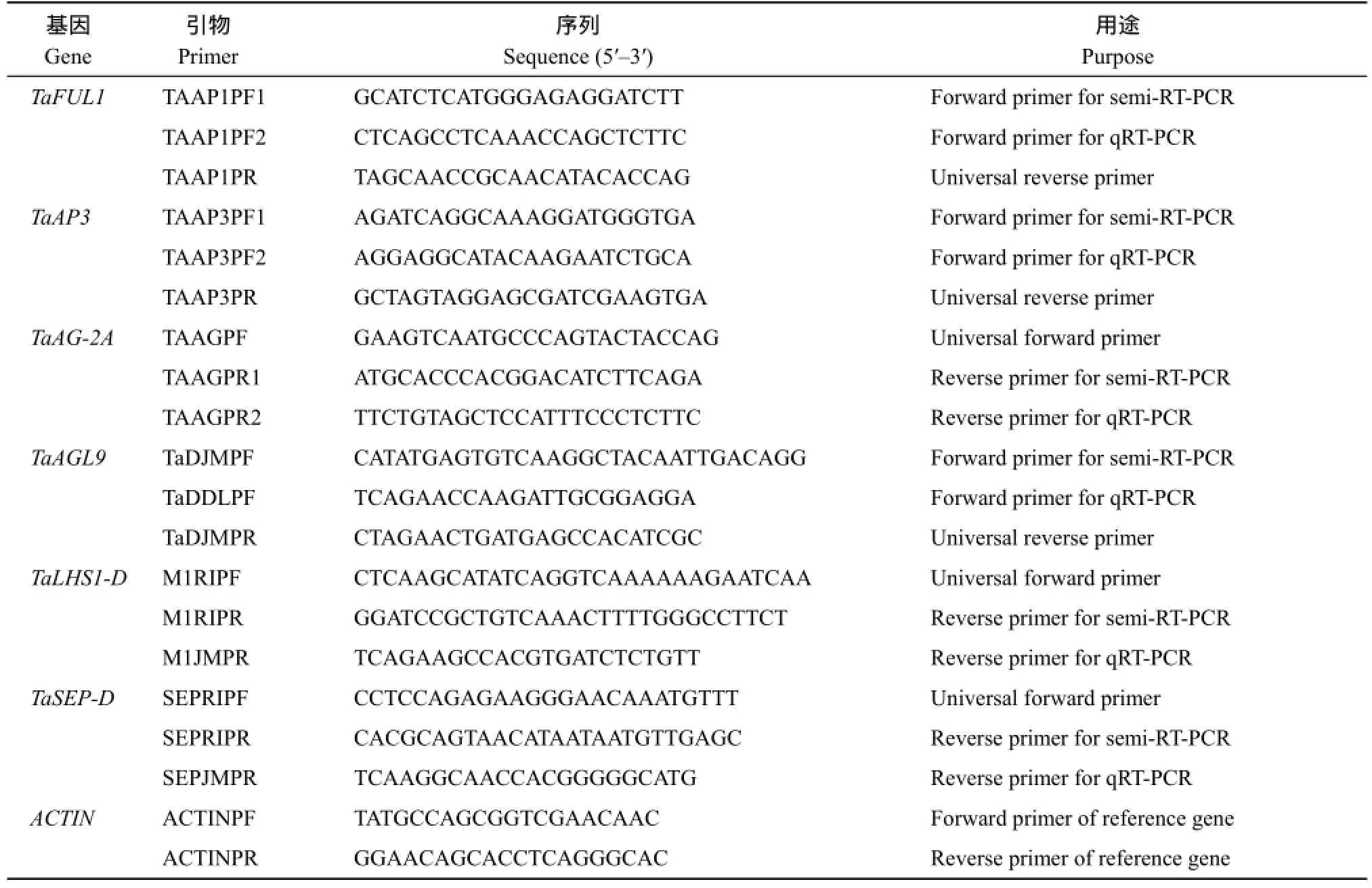
表1 用于表达分析的基因及其引物Table 1 Genes and their primers used in expression analysis
2 结果与分析
2.1小麦花的形态结构
小穗是组成小麦花序的基本单位, 每小穗(图1-A)由2片护颖和若干朵小花组成, 每朵小花从外到内包括4轮花器官, 依次是外稃和内稃(图1-B)、浆片(图1-D)、雄蕊、雌蕊(图1-C)。外稃坚硬, 具有保护内部花器官和种子的作用, 内稃薄而透明; 浆片位于外稃一侧, 由透明细胞和薄壁细胞组成, 在开花时期吸水膨胀, 撑开内、外稃, 使花药伸出; 3枚雄蕊中, 1枚位于浆片之间, 另2枚位于内稃侧(图1-C)。
在整体形态上, 外稃(图1-F)和护颖(图1-E)更相似,而与内稃(图1-G)较易区分。在扫描电镜下, 外稃(图1-I)和护颖(图1-H)表面形态相似, 而与內稃(图1-J)不同。观察小穗的组织切片(图1-K), 发现护颖(图1-L)和外稃(图1-M)都包含多个维管束, 內稃只有2个维管束(图1-N)。在细胞组成上, 外稃(图1-M)和护颖(图1-L)也非常一致,和內稃(图1-O, 1-P)的差别较大。
2.2系统进化树分析
根据文献报道或者NCBI数据库的小麦MADS-box基因序列, 以拟南芥、水稻和短柄草(Brachypodium distachyon)的基因为基础, 构建了系统进化树。从进化树来看, 花发育ABCDE模型的B、C、D和E类基因, 在小麦中都有同源基因存在, 虽然序列的保守程度不同。举例来说, B类基因有和水稻OsMADS16同源的基因WAP3; D类基因有和OsMADS13同源的TaAGL9; E类基因有和OsMADS7同源的TaSEP, 和OsLHS1同源的TaLHS1。另外, AGL6基因在禾本科的花发育中也发挥着重要的作用。在小麦基因组中同样存在AGL6家族基因。小麦中还存在FUL基因(图2)。
2.3相关基因的表达模式
半定量(图3)和定量RT-PCR(图4)结果表明, 小麦中AP1/FUL家族基因TaFUL1/VRN-1在护颖和4轮器官中都有表达, 在护颖和内外稃中表达水平较高, 在雄蕊和雌蕊中表达较低, 在浆片中表达水平最低。这个结果一方面暗示该基因可能具有部分A基因的功能, 同时又和拟南芥中AP1的功能不同。因为在ABCDE模型中, A类基因和C类基因是互相拮抗的, 拟南芥中的AP1在雄蕊中是不表达的, 而在第2轮花器官花瓣中是有较高水平的表达的。

图1 小麦花的结构和器官Fig. 1 Wheat flower structure and floral organsA: 小穗; B: 剥去一半外稃的小花; C: 内轮花器官; D: 浆片; E: 护颖; F: 外稃; G: 內稃; H~J: 护颖(H)、外稃(I )內和稃(J)表层的扫描电镜照片; K: 小穗石蜡横切片; L和M: 护颖(L)和外稃(M)横切片局部放大; N: 內稃的横切片; O和P: 內稃边缘(O)和中间结构(P)的放大。L–N中,*指示维管束。gl: 护颖; le: 外稃; pa: 內稃; lo: 浆片; st: 雄蕊; pi: 雌蕊。A~C和E~G中, bar = 500 µm; D和H~J中, bar = 100 µm。A: spikelet; B: floret with a half of lemma; C: inner floral organs; D: lodicules; E: glume; F: lemma; G: palea; H–J: epidermics SEM observation of glume (H), lemma (I), and palea (J); K: transverse section of one spikelet; L and M: Amplification of glume (L) and lemma (M). N: Transverse section of one palea; O and P: amplification of marginal tissue (O) and main strucuture of palea (P). Asterisks in L–N indicate vascular bundles. gl: glume;le: lemma; pa: palea; lo: lodicule; st: stamen; pi: pistil. Bar = 500 µm in A–C and E–G; bar = 100 µm in D and H–J.
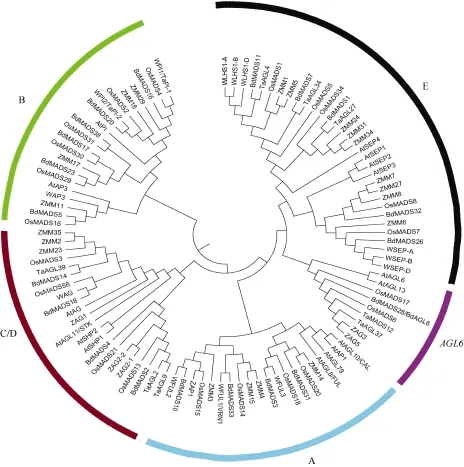
图2 拟南芥、水稻、短柄草、玉米、小麦花发育基因系统进化树Fig. 2 Phylogenetic tree of floral genes in Arabidopsis, Brachypodium distachyon, rice, maize, and wheat不同颜色的圆弧表示不同基因类型。Color circular arcs show the gene classes.
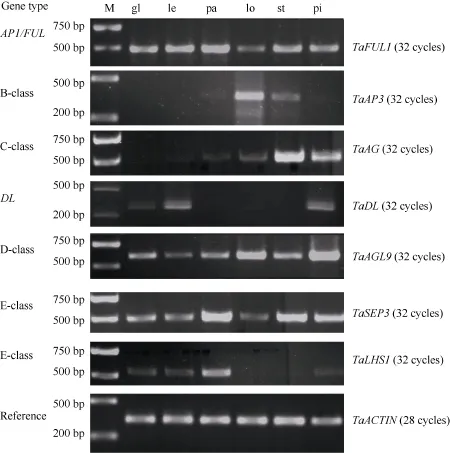
图3 以半定量RT-PCR分析小麦花发育基因在护颖和花器官中的表达模式Fig. 3 Expression patterns of wheat flower genes in glumes and floral organs by semiquantitative RT-PCR assayM: DL 2000 marker; gl: 护颖; le: 外稃; pa: 内稃; lo: 浆片; st: 雄蕊; pi: 雌蕊。M: DL 2000 marker; gl: glume; le: lemma; pa: palea; lo: lodicule; st: stamen; pi: pistil.

图4 以定量RT-PCR分析小麦花发育基因在护颖和花器官中的表达模式Fig. 4 Expression patterns of wheat flower genes in glumes and floral organs by qRT-PCR assaygl: 护颖; le: 外稃; pa: 内稃; lo: 浆片; st: 雄蕊; pi: 雌蕊。gl: glume; le: lemma; pa: palea; lo: lodicule; st: stamen; pi: pistil.
小麦B类基因TaAP3在浆片和雄蕊中表达, 在其他器官基本不表达, 暗示TaAP3的功能比较保守, 和拟南芥中的AP3基因一样, 参与决定第2轮和第3轮花器官的形态建成。小麦C类基因TaAG的表达模式也比较保守, 主要在第3轮和第4轮生殖器官表达, 在雄蕊表达水平特别高, 暗示该基因主要决定雄蕊的特征。小麦中的D类基因,OsMADS13的同源基因在各轮器官都有表达, 主要在雌蕊和浆片中表达。在雌蕊的表达水平最高, 在浆片中也有较高水平的表达。这点和OsMADS13特异在雌蕊(胚珠)表达是不同的[10], 表明该基因的功能可能出现了分化,既决定胚珠的发育, 又参与调控浆片的发育。小麦E类基因TaSEP在內稃、浆片、雄蕊和雌蕊中都有表达。在內稃的表达水平最高, 依次是在雄蕊、浆片和雌蕊中。这和拟南芥及水稻不同。拟南芥中SEP基因在4轮花器官中都表达, OsMADS7和OsMADS8则在第2、第3和第4轮花器官表达, 在内外稃不表达[11]。另外, 水稻中的DL基因特异在心皮和外稃的维管束中表达[10]。小麦基因组中存在DL的同源基因。表达模式分析表明, TaDL基因和水稻一样, 在外稃和雌蕊中高水平表达, 但该基因同时在护颖中有一定的表达。这点和水稻是不同的。这一方面表明该基因的功能比较保守, 同时也出现了一定的分化。
另外, 小麦基因组中存在水稻另一个E类基因LHS1的同源基因。OsLHS1在外稃、内稃和雌蕊中表达[25]。半定量PCR结果表明, TaLHS1除了在内外稃中高水平表达、在雌蕊中低水平表达外, 在护颖中也有一定水平的表达。这点和OsLHS1的表达是不同的。
3 讨论
3.1禾本科植物中的浆片相当于拟南芥中的花瓣
拟南芥小花中, 第2轮有4枚白色的花瓣组成。B类基因AP3在花瓣和雄蕊中表达。该基因突变时, 雄蕊发育成心皮状结构, 花瓣同源异化形成花萼状结构[26]。禾本科作物水稻、玉米和小麦的小花, 组成花被的两轮器官和拟南芥有很大的不同。第1轮是外稃和內稃, 第2轮是2枚白色的浆片, 由透明细胞组成。在开花时, 吸水膨胀, 撑开内外稃。
水稻中AP3的同源基因MADS16/SUPERWOMAN也在第2轮和第3轮表达, 基因突变后, 雄蕊同源异化为心皮状结构, 浆片则同源异化为稃状结构[6]; 玉米中AP3同源基因ZMM16/silk基因突变后, 浆片同样转化为外稃状结构[27]。这些结果暗示浆片等同于双子叶植物中的花瓣。
小麦AP3的表达模式分析结果表明, 该基因同样只在浆片和雄蕊中表达, 而且在浆片中的表达水平远远高于在雄蕊中, 暗示该基因在决定浆片的器官特征方面具有重要的功能, 为单子叶植物的浆片等同于双子叶植物的花瓣假说提供了新的证据。
3.2小麦中的护颖和外稃更相近
在水稻中, 一个小穗只有一朵小花, 在小花的外面有2个属性不确定的器官(organs of unknown identity, OUI)所包被[13]。目前根据不同的解释, 有不同的命名。一种解释是把护颖看作变形的苞叶, 命名为护颖(empty glume); 另一种解释是把护颖看作退化的小花遗留下的花器官, 命名为不育的外稃。护颖和内外稃有一些明显的不同: 护颖只有1个维管束, 外稃有5个, 內稃则有3个; 护颖由1层细胞组成, 内外稃由4层细胞组成; 护颖的结构比较均一, 内外稃除了主体结构, 都还有边缘结构[13]。在分子机制上, 护颖的特征由MADS34[12-13,28]和G1/ELE[29-30]等基因决定; 外稃的特征由DL等基因决定[6,10], 內稃由OsMADS6等基因决定[25]。G1/ELE、OsMADS34是决定OUI特征的2个重要基因, LHS1、DL在OUI中都不表达。对这2个基因突变体的分析也为OUI是退化的外稃这一学说提供了新的证据[28-30]。
在整体形态、细胞组成和表面形态几个方面, 小麦的外稃和护颖更相似(图1)。基因表达模式分析表明, TaDL除了在外稃表达外, 在护颖中也有一定水平的表达; 同样的, LHS1在护颖中也有和在外稃中表达水平一致的表达,而且表达水平都低于内稃; TaSEP在护颖和外稃中都不表达, 但是在內稃中高水平表达(图3和图4)。这些结果进一步在分子水平上提供了证据, 证明护颖和外稃具有更亲缘的关系, 可能具有共同的起源。
References
[1] Coen E S, Meyerowitz E M. The war of the whorls: genetic interactions controlling flower development. Nature, 199, 353: 31–37
[2] Pelaz S, Ditta G S, Baumann E, Wisman E, Yanofsky M F. B and C floral organ identity functions require SEPALLATA MADS-box genes. Nature, 2000, 405: 200–203
[3] Theissen G. Development of floral organ identity: stories from the MADS house. Curr Opin Plant Biol, 2001, 4: 75–85
[4] Wang K, Tang D, Hong L, Xu W, Huang J, Li M, Gu M, Xue Y,Cheng Z. DEP and AFO regulate reproductive habit in rice. PLoS Genet, 2010, 6(1): e1000818
[5] Kobayashi K, Yasuno N, Sato Y, Yoda M, Yamazaki R, Kimizu M,Yoshida H, Nagamura Y, Kyozuka J. Inflorescence meristem identity in rice is specified by overlapping functions of three AP1/FUL-like MAD-box genes and PAP2, a SEPALLATA MADS-box gene. Plant Cell, 2012, 24: 1848–1859
[6] Nagasawa N, Miyoshi M, Sano Y, Satoh H, Hirano H, Sakai H,Nagato Y. SUPERWOMAN1 and DROOPING LEAF genes control floral organ identity in rice. Development, 2003, 130: 705–718
[7] Yamaguchi T, Lee DY, Miyao A, Hirochika H, An G, Hirano H Y. Functional diversification of the two C-class MADS-box genes OsMADS3 and OsMADS58 in Oryza sativa. Plant Cell, 2006, 18: 15–28
[8] Dreni L, Jacchia S, Fornara F, Fornari M, Ouwerkerk P B, An G,Colombo L, Kater M M. The D-lineage MADS-box gene Os-MADS13 controls ovule identity in rice. Plant J, 2007, 52: 690–699
[9] Dreni L, Pilatone A, Yun D, Erreni S, Pajoro A, Caporali E,Zhang D, Kater M M. Functional analysis of all AGAMOUS subfamily members in rice reveals their roles in reproductive organ identity determination and meristem determinacy. Plant Cell,2011, 23: 2850–2863
[10] Li H, Liang W, Yin C, Zhu L, Zhang D. Genetic interaction of OsMADS3, DROOPING LEAF, and OsMADS13 in specifying rice floral organ identities and meristem determinacy. Plant Physiol, 2011, 156: 263–274
[11] Cui R, Han J, Zhao S, Su K, Wu F, Du X, Xu Q, Chong K,
Theissen G, Meng Z. Functional conservation and diversificationof class E floral homeotic genes in rice (Oryza sativa). Plant J,2010, 61: 767–781
[12] Gao X, Liang W, Yin C, Ji S, Wang H, Su X, Guo C, Kong H,Xue H, Zhang D. The SEPALLATA-like gene OsMADS34 is required for rice inflorescence and spikelet development. Plant Physiol, 2010, 153: 728–740
[13] Lin X, Wu F, Du X, Shi X, Liu Y, Liu S, Hu Y, Theissen G, Meng Z. The pleiotropic SEPALLATA-like gene OsMADS34 reveals that the ‘empty glumes’ of rice (Oryza sativa) spikelets are in fact rudimentary lemmas. New Phytol, 2014, 202: 689–702
[14] Yamaguchi T, Nagasawa N, Kawasaki S, Matsuoka M, Nagato Y,Hirano H Y. The YABBY gene DROOPING LEAF regulates carpel specification and midrib development in Oryza sativa. Plant Cell,2004, 16: 500–509
[15] Li H, Liang W, Hu Y, Zhu L, Yin C, Xu J, Dreni L, Kater M M,Zhang D. Rice MADS6 interacts with the floral homeotic genes SUPERWOMAN1, MADS3, MADS58, MADS13, and DROOPING LEAF in specifying floral organ identities and meristem fate. Plant Cell, 2011, 23: 2536–2552
[16] 王兆龙. 小麦小花发育的生理基础及调控研究. 南京农业大学博士学位论文, 江苏南京, 2000. pp 3–4 Wang Z L. Physiological Basis and Regulation of Floret Development in Wheat. PhD Dissertation of Nanjing Agricultural University, Nanjing, China, 2000. pp 3–4 (in Chinese with English abstract)
[17] 刘楠, 李海峰, 窦艳华, 韩德俊. 普通小麦及其近缘物种花序、小穗和小花的形态结构分析. 麦类作物学报, 2015, 35: 293–299 Liu N, Li H F, Dou Y H, Han D J. Morphology and structure analyses on inflorescence, spikelet and floret of bread wheat and its relatives. J Triticeae Crops, 2015, 35: 293–299 (in Chinese with English abstract)
[18] Paolacci A R, Tanzarella O A, Porceddu E, Varotto S, Ciaffi M. Molecular and phylogenetic analysis of MADS-box genes of MIKC type and chromosome location of SEP-like genes in wheat(Triticum aestivum L.). Mol Genet Genomics, 2007, 278: 689–708
[19] Ishikawa M, Ohmori Y, Tanaka W, Hirabayashi C, Murai K,Ogihara Y, Yamaguchi T, Hirano H Y. The spatial expression patterns of DROOPING LEAF orthologs suggest a conserved function in grasses. Genes Genet Syst, 2009, 84: 137–146
[20] Shitsukawa N, Tahira C, Kassai K, Hirabayashi C, Shimizu T, Takumi S, Mochida K, Kawaura K, Ogihara Y, Murai K. Genetic and epigenetic alteration among three homoeologous genes of a class E MADS-box gene in hexaploid wheat. Plant Cell, 2007, 19: 1723–1737
[21] Zhao T, Ni Z, Dai Y, Yao Y, Nie X, Sun Q. Characterization and expression of 42 MADS-box genes in wheat (Triticum aestivum L.). Mol Genet Genomics, 2006, 276: 334–350
[22] Zhao X Y, Cheng Z J, Zhang X S. Overexpression of TaMADS1,a SEPALLATA-like gene in wheat, causes early flowering and the abnormal development of floral organs in Arabidopsis. Planta,2006, 223: 698–707
[23] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 2011, 28: 2731–2739
[24] 窦艳华, 韩萌萌, 孙其信, 李海峰. 二穗短柄草MADS-BOX基因AGL6和FUL1的可变拼接和表达模式分析. 农业生物技术学报, 2015, 23: 459–468 Dou Y H, Han M M, Sun Q X, Li H F. Alternative splicing and expression pattern analyses of two MADS-BOX genes AGL6 and FUL1 in Brachypodium distachyon. J Agric Biotechnol, 2015, 23: 459–468 (in Chinese with English abstract)
[25] Li H F, Liang W Q, Jia R D, Yin C S, Zong J, Kong H Z, Zhang D B. The AGL6-like gene OsMADS6 regulates floral organ and meristem identities in rice. Cell Res, 2010, 20: 299–313
[26] Jack T, Brockman L L, Meyerowitz E M. The homeotic gene APETALA3 of Arabidopsis thaliana encodes a MADS-box and is expressed in petals and stamens. Cell, 1992, 68: 683–697
[27] Ambrose B A, Lerner D R, Ciceri P, Padilla C M, Yanofsky M F,Schmidt R J. Molecular and genetic analyses of the silky1 gene reveal conservation in floral organ specification between eudicots and monocots. Mol Cell, 2000, 5: 569–579
[28] Kobayashi K, Maekawa M, Miyao A, Hirochika H, Kyozuka J. PANICLE PHYTOMER2 (PAP2), encoding a SEPALLATA subfamily MADS-box protein, positively controls spikelet meristem identity in rice. Plant & Cell Physiol, 2010, 51: 47–57
[29] Yoshida A, Suzaki T, Tanaka W, Hirano H Y. The homeotic gene long sterile lemma (G1) specifies sterile lemma identity in the rice spikelet. Proc Natl Acad Sci USA, 2009, 106: 20103–20108
[30] Hong L, Qian Q, Zhu K, Tang D, Huang Z, Gao L, Li M, Gu M,Cheng Z. ELE restrains empty glumes from developing into lemmas. J Genet Genomics, 2010, 37: 101–115
Expression Patterns of MADS-box Genes Related to Flower Development of Wheat
LI Hai-Feng1,2,*, HAN Ying1, LIU Meng-Jia1, WANG Bing-Hua1, SU Ya-Li1, and SUN Qi-Xin1,*1State Key Laboratory of Crop Stress Biology for Arid Areas / College of Agronomy, Northwest A&F University, Yangling 712100, China;2Xinjiang Agriculture Vocational Technology College, Changji 831100, China
The objective of this study was to elucidate the molecular mechanism of wheat (Triticum aestivum L.) flower development. According to the phylogenetic tree of MADS- box genes from different species, we found that wheat contained all kinds of genes involved in the ABCDE model for flower development. The expression patterns of A-, B-, C-, D-, and E-class genes were analyzed by semi-quantitative and quantitative RT-PCR (qRT-PCR). Wheat AP1/FUL gene TaFUL (A-class) was expressed in all floral organs with the highest expression level in lemmas and paleas. Genes TaAP3 (B-class), TaAG (C-class) showed conservative expression patterns in specific organs, i.e., TaAP3 was expressed in lodicules and stamens whereas TaAG was expressed in stamens and pistils. The OsMADS13 homologous gene in wheat (D-class) was expressed in both pistils and lodicules, suggesting its function in lodicule and ovule development simultaneously. Gene TaSEP (E-class) was mainly expressed in paleas and the inner-three whorls. LHS1 is a grass-specific gene family and belongs to E-class. The expression of TaLHS1 was detected in lemmas,paleas, and glumes of wheat. TaDL, the homologous gene of rice DROOPING LEAF (DL) controlling carpel development, was expressed in glumes, lemmas and carpels. These results suggest a conservative molecular mechanism for flower development in wheat, but some genes may have diversified functions due to evolution. The expression evidence of TaDL and TaLHS1 in glumes,in combination with the morphology and structure analyses of glume, lemma and palea, implied that lemma and glume might originate from the same organ in wheat.
Wheat; Flower development; MADS- box gene; Expression pattern
10.3724/SP.J.1006.2016.01067
本研究由国家自然科学基金项目(31571657), 中央高校基本科研业务费项目(2014ZZ009)和新疆农业职业技术学院科研项目(XJNZYKJ201501)资助。
This study was supported by the National Natural Science Foundation of China (31571657), the Fundamental Research Foundation for the Central Universities (2014ZZ009), and the Foundation of Xinjiang Agriculture Vocational Technology College (XJNZYKJ201501).
(Corresponding authors): 李海峰, E-mail: lhf@nwsuaf.edu.cn; 孙其信, E-mail: qxsun@cau.edu.cn
Received(): 2015-11-24; Accepted(接受日期): 2016-03-14; Published online(网络出版日期): 2016-05-09.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20160509.0957.006.html
