茶树PVP申请品种的SSR分子标记鉴定和系谱关系分析
2016-09-18黄丹娟马建强陈亮
黄丹娟,马建强,陈亮
茶树PVP申请品种的SSR分子标记鉴定和系谱关系分析
黄丹娟,马建强,陈亮*
中国农业科学院茶叶研究所国家茶树改良中心,农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008
利用30对SSR引物,对26个植物新品种保护(PVP)申请茶树品种及其13个近似品种与亲本进行了分子鉴定和系谱关系分析,探讨SSR分子标记在茶树新品种保护工作和DUS测试中的应用。研究结果显示,30对SSR引物共检测到131个等位基因,每对引物检测的等位基因数为3~7个,平均4.4个。平均Shannon信息指数(I)和多态信息含量(PIC)分别为1.04和0.51。39个参试品种的遗传距离在0.03~0.70之间。在遗传距离为0.15时,可将39个品种划分为7类,其中地理来源一致或遗传背景相似的材料基本上聚为一类。30对引物的品种鉴定能力差异较大,每对引物能鉴定的品种数为3~16个。通过4对核心引物的组合可以鉴定全部39个参试品种,并依此构建了SSR指纹图谱。
茶树;PVP;SSR标记;指纹图谱
植物新品种保护(Plant variety protection,PVP)是知识产权的一种保护形式。1999年,我国加入国际植物新品种保护联盟(The international union for the protection of new varieties of plants,UPOV),随后开始植物新品种保护工作。山茶属植物与茶组植物分别于1999年和2008年被列入国家林业局和农业部发布的中华人民共和国植物新品种保护名录。随着农业科技创新和知识产权意识的提升,茶树新品种保护日益受到人们的关注和重视。目前,已获得植物新品种权授权的茶树品种有9个,正在进行新品种特异性、一致性和稳定性(DUS)测试的申请品种有20多个。目前现行的茶树DUS测试与品种鉴定通常采用基于外观形态特征的田间观察检测方法,需要茶苗定植后两年以上才能开展,并且测试性状多为多基因控制的数量性状,受环境条件影响,可能导致测试结果不稳定,进一步延长测试周期。同时,近年来茶树新品种的遗传基础越来越集中在少数骨干亲本上,根据形态学进行品种鉴定越来越困难。因此,开发简单快速稳定的测试技术,对于提升茶树新品种保护和管理能力具有重要意义。
基于DNA标记技术构建的分子指纹图谱,具有高度的特异性和环境稳定性,是植物品种鉴定中十分有效的辅助手段。其中,SSR(Simple sequence repeat)标记因具有等位变异高、共显性遗传、操作简便快速等优点[1],在植物指纹图谱研究中被广泛应用。UPOV在其生物化学和分子技术测试指南中已将SSR标记作为DNA指纹数据库构建的推荐标记之一。目前,我国利用SSR标记已构建了玉米[2-3]、小麦[4-5]、水稻[6-7]等作物现有品种的DNA指纹数据库,并且在新品种DUS测试中展现出巨大的应用潜力。
本研究利用30对SSR引物对26个茶树PVP申请品种及其13个近似品种与亲本进行分子鉴定和系谱关系分析,通过筛选特征引物组合,构建参试品种的SSR指纹图谱,探讨其在茶树DUS测试中的应用潜力,以期为我国茶树新品种权保护提供理论依据和技术支撑。
1 材料与方法
1.1 材料
供试的39个茶树品种如表1所示,其中1~26号为已经获得新品种权或正在进行DUS测试的品种,27~39号为对应的近似品种和亲本。从国家种质杭州茶树圃(I)和勐海茶树分圃(II)、贵州省农业科学院茶叶研究所(III)、广东省农业科学院饮料植物研究所(IV)采摘各参试品种无病虫害感染的一芽二叶新梢,液氮迅速冷冻后-80℃保存备用。
1.2 方法
1.2.1 基因组DNA提取
采用改进的CTAB法[8]提取基因组DNA,l%琼脂糖凝胶电泳检测DNA质量,ND-1000 UV-Vis Speetrophotometer检测DNA浓度,稀释到20 ng·μL-1用于PCR扩增。
1.2.2 PCR扩增及产物检测
30对SSR引物的相关信息参见马建强[9]的报道。引物序列由上海生物工程技术服务有限公司合成。PCR反应体系为DNA 2 μL,10×PCR buffer (Mg2+) l μL,2 U·μL-1Taq DNA polymerase 0.2 μL,10 μmol·L-1Forward primer 0.2 μL,10 μmol·L-1Reverse primer 0.2 μL,10 nmol·L-1dNTPs 0.2 μL。PCR反应程序为94℃预变性4 min,然后94℃变性30 s,退火30 s(退火温度根据所用引物确定),72℃延伸30 s,重复35个循环;随后72℃下延伸7 min。PCR产物用10%的聚丙烯酰胺凝胶和银染法进行检测[10]。
1.2.3 数据统计与分析
采用人工读带方法,每对引物只分析目标条带附近的扩增位点,等位基因按照电泳图谱从上到下依次命名为A、B、C、D、E等。利用POPGENE[11]统计每对引物在39个茶树品种中扩增等位基因数(Observed number of alleles,Na),计算有效等位基因(Effective number of alleles,Ne)、等位基因频率(Allele frequency)、观测杂合度(Observed heterozygosity,HO)、期望杂合度(Expected heterozygosity,HE)、Shannon信息指数(Shannon’s information index,I)。采用PowerMarker[12]计算基因型数(Genotype)、多态性信息量(Polymorphism information content,PIC),并根据Nei′s[13]遗传距离,进行Neighbor-Joining聚类分析。根据所选SSR引物的扩增条带清晰程度、基因型个数、PIC值高低等因素,筛选出能鉴别所有品种的核心引物。将核心引物扩增出的基因型转换为[0, 1]数据矩阵,利用在线二维码生成器(http://cli.im)构建DNA指纹图谱。
2 结果与分析
2.1 SSR引物的多态性
利用30对SSR引物对39份供试材料进行扩增,结果如表2所示。30对SSR引物共扩增出131个等位基因,每对引物扩增出的等位基因数为2~7个,平均4.4个;有效等位基因变异范围1.29~4.49,平均2.53个;共检测到239个基因型,平均达8个。引物观测杂合度(HO)最大为0.76,最小为0.21,平均值为0.47;期望杂合度(HE)最大为0.79,最小为0.23,平均值为0.56。Shannon信息指数(I)变化范围较大,为0.38~1.60,平均1.04。多态信息含量(PIC)最小为0.20,平均多态信息含量为0.51,引物TM391的PIC信息最高为0.74。
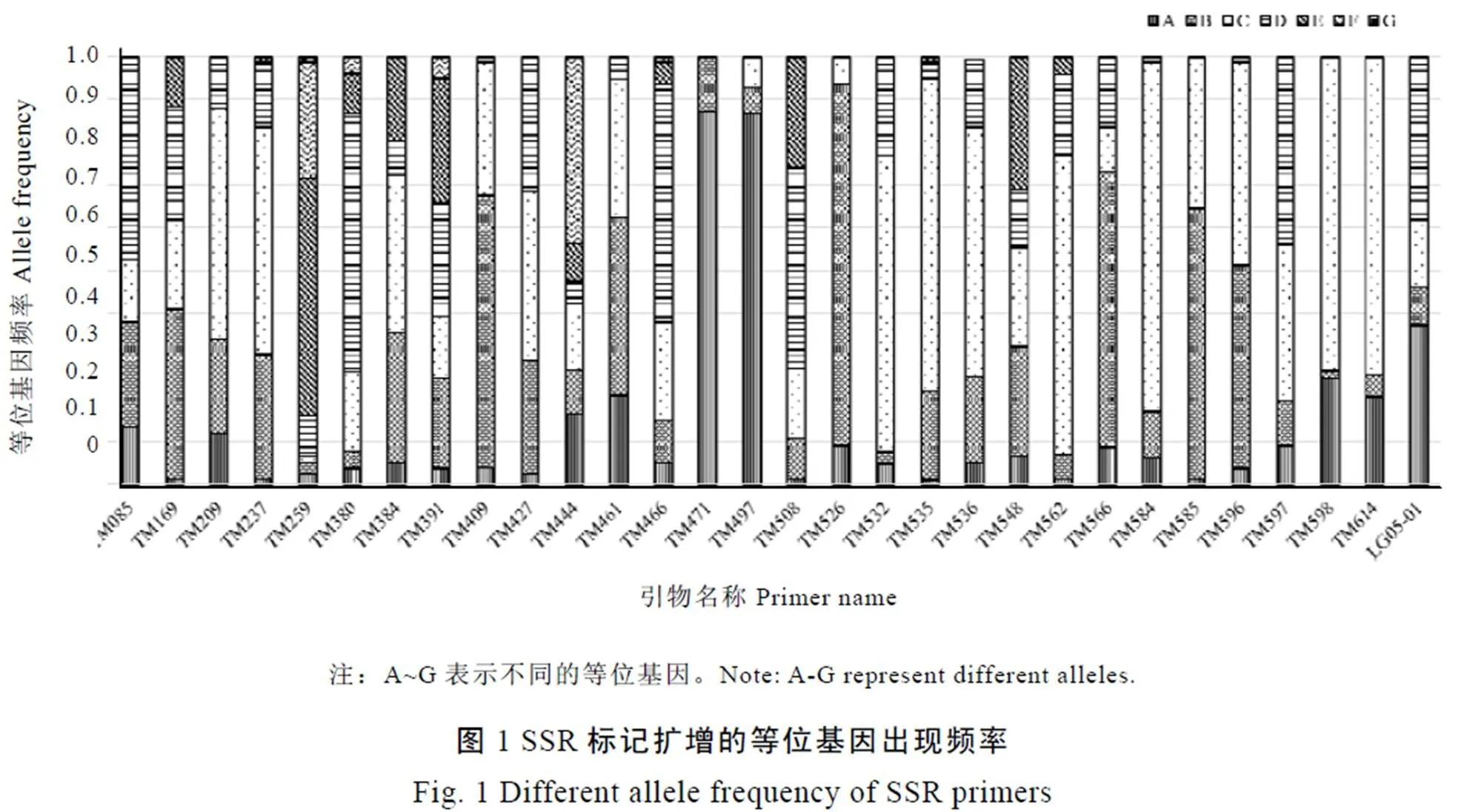
30对SSR引物在供试品种中共检测出了131个等位基因,不同等位基因出现频率差异很大。TM526的等位基因B出现频率最高(84%),其次为TM584的等位基因C(82%)。出现频率最低的为TM535的等位基因A和E,都只有1%(图1)。
2.2 39个新品种DNA指纹图谱的构建
将30对SSR引物的扩增谱带转化为[0, 1]数据进行分析,根据SSR引物扩增结果,30个茶树品种中有23个具有特征谱带(表3)。
利用特征引物可以快速准确地鉴定这些茶树品种。

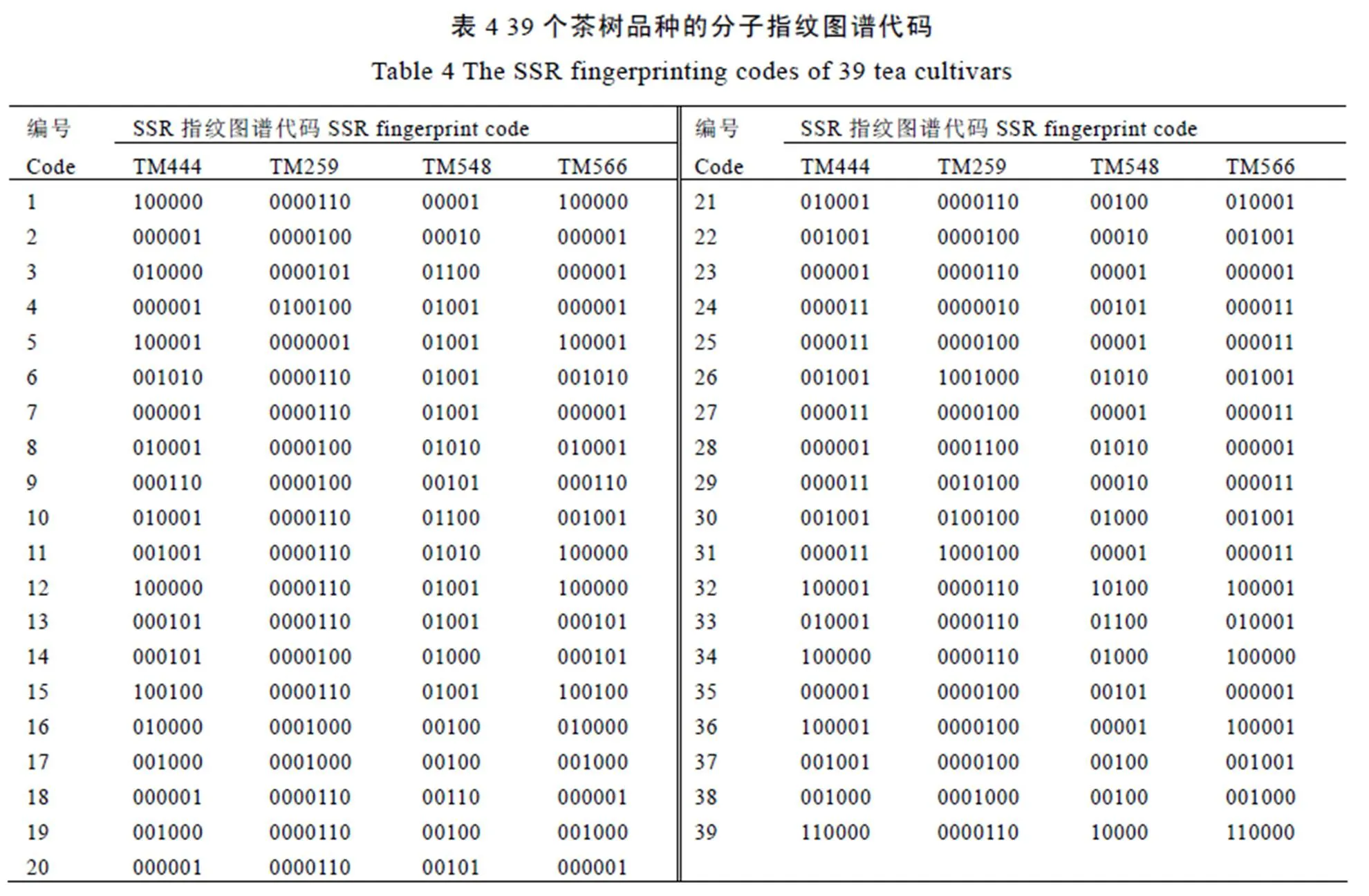
根据特征谱带统计结果可以看出,任何1对引物都不能区分开所有品种,只有采用引物组合鉴定的方式才能将39个品种全部区分开来。综合考虑所选SSR引物的扩增条带清晰程度、基因型个数、PIC值高低等因素,将引物进行排序,通过组合的方式,本研究采用最少4对引物(TM444、TM548、TM259、TM566),便可将参试品种全部区分开来。利用这4个标记的扩增信息,将参试品种的等位基因转换为l和0字符串,作为每一品种的分子身份证,并利用在线二维码生成器(http://cli.im)建立39份供试材料的指纹图谱(表4,图2)。

2.3 品种系谱关系分析
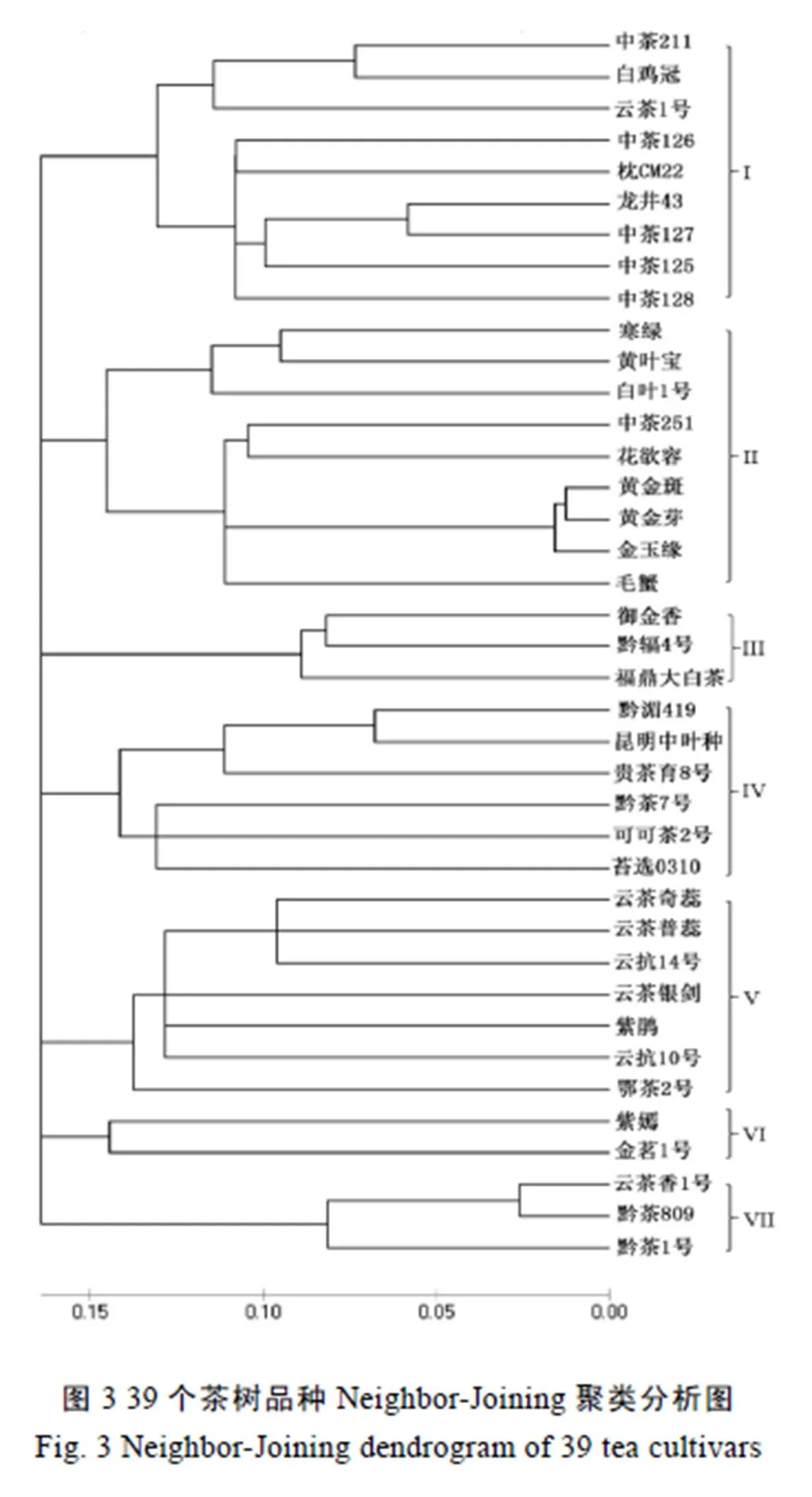
根据30对SSR引物等位基因出现的频率计算Nei′s遗传距离(D),结果显示39个品种的遗传距离为0.03~0.70。其中来源于浙江的品种黄金斑和金玉缘的遗传距离最小,来源于浙江的品种花欲容和来源于云南的品种云
茶奇蕊的遗传距离最大。
根据Nei′s遗传距离(D),绘制39份材料的Neighbor-Joining聚类图。当D为0.15时可以将所有供试材料分为7类,相同地理来源或遗传背景的品种基本上聚为一类。例如,第I和第II类群主要聚类了浙江省的品种,第IV和第V类群分别聚类了贵州和云南的品种。从遗传背景来看,中茶125、中茶126和中茶128都是从龙井43的杂交后代中选育而来,黄金斑和金玉缘分别为黄金芽自然芽变后代培育获得,云抗14号和云抗10号均选自南糯山群体,它们分别聚在了一起。
3 讨论
3.1 SSR引物的多态性
利用分子标记构建指纹图谱的原则之一就是利用尽量少的引物分开尽量多的品种,以降低成本,提高检测效率,这对所用标记的多态性有一定要求。段艳凤等[14]从138对SSR引物中筛选出6对多态性高、谱带清晰的引物,构建了88个马铃薯品种的指纹图谱,引物多态信息含量均大于0.7。罗忠霞等[15]从342对SSR引物中筛选出了16对SSR核心引物,其平均多态信息含量达0.79,最终用2对引物即可鉴别所有52份甘薯种质资源。本研究所用的30个SSR标记是前期经筛选过的,扩增效果都比较清晰、稳定,大部分样品获得了1~2条谱带,平均多态信息含量为0.51。多态性较低的标记(PIC<0.25)有3个;多态性适中(0.25<PIC<0.5)的有9个;多态性高(PIC>0.5)的达18个,占所有引物总数的60%。上述结果说明,本研究选用的30个SSR标记多态信息含量丰富,在品种鉴定方面具有较高的应用潜力。
3.2 DNA指纹图谱的构建
核心标记组合法在构建作物DNA指纹图谱时十分有效。韩宗福等[16]从63对SSR引物中筛选出5对引物的组合,构建了黄河流域棉花主栽品种的分子指纹图谱。陆徐忠等[5]从前人研究的基础上筛选出一套12对SSR核心引物(每条染色体1对)对127份水稻品种进行了分子指纹分析。在茶树DNA指纹图谱构建中,Ujihara等[17]、杨阳等[18]、刘本英等[19]、王让剑等[20]进一步验证了核心SSR引物组合的适用性。本研究筛选出TM444、TM548、TM259和TM566等4对核心引物可区分开所有的品种。这4对核心引物单独可鉴定的品种数分别是13、11、9、7。根据刘峰等[21]提出的核心引物理论,可区分的最大品种数N=G1×……×Gn,Gn为n个引物在参试品种中检测到的基因型。这4对核心引物组合理论上可鉴定的最大品种数为9009,理论上可以满足现有茶树PVP申请品种鉴定的需要。
3.3 DUS测试中近似品种选择
在DUS测试中,需要将申请品种与近似品种进行多方面比较,当两个品种存在明显和稳定的差异时,申请品种才具有特异性。因此,近似品种的选择至关重要,必须以科学、严谨的方法筛选。系谱真实反映了品种之间的亲缘关系,具有相同遗传背景的品种,在表型性状和SSR扩增电泳图谱上均具有相似性,因此,近似品种的选择可以参考亲缘关系分析[22]。本研究中,遗传背景相似的品种大多聚在一起。例如黄金斑和金玉缘都是由黄金芽的自然突变单株选育而来,二者遗传距离在所有参试品种中最小,只有0.03。有的品种与父母本中的一个亲本聚在一起,而与另一个亲本则相距甚远。如中茶126为毛蟹与龙井43的杂交后代,它与毛蟹分在了不同类群,而与龙井43聚在一起。综上所述,SSR标记应用于茶树DUS测试近似品种辅助选择是可行的。
本研究通过30对SSR引物对26个茶树PVP申请品种及其13个近似品种和亲本进行分子鉴定和系谱关系分析,30对SSR引物中60%的引物多态性高,聚类分析将地理来源一致或遗传背景相似的材料基本上聚为一类。通过4对核心引物的组合可以清晰地区分所有39个参试品种,并依此构建了SSR指纹图谱,为茶树PVP申请品种鉴定、DUS测试和知识产权保护等方面提供一定的理论依据。
[1] 黄丹娟,马建强,陈亮. 茶树DNA分子指纹图谱研究进展 [J]. 茶叶科学, 2015, 35(6): 513-519.
[2] 吴渝生, 杨文鹏, 郑用琏. 3个玉米杂交种和亲本SSR指纹图谱的构建[J]. 作物学报, 2003, 29(4): 496-500.
[3] 张金渝, 张建华, 杨晓洪, 等. 玉米DUS 测试标准品种的SSR分子指纹图谱的构建[J]. 玉米科学, 2006, 14(4): 47-52.
[4] 李根英, Dreisigacker S, Warburton M L, 等. 小麦指纹图谱数据库的建立及SSR分子标记试剂盒的研发[J]. 作物学报, 2006, 32(12): 1771-1778.
[5] 张晗, 姚凤霞, 刘永杰, 等. EST-SSR 标记在冬小麦品种DUS 测试中的应用[J]. 麦类作物学报, 2010, 30(5): 801-806.
[6] 陆徐忠, 倪金龙, 李莉, 等. 利用SSR分子指纹和商品信息构建水稻品种身份证[J]. 作物学报, 2014, 40(5): 823-829.
[7] 唐浩, 余汉勇, 张新明, 等. 水稻新品种测试的标准品种DNA指纹图谱多样性分析[J]. 植物遗传资源学报, 2015, 16(1): 100-106.
[8] Dellaporta S L, Wood J, Hicks J B. A plant DNA mini preparation: version II [J]. Plant molecular biology reporter, 1983(1): 19-21.
[9] 马建强. 茶树高密度遗传图谱的构建及重要性状QTL定位[D]. 北京: 中国农业科学院, 2013: 88-104.
[10] Charters Y M, Wilkinson M J. Theuseofself-pollinated progeniesas ‘in-groups’ for the genetic characterization of cocoa germplasm [J]. Theoretical and applied genetics, 2000, 100: 160-166.
[11] Yeh F C, Yang R C, Boyle T B,et al. POPGENE, the user-friendly shareware for population genetic analysis [R]. Edmonton: Molecular biology and biotechnology centre, University of Alberta, 1997: 112-118.
[12] Liu K, Muse S V. PowerMarker an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 2005, 21: 2128-2129.
[13] Takezaki H, Nei M. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA [J]. Genetics, 1996, 144: 389-399.
[14] 段艳凤, 刘杰, 卞春松, 等. 中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析[J]. 作物学报, 2009, 35(8): 1451-1457.
[15] 罗忠霞, 房伯平, 李茹, 等. 基于EST-SSR标记的甘薯种质资源DNA指纹图谱构建[J]. 植物遗传资源学报, 2014, 15( 4): 810-814.
[16] 韩宗福, 王景会, 申贵芳, 等. 黄河流域棉花主要品种SSR指纹图谱构建及遗传差异分析[J]. 棉花学报, 2011, 23(6): 545-551.
[17] Ujihara T, Ohta R, Hayashi N,et al. Identification of Japanese and Chinese green tea cultivars by using simple sequence repeat markers to encourage proper labeling [J]. Bioscience, biotechnology, and biochemistry, 2009, 73(1): 15-20.
[18] 杨阳, 刘振, 赵洋, 等. 湖南省主要茶树品种分子指纹图谱的构建[J]. 茶叶科学, 2010, 30(5): 367-373.
[19] 刘本英, 孙雪梅, 李友勇, 等. 基于EST-SSR标记的云南无性系茶树良种遗传多样性分析及指纹图谱构建[J]. 茶叶科学, 2012, 32(3): 261-268.
[20] 王让剑, 杨军, 孔祥瑞, 等. 福建部分茶树品种SSR遗传差异分析与指纹图谱建立[C]//中国茶叶学会. 第十六届中国科协年会——分12茶学青年科学家论坛论文集. 昆明: 中国科学技术协会出版, 2014: 22.
[21] 刘峰, 冯雪梅, 钟文, 等. 适合棉花品种鉴定的SSR核心引物的筛选[J]. 分子植物育种, 2009, 6(7): 1160-1168.
[22] 赖运平, 张浙峰, 王丽容, 等. 利用SSR标记筛选DUS测试中甘蓝型油菜近似品种[J]. 分子植物育种, 2013, 11(2): 174-184
.
SSR Identification and Pedigree Analysis of PVP Application Cultivars in Tea Plant
HUANG Danjuan, MA Jianqiang, CHEN Liang*
Tea Research Institute of the Chinese Academy of Agricultural Sciences, National Center for Tea Improvement, Key Laboratory of Tea Biology and Resource Utilization, Ministry of Agriculture, Hangzhou 310008, China
Thirty SSR markers were used in this study for molecular identification and pedigree analysis of 26 PVP application tea cultivars, and 13 similar cultivars and parents, for probing the possible application of PVP and DUS testing using SSR markers in tea plant. A total of 131 alleles were detected, the number of alleles detected by each SSR marker ranged from 3 to 7, with a mean of 4.4. The average values of Shannon index and polymorphism information content were 1.04 and 0.51, respectively. The genetic distance ranged from 0.03 to 0.70 between 39 cultivars. When the genetic distance was 0.15, they could be classified into 7 groups. Tea cultivars from the same province and genetic background were clustered into one group to some extent. The identification ability of the 30 SSR markers was quite different, each marker could identify 3-16 cultivars. The 39 tea cultivars could be clearly distinguished by 4 core primers which were used to construct the molecular fingerprinting of all cultivars.
tea cultivars, PVP, SSR markers, fingerprinting
S571.1;Q52
A
1000-369X(2016)01-068-09
2015-08-12
2015-09-14
国家茶叶产业技术项目(CARS-023)、中国农业科学院科技创新工程(CAAS-ASTIP-2014-TRICAAS)、浙江省农业新品种选育重大科技专项(2012C12905)资助。
黄丹娟,女,硕士研究生,主要从事茶树资源育种研究。*通讯作者:liangchen@tricass.com
