枯草芽孢杆菌QM11漆酶基因克隆表达及序列分析
2016-09-16哈尔滨商业大学食品工程学院黑龙江哈尔滨150076
由 庆,徐 伟(哈尔滨商业大学食品工程学院,黑龙江哈尔滨150076)
枯草芽孢杆菌QM11漆酶基因克隆表达及序列分析
由 庆,徐 伟*
(哈尔滨商业大学食品工程学院,黑龙江哈尔滨150076)
为获得高效表达的产漆酶工程菌,将枯草芽孢杆菌(Bacillus subtilis)的漆酶(cotA)基因在大肠杆菌中表达。用PCR的方法从枯草芽孢杆菌(Bacillus subtilis)QM11基因组中扩增cotA基因,连接载体pET22b构建成表达载体pET22b-cotA,转化至E.coli BL21(DE3)进行诱导表达,最终通过SDS-PAGE检测蛋白表达情况。克隆得到的cotA基因含有1542个核苷酸,由513个氨基酸组成,预测相对分子量为58 ku,理论等电点为5.91,无信号肽,二级结构以β-折叠和无规卷曲为主,其中β-折叠占26.71%,无规卷曲占52.05%,与NCBI已公布的Bacillus subtilis漆酶cotA基因(GenBank:GQ184294.1)碱基序列有12个碱基的差异,其编码的氨基酸序列有4个发生了改变。通过SDS-PAGE检测,目标蛋白相对分子质量约为54 ku,与预测相对分子量基本相符。
枯草芽孢杆菌,漆酶,基因克隆,序列分析,表达载体构建,诱导表达
漆酶(EC 1.10.3.2)是一种含铜的多酚氧化酶,能够催化多种酚类化合物氧化,同时将分子氧还原为水[1],广泛应用于造纸及食品工业等。秸秆是我国主要的农业废弃物[2],主要由木质素、纤维素和半纤维素组成,秸秆降解的关键就是处理包裹在纤维素和半纤维素外面的木质素[3]。木质素是由带有苯环结构的单体交联组成的三维高聚物,漆酶可以有效的降解木质素。
在自然界中能够降解木质素并产生相应酶类的生物只占少数,主要是真菌和细菌[4-6]。由于与真菌漆酶结构不同,细菌漆酶无需糖基化、热稳定性高、可耐受更高的pH及Cu2+抗性[7]等特点比真菌漆酶更有优势。Suzuki等[8]发现一种来源于细菌的漆酶可在70℃条件下保温20 min依然保持较高的酶活。Ruijssenaars等[9]也发现了一种最适pH在7.5~8的细菌漆酶。
本研究以从森林土壤中分离得到的高产漆酶细菌枯草芽孢杆菌QM11为研究对象,通过设计特异性引物扩增出cotA基因,进行序列分析,通过构建pET22bcotA重组表达载体,将其转入E.coli BL21(DE3)菌株中,以期实现重组漆酶cotA的高效表达,提高漆酶cotA的比活力,进一步增加细菌漆酶的产量,提高秸秆的降解效率,为实现农业废弃物资源化奠定基础。
1 材料与方法
1.1 材料与仪器
大肠杆菌E.coli BL21(DE3)感受态细胞 购自北京全式金生物技术有限公司;克隆载体pMD18-T购自大连宝生物工程有限公司;表达载体pET22b 购自于美国Novagen公司;枯草芽孢杆菌(Bacillus subtilis)QM11 本实验室保藏;限制性内切酶 购自美国NEB公司;Taq DNA聚合酶、T4 DNA连接酶购自北京全式金生物技术有限公司;Ezup柱式细菌基因组DNA抽提试剂盒 购自上海生工生物工程股份有限公司;质粒小提试剂盒、普通琼脂糖凝胶DNA回收试剂盒 购自北京天根生化科技有限公司;其他试剂 均为国产分析纯。
PCR仪 美国BIO-RAD公司;凝胶成像系统 美国SIM公司;电泳仪和电泳槽 北京六一仪器厂。
1.2 实验方法
1.2.1 QM11基因组DNA的提取 QM11基因组DNA提取按照Ezup柱式细菌基因组DNA抽提试剂盒说明书操作。采用1.0%琼脂糖凝胶电泳,121 V电压,20 min,检验完整性并采用凝胶成像系统观察并拍照,总DNA保存于-80℃备用。
1.2.2 引物设计 根据NCBI中已公布的枯草芽孢杆菌cotA基因序列(GenBank登录号GQ184294),用Primer 5.0软件设计引物QM-F 5'-CGGGATCCATGA CACTTGAAAAATTTGTGGATGC-3';QM-R 5'-ATA AGAATGCGGCCGCTTTATGGGGATCAGTTATATCC ATCG-3',并在其上下游分别引入酶切位点BamHⅠ和NotⅠ(下划线部分所示),上下游引物由上海生工生物工程有限公司合成。
1.2.3 QM11 cotA基因扩增 PCR反应采用50 μL体系,模版DNA 1 μL、10 μmol/L QM-F及QM-R各1 μL、10×PCR Buffer 5 μL、25 mmol/L MgCl23μL、dNTP mixture 3 μL、Taq DNA聚合酶(5 U/μL)1 μL、ddH2O补足至50 μL。PCR反应参数:95℃5 min;95℃30 s,55℃30 s,72℃2 min,30个循环;72℃10 min。扩增产物用1%的琼脂糖凝胶电泳检测,切胶纯化目的条带,PCR产物纯化后与pMD18-T载体连接,转化JM109感受态细胞,蓝白斑筛选出阳性克隆,挑取阳性克隆进行测序,基因序列由上海生工生物工程公司进行测定。
1.2.4 QM11 cotA基因的生物信息学分析 测序结果采用Vector NTI 10.0进行序列比对分析。相对分子量与理论等电点预测采用ExPASy在线服务器的Compute PI/Mw工具(http://web.expasy.org/compute_pi/)。采用在线软件SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽序列的预测。采用http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page= npsa_sopm.html预测蛋白质的二级结构,采用SWISSS MODEL(http://swissmodel.expasy.org/)进行蛋白质结构三维建模。
1.2.5 重组表达载体的构建及重组子的筛选 将pMD18-cotA重组质粒与表达质粒pET22b分别用BamHⅠ和NotⅠ双酶切,经1%琼脂糖凝胶电泳,121 V,20 min,回收目的片段,将目的基因的酶切片段与酶切处理后的表达质粒pET22b进行连接。将连接产物转入E.coli BL21(DE3),在含有氨苄青霉素(50 μg/mL)的LB抗性平板上37℃培养过夜,挑取单克隆至LB液体培养基中,37℃150 r/min摇菌过夜,并进行菌落PCR验证。
1.2.6 重组载体在大肠杆菌中的诱导表达 挑取正确的转化子过夜活化后,以1%的接种量转接至氨苄青霉素(50 mg/mL)的LB液体培养基中,37℃、150 r/min摇床培养,当培养至OD600达到0.6时,加入IPTG至终浓度1 mmol/L开始诱导,于诱导的1、2、3、4 h取样,同时未诱导的作为对照。
1.2.7 重组蛋白的SDS-PAGE 收集诱导表达后的菌液,12000 r/min离心10 min,沉淀加入5 mL PBS溶液,进行超声破碎。破碎后的溶液12000 r/min离心10 min,取上清进行SDS-PAGE电泳,浓缩胶浓度为5%,分离胶浓度为12%。
2 结果与分析
2.1 QM11总DNA提取及目的基因的扩增

图1 QM11基因组DNA提取结果Fig.1 Electrophoresis of total DNA extracted from QM11 cotA

图2 基因组DNA扩增结果Fig.2 Electrophoretic profile of PCR products of cotA
如图1所示,QM11基因组DNA经1%琼脂糖凝胶电泳可知,条带单一,清晰可见,说明DNA完整性较好。以提取的基因组DNA为模版,利用QM-F及QM-R进行PCR扩增,得到1500 bp左右的一条带,如图2所示,大小与预期结果相符。
2.2 cotA基因序列的测定及分析
将扩增获得的cotA基因进行TA克隆,获得重组的pMD18-T-cotA,转化JM109后,蓝白斑筛选后,将重组子送于上海生工生物工程有限公司进行测序。
将测得的序列进行生物信息学分析结果表明,cotA基因含1542个核苷酸,其中GC含量为48.02%,编码513个氨基酸组成(图3),相对分子量为58436.07,理论等电点为5.91。在线软件SignalP 4.1 Server分析结果表明cotA基因无信号肽。将推导的氨基酸序列提交http://blast.ncbi.nlm.nih.gov/Blast.cgi进行同源比对,结果显示该序列与Bacillus sp.WN01和Bacillus sp.LS02等cotA基因同源性较高,均达到99%。漆酶cotA碱基序列与已公布的Bacillus subtilis漆酶基因(NCBI GenBank:GQ184294.1)碱基序列有12个碱基的差异,其编码的氨基酸序列有4个发生了改变。

图3 cotA基因序列及对应的氨基酸序列Fig.3 DNA sequence of cotA gene and deduced amino acid sequence of cotA protein
2.3 cotA蛋白二级结构分析
采用SOPM二级结构预测程序(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopm.pl)进行cotA蛋白二级结构预测。由图4可知,α-螺旋占14.04%(72个),β-折叠为26.71%(137个),β-转角为7.21%(37个),无规则卷曲为52.05%(267个)。
2.4 cotA三维结构建模

图4 cotA蛋白二级结构预测Fig.4 Deduced secondary structure of cotA protein
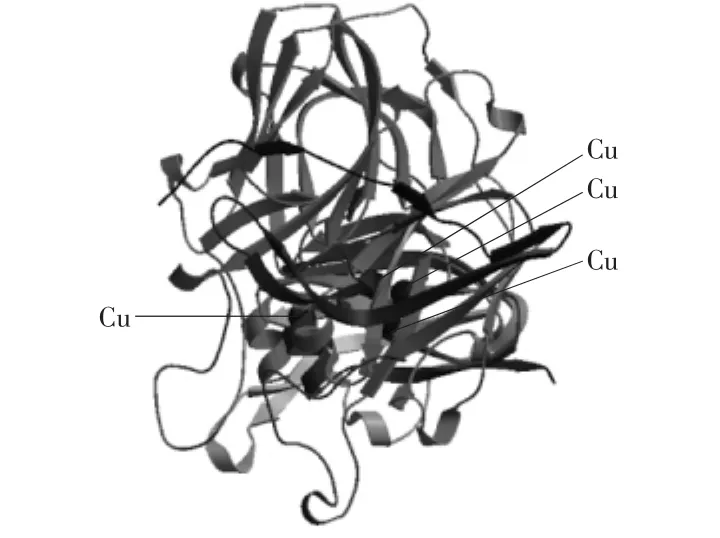
图5 cotA蛋白的三级结构Fig.5 Deduced 3-D structure of cotA protein
进入Swiss-Model三维结构预测服务器,提交cotA基因编码的氨基酸序列,所得结果见图5。cotA蛋白三级结构模型的建立是以4akq.1.A为模板,一致性达到99.22%,QMEAN4值为0.45。观察cotA蛋白三级结构,图5中的4个黑色球为蛋白中含有的4个铜原子,具有由3个杯状结构域紧密组成的漆酶的球状构象。
2.5 原核表达载体的构建及重组子的筛选
将pMD18T-cotA和载体pET22b分别用BamHⅠ、NotⅠ进行双酶切,回收目的基因片段和载体片段,用T4 DNA连接酶进行连接,将pET22b-cotA表达载体转入感受态的E.coli BL21宿主菌,涂布于含氨苄青霉素的LB平板,挑取8个阳性克隆,进行菌落PCR鉴定,经菌落PCR均扩增出一段1500 bp左右的片段(图6),证明为正确的转化子。

图6 重组子鉴定结果Fig.6 Identification of constructed plasmid
2.6 cotA融合蛋白的原核表达
分别将未诱导和诱导过夜的E.coli BL21的总蛋白进行SDS-PAGE蛋白电泳,重组子经IPTG诱导后,与对照比较,1、2、3、4 h后在54 ku处均有明显条带(图7)。该条带所示的蛋白分子量与理论值相符,表明成功表达了cotA蛋白。

图7 表达产物SDS-PAGE分析Fig.7 SDS-PAGE profile of expressed products
3 结论
漆酶是一种含铜的多酚氧化酶,能够催化多种酚类物质的氧化,过去认为漆酶主要存在于植物以及真菌中,且研究主要集中在真菌漆酶上,而关于细菌漆酶的研究在近几年才逐渐开展。
本研究从Bacillus subtilis QM11中克隆得到了漆酶基因cotA,以基因组为模板,PCR扩增出的核酸序列全长为1542个核苷酸,编码513个氨基酸组成,预测相对分子量为58436.07,理论等电点为5.91,无信号肽,cotA蛋白二级结构预测:其中α-螺旋占14.04% (72个),β-折叠为26.71%(137个),β-转角为7.21% (37个),无规卷曲为52.05%(267个)。经过BLAST比对,该序列与Bacillus sp.WN01和Bacillus sp.LS02等cotA基因同源性较高,均达到99%。漆酶cotA碱基序列与已公布的Bacillus subtilis漆酶基因(NCBI GenBank:GQ184294.1)碱基序列有12个碱基的差异,其编码的氨基酸序列有4个发生了改变。将Bacillus subtilis的漆酶基因克隆到表达载体pET22b,转化E.coli BL21(DE3),通过含有Amp的LB平板筛选单克隆,使用IPTG进行诱导表达,最终通过SDS-PAGE进行检测,表达产物分子量约为54 ku,与预测分子量基本一致。为获得高效表达的产漆酶工程菌以及提高漆酶产量及酶活力奠定了基础。
[1]K Nakamura.Function and molecular evolution of multicopper blue proteins[J].Cell Mol Life Sci,2005,62:2050-2066.
[2]王勇,孟晓林.秸秆废弃物的生物学特性及其开发利用[J].山西农业科学,2009,37(2):42-44.
[3]Gai YP,Zhang WT,Mu ZM,et al.Involvement of ligninolytic enzymes in degradation of wheat straw by Trametes trogii[J].J Appl Microbiol,2014,117(1):85-95.
[4]丛立新.白腐真菌生物降解秸秆及饲料化的研究进展[J].饲料研究,2014,7:11-13.
[5]郑苗苗,邵淑丽,张东向,等.红平菇漆酶基因异源表达及对染料脱色的研究[J].纺织学报,2014,35(12):84-89.
[6]李红亚,李术娜,王树香,等.产芽孢木质素降解菌MN-8的筛选及其对木质素的降解[J].中国农业科学,2014,47(2):324-333.
[7]Hullo MF,Moszer I,Danchin A,et al.CotA of Bacillus subtilis is a copper-dependent laccase[J].J Bacteriol,2001,183(18):5426-5430.
[8]Suzuki T,Endo K,Ito M,et al.A thermostable laccase from Streptomyces lavendulae REN-7:purification,characterization,nucleotidesequence,andexpression[J].Biosci Biotechnol Biochem,2003,67(10):2167-2175.
[9]Ruijssenaars HJ,Hartmans S.A cloned Bacillus halodurans multicopper oxidase exhibiting alkaline laccase activity[J].Appl Microbiol Biotechnol,2004,65(2):177-182.
Cloning,expression and characterization of laccase from Bacillus subtilis
YOU Qing,XU Wei*
(College of Food Engineering,Harbin University of Commerce,Harbin 150076,China)
To express laccase cotA gene from Bacillus subtilis in Escherichia coli expression system,cotA gene was cloned from Bacillus subtilis QM11 genome DNA by PCR.Plasmid pET22b containing cotA gene was constructed and transferred into competent Escherichia coli BL21(DE3)and the expression of recombinant protein was proved by SDS-PAGE.The gene cotA contained 1542 nucleotides,comprising one open reading frame encoding a polypeptide of 513 amino acids with 58 ku of relative molecular weight and predicted pI value of 5.91 and there was no signal peptide in cotA.The secondary structure of cotA was mainly β-sheet and random coil,β-sheet was 26.71%and random coil was 52.05%.There were twelve-site mutations between the nucleotide sequence from Bacillus subtilis QM11 and cotA(NCBI GenBank:GQ184294.1)from Bacillus subtilis,which could result in four amino acid substitution.The molecular weight of cotA gene protein was estimated to be 54 ku by SDS-PAGE.
Bacillus subtilis;laccase;gene cloning;sequence analysis;construction of expression vector;induced expression
TS201.1
A
1002-0306(2016)06-0225-04
10.13386/j.issn1002-0306.2016.06.038
2015-08-19
由庆(1990-),男,在读硕士研究生,研究方向:微生物学与发酵工程,E-mail:youq_will@126.com。
徐伟(1963-),女,博士,教授,研究方向:微生物学与发酵工程,E-mail:xuw@hrbcu.edu.cn。
哈尔滨商业大学研究生创新科研项目(JYSCX2014-333HSD)。
