四川泡豇豆原料表皮附生乳酸菌抗生素抗性及抗性基因污染分析
2016-09-14徐顾榕宋菲菲向文良西华大学食品与生物工程学院四川省食品生物技术重点实验室西华大学古法发酵酿造生物技术研究所四川成都610039
徐顾榕,蔡 婷,林 凯,宋菲菲,张 庆,向文良(西华大学食品与生物工程学院,四川省食品生物技术重点实验室,西华大学古法发酵(酿造)生物技术研究所,四川成都610039)
四川泡豇豆原料表皮附生乳酸菌抗生素抗性及抗性基因污染分析
徐顾榕,蔡 婷,林 凯,宋菲菲,张 庆,向文良*
(西华大学食品与生物工程学院,四川省食品生物技术重点实验室,西华大学古法发酵(酿造)生物技术研究所,四川成都610039)
以鲜豇豆为研究对象,利用生理生化特征、16S rRNA、抗生素抗性和抗性基因检测分析其表皮附生乳酸菌对氯霉素(CHL)、四环素(TET)和氨苄青霉素(AMP)的抗性与抗性基因。结果表明:从鲜豇豆表皮共分离得到182株乳酸菌,分别属于Weissella confuse(50.0%)、Weissella cibaria(15.4%)、Enterococcus sulfurous(7.1%)和Lactococcus lactis subsp.lactis(27.5%)。32株(17.6%)乳酸菌对CHL和TET有抗性,其中,6株W.confuse(3.3%)和4株L.lactis subsp.Lactis (2.1%)对CHL单一抗性,4株W.confuse(2.1%)和5株L.lactis subsp.Lactis(2.7%)对TET单一抗性,6株W.confuse (3.2%)、2株W.cibaria(1.1%)、1株E.sulfurous(0.5%)和4株L.lactis subsp.Lactis(2.1%)对CHL和TET二重抗性;编码外排泵基因efrA、efrB、norB、norC、norE和sugE中,efrB的检出率最高,达31.3%,efrB和sugE的检出率最低,为3.1%。TET被检抗性基因tet(A)、tet(B)、tet(C)、tet(D)、tet(H)和tet(L)中,tet(C)的检出率最高,达90.9%,tet(B)的检出率最低,为4.5%。
豇豆,附生乳酸菌,抗生素抗性,抗生素抗性基因
抗生素抗性基因是一种新型的环境污染物,在环境介质中的持久残留以及在不同宿主间的水平转移往往比抗生素本身危害更大,因此其对公共健康和食品安全构成了威胁,目前已成为研究的热点[1-3]。
研究表明,从抗生素发现的20世纪30年代至20世纪90年代,土壤微生物对常用抗生素的抗性明显增加,极易污染食物资源和食品加工环境[4-5]。迄今为止,已有16种四环素类抗性基因、3种磺胺类抗性基因、10种β-内酰胺类抗性基因在河流、海洋、土壤、空气等环境中被发现[2,6-7]。随着环境抗生素抗性基因污染的加剧,中国传统发酵蔬菜产品中也发现了可转移的抗性基因,为中国传统发酵蔬菜的食品安全埋下了隐患。
附着在鲜豇豆表皮的乳酸菌是泡豇豆自然发酵微生物的主要来源。长期以来,发酵食品中的乳酸菌被认为是安全的,因此乳酸菌对抗生素的抗性常被忽视。但近年来研究发现,发酵食品中使用的乳酸菌对抗生素也产生了抗性,并有上升趋势。豇豆在种植过程中不可避免会污染环境中的抗性基因,尽管其污染浓度很低,但一旦它们进入发酵系统,就可能向其他微生物水平转移,给泡豇豆的安全埋下隐患。因此,本研究以新鲜豇豆为原料,以常用农用抗生素为筛选因子,分析其表皮附生的抗生素抗性乳酸菌,评估其抗性,并检测抗性基因,以期为传统发酵食品四川泡豇豆生产中的潜在安全风险控制提供参考。
1 材料与方法
1.1 材料与仪器
豇豆 四川某泡菜企业的鲜豇豆10 kg,产地分布在西南和东南省份;MRS肉汤 杭州微生物试剂有限公司;细菌基因组DNA提取试剂盒 天根生化科技有限公司;DH5α感受态细胞 TaKaRa Biotechnology(Dalian);pGM-T试剂盒、MarkerⅦ 天根生化科技有限公司;氯霉素、四环素和氨苄青霉素 北京索莱宝科技有限公司,纯度为98%;NEST Biotech Co.,Ltd.,96孔板 无锡耐思生物科技有限公司。
TB-214电子天平 北京赛多利斯仪器有限公司;Allegra X-15R冷冻离心机 贝克曼库尔特公司;720BR/01492电泳凝胶成像分析系统 BIO-RAD公司;Biometra T1 PCR仪 BIO-RAD公司;SGSP-02恒温培养箱 黄石恒丰器械有限公司;DYY-8C型电泳仪 北京六一仪器厂;Tecan Infinite F200型多功能酶标仪 上海闪谱生物科技有限公司。
1.2 实验方法
1.2.1 乳酸菌的分离、纯化与生化特征分析 随机选取豇豆25 g,加入225 mL无菌生理盐水的无菌均质袋中,用拍击式均质器Bagmixer均质1 min。取适量均质液用无菌生理盐水10倍梯度稀释后涂布于改良MRS琼脂培养基,37℃倒置培养48 h,挑取有明显溶钙圈的菌落于MRS琼脂培养基上划线纯化3次,得到纯菌落。分离得到的所有菌株的生化特征按《乳酸细菌分类鉴定及实验方法》中的鉴定方法进行[8]。
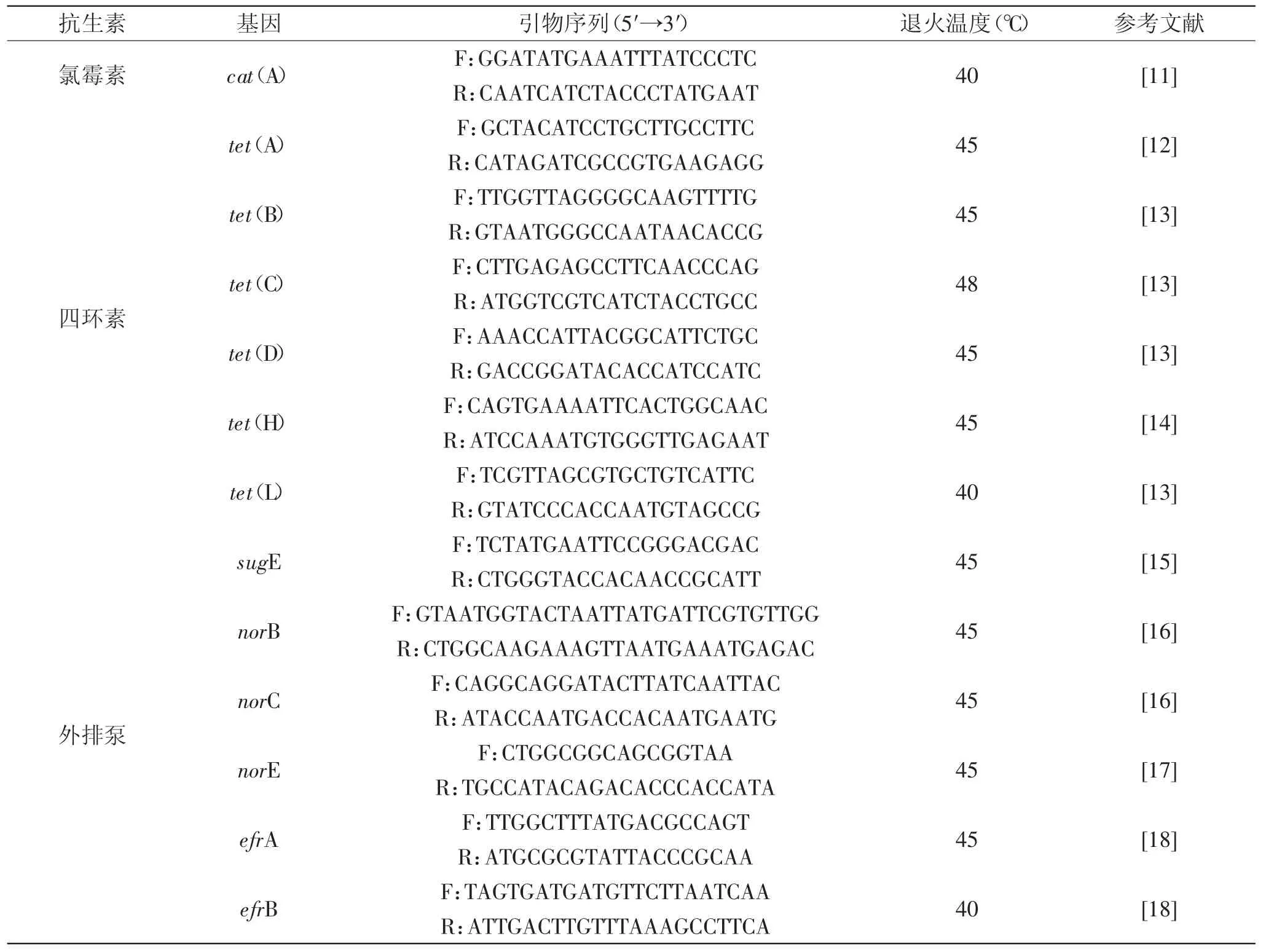
表1 被检抗生素耐药基因的PCR引物序列和退火温度Table1 The PCR primer and anneal temperature of antibiotic resistance genes in current study
1.2.2 16S rRNA序列分析 根据Xiang等[9]的方法提取分离得到的所有菌株DNA。以细菌DNA为模板,细菌通用引物Eu27F(5'-AGAGTTTGATCCTGGCTCAG-3')和原核微生物特异性引物1490R(5'-GGTTACCT TGTTACGACTT-3')进行16S rRNA扩增。PCR扩增条件:95℃预变性5 min,然后95℃变性1 min、50℃退火1 min、72℃延伸2 min,30个周期,最后72℃保持10 min。PCR产物连接到pGM-T载体后克隆入感受态细胞E.coli DH5α中,筛选阳性克隆子,提取重组质粒对16S rRNA测序,无嵌合体的序列与RDP数据库中的模式菌株进行相似性比较,确定菌株的分类地位。
1.2.3 抗生素抗性分析 依据欧洲微生物药物敏感委员会(http://www.eucast.org)关于微生物对抗生素的敏感阈值X(http://www.eucast.org/mic_distributions/),分析菌株的抗生素抗性。当分离菌株的最小抑菌质量浓度(Minimal Inhibitory Concentration,MIC)≤X μg/mL时,为敏感菌株;反之,则为抗性菌株。菌株Enterococcus faecalis ATCC 29212作为质控菌株。
最小抑菌浓度测定采用微量肉汤稀释法[10]。分别将CHL、TET和AMP配制成2048 μg/mL的贮存液,MRS液体培养基2倍梯度稀释成使用液。CHL、TET和AMP的梯度稀释浓度为1~512 μg/mL。向96孔板中加入198 μL含不同浓度抗生素的MRS液体培养基后,接种2 μL菌株的培养液,37℃静止培养24 h后,统计不同菌株的MIC。每组重复3次,并设置对照组。
1.2.4 抗生素抗性基因分析 PCR检测菌株的氯霉素抗性基因cat(A)[11];四环素抗性基因tet(A)、tet(B)、tet(C)、tet(D)、tet(H)和tet(L)[12-14];编码外排泵基因efrA、efrB、norB、norC、norE和sugE[15-18]。PCR扩增体系:PCR mixture 25 μL,ddH2O 22 μL,DNA模板1 μL,引物各为1 μL;PCR程序:95℃预变性5 min,95℃变性30 s,待检基因引物的退火温度退火30 s,72℃延伸2 min,30个循环后72℃延伸5 min。引物及退火温度见表1。1.0%的琼脂糖电泳检测扩增产物后,连接到pGM-T载体后,克隆入感受态E.coli DH5α中,筛选阳性克隆子测序,测序结果在NCBI中利用BlastX程序比对扩增序列,进一步判断分离菌株是否含有上述被检抗性基因。
1.3 数据处理
统计分离菌株抗性基因,含有该种抗性基因赋值为“1”、不含该种抗性基因赋值为“0”,构建分离菌株抗生素抗性基因表型的“0”和“1”的矩阵。利用NTSYS PC 2.11软件做UPGMA聚类分析[19-20],并以各分离菌株抗性基因构建点阵图,黑色图表示含有该种抗性基因,灰色图表示不含该种抗性基因。
2 结果与分析
2.1 乳酸菌的分离与生化特征
本研究从鲜豇豆表皮共分离得到182个溶钙圈菌落,根据糖和醇发酵实验结果,这些菌株被分成4个簇群,结果见表2。其中,菌株XC001代表的簇群最大,约占总菌落数的50.0%;菌株XC144代表的簇群最小,仅占7.1%。

表2 豇豆表皮附生乳酸菌的生理生化特征Table2 Physiological and biochemical characteristics of epibiotic lactic acid bacteria on the surface of the fresh cowpea
2.2 16S rRNA分析

表3 豇豆表皮附生乳酸菌的分类地位与群落构成Table3 The community structure and classify of epibiotic lactic acid bacteria on the surface of the fresh cowpea
为进一步确定乳酸菌的分类学地位,将分离得到的所有菌株进行16S rRNA序列分析,结果见表3。Stackebrandt等[21]认为:当16S rRNA序列相似性≥97%时可以认为是同一个属,序列相似性≥98%时则可以认为是同一个种。豇豆表皮附生乳酸菌的16S rRNA序列结果表明:其中,119株乳酸菌均属于Weissella属,XC001和XC108群中所分析菌株的16S rRNA序列分别与W.confuse JCM1093T和W.cibaria LMG17699T 的16S rRNA序列相似性为100%和99%,对糖或醇的利用也分别与各自簇群的相似菌株相同,因此XC001 和XC108分别被鉴定为W.confuse和W.cibaria,各占50.0%和15.4%。XC144代表的3株菌属于Enterococcus属,16S rRNA序列与E.sulfurous ATCC49903T的16S rRNA序列相似性为100%,在糖或醇的利用方面,XC144群与ATCC49903T一致(表3),因此被鉴定为E.sulfurous,占所有附生乳酸菌的7.1%。XC156代表的50株菌属于Lactococcus属,16S rRNA序列皆与L.lactis subsp.lactis NCDO604T的16S rRNA序列相似性为100%,这些菌株对糖或醇的利用与NCDO604T一致(表3),因此XC156群被鉴定为L.lactis subsp.lactis,占27.5%。
2.3 抗生素抗性分析
根据欧洲抗微生物药物敏感委员会对乳酸菌阈值X的规定,在182株乳酸菌株中,有32株乳酸菌具有抗性,约占总菌株数的17.6%(见表4)。其中CHL单一抗药菌株10株(5.5%),TET单一抗药菌株9株(5.0%),CHL和TET的二重抗药菌株13株(7.1%),无菌株表现出对AMP抗药性。在32株CHL和TET的单一或二重抗药菌株中,同种的不同菌株对CHL或TET的MIC值不同,表现出抗药多样性。其中W.confusa XC001对CHL和TET的抗药性最强,其对CHL和TET的MIC分别达到128 μg/mL(阈值X=4 μg/mL)和256 μg/mL(阈值X=8 μg/mL)。

表4 豇豆表皮附生的部分乳酸菌对CHL、TET和STR的MIC与抗药性(μg/mL)Table4 The CHL,TET and STR antibiotic resistance and MICs for epibiotic lactic acid bacteria on the surface of the fresh cowpea(μg/mL)
在91株W.confuse中,有6株菌(6.6%)表现出对CHL的单一抗药性,有4株菌(4.4%)表现出对TET的单一抗药性,有6株菌(6.6%)表现出对CHL和TET的二重抗药性;在28株W.cibaria中,有2株菌(7.1%)表现出对CHL和TET二重抗药;在13株E.sulfurous中,有1株菌(7.7%)表现出对CHL和TET的二重抗药性;在50株L.lactis subsp.lactis菌株中,有4株菌(8.0%)表现出对CHL的单一抗药性,有5株菌(10.0%)表现出对TET的单一抗药性,有4菌株(8.0%)表现出对CHL和TET的二重抗药性。
2.4 抗生素抗性基因分析
在32株CHL和TET的单一或二重抗药菌株中,除CHL单一抗药菌株L.lactis subsp.lactis XC172未被检出任何抗药基因外,其他抗药菌株都被检出1个或多个抗药基因,结果见图1。L.lactis subsp.lactis XC172未检出当前的CHL被检抗药基因和外排泵基因,表明XC172可能存在其他抗药基因。在32株CHL 和TET的单一或二重抗药菌株中,编码外排泵基因efrB的检出率最高,达31.3%,其次是norC,检出率为28.1%;efrA、norB、norE和sugE的检出率分别为12.5%、3.1%、18.8%和3.1%。在TET抗药菌株中,tet(C)的检出率最高,达90.9%,其次是tet(L),检出率为36.4%,检出率最低的是tet(B),为4.5%;tet(A)、tet(D)和tet(H)的检出率分别为22.7%、27.3%和13.6%。

图1 豇豆中CHL和TET抗药附生乳酸菌的CHL、TET和外排泵基因分布Fig.1 The CHL and TET resistance gene distribution of CHL,TET and efflux pump genes resistant epibiotic LABs on the fresh cowpea
菌株W.confuse XC012、XC096和XC081检测出携带的抗药性基因种类最多,分别包括3种TET基因、3种外排泵基因和4种TET基因、2种外排泵基因;菌株W.confuse XC043、W.confuse XC131、L.lactis subsp.lactis XC148、L.lactis subsp.lactis XC151、L.lactis subsp.lactis XC156、L.lactis subsp.lactis XC157、L.lactis subsp.lactis XC158、L.lactis subsp.lactis XC168和L.lactis subsp.lactis XC171都仅被检出1种 TET抗药基因。
在TET抗药W.confuse菌株中,在同种内的不同菌株被检出抗药基因的种内和数量不尽相同,在具有相同MIC的TET抗药W.confuse菌株中,被检出抗药基因的种内和数量也不尽相同(图1)。W.cibaria 和E.sulfureus中的不同抗药菌株对TET的抗药基因分布也表现出了与W.confuse类似的情况。上述结果表明:当前被检菌株中,同种内的不同菌株对TET的抗药性与相应抗药基因的种类和数量多少无相关性。
3 结论
传统意义上合格的蔬菜主要关注农药残留和重金属含量等这些理化指标,却很少关注其中的微生物指标,特别是微生物对抗生素抗性指标[1]。因此,当蔬菜表面存在抗性菌株的蔬菜作为四川泡菜原料时,将会给不经二次灭菌即可直接食用的四川泡菜带来潜在的安全风险。本研究从鲜豇豆表皮共分离得到182株乳酸菌,主要属于W.confuse(50.0%)、W.cibaria(15.4%)、E.sulfurous(7.1%)和L.lactis subsp.lactis(27.5%)。在182株乳酸菌株中,无AMP抗药菌株,有32株乳酸菌表现出了对CHL和TET的抗药性,约占总菌落数的17.6%。其中CHL单一抗药菌株10株(5.5%),TET单一抗药菌株9株(5.0%),CHL和TET的二重抗药菌株13株(7.1%)。在91株W.confuse中,6株CHL单一抗药,4株TET单一抗药性,6株CHL和TET二重抗药;在28株W.cibaria中,2株CHL和TET二重抗药;在13株E.sulfurous中,1株CHL和TET的二重抗药;在50株L.lactis subsp.lactis菌株中,4株CHL单一抗药,5株TET单一抗药,4株CHL和TET二重抗药。
抗生素抗性基因分析发现,除CHL单一抗药菌株L.lactis subsp.lactis XC172未检出任何抗药基因和外排泵基因外,其他31株CHL和TET单一或二重抗药菌株中,都有1个或多个相应抗药基因被检测。其中编码外排泵基因efrA、efrB、norB、norC、norE和sugE中,efrB的检出率最高,达31.3%;efrB和sugE的检出率最低,为3.1%。TET被检抗药基因tet(A)、tet(B)、tet(C)、tet(D)、tet(H)和tet(L)中,tet(C)的检出率最高,达90.9%;tet(B)的检出率最低,为4.5%。
四川泡豇豆原材料豇豆中抗生素抗药菌的发现为四川泡菜的原料安全敲响了警钟。因此,对于用于四川泡豇豆的豇豆原料,需建立抗生素抗药性附生乳酸菌的检测与防控体系,确保未经二次灭菌即可直接食用的四川泡菜的食品安全,维护广大消费者的健康权益。
[1]Martínez J L.Antibiotics and antibiotic resistance genes in natural environments[J].Science,2008,321(5887):365-367.
[2]Su J Q,Wei B,Xu C Y,et al.Functional metagenomic characterization of antibiotic resistance genes in agricultural soils from China[J].Environment International,2014,65:9-15.
[3]Sharma P,Tomar S K,Goswami P,et al.Antibiotic resistance among commercially available probiotics[J].Food Research International,2014,57:176-195.
[4]Campos J,Mourao J,Pestana N,et al.Microbiological quality of ready-to-eat salads:an underestimated vehicle of bacteria and clinically relevant antibiotic resistance genes[J].Int J Food Microbiol,2013,166(3):464-470.
[5]Fernandez-Fuentes M A,Abriouel H,Morente E O,et al.Genetic determinants of antimicrobial resistance in Gram posistiver bacteria from organic foods[J].Int J Food Microbiol,2014,172:49-56.
[6]Zhang X X,Zhang T,Fang H H.Antibiotic resistance genes in water environment[J].Appl Microbiol Biot,2009,82(3):397-414.
[7]Jiang L,Hu X L,Xu T,et al.Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources[J].Sci Total Environ,2013,458-460:267-272.
[8]凌代文.乳酸细菌分类鉴定及实验方法[M].北京:中国轻工业出版社,1999.
[9]Xiang W L,Liang H Z,Liu S,et al.Isolation and performance evaluation of halotolerant phosphate solubilizing bacteria from the rhizospheric soils of historic Dagong Brine Well in China[J].World Journal of Microbiology and Biotechnology,2011,27(11):2629-2637.
[10]Klare I,Konstabel C,Muller-Bertling S,et al.Evaluation of new broth media for microdilution antibiotic susceptibility testing of Lactobacilli,Pediococci,Lactococci,and Bifidobacteria[J].Applied and Environmental Microbiology,2005,71(12):8982-8986.
[11]Beatriz Rojo-Bezares,Yolanda S,Patricia P,et al.Assessment of antibiotic susceptibility within lactic acid bacteria strains isolated from wine[J].International Journal of Food Microbiology,2006,111:234-240.
[12]杨芳,陶然,杨扬,等.人工湿地中抗生素抗性大肠杆菌和抗性基因的去除与分布[J].环境工程学报,2013,7(6):2057-2062.
[13]Jia S Y,He X W,Bu Y Q,et al.Environmental fate of tetracycline resistance genes originating from swine feedlots in river water[J].Journal of Environmental Science and Health,2014,49(8):624-631.
[14]Aminov R I,Chee-Sanford J C,Garrigues N,et al.Development,validation,and application of PCR primers for detection of tetracycline efflux genes of gram-negative bacteria [J].Applied and Environmental Microbiology,2002,68(4):1786-1793.
[15]He G X,Zhang C,Crow R R,et al.SugE,a new member of the SMR family of transporters,contributes to antimicrobial resistance in Enterobacter cloacae[J].Antimicrobial Agents and Chemotherapy,2011,55(8):3954-3957.
[16]Patel D,Kosmidis C,Seo,et al.Ethidium bromide MIC screening for enhanced efflux pump gene expression or efflux activity in Staphylococcus aureus[J].Antimicrobial Agents and Chemotherapy,2010,54(12):5070-5073.
[17]Swick M C,Morgan-Linnell S K,Carlson K M,et al.Expression of multidrug efflux pump genes acrAB-tolC,mdfA,and norE in Escherichia coli clinical isolates as a function of fluoroquinolone and multidrug resistance[J].Antimicrobial Agents and Chemotherapy,2011,55(2):921-924.
[18]Lavilla L L,Benomar N,Sanchez V A,et al.Role of efrAB efflux pump in biocide tolerance and antibiotic resistance of Enterococcus faecalis and Enterococcus faecium isolated from traditional fermented foods and the effect of EDTA as efrAB inhibitor[J].Food Microbiology,2014(44):249-257.
[19]Cytryn E,Rijn J V,Schramm A,et al.Identification of bacteria potentially responsible for oxic and anoxic sulfide oxidation in biofilters of a recirculating mariculture system[J].Applied and Environmental Microbiology,2005,71(10):6134-6141.
[20]Nayak S,Prasanna R,Prasanna B M,et al.Analysing diversity among Indian isolates of Anabaena(Nostocales,Cyanophyta)using morphological,physiological and biochemical characters[J].World Journal of Microbiology Biotechnology,2007,23(11):1575-1584.
[21]Stackebrandt E,Goebel B M.Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic Bacteriology,1994,44(4):846-849.
Investigation of antibiotic resistance and resistant genes of epibiotic lactic acid bacteria on the surface of Sichuan pickled cowpea
XU Gu-rong,CAI Ting,LIN Kai,SONG Fei-fei,ZHANG Qing,XIANG Wen-liang*
(College of Food and Bioengineering,Xihua University,Institute of Ancient Brewing,Xihua Uniwersity,Chengdu 610039,China)
The epibiotic lactic acid bacteria(LABs)on the fresh cowpea,which resist to chloramphenicol(CHL)tetracycline(TET)and ampicillin(AMP),were investigated by the physiological and biochemical characteristics,16S rRNA,antibiotic resistance and resistant genes.182 isolates in current study were assigned to Weissella confuse(50.0%),Weissella cibaria(15.4%),Enterococcus sulfurous(7.1%)and Lactococcus lactis subsp.lactis (27.5%),respectively.And 32 isolates(17.6%)were found to be against CHL and TET with one or two resistance,including 6 W.confuse(3.3%)and 4 L.lactis subsp.lactis(2.1%)with resistance to CHL,and 4 W.confuse(2.1%)and 5 L.lactis subsp.lactis(2.7%)with resistance to TET.6 W.confuse(3.2%),2 W.cibaria (1.1%),1 E.sulfurous(0.5%)and 4 L.lactis subsp.lactis(2.1%)with resistances to CHL and TET.In the effux pump genes efrA,efrB,norB,norC,norE and sugE,efrB had the highest detection rate with 31.3%,but efrB and sugE with only 3.1%.Among the TET resistant genes tet(A),tet(B),tet(C),tet(D),tet(H)and tet(L),tet(C)had the highest detection rate,90.9%,and tet(B)was found with 4.5%lowest detection rate.
cowpea;epibiotic lactic acid bacteria;antibiotic resistance;antibiotic resistant genes
TS201.3
A
1002-0306(2016)02-0170-06
10.13386/j.issn1002-0306.2016.02.026
2015-07-02
徐顾榕(1992-),女,硕士研究生,研究方向:食品微生物分子生态,E-mail:xuluffy@sina.com。
*通讯作者:向文良(1973-),男,博士,教授,研究方向:中国西南地区特色发酵食品微生物过程学,E-mail:xwllm7687@sina.com。
国家自然科学基金(31571935);教育部春晖计划(Z2014061);四川省应用基础(2014JY0045);四川省教育厅重点(14ZA0110)。
