谷类食品中克罗诺杆菌的分离与鉴定
2016-09-12李远宏张逸飞张庆成邹小倩裴尚飞赵珉生
李远宏,张逸飞,张庆成,徐 湾,邹小倩,裴尚飞,赵珉生,姜 华
(1.徐州医科大学公共卫生学院,江苏徐州 221004;2.空军勤务学院航空军需系,江苏徐州 221000)
谷类食品中克罗诺杆菌的分离与鉴定
李远宏1,张逸飞1,张庆成2,徐湾1,邹小倩1,裴尚飞1,赵珉生1,姜华1
(1.徐州医科大学公共卫生学院,江苏徐州 221004;2.空军勤务学院航空军需系,江苏徐州 221000)
为了解谷类食品中克罗诺杆菌(Cronobacterspp.)的污染情况。本研究采集了100份谷类食品样品,采用国家标准GB 4789.40-2010分离克罗诺杆菌,并利用基于ITS和OmpA特异基因片段的PCR鉴定方法、16S rDNA基因序列分析等方法对可疑克罗诺杆菌进行了初步的鉴定。结果发现谷类食品中克罗诺杆菌的污染率为21.0%,并分离出21株可疑克罗诺杆菌,其中17株(占总数的81.0%)被初步鉴定为阪崎克罗诺杆菌(C.sakazakii),3株(占总数的14.2%)为都柏林克罗诺杆菌(C.dublinensis),1株(占总数的4.8%)可能为尤尼沃斯克罗诺杆菌(C.universalis)或苏黎世克罗诺杆菌(C.turicensis)。研究结果表明市售谷类食品中存在食源性克罗诺杆菌的污染,应该加强谷类食品中克罗诺杆菌的流行病学监测。
克罗诺杆菌,谷类食品,分离,鉴定
克罗诺杆菌(Cronobacterspp.),原阪崎肠杆菌(Enterobactersakazakii),是近年来婴幼儿配方奶粉中引起广泛关注的一种食源性条件致病菌。它是一种周生鞭毛、能运动、无芽孢的革兰阴性细菌,在一定条件下可引起人和动物致病[1]。近期的分类学研究结果表明该菌包括7个种:阪崎克罗诺杆菌(C.sakazakii),丙种二酸盐阳性克罗诺杆菌(C.malonaticus),苏黎世克罗诺杆菌(C.turicensis),都柏林克罗诺杆菌(C.dublinensis),穆汀斯克罗诺杆菌(C.muytjensis),康迪蒙提克罗诺杆菌(C.condimenti)和尤尼沃斯克罗诺杆菌(C.universalis)[2]。克罗诺杆菌主要通过婴幼儿配方奶粉感染新生儿及婴幼儿,特别是早产儿、低出生体重的婴幼儿,引起脑膜炎、菌血症和坏死性小肠结肠炎等疾病,死亡率高达20%~50%[3-4]。此外,研究表明该菌也能导致老年人和免疫力低下的人群致病[5]。
很多报道证实克罗诺杆菌主要的传染源和传播媒介是婴儿配方粉,然而在奶酪、肉制品、谷物制品、香肠、香辛料、蔬菜等各种食品中也分离出了克罗诺杆菌[6-9]。此外,克罗诺杆菌还广泛存在于食品生产企业的各种加工设备及生产环境中[10]。目前,不同学者对克罗诺杆菌的真正宿主、传播途径和致病机制还没达成统一的认识。一些学者认为婴幼儿配方奶粉及其加工环境、面粉及其生产环境可能是克罗诺杆菌的天然储藏场所,在克罗诺杆菌的传播过程中起重要作用[11-12]。
董晓晖等[2]对非奶粉类食品中克罗诺杆菌的污染情况做定量检测,并从冷冻面条中分离出了克罗诺杆菌,检出的克罗诺杆菌数量为0.62 MPN/g。张翼等[13]对婴幼儿食品中克罗诺杆菌分离株的生物被膜形成能力进行了研究,结果表明从婴儿米粉和婴儿面条等样品中分离的克罗诺杆菌均具有一定生物膜成膜能力。污染了克罗诺杆菌的谷类食品将可能导致新生儿及婴幼儿致病,但目前国内外尚未有较为全面的关于谷类食品中克罗诺杆菌污染情况的研究资料。进一步了解各类食品中克罗诺杆菌的分布特征和污染状况可为预防该菌引起的疾病的爆发、流行及追踪污染源提供理论依据,具有重要的公共卫生学意义。本文主要对常见谷类食品中克罗诺杆菌的污染状况进行了检测,并利用基于ITS(Internal Transcribed Spacer,内转录间隔区)和OmpA(Outer membrane protein A,外膜蛋白A)基因片段的特异性PCR鉴定和16S rDNA基因序列分析等方法对可疑克罗诺杆菌进行了初步的鉴定。
1 材料与方法
1.1材料与仪器
谷类食品2014年9月至2015年6月,从湖南、湖北、江苏、上海等地共采集谷类食品样品100份,包括玉米碴13份,大米9份,红豆8份,燕麦片和绿豆7份,黄豆、薏米、小黄米、荞麦粉、小麦粉、糯米和黑米各6份;高粱米和黑豆各5份,西米4份(表1)。样品采集完毕后立即送回实验室检测。
克罗诺杆菌标准菌株阪崎克罗诺杆菌C.sakazakiiCICC 21560和穆汀斯克罗诺杆菌C.muytjensiiCICC 21563由南京农业大学食品科技学院酶工程实验室馈赠;缓冲蛋白胨水(BPW)、改良月桂基硫酸盐胰蛋白胨肉汤(mLST)、万古霉素、胰蛋白胨大豆琼脂(TSA)北京陆桥技术有限责任公司;克罗诺杆菌显色培养基HiCromeTMCronobacterspp. agar(配方:酪蛋白15.0 g/L,大豆蛋白胨5.0 g/L,显色混合物10.17 g/L,氯化钠5.0 g/L,琼脂15.0 g/L)美国sigma公司;细菌基因组DNA提取试剂盒美国Omega公司;2× Taq Master Mix南京诺唯赞生物科技有限公司;引物由上海生工生物工程有限公司合成。
DNP9162电热恒温培养箱上海精宏;SW-CJ-2FD超净工作台苏州安泰;Veriti 96 PCR仪美国ABI公司;5418型小型高速离心机德国Eppendorf公司;Gel Doc XR凝胶成像仪美国BIO-RAD公司。
1.2实验方法
1.2.1分离方法按照国家标准GB 4789.40-2010《食品卫生微生物学检验阪崎肠杆菌检验》中的方法对100份谷类食品中的克罗诺杆菌进行分离鉴定[14]。
1.2.2基因组DNA提取将纯化后的可疑分离株接种于LB培养基中,置于摇床中于37 ℃,180 r/min过夜培养,取过夜培养液1~2 mL,参照美国Omega公司的细菌基因组DNA提取试剂盒的操作说明书提取细菌总DNA。
1.2.3PCR扩增ITS基因特异性片段按照参考文献[15]的方法采用PCR方法扩增克罗诺杆菌ITS基因特异性片段。正向引物SG-F:5′-GGGTTGTCTG CGAAAGCGAA-3′;反向引物SG-R:5′-GTCTTC GTGCTGCGAGTTTG-3′。PCR反应体系:2×Taq Master Mix 25 μL,引物SG-F和SG-R各2 μL,DNA模板1 μL,加水补足至50 μL。PCR反应条件:95 ℃预变性3 min;95 ℃变性30 s,57 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸7 min。反应结束后,取PCR产物经2%琼脂糖凝胶电泳,在凝胶成像系统下观察并记录实验结果。
1.2.4PCR扩增OmpA基因特异性片段按照参考文献[16]的方法采用PCR方法扩增克罗诺杆菌OmpA基因特异性片段。正向引物ESSF:5′-GGATTTAACCGTGAACTTTTCC-3′;反向引物ESSR:5′-CGCCAGCGATGTTAGAAGA-3′。PCR反应体系:2×Taq Master Mix 25 μL,引物ESSF和ESSR各2 μL,DNA模板1 μL,加水补足至50 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火45 s,72 ℃延伸50 s,35个循环;72 ℃延伸7 min。反应结束后,取PCR产物经1%琼脂糖凝胶电泳,在凝胶成像系统下观察并记录实验结果。
1.2.516S rDNA基因扩增及测序以细菌总DNA为模板,用细菌16S rDNA通用引物进行PCR扩增[17]。正向引物fD1:5′-AGAGTTTGATCCTGG CTCAG-3′和反向引物rP1:5′-ACGGTTACCTTGTT ACGACTT-3′。PCR反应体系:2×Taq Master Mix 25 μL,引物(10 μmol/L)各2 μL,DNA模板1 μL,加无菌双蒸水至50 μL。PCR反应条件:95 ℃预变性2 min;95 ℃变性45 s,55 ℃退火55 s,72 ℃延伸2 min,35个循环;72 ℃延伸7 min。PCR产物经1%琼脂糖凝胶电泳检测后送南京金思瑞生物科技有限公司测序。将测序获得的16S rDNA基因序列提交至GenBank数据库(http://www.ncbi.nlm.nih.gov/),用Blastn进行序列同源性比对。采用Clustalx 2.0软件将16S rDNA基因序列进行多重比较后,利用MEGA 4.0软件采用邻近法构建系统发育树。
2 结果与分析
2.1克罗诺杆菌污染情况
将在克罗诺杆菌显色培养基上生长呈蓝绿色,且在TSA平板上于25 ℃培养48 h呈黄色的菌落初步确定为可疑克罗诺杆菌分离株。通过显色平板及TSA平板共检出24份谷类食品样本污染了克罗诺杆菌,检出率为24.0%(表1)。对可疑分离株进行了16S rDNA测序鉴定,结果最终证实21份谷类食品样品污染了克罗诺杆菌,检出率为21.0%(表1)。本研究从绿豆、红豆、玉米、黄豆、荞麦粉、小黄米、黑豆、小麦粉、糯米、燕麦、黑米和薏米等样品中分离出了克罗诺杆菌,其中污染率较高的样品有荞麦粉(50%)、小麦粉(50%)和绿豆(42.9%)(表1)。
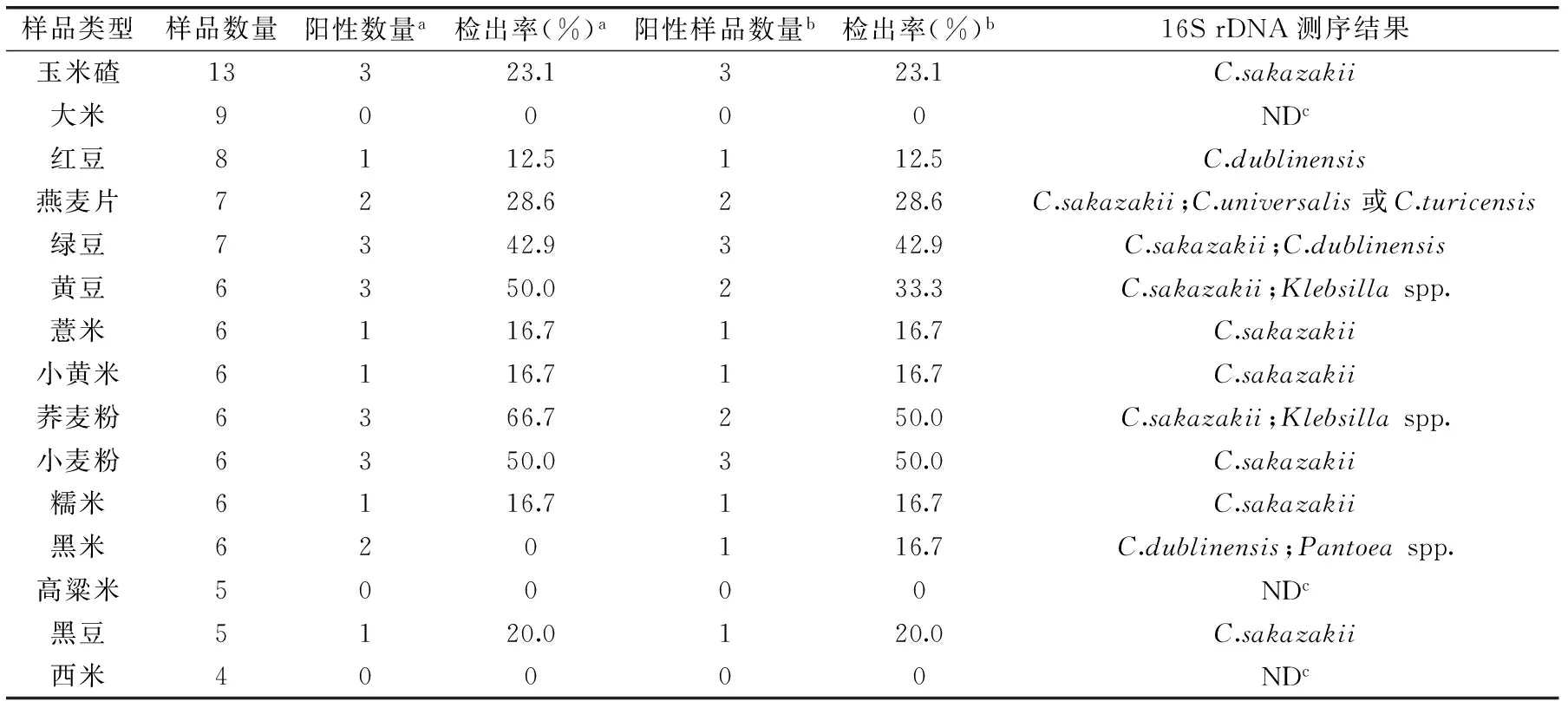
表1 不同谷类食品中克罗诺杆菌的污染率Table 1 Prevalence of Cronobacter isolates screened from various cereal samples
注:a:显色平板法;b:16S rDNA测序结果;c:ND,未检测(Not detected)。
2.2基于ITS基因的PCR验证结果
经琼脂糖凝胶电泳检测,结果显示24株可疑克罗诺杆菌分离株PCR检测全部为阳性,在250 bp附近出现目的条带,和预期片段大小(282 bp)相吻合,而阴性对照(双蒸水替代基因组DNA)未扩增出目的片段(图1)。

图1 基于ITS基因的克罗诺杆菌分离株PCR检测结果Fig.1 Detection of Cronobacter spp. isolated from cereal samples by PCR based on ITS gene注:Lane M:DL2000 DNA ladder;lane P1:阳性对照(C. sakazakii CICC 21560);lane P2:阳性对照(C. muytjensii CICC 21563);lane N:阴性对照;lanes 1~24:可疑克罗诺杆菌分离株。
2.3基于OmpA基因的PCR验证结果
从琼脂糖凝胶电泳图谱可知:经PCR扩增后,有21株分离株能扩增出片段大小约为469 bp的目标基因片段(图2),表明这些分离株可能为克罗诺杆菌。然而分离株N1(17)、分离株N2(19)和分离株N3(21)未扩增出目的片段,表明这些分离株可能不是克罗诺杆菌,分析可能的原因为基于ITS基因的PCR检测方法产生了假阳性结果或基于OmpA基因的PCR检测方法产生了假阴性结果。为了进一步验证本研究获得的分离株是否为克罗诺杆菌,本研究扩增了上述可疑分离株的16S rDNA基因片段,并对其进行测序验证和BLAST在线比对分析。
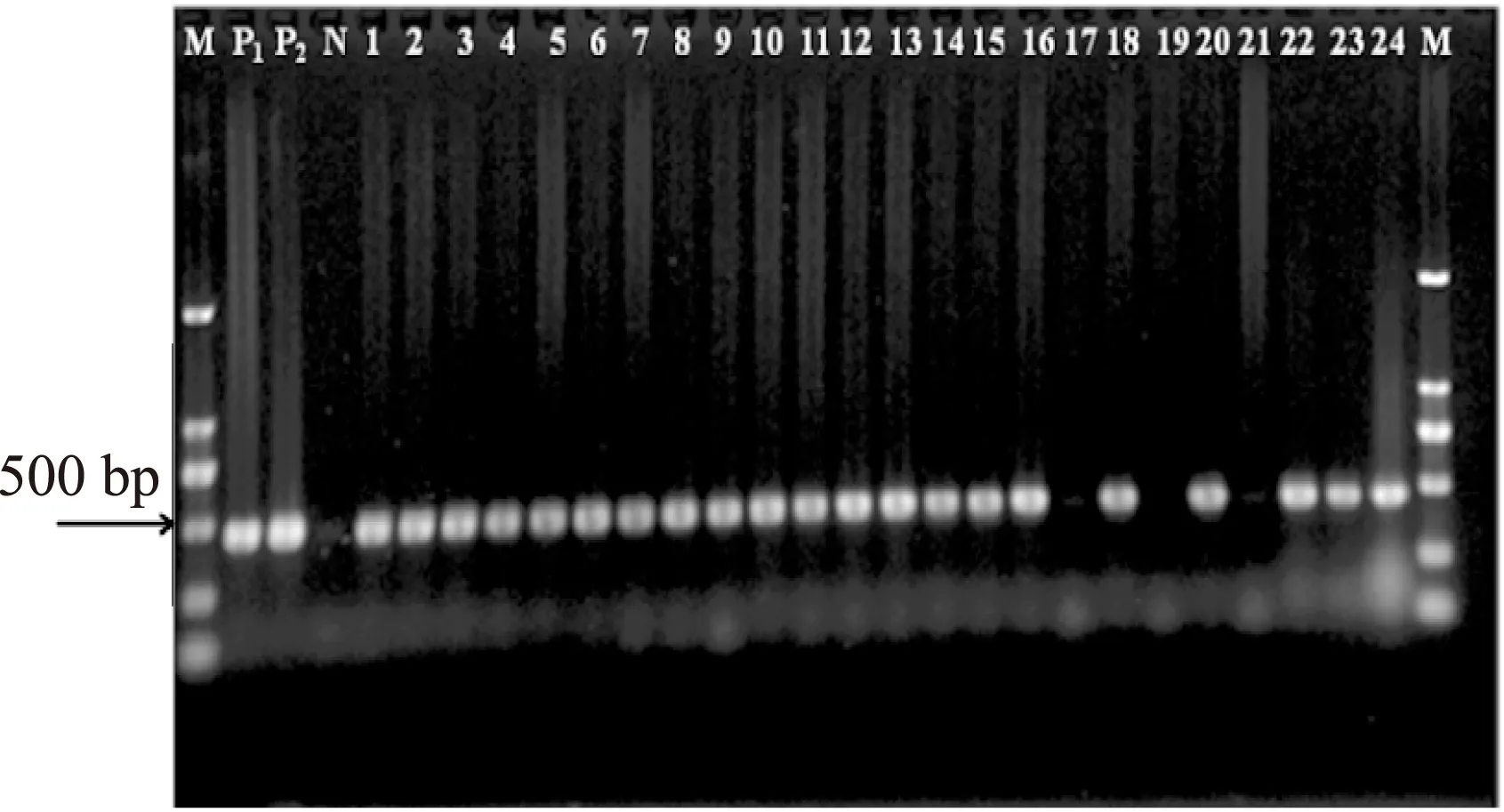
图2 基于OmpA基因的克罗诺杆菌 分离株PCR检测结果Fig.2 Detection of Cronobacter spp. isolated from cereal samples by PCR based on OmpA gene注:Lane M:DL2000 DNA ladder;lane P1:阳性对照(C. sakazakii CICC 21560);lane P2:阳性对照(C. muytjensii CICC 21563);lane N:阴性对照;lanes 1~24:可疑克罗诺杆菌分离株。
2.4分离株16S rDNA测序结果
利用BLAST在线比对分析(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分离株16S rDNA基因序列,结果显示21株可疑分离株均为Cronobacterspp.,其中大部分分离株(17株)的16S rDNA基因序列与C.sakazakii的相似性较高,序列相似性达99%以上;3株分离株的16S rDNA基因序列与C.dublinensis的相似性最高,序列相似性达99%以上;1株分离株的16S rDNA基因序列与C.universalis和C.turicensis的相似性高达99%以上(表2)。由于克罗诺杆菌属内不同种间菌株的16S rDNA基因序列相似性很高,通常无法通过16S rDNA基因序列比对的方法将其鉴定到种的水平,因而将本研究获得的上述21株分离株初步鉴定为Cronobacterspp.。此外,BLAST比对分析结果表明分离株N1和N2为克雷伯菌属(Klebsillaspp.),N3 为泛菌属(Pantoeaspp.)(表2)。该结果与基于OmpA基因的PCR验证结果一致,进一步证实了分离株N1、N2和N3不是克罗诺杆菌,同时也表明基于ITS基因的特异性PCR验证方法产生了假阳性结果。
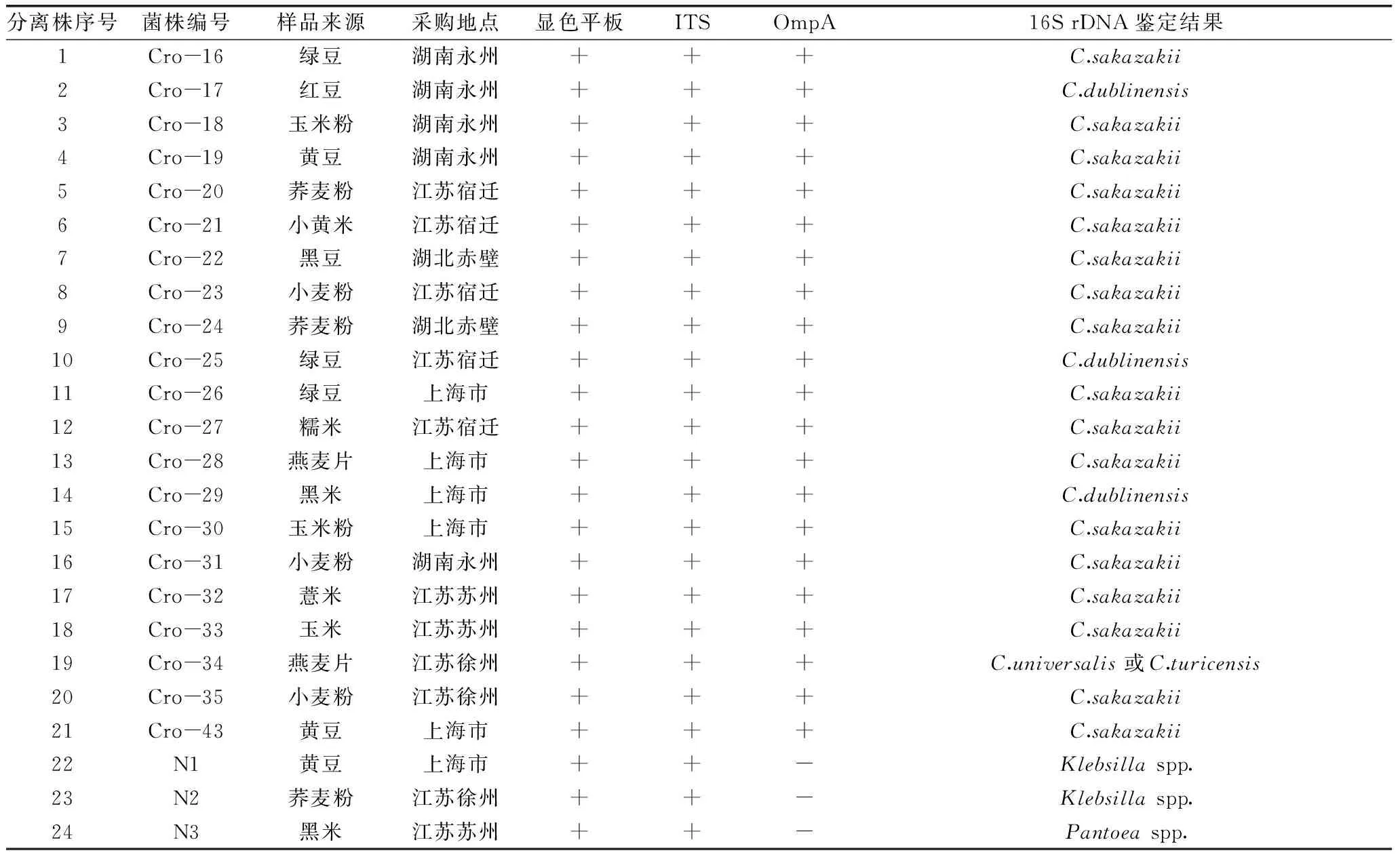
表2 克罗诺杆菌分离株形态学和分子生物学鉴定结果Table 2 Morphology and molecular identification of Cronobacter isolates screened from cereal samples
注:+,阳性;-,阴性。
2.5分离株系统发育分析
为了进一步分析克罗诺杆菌分离株的变异及系统进化关系,利用Mega 4.0软件以16S rDNA基因序列为基础构建系统发育树。分析结果表明,21株分离菌株与克罗诺杆菌标准菌株(C.sakazakiiATCC 29544、C.malonaticusLMG 23826、C.turicensisLMG 23827、C.dublinensissubsp.lactaridiLMG 23823、C.dublinensissubsp.lactaridiLMG 23824、C.dublinensissubsp.lactaridiLMG 23825、C.universalisNCTC 9529和C.muytjensiiATCC 51329)位于同一簇群,亲缘关系较近,而与其他肠杆菌科细菌的亲缘关系较远(图4),进一步推断本研究分离的上述21株可疑菌株为Cronobacterspp.。此外,分离株N1、N2和N3与克罗诺杆菌不在同一簇群,亲缘关系较远,进一步说明分离株N1、N2和N3不是克罗诺杆菌(图3)。
3 结论与讨论
采用准确可靠的检测方法是实现对食品中克罗诺杆菌的检测和监测的关键。常见的克罗诺杆菌分离鉴定方法主要有基于显色平板的微生物培养法、基于特异性基因片段进行PCR鉴定方法等,但是这2种检测方法往往会导致假阳性或假阴性结果[18]。因此,有必要结合16S rDNA基因序列分析方法、rpoB基因序列分析方法、脉冲场凝胶电泳(PFGE)和多位点序列分型(MLST)等方法对分离株进行进一步的验证[19-20]。本研究采用GB 4789.40-2010分离了谷类食品中的克罗诺杆菌,并利用了基于ITS基因和OmpA基因的特异性PCR以及16S rDNA基因序列分析的方法对分离株进行了初步的鉴定,最终从100份谷类食品样品中分离出21株克罗诺杆菌,其中17株被初步鉴定为C.sakazakii,3株为C.dublinensis,1株为C.universalis或C.turicensis。国内外关于食品中克罗诺杆菌污染状况的调查结果表明食品中污染的克罗诺杆菌主要为C.sakazakii,与本研究的结果一致[21-22]。
不同种的克罗诺杆菌在致病性、耐药性和耐干燥特性等方面具有较大差异。将分离株精确到种的水平对于追溯污染源、疾病的诊断和控制具有重要的意义。由于克罗诺杆菌不同种间菌株尤其是C.sakazakii和C.malonaticus之间的亲缘关系较近,16S rDNA基因序列分析技术无法将其准确鉴定到种,可通过PCR-限制酶片段长度多态性(PCR-RFLP)、PFGE和MLST等基因分型方法予以解决[22-24]。
[1]Vojkovska H,Karpiskova R,Orieskova M,et al. Characterization ofCronobacterspp. isolated from food of plant origin and environmental samples collected from farms and from supermarkets in the Czech Republic[J]. International Journal of Food Microbiology,2016,217:130-136.
[2]董晓辉,李程思,吴清平,等.食品污染克罗诺杆菌(阪崎肠杆菌)的分离鉴定[J].微生物学报,2013,53(5):429-436.
[3]Caubilla-Barron J,Hurrell E,Townsend S,et al. Genotypic and phenotypic analysis ofEnterobactersakazakiistrains from an outbreak resulting in fatalities in a neonatal intensive care unit in France[J]. Journal of Clinical Microbiology,2007,45(12):3979-3985.
[4]Lai KK.Enterobactersakazakiiinfections among neonates,infants,children,and adults:Case reports and a review of the literature[J]. Medicine,2001,80(2):113-122.
[5]龚燕,顾学章. 阪崎肠杆菌肺炎 64 例临床及药敏分析[J]. 上海医学,2005,28(12):1047-1048.
[6]Li Y,Chen Q,Zhao J,et al. Isolation,identification and antimicrobial resistance ofCronobacterspp. isolated from various foods in China[J]. Food Control,2014,37:109-114.
[7]陈万义,任婧,吴正钧,等. 生鲜蔬菜中阪崎克罗诺杆菌的分离与鉴定[J].食品科技,2014,39(1):304-308.
[8]Jung K,Park H. Prevalence and thermal stability ofEnterobactersakazakiifrom unprocessed ready-to-eat agricultural products and powdered infant formulas[J]. Food Science and Biotechnology,2006,15:152-157.
[9]Iversen C,Lane M,Forsythe J. The growth profile thermotolerance and biofilm formation ofEnterobactersakazakiigrown in infant formula milk[J]. Letters in Applied Microbiology,2004,38:378-382.
[10]Shaker R,Osaili T,Al-Omary W,et al. Isolation ofEnterobactersakazakiiand otherEnterobactersp from food and food production environments[J]. Food Control,2007,18:1241-1245.
[11]Jacobs C,Braun P,Hammer P. Reservoir and routes of transmission ofEnterobactersakazakii(Cronobacterspp.)in a milk powder-producing plant[J]. Journal of Dairy Science,2011,94(8):3801-3810.
[12]Lou X,Si G,Yu H,et al. Possible reservoir and routes of transmission ofCronobacter(Enterobactersakazakii)via wheat flour[J]. Food Control,2014,43:258-262.
[13]张翼,陈雅蘅,周帼萍,等. 克罗诺杆菌的生物膜检测和药敏性分析[J].食品科学,2015,36(21):129-134
[14]GB 4789.40-2010.食品微生物学检验 阪崎肠杆菌检验[S].北京:中国标准出版社,2010.
[15]Liu Y,Gao Q,Zhang X,et al. PCR and oligonucleotide array for detection ofEnterobactersakazakiiin infant formula[J]. Mol Cell Probe,2006,20:11-17.
[16]Nair M,Vekitanarayanan S. Cloning and sequencing of the
ompA gene ofEnterobactersakazakiiand development of an ompA-targeted PCR for rapid detection ofEnterobactersakazakiiin infant formula[J].Applied and Environmental Microbiology,2006,72:2539-2546.
[17]Weisburg G,Barns M,Pelletier A,et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology,1991,173(2):697-703.
[18]Jaradat Z,Ababneh Q,Saadoun I,et al. Isolation ofCronobacterspp.(formerlyEnterobactersakazakii)from infant food,herbs and environmental samples and the subsequent identification and confirmation of the isolates using biochemical,chromogenic assays,PCR and 16S rRNA sequencing[J]. BMC Microbiology,2009,9:225.
[19]Li Y,Cao L,Zhao J,et al. Use of rpoB gene sequence analysis for phylogenetic identification ofCronobacterspecies[J]. Journal of Microbiological Methods,2012,88:316-318.
[20]Joseph S,Sonbol H,Hariri S,et al. Diversity of theCronobactergenus as revealed by multilocus sequence typing[J]. Journal of Clinical Microbiology,2012,50(9):3031-3039.
[21]Lee D,Ryu W,Chang I,et al. Identification and classification ofCronobacterspp. isolated from powdered food in Korea[J]. Journal of Microbiology and Biotechnology,2010,20(4):757-762.
[22]Strydom A,Cameron M,Witthuhn R. PCR-RFLP analysis of the rpoB gene to distinguish the five species ofCronobacter[J]. Food Microbiology,2011,28:1472-1477.
[23]Molloy C,Cagney C,O’Brien S,et al. Surveillance and characterisation by pulsed-field gel electrophoresis ofCronobacterspp. in farming and domestic environments,food production animals and retail foods[J]. International Journal of Food Microbiology,2009,136(2):198-203.
[24]Cui J,Du X,Wei R,et al. Multilocus sequence typing analysis ofCronobacterspp. isolated from China[J]. Arch Microbiology,2015,197:665-672.
Isolation and identification ofCronobacterspp. from cereal foods
LI Yuan-hong1,ZHANG Yi-fei1,ZHANG Qing-cheng2,XU Wan1,ZOU Xiao-qian1,PEI Shang-fei1,ZHAO Min-sheng1,JIANG Hua1
(1.School of Public Health,Xuzhou Medical University,Xuzhou 221004,China;2.Department of Aviation Quartermaster,Air Force Logistics College,Xuzhou 221000,China)
To investigate the presence ofCronobacterspp. in cereal foods,a total of 100 cereal food samples were analyzed based on the national standard method of GB 4789.40-2010. The presumptive isolates was further identified by biochemical identification,PCR amplification of specific gene of ITS(internal transcribed spacer sequences)and OmpA,and sequence analysis of 16S rDNA. Twenty-one samples(21.0%)were positive forCronobacterspp.,and 21 isolates ofCronobacterwas collected in total. Most of theCronobacterisolates(81.0%)was identified asC.sakazakii,followed byC.dublinensis(14.2%),andC.universalisorC.turicensis(4.8%). The results indicated that commercially available cereal foods were possible reservoir ofCronobacterspp.,and therefore the epidemiological surveillance ofCronobacterspp. should be strengthened in cereal foods.
Cronobacterspp.;cereal foods;isolation;identification
2016-02-01
李远宏(1984-),男,博士,讲师,研究方向:食品微生物与生物技术,E-mail:lhy@xzmc.edu.cn。
国家自然科学基金青年科学基金项目(31401595);国家级大学生创新创业训练计划(20140313012);江苏省高等学校大学生创新创业训练计划(201410313012Z)。
TS201.3
A
1002-0306(2016)15-0154-06
10.13386/j.issn1002-0306.2016.15.022
