不同类型白癜风基因谱表达差异的初步研究
2016-09-10聂慧琼张小燕侯秀丽邵琼琰
聂慧琼,王 平,,张小燕,侯秀丽,许 文,邵琼琰
不同类型白癜风基因谱表达差异的初步研究
聂慧琼1,王平1,2,张小燕2,侯秀丽1,许文2,邵琼琰3
目的 探讨白癜风临床类型与基因表达差异的相关性。方法 分别选择节段型白癜风(SV)、非节段型白癜风(NSV)和健康对照(HI)各4例,应用Phalanx人全基因组表达谱芯片检测外周血淋巴细胞基因谱表达差异,并通过RTPCR进行验证。结果 与HI相比,SV有239个上调基因和175个下调基因,主要涉及适应性免疫反应、细胞因子受体相互作用、趋化因子信号通路,NSV有88个上调基因和560个下调基因,涉及先天性免疫、细胞自噬与凋亡、黑素细胞生物学、泛素介导的蛋白裂解与酪氨酸酶代谢。SV和NSV共同基因表达差异包括60个上调基因和60个下调基因;相对于SV,NSV表达223个上调基因和129个下调基因,主要包括嘌呤、嘧啶、鞘脂类代谢。结论 SV有不同于NSV的遗传背景与发病机制。
白癜风,节段型;白癜风,非节段型;基因表达
网络出版时间:2016-4-19 11:04:48 网络出版地址:http://www.cnki.net/kcms/detail/34.1065.R.20160419.1104.044.html
白癜风是一种常见的皮肤黏膜色素脱失性疾病,其病因和确切发病机制至今尚不完全清楚,涉及遗传、免疫炎症、神经体液、细胞毒性、氧化应激、功能性黑素细胞凋亡和(或)丢失及环境因素等假说[1-2]。根据皮损分布特点,白癜风分为两种类型,节段型白癜风(segmental vitiligo,SV):白斑沿某一皮神经节段支配的皮肤区域走向分布,通常皮损不超过中线;非节段型白癜风(non-segmental vitiligo,NSV):又称寻常型白癜风,为广泛、局灶、或散在白斑。SV和NSV不仅在皮损分布上各具特点,对治疗选择与治疗反应也存在较大差异,两者存在不同的病理机制[3-4]。该研究采用高通量的基因芯片技术,筛选SV和NSV患者外周血差异表达基因,探讨不同类型白癜风发病相关基因的差异,为进一步揭示白癜风发病机制和采用个体化治疗方案提供新思路。
1 材料与方法
1.1标本收集 选取杭州市第三人民医院皮肤科SV和NSV各4例(表1),病例均有典型白斑分布特点,并通过Wood灯和激光共聚焦显微镜再次确诊。患者均无伴发其他器质性疾病、斑秃、桥本甲状腺炎等自身免疫性疾病及感染性疾病。入选者均为初次诊治,均未系统应用糖皮质激素、免疫抑制剂、光敏剂及紫外线治疗。两组的性别、年龄及病程差异均无统计学意义,有可比性。健康对照组均来自我院健康体检者。本研究获得杭州市第三人民医院医学伦理委员会批准,入选病例均签署知情同意书。
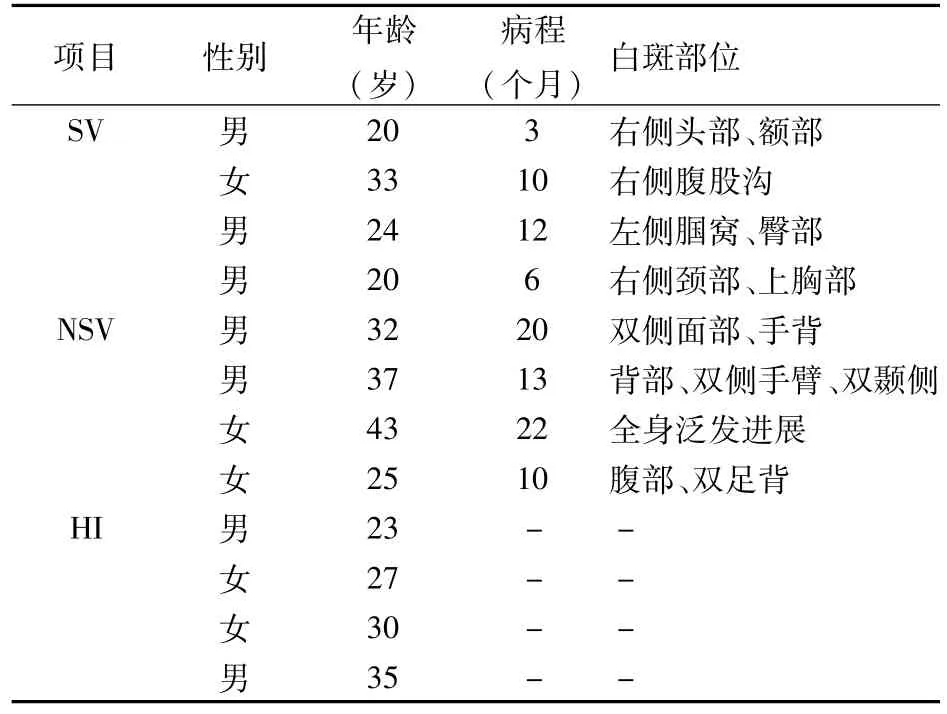
表1 病例资料
1.2全基因表达芯片检测 抽取浅静脉抗凝血6 ml,常规分离外周血淋巴细胞,TRIzol抽提RNA后-20℃保存,送上海仪方生物技术有限公司进行Phalanx人全基因组表达谱芯片(人32 679个基因)检测。每组重复检测4次。基因芯片数据利用Rosetta Resolver软件(USA)进行处理,筛选全基因组表达谱芯片获取的基因差异表达水平≥0.585,且经T-test统计分析显著差异表达的基因(P<0.05)判定为差异表达基因。
1.3RT-PCR验证 选取6条基因产物进行RTPCR验证,其引物序列见表2。操作步序按常规操作,应用2-ΔΔCt法计算相对表达量。

表2 RT-PCR引物设计
1.3统计学处理 采用SPSS 13.0统计软件进行分析,数据以 ¯x±s表示,组间比较采用两样本 t检验;系统聚类分析法将差异性表达基因数据进行对数变换,应用Version3.0软件进行数据分析。
2 结果
2.1基因芯片检测结果
2.1.1基因表达情况 相对于HI,SV中239个基因表达上调,175个表达下调,差异性表达基因发生聚类(图1A)。利用基因本体论(gene ontology,GO)数据库提供的3种本体论(生物过程、细胞组分、分子功能)对差异表达基因进行GO分类,生物学过程中(表3),主要涉及以下3个富集项目:信号转导、生物合成过程及细胞营养代谢过程;细胞组分中(表4),主要是25%的细胞质,19%细胞核,12%细胞支架等;分子功能中(表5),差异性表达基因主要分为离子的结合、脱氧核糖核酸(DNA)的结合、信号转导活性、酶的结合。这些差异性表达的基因主要作用的通路包括细胞因子-细胞因子受体相互作用,肿瘤通路、趋化因子信号通路、MAPK通路(表6)。
2.1.2NSV基因表达情况 相对于HI,NSV表达688个上调基因和560个下调基因。通过用聚类cluster软件对差异表达的基因进行聚类分析的结果显示分析(图1B),左侧探针聚类区主要分上调和下调基因,这些基因参与多种功能如免疫反应,信号通路,转录因子。通过GO分析,生物学过程中(表3),主要富集项目包括转录调节,信号转导,固有免疫反应,氧化还原过程;细胞组分中(表4),24%细胞膜,20%细胞核,19%细胞液等物质参与此过程;分子功能中(表5),差异性基因包含脱氧核糖核酸(DNA)的结合,受体转录因子、转移酶、GTP酶活性。通过KEGG通路分析,这些差异性表达基因主要涉及的通路如下:MAPK信号通路,Toll样受体信号通路、Jak-STAT信号通路,趋化因子通路、泛素水解蛋白及C-C受体反应(表6)。
2.1.3SV和NSV基因表达差异比较 相对于HI,SV和NSV共同表达60个上调节基因和60个下调基因。相对于SV,NSV表达223个上调基因和129个下调基因。聚类分析差异性表达基因主要分为3种类型(图1C)。通过 GO分析,生物学过程中(表3),主要分为信号转导、生物合成过程细胞分化和免疫系统过程;细胞组分中(表4),蛋白复合物,质膜,细胞浆、细胞核和细胞液占75%以上;分子功能中(表5),包含19%信号转导蛋白活性,16%酶的结合和10%氧化还原酶活性。基因通路分析显示,这些差异性表达基因参与的过程包括嘌呤、嘧啶、鞘脂类和色氨酸代谢,免疫适应(表6)。

表3 主要生物学过程 GO分析结果
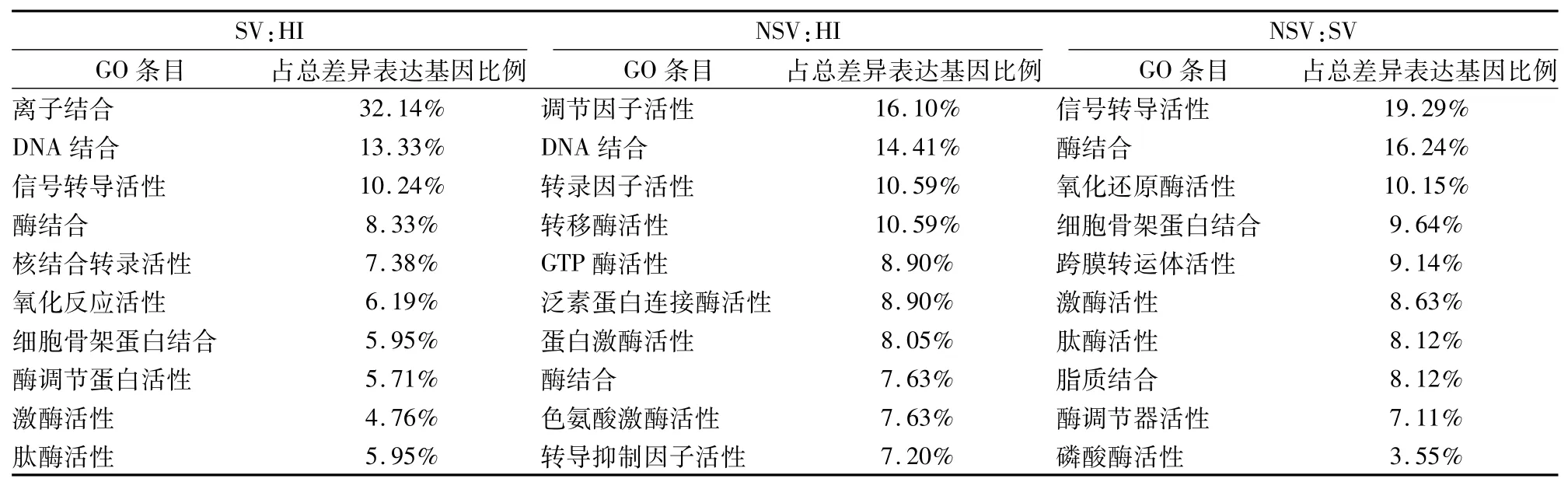
表5 主要分子功能 GO分析结果
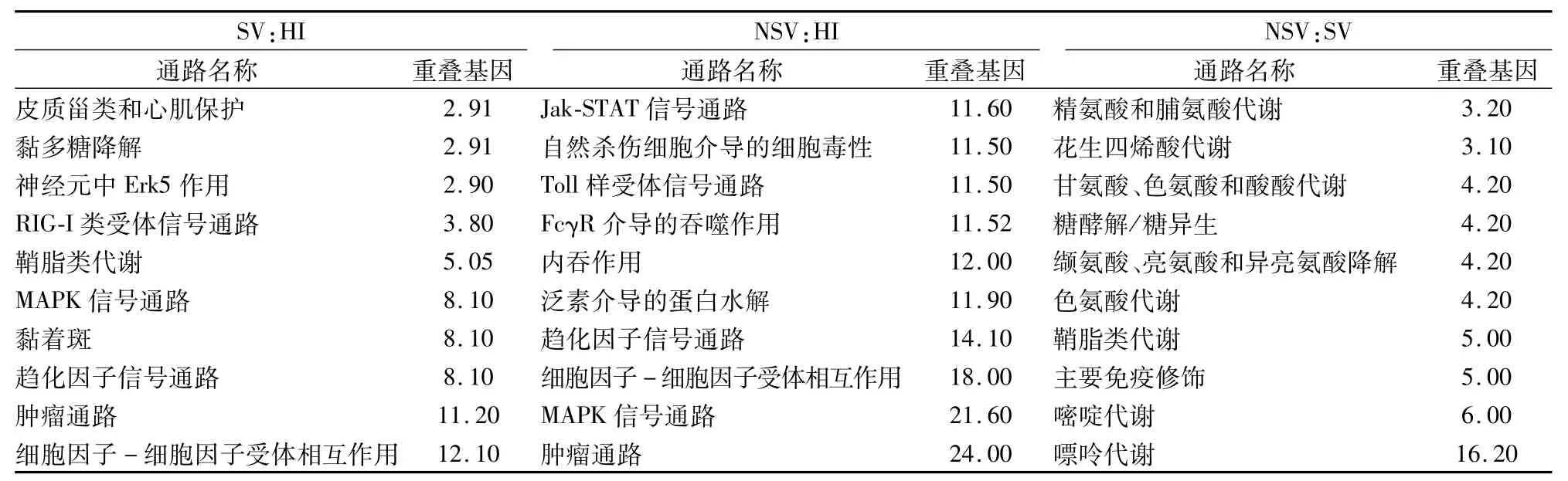
表6 差异表达基因主要pathway通路分析结果

图1 聚类分析A:SV和HI前5%差异性表达基因聚类分析;B:NSV和HI前5%差异性表达基因聚类分析;C:SV和NSV5%差异性表达基因聚类分析
2.2RT-PCR结果验证 RT-PCR试验验证6条基因产物见表2。基因芯片与RT-PCR检测结果一致(图2)。相对于 HI,NSV中 EGR2、UBE2D1、KIR3DS1的mRNA表达上调;SV中,UBE2D1、MMP8表达上调,CCL3、KIR3DS1和MRC2表达下调;相对于SV,NSV中EGR2、CCL3和KIR3DS1的mRNA表达上调,MMP8表达下调。
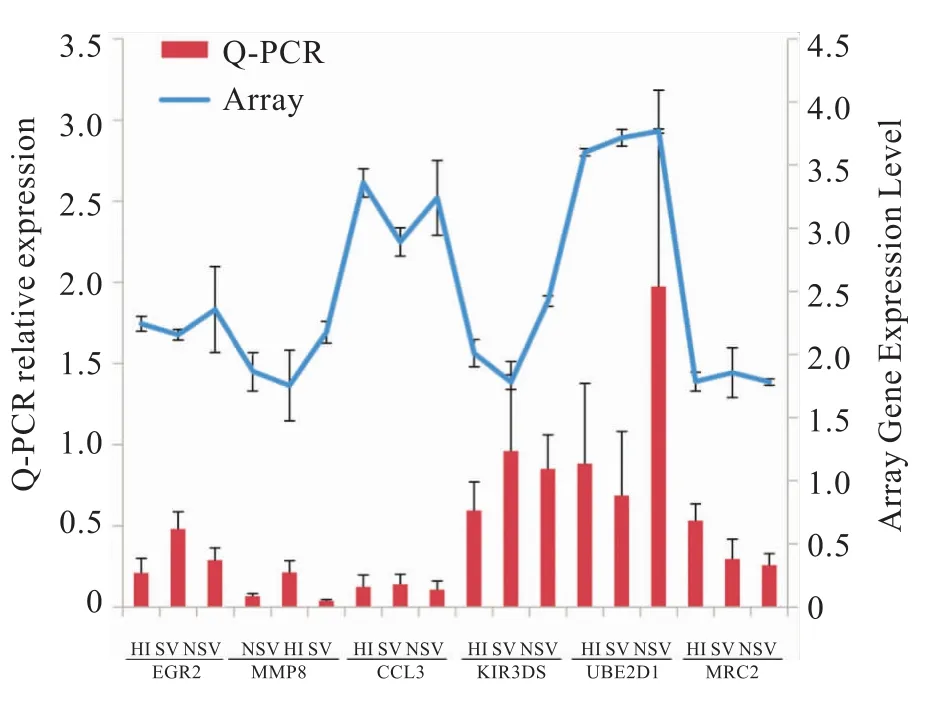
图2 RT-PCR验证对比基因芯片结果
3 讨论
免疫反应在白癜风发病机制中起着重要作用,既往研究[5]表明,白癜风患者存在着体液免疫、细胞免疫、细胞因子及其受体功能的异常以及自身免疫耐受状态的破坏。本研究结果表明,SV、NSV及HI均有涉及Toll样受体信号通路,B细胞和T细胞分化,淋巴细胞增殖,免疫反应激活等免疫反应。但是,SV主要是适应性免疫反应起着重要作用,而NSV则是固有免疫反应及B细胞分化、活化占主导作用,提示两种类型白癜风的黑素细胞破坏过程可能由不同免疫过程参与。
Jeong et al[6]对225名NSV和439名健康对照组进行单核苷酸多态性(SNP)研究,发现自噬相关基因紫外线辐射抵抗相关基因(ultraviolet radiation resistance-associated gene,UVRAG)多态性和NSV密切相关,推测 UVRAG可能和 NSV易感性相关。本研究则有类似结果,即NSV与自噬相关性较更高。相对于HI,NSV存在14个自噬相关的差异性表达基因,而只有一个自噬相关基因PAFAH1B2有差异性表达,提示自噬可能在NSV的病理机制中起着重要作用。
过量的氧化应激产物可通过对黑素细胞的直接毒性作用、干扰表皮细胞生物嘌呤代谢、抑制细胞黑素合成酶功能等多种途径,损伤或破坏黑素细胞,影响黑素代谢,甚至导致黑素细胞的死亡[7]。在进展期白癜风患者中,外周血单核细胞超氧化歧化酶活性增强,谷胱甘肽和维他命E水平减低[8]。本研究显示两组白癜风均存在氧化酶活性相关基因如SOD1和SOD2表达上调,表明氧化应激在SV和NSV发病中作用相当。
功能性黑素细胞凋亡和(或)丢失,是白癜风发病机制的假说之一[9-10]。白癜风患者受损皮肤中,可发现凋亡角质形成细胞,且抗凋亡蛋白Bcl-2、FLIP低表达,而凋亡多肽抗原和P53则高表达[10]。本研究中,凋亡相关基因多高表达在NSV,而SV几乎不表达,提示NSV与细胞凋亡关系密切。
黑素细胞产生黑素受酪氨酸酶相关家族调控,包括酪氨酸酶和酪氨酸酶相关蛋白,其受小眼畸形转录因子调控[11]。本研究表明,NSV有45个酪氨酸代谢和酪氨酸激酶启动子活性相关基因差异表达,而不见于SV,推测NSV存在酪氨酸代谢异常。另外,NSV还存在几种参与泛素介导的蛋白水解差异性表达基因。这些基因可能参与NSV中相关蛋白异常水解过程,从而影响其黑素细胞功能。
综上所述,本研究显示SV和NSV均存在免疫异常和高氧化应激反应过程,而相对于SV,NSV特异性表达自噬、凋亡、蛋白水解和酪氨酸酶代谢相关基因,提示两者有不同的遗传背景和发病机制。本研究对可能与白癜风相关的6个基因进行了RTPCR验证,验证结果显示与基因芯片结果相符,这也从另一方面说明了基因芯片结果的可信度。期待今后多环境多样本交叉研究资料,为SV和NSV靶基因筛选及靶向治疗提供分子基础。
[1] Passeron T,Ortonne JP.Physiopathology and genetics of vitiligo[J].JAutoimmun,2005,25:63-8.
[2] Guerra L,Dellambra E,Brescia S,et al.Vitiligo:pathogenetic hypotheses and targets for current therapies[J].Curr Drug Metab,2010,11(5):451-67.
[3] 王 平,洪为松,章莉,等.白癜风表皮黑素细胞超微结构及小眼畸形相关转录因子转录调控研究[J].中华皮肤科杂志,2010,43(7):37-40.
[4] 倪亚杰,王平,洪为松,等.小眼畸形相关转录因子转录调控与白癜风临床类型的相关性研究[J].医学研究杂志,2013,42(3):52-5.
[5] Le Poole IC,Luiten R M.Autoimmune etiology of generalized vitiligo[J].Curr Dir Autoimmun,2008,10:227-43.
[6] Jeong T J,Shin M K,Uhm Y K,et al.Association of UVRAG polymorphismswith susceptibility to non-segmental vitiligo in a Korean sample[J].Exp Dermatol,2010,19(8):e323-5.
[7] Glassman S J.Vitiligo,reactive oxygen species and T-cells[J]. Clin Sci(Lond),2011,120(3):99-120.
[8] Dell'Anna M L,Maresca V,Briganti S,etal.Mitochondrial impairment in peripheral blood mononuclear cells during the active phase of vitiligo[J].J Invest Dermatol,2001,117(4):908-13.
[9] Moretti S,Fabbri P,Baroni G,et,al.Keratinocyte dysfunction in vitiligo epidermis:cytokinemicroenvironment and correlation tokeratinocyte apoptosis[J].HistolHistopathol,2009,24(7):849-57.
[10]Lee A Y,Youm Y H,Kim N H.Keratinocytes in the depigmented epidermis of vitiligo aremore vulnerable to trauma(suction)than keratinocytes in the normally pigmented epidermis,resulting in their apoptosis[J].Br JDermatol,2004,151(5):995-1003.
[11]Murisier F,Beermann F.Genetics of pigment cells:lessons from the tyrosinase gene family[J].Histol Histopathol,2006,21(5):567-78.
Prelim inary study of differential gene expression profiling between segmental vitiligo and non-segmental vitiligo
Nie Huiqiong1,Wang Ping1,2,Zhang Xiaoyan2,et al
(1Clinical College of Hangzhou,Anhui Medical University,Hangzhou 310000;2Dermatological Dept of Hangzhou Third People's Hospital,Hangzhou 310000)
Objective To investigate the correlation between clinical pattern and differential gene expression in patientswith vitiligo.Methods Peripheral blood lymphocytes obtained from four cases of segmental vitiligo(SV),non-segmental vitiligo(NSV)and healthy individual(HI)respectively.Whole genome expression microarrays were used to assay the gene expression profiles between SV,NSV and HI.Quantitative PCR assay was used to validate the gene expression of array.Results Compared to HI,239 over-expressed and 175 down-expressed genes were detected in SV,which weremainly involved in the adaptive immune response,cytokine-cytokine receptor interaction and chemokine signaling.In NSV,88 over-expressed and 560 down-expressed geneswere found and were mainly involved in the innate immune,autophagy,apoptosis,melanocyte biology,ubiquitin mediated proteolysis and tyrosine metabolism,which were different from SV.Sixty over-expressed and sixty down-expressed genes shared similar tendency in SV and NSV.Compared to SV,223 over-regulated and 129 down-regulated geneswere found in NSV,which weremainly involved in themetabolism of purine,pyrimidine and sphingolipid.Conclusion Theremay exit different genetic background and pathogenesis between SV and NSV.
segmental vitiligo;non-segmental vitiligo;gene expression
R 75
A
1000-1492(2016)05-0707-05
2016-02-22接收
杭州市医学重点专科专病项目(编号:20140733Q19);浙江省医药卫生科技计划项目(编号:2014KYB200)
1安徽医科大学附属杭州临床学院皮肤科,杭州 310000
2杭州市第三人民医院皮肤科,杭州 310000
3浙江中医药大学附属杭州市第三人民医院皮肤科,杭州
310000
聂慧琼,女,硕士研究生;
王 平,男,主任医师,硕士生导师,责任作者,E-mail:dermwang@aliyun.com
