红鳍东方鲀去乙酰化酶SIRT3基因的克隆及原核表达
2016-08-10张伟姬明丽张军华千智斌
张伟,姬明丽,张军华,千智斌
(新乡医学院基础医学院,河南 新乡 453003)
红鳍东方鲀去乙酰化酶SIRT3基因的克隆及原核表达
张伟,姬明丽,张军华,千智斌
(新乡医学院基础医学院,河南 新乡 453003)
从红鳍东方鲀(Takifugu rubripes)肝脏组织中提取总RNA,通过RT-PCR扩增,获得SIRT3基因的编码阅读框(ORF),用原核表达载体pET32a(+)构建pET32a/SIRT3重组质粒,用异丙基-β-D-硫代半乳糖苷(IPTG)将重组质粒pET32a/STRT3在大肠杆菌Rosetta (DE3)进行诱导表达,并用镍柱纯化融合表达蛋白。结果显示,红鳍东方鲀SIRT3基因(trSIRT3)ORF区大小为1 263 bp大小,编码420个氨基酸。经IPTG诱导表达后,获得1个带组氨酸(His)标签的融合蛋白,目的蛋白主要存在于上清溶液中,为可溶性表达。用镍离子亲和层析柱对重组蛋白进行纯化,获得了相对分子质量约66 000的重组蛋白。
红鳍东方鲀;去乙酰化酶;SIRT3基因;基因克隆;原核表达
投稿网址:http://xb.ijournal.cn
信息沉默调节因子 2(The silent information regulator 2,Sir2或Sirtuins)蛋白最初是在酵母细胞中发现的,是一种高度保守的NAD+依赖性去乙酰化酶和ADP-核糖基转移酶,广泛存在于原核生物和真核生物中[1],有7个家族成员为Sirt1-7[2]。Sirtuins蛋白家族有很多生物学功能,如细胞周期调节[3]、基因表达沉默[4]、新陈代谢[5]、寿命延长[6]和细胞凋亡[7]等。SIRT3是Sirtuins家族中主要定位在线粒体内的去乙酰化酶,调节线粒体内的活力及能量代谢等[3]。与野生型相比,在SIRT3敲除的小鼠中,脂肪酸代谢中间产物的浓度发生异常,肝脏中甘油三酯也明显增多,但是其肝脏中的脂肪酸吸收以及分解作用正常[8];用SIRT3缺失小鼠进行饥饿试验,48 h后,发现小鼠肝脏和血浆中的酯酰肉碱和棕榈酰肉碱水平明显升高,脂肪酸β-氧化发生障碍[9];对小鼠饲喂高脂食物,发现肝脏组织中SIRT3的表达量明显下降,而线粒体蛋白质的乙酰化水平升高[10];在棕色脂肪细胞的分化中会存在 SIRT3的诱导表达[11]等。总之,SIRT3在调节脂肪代谢过程中发挥着非常重要的作用。
目前,关于鱼类Sirtuins基因中SIRT1的研究较多,而关于SIRT3的研究尚少,仅研究了斑马鱼SIRT3基因的克隆及其在肝脏中的表达模式[12]。红鳍东方鲀是中国沿海地区养殖的主要品种之一,其肉质极为鲜美,营养丰富,经济价值较高。笔者以红鳍东方鲀为研究对象,用RT-PCR技术克隆了红鳍东方鲀SIRT3基因的ORF区序列,并对其进行原核表达及蛋白纯化,旨在为深入研究TrSIRT3基因调节鱼类脂肪代谢的分子机制提供参考依据。
1 材料与方法
1.1材料
以红鳍东方鲀为试验材料,取其肝脏组织,用灭菌DEPC处理水进行冲洗,在液氮速冻1 h后取出,放-80 ℃冰箱备用。
1.2方法
1.2.1SIRT3基因的克隆
以大黄鱼SIRT3的氨基酸序列(XP_010752299.1)为探针,在河鲀基因组数据库(http://genome.jgi-psf.org/Takru4/Takru4.home.html)中进行BLAST分析,获得预测的红鳍东方鲀的SIRT3 cDNA序列。
用Trizol试剂(Invitrogen,美国)从肝脏组织中抽提总RNA。以总RNA为模板,用SuperScript Ⅲreverse transcriptase(Invitrogen Corp,Carlsbad,CA,美国)合成cDNA第1条链。根据预测的SIRT3基因序列,在ORF区两端设计引物(上游引物为5'-ATGAACAAAAAAGCCCCCCAG-3';下游引物为5'-TCAGCTGCTGAGCGTGGAC-3')。RT-PCR扩增条件:95 ℃变性40 s,60 ℃退火40 s,72 ℃延伸90 s,35个循环,最后72 ℃延伸10 min。将PCR产物连接于pGEM T-easy载体(Promega,USA),连接产物转化Escherichia coli JM109,在含Amp、X-gal和IPTG的LB平板上筛选用菌落PCR鉴定的阳性克隆。抽提质粒后,在生工生物工程(上海)股份有限公司进行测序。
1.2.2生物信息学分析
用DNA Star软件中的Editseq程序分析核苷酸和氨基酸序列理化性质。用http://www.expasy.ch/网站分析SIRT3氨基酸序列的亲/疏水性。用DNA Star软件子程序MegAlign和ClustalW2.1 Version (http:// clustalw.ddbj.nig.ac.jp/)对不同物种SIRT3的氨基酸序列进行多序列比对与同源性分析。采用MEGA5.05软件构建Neighbor-Joining分子系统进化树。用CBS的ProtComp Version 9.0软件预测蛋白质亚细胞定位。
1.2.3原核表达载体的构建及鉴定
根据克隆得到的SIRT3的ORF序列设计特异引物,上游ES3-F为5'-GAATTCATGAACAAAAA AGCCCCCCAG-3',下游ES3-R为5'-CTCGAGTC AGCTGCTGAGCGTGGAC-3',引物对的划线部分分别为 EcoRⅠ酶切位点和 XhoⅠ酶切位点。RT-PCR反应体系:cDNA模版2.0 μL、10 μmol/L ES3-F 2.0 μL、10 μmol/L ES3-R 2.0 μL、10×PCR Buffer 5.0 μL、dNTP Mixture 4.0 μL、ddH2O 34.0 μL、高保真DNA聚合酶 1.0 μL,总计50 μL。反应程序:95 ℃变性4 min;94℃变性40 s,60 ℃退火40 s,72 ℃延伸90 s,35个循环。对得到的SIRT3基因PCR产物与pET32a质粒用EcoRⅠ和XhoⅠ酶切3 h,用琼脂糖凝胶回收目的基因和载体后用T4DNA连接酶连接,再转化进E.coli JM109,在含氨苄抗性的LB平板上涂布后过夜培养。菌落PCR和双酶切鉴定筛选阳性克隆后,对重组质粒进行测序验证。将构建成功的重组质粒命名为pET-32a/SIRT3。
1.2.4重组质粒pET32a/SIRT3的诱导表达、纯化及SDS–PAGE分析
将测序后被鉴定为正确的pET-32a/SIRT3重组质粒转化到大肠杆菌 Rosetta(DE3)中,涂布于平板后过夜培养,用菌落PCR鉴定阳性克隆,将克隆接种到含氨苄的 LB液体培养基,37 ℃振荡培养12~16 h,按1∶50的比例转接到20 mL培养基中振荡培养至OD值为0.6~0.8,加入IPTG至终浓度为0.8~1.0 mmol/L,37 ℃诱导5 h后离心,收集菌体,将菌体放入冰上用超声波处理后,在 4 ℃下10 000×g离心 20 min,将上清液转到新离处理心管。上清液部分按His-Bind Purification Kit试剂盒操作说明进行纯化,将表达产物和纯化产物同时用SDS-PAGE进行电泳检测。
2 结果与分析
2.1SIRT3基因的序列测定结果
根据电子克隆获得的序列设计特异性引物,经RT-PCR扩增后,电泳检测到目的条带(图1)符合预期SIRT3基因的ORF区的大小。测序结果表明,该序列的全长为1 263 bp。用DAN Star软件推测获得的红鳍东方鲀SIRT3基因ORF序列的氨基酸序列(图2),发现其编码氨基酸420个,该蛋白质的理论等电点为8.10,预测红鳍东方鲀SIRT3蛋白的理论相对分子质量为45 480。氨基酸成分分析结果显示,酸性氨基酸有42个,占10.0%;碱性氨基酸有45个,占10.71%;极性氨基酸(S、T、Y、C、Q、 N)有121个,占28.81%,非极性氨基酸(A、F、I、L、V、W)有136个,占32.38%。由以上结果可知,非极性氨基酸的比例略高。TrSIRT3蛋白中有保守KIV与GIP重要的功能域(图1)。用ProScale在线软件进行分析,可以判定SIRT3蛋白为亲水性蛋白。用ProtComp Version 9.0在线软件分析亚细胞定位,推断红鳍东方鲀SIRT3蛋白定位于线粒体(表略),与哺乳动物的亚细胞定位一致。
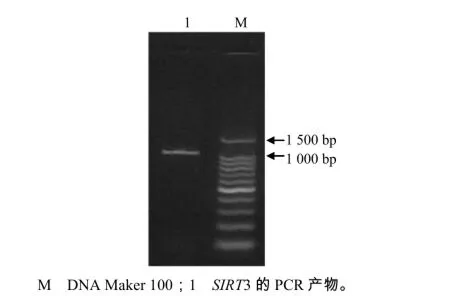
图1 RT–PCR扩增电泳图谱Fig. 1 Electrophoretogram from PCR amplification of gene SIRT3

图2 红鳍东方鲀与其他物种SIRT3的氨基酸比对Fig.2 Alignment of amino acid sequences of trSIRT3 to others
2.2红鳍东方鲀SIRT3的同源性及系统进化分析结果
将红鳍东方鲀SIRT3蛋白的氨基酸序列与其他物种进行同源性分析,发现红鳍东方鲀与大黄鱼、尼罗罗非鱼、梭子鱼、半滑舌鳎、绿头鸭、兔子、小鼠、猪、牛、裸鼢鼠及人的同源性分别为78.3%、76.5%、73.8%、69.5%、55.1%、55.1%、49.5%、49.4%、48.0%、47.1%及46.6%(表1)。用Mega5.0以邻位相接(N-J)构建分子系统进化树(图3),进化树总体分为2支:鱼类为1支;哺乳类动物聚为另外1支。在鱼类这1支中,红鳍东方鲀与大黄鱼先聚在一起,故红鳍东方鲀与鱼类的亲缘关系较近(与大黄鱼的亲缘关系最近),与哺乳动物的亲缘关系较远。

表1 红鳍东方鲀与其他物种SIRT3的同源性Table 1 Homology comparison between amino acid sequence of SIRT3 from Takifugu rubripes and other species

图3 红鳍东方鲀SIRT3与其他生物的系统进化树Fig.3 Phylogenetic tree for the evolutionary relationships between various SIRT3
2.3重组表达载体的构建及鉴定结果
用EcoRⅠ和XhoⅠ对pET32a/SIRT3重组表达质粒进行双酶切鉴定(图4),1条为载体,1条为目的条带。测序结果表明,所测序列没有碱基缺失及变异,故初步确认所构建的pET32a/SIRT3重组表达质粒是成功的。

图4 重组质粒pET32a/SIRT3的酶切分析结果Fig.4 Restriction analysis of plasmid pET32a/SIRT3
2.4融合蛋白及纯化蛋白的SDS–PAGE电泳结果
将pET-32a/SIRT3转化到大肠杆菌Rosetta (DE3),与空质粒pET32a的阴性对照相比,经IPTG诱导后,在上清液和沉淀中均出现相对分子质量约66 000的蛋白条带,与推测的融合蛋白的相对分子质量相当,而转入空质粒中未见相应的蛋白条带(图5)。表达的目的蛋白主要以可溶蛋白形式存在,在沉淀中含有部分目的蛋白,据此,可初步推断SIRT3在大肠杆菌中获得成功表达。用镍柱纯化的融合蛋白通过SDS-PAGE检测,发现只有1条相对分子质量约66 000的目的蛋白(图5),说明纯化成功,已获得重组表达蛋白。
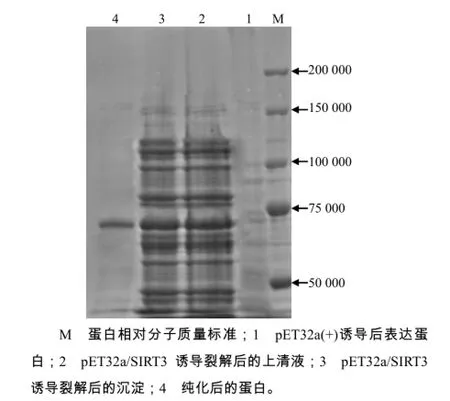
图5 重组质粒表达产物的SDS–PAGE分析结果Fig.5 SDS–PAGE analysis on the expression of SIRT3 in Rosetta (DE3)
3 结论与讨论
SIRT3是Sirtuins家族中定位于线粒体的NAD+依赖性去乙酰化酶[13],参与调控动物的脂肪代谢。目前,国内对鱼类Sirtuins基因家族的研究尚少,张伟等[14-15]克隆红鳍东方鲀SIRT1基因的ORF区并对其进行了原核表达,并获得了红鳍东方鲀FoxO1的融合表达蛋白。红鳍东方鲀SIRT3的氨基酸序列上含2个特异性保守结构区域,即KIV和GIP,它们是SIRT3蛋白调控基因沉默关键的功能结构域[16]。该研究进行亚细胞定位预测,红鳍东方鲀SIRT3同样定位在线粒体中,与哺乳类动物一样,因此,推测红鳍东方鲀SIRT3基因可能在线粒体中调控能量代谢及ATP的产生,从而影响着糖脂代谢。
pET系统因其所带标签小,纯化方便,pET-32a(+)载体的N端含有编码6个组氨酸标签(6×His-Tag)的序列,鉴定与纯化蛋白较为方便[17-18]。在本研究中,将红鳍东方鲀SIRT3基因克隆到原核表达载体pET-32a(+),构建了重组表达质粒pET-32a/SIRT3,然后用IPTG诱导其在大肠杆菌中的表达,通过原核表达,为进一步研究红鳍东方鲀SIRT3蛋白的结构和功能提供了参考依据。
[1] Dutnall RN,Pillus L.Deciphering NAD-dependent deacetylases[J].Cell,2001,105(2):161-164.DOI:10. 1016/s0092-8674(01)00305-1.
[2] North B J,Verdin E.Sirtuins:Sir2-related NAD-dependent protein deacetylases[J].Genome Biol,2004,5(5):224.DOI:10.1186/gb-2004-5-5-224.
[3] Ardestani P M,Liang F.Sub-cellular localization,expression and functions of Sirt6 during the cell cycle in HeLa cells[J].Nucleus,2012,3(5):442-451.DOI:10.4161/nucl.21134.
[4] Rusche L N,Kirchmaier A L,Rine J.The establishment,inheritance,and function of silenced chromatin in Saccharomyces cerevisiae[J].Annu Rev Biochem,2003,72:481-516.DOI:10.1146/annurev.biochem.72.121801. 161547.
[5] Schwer B,Verdin E.Conserved metabolic regulatory functions of sirtuins[J].Cell Metab,2008,7(2):104-112.DOI:10.1016/j.cmet.2007.11.006.
[6] Wątroba M,Szukiewicz D.The role of sirtuins in aging and age-related diseases[J].Adv Med Sci,2016,61(1):52-62.DOI:10.1016/j.advms.2015.09.003.
[7] Cattelan A,Ceolotto G,Bova S,et al.NAD(+)- dependent
SIRT1 deactivation has a key role on ischemia-reperfusioninduced apoptosis[J].Vascul Pharmacol,2015,70:35-44. DOI:10.1016/j.vph. 2015.02.004.
[8] Hirschey M D,Shimazu T,Capra J A,et al.SIRT1 and SIRT3 deacetylate homologous substrates:AceCS1,2 and HMGCS1,2[J].Aging (Albany NY),2011,3(6):635-642.DOI:10.18632/aging.100339.
[9] Hallows W C,Yu W,Smith B C,et al.Sirt3 promotes the urea cycle and fatty acid oxidation during dietary restriction[J].Mol Cell,2011,41(2):139-149.DOI:10.1016/j.molcel.2011.01.002.
[10] Finley L W,Haigis M C.Metabolic regulation by SIRT3:implications for tumorigenesis[J].Trends Mol Med,2012,18(9):516-523.DOI:10.1016/j.molmed.2012. 05.004.
[11] Giralt A,Hondares E,Villena J A,et al.Peroxisome proliferators-activated receptor-gamma coactivator-1alpha controls transcription of the Sirt3 gene,an essential component of the thermogenic brown adipocyte phenotype[J].J Biol Chem,2011,286(19):16958-16966.DOI:10.1074/jbc.M110.202390.
[12] Schirmer H,Pereira T C,Rico E P,et al.Modulatory effect of resveratrol on SIRT1,SIRT3,SIRT4,PGC1α and NAMPT gene expression profiles in wild-type adult zebrafish liver[J].Mol Biol Rep,2012,39(3):3281-3289.DOI:10.1007/s11033-011-1096-4.
[13] Lombard D B,Alt F W,Cheng H L,et al.Mammalian Sir2 homolog SIRT3 regulates global mitochondrial lysine acetylation[J].Mol Cell Biol,2007,27(24):8807-8814.DOI:10.1128/MCB.01636-07.
[14] 张伟,杨志军,王群,等.红鳍东方鲀Sirt1基因的克隆及其原核表达[J].西北农林科技大学学报(自然科学版),2012,40(8):54-59.
[15] 张伟,李翠萍,杨志军,等.红鳍东方鲀FoxO1基因的原核表达及纯化[J].湖南农业大学学报(自然科学版),2016,42(1):81-84.DOI:10.13331/j.cnki.jhau.2016. 01.016.
[16] Onyango P,Celic I,McCaffery J M,et al.SIRT3,a human SIR2 homologue, is an NAD-dependent deacetylase localized to mitochondria[J].Proc Natl Acad Sci USA,2002,99(21):13653-13658.DOI:10.1073/pnas. 222538099.
[17] Ribas A V,Ho P L,Tanizaki M M,et al.High-level expression of tetanus toxin fragment C-thioredoxin fusion protein in Escherichia coli[J].Biotechnol Appl Biochem,2000,31(2):91-94.DOI:10.1042/ ba19990084.
[18] Zhao J H,Xu Z,Hua Z C.Expression of human cardiac-specific homeobox protein in Escherichia coli[J].Protein Expr Purif,2000,18(3):316-319. DOI:10.1006/prep.2000.1198.
责任编辑:王赛群
英文编辑:王 库
Cloning and prokaryotic expression of silencing deacetylase SIRT3 gene from Takifugu rubripes
Zhang Wei, Ji Mingli, Zhang Junhua, Qian Zhibin
(School of Basic Medical Science, Xinxiang Medical University, Xinxiang, Henan 453003, China)
In order to further study the function of gene SIRT3 in Takifugu rubripes, the full-length open reading frame(ORF) of gene T. rubripes SIRT3 (trSIRT3) was amplified from total RNA in their liver tissue through reverse transcription-polymerase chain reaction (RT-PCR). The recombinant expression vector pET32a/SIRT3 was generated from the prokaryotic expression vector pET32a (+) derived from sub-cloned ORF, then, it was transformed into E.coli Rosetta (DE3). As the result of IPTG induction, SIRT3 was successfully expressed in E.coli Rosetta (DE3). Result of sequence analysis showed that the ORF of SIRT3 was 1 263 bp and encoded with 420 amino acids. The prokaryotic expression system of recombined vector pET-32a/SIRT3 was successfully constructed. From the results of sodium dodecyl sulfate-polyacrylamide gel electrophoresis showed that the recombinant protein had an approximate molecular weight of 66 000, which was consistent with the theoretical molecular weight. The fusion protein with tag His, was mainly detected in the supernatant.
Takifugu rubripes; silencing deacetylase; SIRT3 gene; gene cloning; prokaryotic expression
张伟(1977—),男,河南新乡人,博士,讲师,主要从事水产化学与生物技术研究,zhangwei0920@163.com
Q786;S917
A
1007-1032(2016)04-0424-05
2016-01-15 修回日期:2016-04-20
河南省教育厅重点项目(14A310027);新乡医学院高学历人才启动基金(505001)
