3个群体方斑东风螺线粒体COI基因的遗传多样性分析
2016-08-10朱丹丽王晓清曾志南秦溱熊钢曾丹严璐琪
朱丹丽,王晓清*,曾志南,秦溱,熊钢,曾丹,严璐琪
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.水产高效健康生产湖南省协同创新中心,湖南 常德415000;3.福建省水产研究所,福建 厦门 361000;4.湖南生物机电职业技术学院,湖南 长沙 410127)
3个群体方斑东风螺线粒体COI基因的遗传多样性分析
朱丹丽1,2,王晓清1,2*,曾志南3*,秦溱1,2,熊钢1,4,曾丹1,2,严璐琪1,2
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.水产高效健康生产湖南省协同创新中心,湖南 常德415000;3.福建省水产研究所,福建 厦门 361000;4.湖南生物机电职业技术学院,湖南 长沙 410127)
采用 PCR产物直接测序法对来自福建东山、广东湛江和广西北海的 3个群体方斑东风螺(Babylonia areolata)的COI基因进行比较分析。结果表明:COI基因片段长度为680 bp,碱基组成显示A+T比例较高,约为62.42%;种群间多态位点数为32,单倍型多样性指数为0.707±0.092,核苷酸多态性指数为0.001 85±0.000 39,平均核苷酸差异数为1.252。以泥东风螺(Babylonia lutosa)和织纹螺(Nassarius)为外群,构建了NJ系统发育树,其拓扑结构显示,3个群体的方斑东风螺首先聚类到一起,然后与泥东风螺聚为一支,与织纹螺形成不同的分支。
方斑东风螺;COI基因;单倍型;遗传多样性
投稿网址:http://xb.ijournal.cn
方斑东风螺(Babylonia areolata)简称东风螺,俗称花螺,隶属于腹足纲,新腹足目(Neogastropoda),蛾螺科(Buccinidae),东风螺属(Babylonia)[1]。在中国主要分布于福建、广东、广西和海南等省的沿海地区[2],是中国境内分布的几种东风螺中个体较大、生长较快、抗病能力强的一种优质贝类。近年来,由于市场需求量增加,过度捕捞造成方斑东风螺的自然资源锐减[3]。方斑东风螺现已发展成为新兴的海水养殖对象,在中国的海南、广东和福建等省,方斑东风螺养殖行业呈上升趋势。
线粒体DNA(mitochondrial DNA,mtDNA)是承载线粒体遗传密码的物质,主要呈双链环状。mtDNA表现为母系遗传,进化速度快且不易发生重组现象。动物体内的线粒体基因(mtDNA)是唯一的核外遗传信息载体,严格遵守母系遗传,已被广泛应用于多种动物类群的系统进化研究。细胞色素氧化酶I亚基(cytochrome oxidasesubunit I,COI)是线粒体氧化呼吸链的重要成员。由于COI基因变异较大[4],其序列已被广泛应用于贝类的种质鉴定[5-6]、种群遗传结构分析[7]和分子系统发育分析[8]等领域。
笔者对福建东山、广东湛江和广西北海3个群体方斑东风螺的线粒体COI基因进行克隆和分析,并对其遗传变异进行比较研究,分析其序列组成特性、系统关系以及序列多态性,旨在为进一步研究其遗传分析、种质鉴定和系统进化等提供参考依据。
1 材料与方法
1.1材料
1.1.1试验动物
试验用方斑东风螺采自福建东山(DSYF)、广东湛江(ZJYF)和广西北海(BHYF)3个地区,采集数量分别为 27、25、22个,且均为养殖群体。测定采集样品的形态指标,然后进行编号,放入-20 ℃冰箱保存。
1.1.2试剂
总DNA提取采用索莱宝柱式动物DNA OUT试剂盒,PCR扩增用Mix为全式金提供的2*Easy Taq PCR Supermix(+dye),切胶回收为全式金提供的EasyPure Quick Gel Extraction Kit琼脂糖凝胶回收试剂盒。
1.1.3引物
COI基因引物序列为L1490(5'-GGTCAACAA ATCATAAAGATATTGG-3')和H2198(5'-TAAAC TTCAGGGTGACCAAAAAATCA-3')[9],均由上海铂尚生物技术有限公司合成。
1.2方法
1.2.1基因组DNA的提取
取方斑东风螺腹足约100 mg,按照索莱宝柱式动物 DNA OUT 试剂盒提示的操作步骤提取总DNA。总DNA 用1% 琼脂糖凝胶电泳检测,-20 ℃保存,备用。
1.2.2目的片段的PCR扩增和测序
PCR反应体系总体积25 μL,其中DNA模板1 μL,正反引物各0.5 μL,Mix 12.5 μL,加水至25 μL。
PCR扩增反应条件:94 ℃预变性5 min;94 ℃变性45 s,52 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸5 min。
PCR产物经1% 琼脂糖电泳、切胶和回收纯化后,送上海铂尚生物技术有限公司进行双向测序。
1.2.3序列分析
测序获得的片段先用BLAST在线比对,确定片段的可靠性,然后由 Clustral[10]进行多重比对分析,并用DNAman软件分析序列长度和计算碱基组成,用DnaSp 4.0[11]软件分析多态数位点(singleton polymorphic sites)、单倍型多样性(haplotype diversity,Hd) 、核苷酸多样性(nucleotide diversity,Pi) 等遗传多样性参数,最后以泥东风螺(Babylonia lutosa)和织纹螺(Nassarius)为外群,用MEGA6.0[12]软件构建NJ系统发育树,并进行同源性分析和遗传距离分析。
2 结果与分析
2.13个群体方斑东风螺COI基因片段的碱基组成
通过PCR扩增,经过双向直接测序,测得COI基因的序列长度约为710 bp。通过在线BLAST比对,证实所得序列为方斑东风螺COI基因片段。通过Clustal X多重序列比对分析后,除去引物及部分杂合序列,最终获得大小约为680 bp的COI基因片段。
由表1可知,3个群体方斑东风螺COI基因序列中的碱基组成均显示A+T所占比例较高,存在着明显的A/T碱基偏向,并且C的含量最低。

表1 3个群体方斑东风螺COI基因片段的碱基组成Table 1 The base composition of COI gene fragments from three different groups of Babylonia areolata
2.2序列多态性分析结果
遗传多样性检测结果如表2所示。3个群体间存在的多态位点数为 7~18,单倍型多态性指数为(0.459±0.119)~(0.840±0.078),核苷酸多态性指数为(0.000 76±0.000 23)~(0.002 52±0.000 49),平均核苷酸差异数为 0.519~1.710。遗传多样性分析结果显示:3个群体方斑东风螺的核苷酸多样性指数以东山方斑东风螺的最低,以北海方斑东风螺的最高。北海养殖群体的遗传多样性最高,其次是湛江养殖群体,东山养殖群体的遗传多样性最低。

表2 3个群体方斑东风螺COI基因片段的遗传多样性参数Table 2 Genetic diversity parameters of COI gene fragments among three different groups of Babylonia areolata
由表3可知,在3个群体方斑东风螺的COI基因序列中检测出了28个单倍型,不计插入和缺失,共发现了32个变异位点,其中简约信息位点5个,转换位点数30个,颠换位点数2个,平均转颠换比为15。转换方式以A-G(12个)为主,C-T(8个)其次,然后是T-C(6个),G-A(4个)最少;颠换方式只有G-C(2个)。由表4可知,具有单倍型Hap1的个体有40个,为优势单倍型;具有单倍型Hap26的个体有4个;具有单倍型Hap10和Hap16的个体均有3个;其余单倍型均为1个个体。从方斑东风螺COI基因序列的组成来看,群体间的单倍型交流较少。东山群体的样本数虽然最多,但是其单倍型数最少;北海群体样本数虽然最少,但其单倍型数最多。

表3 3个群体方斑东风螺COI基因的单倍型及变异位点位置Table 3 Haplotypes and variable loci of COI gene in three different groups of Babylonia areolata

表3 (续)
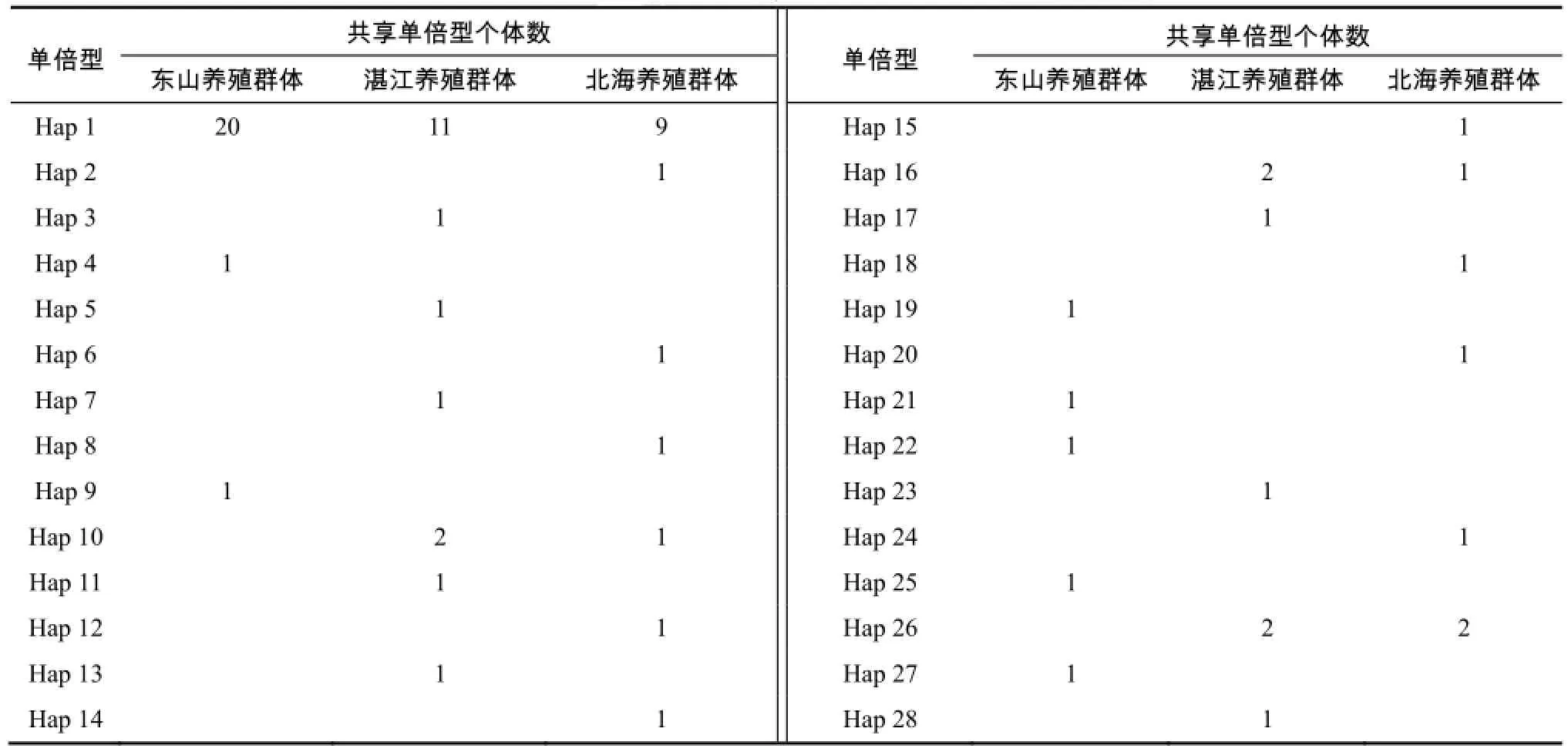
表4 3个群体方斑东风螺COI基因的共享单倍型个体数Table 4 Number of shared haplotypes of COI gene in three different groups of Babylonia areolata
2.3遗传距离与聚类分析结果
由表5可知,东山群体内遗传距离为0.001,湛江群体内遗传距离为 0.002,北海群体内遗传距离为0.003,3个群体间的遗传距离为0.002~0.005。3个群体方斑东风螺与泥东风螺的遗传距离为0.127~0.129,与织纹螺的遗传距离为0.181~0.183。

表5 3个群体方斑东风螺及其外群之间COI基因的遗传距离Table 5 Distance matrix among three different groups of Babylonia areolata and outgroups on COI gene fragments
拓扑结构显示3个养殖群体的方斑东风螺首先聚类到一起,然后与泥东风螺聚为一支,与织纹螺形成不同的分支。3个群体之间无明显分化现象。本结果与遗传多样性指数分析结果相一致。贝叶斯推断树相应节点的后验概率值为0.001~0.099。

图1 3个群体方斑东风螺COI基因的NJ系统发育树Fig.1 NJ phylogenetic trees based on COI gene fragments from three different groups of Babylonia areolata and outgroups
3 结论与讨论
对于任何一个物种而言,遗传多样性越丰富,说明该物种对环境变化的适应能力和扩展种群分布范围的能力越强[13]。3个养殖群体方斑东风螺的COI基因序列碱基组成均显示较高的A+T比例,这与苏天凤等[5]对来自粤东和粤西方斑东风螺和来自台湾东风螺的线粒体16S rRNA和COI基因片段的序列分析结果基本一致,且与其他贝类COI基因的分析结果也一致[13-17]。Folmer等[9]认为,A+T含量高的线粒体基因在进化中更具优势。本研究中,3个群体方斑东风螺的A+T占比为62.42 %,说明该物种具有较高的遗传变异能力和进化优势,但其遗传多样性指数分析结果显示其具有较低的遗传多样性。导致这一现象的原因:一方面是由于长期人工繁殖导致亲螺的亲缘关系越来越接近,减少了地理隔离造成的遗传分化;另一方面是因为方斑东风螺自身的奠基者效应或近期发生的瓶颈效应。此外,人类活动对方斑东风螺生存环境的影响也不容忽视,如对方斑东风螺栖息环境的破坏和近年来的过度捕捞,使野生种群日趋缩小[18],导致方斑东风螺遗传多样性水平较低。
3个群体方斑东风螺的核苷酸多样性指数为0.001 85±0.000 39。当核苷酸多样性指数为0.001~0.004 7时,表明该物种种群的遗传多样性较低[19]。从遗传距离来看,3个群体间遗传距离的差异不明显。聚类分析结果显示,东山群体与湛江群体首先聚类到一起,然后再与北海群体聚到一起,且三者之间无明显分化。从地理位置看,福建东山临东海,而广东湛江和广西北海地处南海,尽管这与遗传多样性指数和聚类分析结果不相符,但从整体而言,东海与南海以台湾岛南端和闽粤两省交界处的连线为界,两海其实南北相连,都属北太平洋西部的陆缘海。随着方斑东风螺苗种繁育技术和成螺养殖技术的日趋成熟,长期人工养殖导致方斑东风螺群体内的遗传分化较少,因此,体现出较小的遗传差异。本研究中的3个群体均为养殖群体,其遗传差异较小也是符合常理的。
综上所述,开展方斑东风螺种群遗传多样性的研究和资源保护刻不容缓。首先,要加强渔政执法,规定起捕规格,控制捕捞强度,实行严格的禁渔期制度,以逐步恢复方斑东风螺的野生资源;其次,应开展方斑东风螺种群遗传多样性研究,以便为其种群保护和遗传多样性监测提供依据;最后,建议每年多进行一些野外放流,在苗种养殖场进行人工繁育时尽量采用遗传距离较远的亲本,避免近交繁育带来的遗传衰退,以利于保护其遗传多样性。
[1] 王如才.中国水生贝类原色图鉴[M].杭州:浙江科学技术出版社,1988:92.
[2] 张汉华,吴进锋,陈利雄,等.东风螺人工育苗、养殖及产业化发展前景[J].南方水产科学,2004(11):2-5.
[3] 陈利雄,吴进锋.东风螺的增养殖技术及产业化前景[J].齐鲁渔业,2004,21(10):9-11.
[4] Hoeh WR,Stewart DT,Sutherland BW,et al. Cytochrome c oxidase sequence comparisons suggest an unusually high rate of mitochondrial DNA evolution in Mytilus (Mollusca:Bivalvia)[J].Mol Biol Evol,1996,13(2):418-421.DOI:10.1093/oxfordjournals.molbev.a025600.
[5] 苏天凤,黄建华,吴进锋,等.2种东风螺线粒体基因序列的多态性研究[J].中国水产科学,2007,14(3):369-376.DOI:10.3321/j.issn:1005-8737.2007.03.004.
[6] 陈丽梅,孔晓瑜,喻子牛,等.3种蛏类线粒体16S rRNA 和 COI基因片段的序列比较及其系统学初步研究[J].海洋科学,2005,29(8):27-32.DOI:10.3969/j.issn. 1000-3096.2005.08.008.
[7] King TL,Eackles MS,Gjetvaj B,et al.Intraspecific phylogeography of Lasmigona subviridis (Bivalvia:Unionidae): conservation implications of range discontinuity[J].Mol Ecol,1999,8(12 Suppl 1):65-78.DOI:10.1046/j.1365-294x.1999.00784.x.
[8] An HS,Jee YJ,Min KS,et al.Phylogenetic analysis of six species of Pacific abalone (Haliotidae) based on DNA sequences of 16s rRNA and cytochrome c oxidase subunit I mitochondrial genes[J].Mar Biotechnol,2005,7(4):373-380.DOI:10.1007/s10126-004-4405-2.
[9] Folmer O,Black M,Hoeh W,et al.DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J].Mol Marine Biol Biotechnol,1994,3(5):294-299.
[10] Thompson JD,Gibson TJ,Plewniak F,et al.The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24):4876-4882.DOI:10.1093/nar/25.24.4876.
[11] Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.DOI:10.1093/bioinformatics/btp187.[PubMed]
[12] Kumar S,Tamura K,Jakobsen IB,et al.MEGA2:molecular evolutionary genetics analysis software[J]. Bioinformatics,2001,17(12):1244-1245.
[13] 陈灵芝.中国的生物多样性现状及其保护对策[M].北京:科学出版社,1993:99-113.
[14] 苏天凤,江世贵,朱彩艳,等.广西钦洲湾养殖牡蛎线粒体16SrRNA基因片段序列变异分析[J].中国水产科学,2005,12(1):1-4.
[15] 王琳楠,闫喜武,秦艳杰,等.中国帘蛤目16种经济贝类DNA条形码及分子系统发育的研究[J].大连海洋大学学报,2013,28(5):431-437.DOI:10.3969/j.issn. 2095-1388.2013.05.004.
[16] 刘亚军,喻子牛,姜艳艳,等.栉孔扇贝 16S rRNA基因片段序列的多态性研究[J].海洋与湖沼,2002,33(5):477-483.DOI:10.3321/j.issn:0029-814X .2002 .05.004.
[17] Zheng Xiaodong,Wang Rucai,Xiao Shu,et al.Genetic diversity in populations of sepiella maindroni using 16S rRNA gene sequence analysis[J] .High Technology Letters,2003,9(1):1-4.
[18] 董津茂,蔡如星.浙江动物志(软体动物)[M].杭州:浙江科学技术出版社,1991:17-115.
[19] Lan H,Shi L.The origin and genetic differentiation of native breeds of pigs in southwest China:an approach from mitochondrial DNA polymorphism[J].Biochem Genet,1993,31(1-2):51-60.DOI:10.1007/bf02399819.
责任编辑:王赛群
英文编辑:王 库
Genetic polymorphisms of three different groups of Babylonia areolata based on COI gene
Zhu Danli1,2, Wang Xiaoqing1,2*, Zeng Zhinan3*, Qin Qin1,2, Xiong Gang1,4, Zeng Dan1,2, Yan Luqi1,2
(1.College of Animal Science and Technology, Hunan Agricultural University, Changsha 410128, China; 2.Collaborative Innovation Center for Aquatic Efficient Health Production of Fisheries in Hunan Province, Changde, Hunan 415000,China; 3.Fisheries Research Institute of Fujian, Xiamen, Fujian 361000, China; 4.Hunan Biological Electromechanical Vocational and Technical College, Changsha, 410127, China)
We analyzed and compared the polymorphism of mitochondrial COI gene sequence derived from three different groups of Babylonia areolata in Dongshan, Zhanjiang and Beihai by means of PCR. The results showed that the nucleotide sequence of COI gene was 680 bp with a high percentage of A+T base composition (about 62.42%); there were 32 singleton poylmorphic sites, 0.707±0.092 haplotype diversity, 0.001 85±0.000 39 nucleotide polymorphisms index, and 1.252 average number of nucleotide differences. Taking Babylonia lutosa and Nassarius as outgroups, we built NJ phylogenetic tree, its topology suggested that all the Babylonia areolata clustered together first, then mixed with Babylonia lutosa for one, and finally Nassarius formed independent.
Babylonia areolata; COI gene; phyletic evolution; genetic polymorphism
朱丹丽(1991—),女,上海人,硕士研究生,主要从事水产养殖研究,463609105@qq.com;*通信作者,王晓清,博士,教授,主要从事水产动物育种研究,wangxiao8258@126.com;*通信作者,曾志南,博士,研究员,主要从事海水养殖和贝类遗传育种研究,xmzzn@sina.com
Q959.212
A
1007-1032(2016)04-0429-06
2015-11-02 修回日期:2016-06-20
海洋公益性行业科研专项经费项目(201205021-1);国家自然科学基金项目(31372530)
