广西普通野生稻遗传多样性中心的确定与核心种质构建
2016-08-05薛艳霞梁燕理黄金艳覃宝祥邱永福李容柏
薛艳霞, 梁燕理, 冯 璇, 黄金艳, 刘 芳, 覃宝祥, 邱永福, 李容柏
(广西大学 农学院/亚热带农业生物资源保护与利用国家重点实验室, 广西 南宁530004)
广西普通野生稻遗传多样性中心的确定与核心种质构建
薛艳霞, 梁燕理, 冯璇, 黄金艳, 刘芳, 覃宝祥, 邱永福, 李容柏
(广西大学 农学院/亚热带农业生物资源保护与利用国家重点实验室, 广西 南宁530004)
摘要:【目的】确定广西普通野生稻Oryza rufipogon Griff.遗传多样性中心,构建普通野生稻核心种质资源,为广西普通野生稻资源保护利用提供参考资料。【方法】利用24 对微卫星标记分析来自郁江流域、红水河流域、南流江流域和桂北山区的普通野生稻623份材料的遗传多样性;采用逐步聚类法构建10%和5%的广西普通野生稻核心种质。【结果】24个SSR位点总共检测到114个等位基因。平均等位基因数为4.75,平均有效等位基因数为3.000 1,Shannon信息指数为1.180 1,平均期望杂合度为0.638 8。9个居群遗传多样性指数为:邕宁居群>临桂居群>扶绥居群>容县居群>贵港居群>平南居群>古棚居群>五里塘居群>博白居群。4个区域的多样性指数为:郁江流域>桂北山区>南流江流域>红水河流域。广西普通野生稻资源5%的核心样本共31份,其中邕宁居群有14份,扶绥居群有12份;邕宁居群和扶绥居群分别占本居群分析样本的5.76%和18.75%,是核心样本的主要来源。【结论】郁江流域是广西普通野生稻多样性中心;邕宁居群的普通野生稻地理分布广、种类丰富,而扶绥居群遗传多样性异常丰富,它们是核心样本的主要来源,也是值得特别关注和重点保护的重要区域。
关键词:普通野生稻; SSR; 遗传多样性; 核心种质; 广西
普通野生稻OryzarufipogonGriff. 是亚洲栽培稻O.sativaL.的祖先种,经过长期的自然选择,蕴藏着大量可供育种利用的基因资源[1]。因此,保护和利用普通野生稻是该领域科研工作者关注的重点。许多研究表明,我国普通野生稻遗传多样性丰富且地域特性明显[2]。王美兴等[3]利用36对SSR引物对889份中国普通野生稻进行遗传多样性分析,平均多态性指数为0.782 7,提出广东和广西的普通野生稻具有较高的遗传多样性。韩东飞[4]按照纬度高低对中国境内20个居群的628 份野生稻材料进行遗传多样性研究,发现我国普通野生稻具有较高的遗传多样性并提出两广南部北回归线以南的居群遗传多样性较高。对全国不同省份普通野生稻遗传多样性的估值也表明,广西普通野生稻具有丰富的遗传多样性[5-10]。近年来,一些学者对广西普通野生稻遗传多样性有不同程度的研究[11-17]。余萍等[11]利用34对SSR引物对中国普通野生稻初级核心种质中223份广西普通野生稻进行遗传多样性分析,平均等位变异数为24.91。黄娟等[12]利用36对SSR引物对广西27个居群690份普通野生稻材料进行遗传多样性分析,平均等位基因数为9.86,并把广西区域的普通野生稻划分为3个类群4个特征区域。但由于所研究群体的规模、分布及材料代表性等的不同, 导致不同学者对广西普通野生稻遗传多样性的评价各异。在遗传多样性评价的基础上建立普通野生稻核心种质的研究鲜见报道。2012年Huang等[18]比较了世界各国和我国各省区的普通野生稻资源,认为广西是我国普通野生稻遗传多样性中心和栽培稻的起源中心,但广西的普通野生稻多样性中心具体在哪里没有进行追踪。
本研究对来自广西的郁江流域、红水河流域、南流江域以及桂北山区的9个居群进行遗传多样性分析,并在此基础上构建了广西普通野生稻的核心种质。以期对广西野生稻遗传多样性进行有效的保护和利用,为普通野生稻遗传多样性形成机制的研究、以及优异基因资源的发掘和利用提供理论依据和核心材料。
1材料与方法
1.1材料
从郁江流域、红水河流域、南流江域和桂北山区的9个居群收集623份材料(表1)。
表1来自广西4个区域的普通野生稻材料
Tab.1Materials of common wild rice from four regions in Guangxi
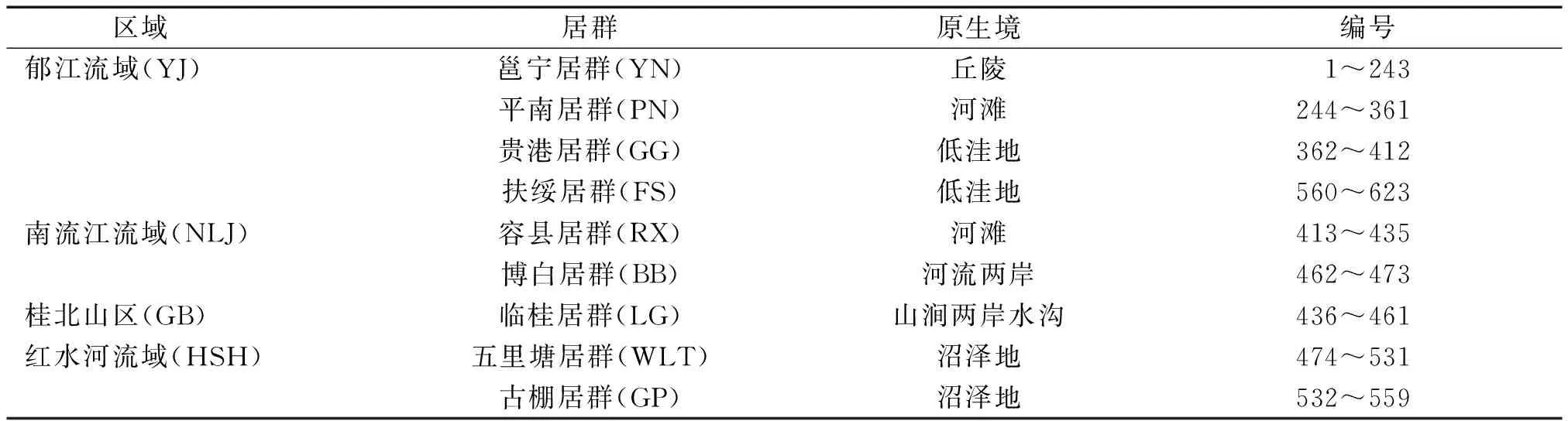
区域居群原生境编号郁江流域(YJ)邕宁居群(YN)丘陵1~243平南居群(PN)河滩244~361贵港居群(GG)低洼地362~412扶绥居群(FS)低洼地560~623南流江流域(NLJ)容县居群(RX)河滩413~435博白居群(BB)河流两岸462~473桂北山区(GB)临桂居群(LG)山涧两岸水沟436~461红水河流域(HSH)五里塘居群(WLT)沼泽地474~531古棚居群(GP)沼泽地532~559
1.2方法
1.2.1DNA提取水稻叶片DNA提取参考Chen等[ 19 ]的CTAB法。
1.2.2多态性标记筛选从水稻基因组SSR图谱中,挑选出均匀分布在水稻12条染色体上的且能扩增出多态性的24对引物(表2)进行PCR分析。SSR标记引物由上海生工生物工程技术服务有限公司合成。
1.2.3PCR扩增PCR体系20 μL: 10×PCR Buffer(含15 mmol·L-1MgCl2) 2.0 μL, 10 mmol·L-1dNTP 0.4 μL,15 μmol·L-1正反引物各1.0 μL,5 U·μL-1Taq酶0.2 μL, 25 ng ·μL-1DNA 1.0 μL, ddH2O 14.4 μL。PCR反应程序:94 ℃预变性5 min, 94 ℃变性30 s, (50~57) ℃退火45 s, 72 ℃延伸30 s,共40个循环, 72 ℃延伸5 min。扩增产物在60 g·L-1的非变性聚丙烯酰胺凝胶上恒压电泳, 用银染法染色后拍照。用图形处理软件Photoshop中的标尺工具和参考工具分析图片中的DNA片段,扩增谱带均以“0”或“1”建立数据库,扩增带存在时记为“1”,不存在时记为“0”,最后将条带数据转换为基因型数据。
1.2.4多样性分析根据每个个体产生的条带位置确定基因型,采用Popgene统计各居群的平均等位基因数(A), 有效等位基因数(Ae),Shannon 信息指数(I),平均期望杂合度(He),实际观察杂合度(Ho),平均多态信息含量(PIC)。运用Popgen软件进行了遗传距离和遗传一致度分析。采用Upgma方法运行Ntsys2.10软件的Shan程序进行聚类分析。
1.2.5核心种质构建核心种质构建参考Hu等[20]提出的逐步聚类法。在利用24对SSR标记对623份材料进行遗传多样性分析的基础上,进行第1次聚类分析,遗传距离相同或极相近的同类材料只选1个代表编号,得到初次聚类核心样本;利用96对SSR标记对初次核心样本进行第2次聚类分析,得到10%核心样本,构建出10%核心聚类图;对第2次的10%核心样本进行第3次聚类分析,得到5%核心样本,构建出5%核心聚类图。进一步分析核心样本的遗传多样性特征、地理分布、来源等。
2结果与分析
2.1广西普通野生稻不同基因位点的遗传多样性
利用筛选出的24对SSR引物对623份材料进行遗传多样性分析,相关结果列于表2。 从表2可以看出,24个SSR位点共检测到114个等位变异,每个位点的平均等位基因数(A)最高为7(RM586),最低为3(RM537),平均为4.75;有效等位基因数(Ae)最高为4.476 3(RM257),最低为1.161 7(RM330A),平均为3.000 1;Shannon信息指数(I)最高为1.544 3(RM257),最低为0.313 0 (RM330A),平均为1.180 1;实际观察杂合度(Ho)最高为0.566 6(RM257),最低为0.041 7(RM528),平均为0.175 8;平均期望杂合度(He)最高为0.777 2(RM257),最低为0.139 3(RM330A),平均为 0.638 8。
表2广西普通野生稻遗传多样性参数1)
Tab.2Parameters of genetic diversity of common wild rice in Guangxi
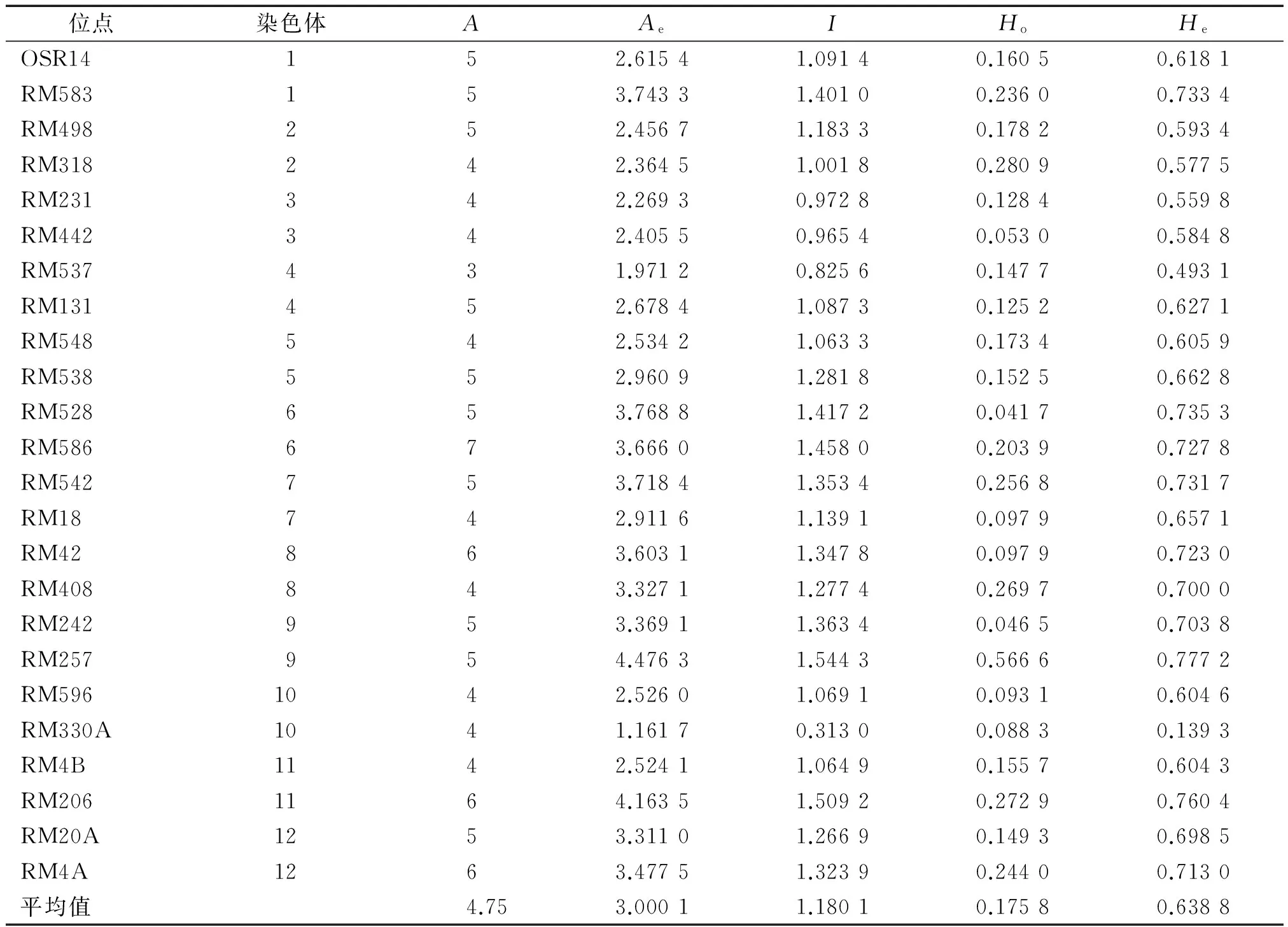
位点染色体AAeIHoHeOSR14152.61541.09140.16050.6181RM583153.74331.40100.23600.7334RM498252.45671.18330.17820.5934RM318242.36451.00180.28090.5775RM231342.26930.97280.12840.5598RM442342.40550.96540.05300.5848RM537431.97120.82560.14770.4931RM131452.67841.08730.12520.6271RM548542.53421.06330.17340.6059RM538552.96091.28180.15250.6628RM528653.76881.41720.04170.7353RM586673.66601.45800.20390.7278RM542753.71841.35340.25680.7317RM18742.91161.13910.09790.6571RM42863.60311.34780.09790.7230RM408843.32711.27740.26970.7000RM242953.36911.36340.04650.7038RM257954.47631.54430.56660.7772RM5961042.52601.06910.09310.6046RM330A1041.16170.31300.08830.1393RM4B1142.52411.06490.15570.6043RM2061164.16351.50920.27290.7604RM20A1253.31101.26690.14930.6985RM4A1263.47751.32390.24400.7130平均值4.753.00011.18010.17580.6388
1)A:平均等位基因数;Ae:有效等位基因数;I:Shannon信息指数;Ho:实际观察杂合度;He:平均期望杂合度。
2.2广西普通野生稻不同居群间遗传多样性比较
9个居群的遗传多样性和地理分布见图1和表3。从图1可以看出:邕宁、扶绥、贵港和平南属于郁江流域,临桂属于桂北山区,古棚和五里塘属于红水河流域,博白和容县属于南流江域。从表3可以看出,9个普通野生稻居群的遗传多样性指数大小顺序为:邕宁居群(YN)>临桂居群(LG)>扶绥居群(FS)>容县居群(RX)>贵港居群(GG)>平南居群(PN)>古棚居群(GP)>五里塘居群(WLT)>博白居群(BB)。不同流域居群A的变化范围为2.946 7(HSH)~4.010 8(YJ),Ae的变化范围为0.139 3(HSH)~9.512 1(YJ),He的变化范围0.001 3(HSH)~2.053 9(YJ)。A最大的为郁江流域(YJ),最小的为红水河流域(HSH),表明郁江流域(YJ)是广西普通野生稻的遗传多样性中心(表2)。
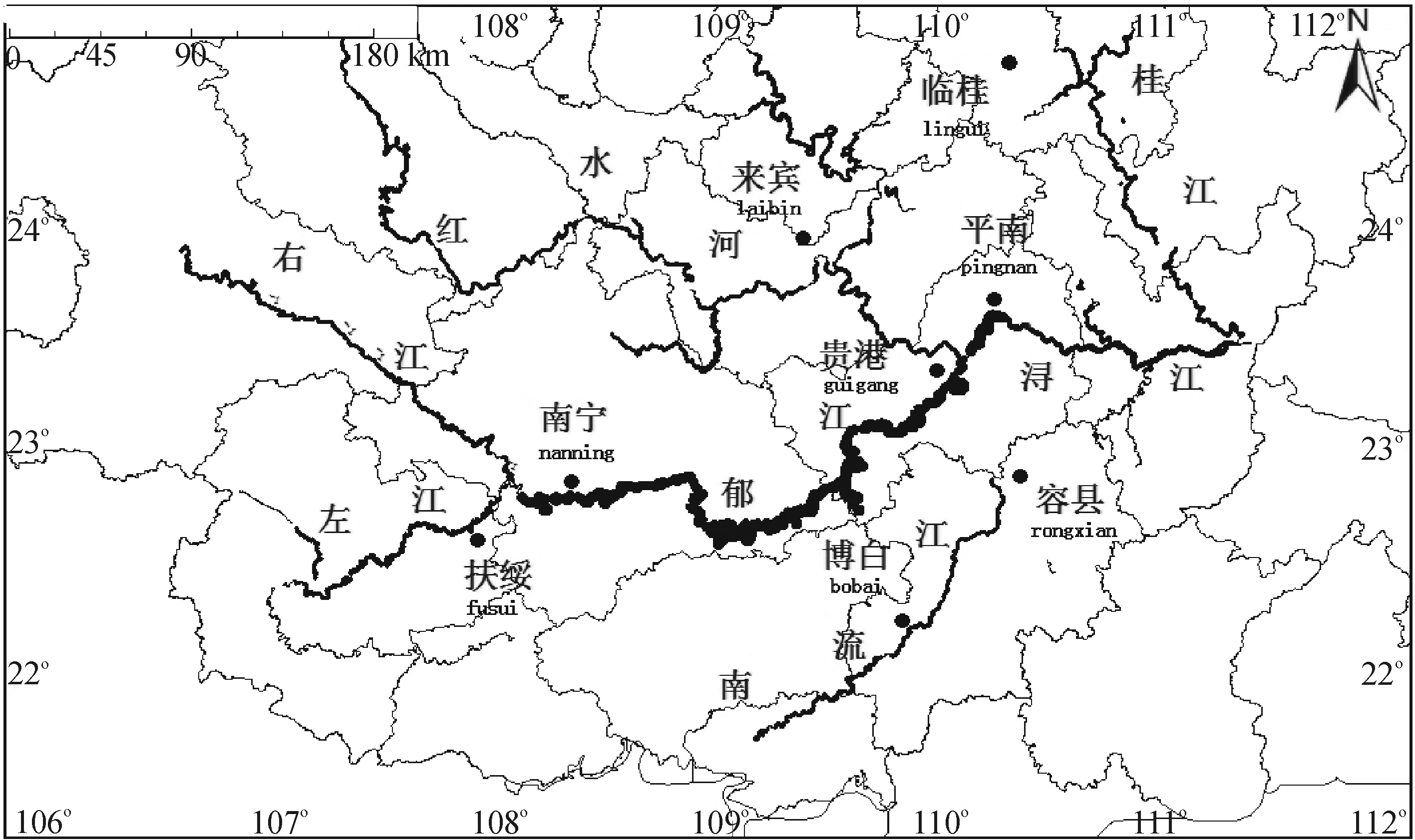
图1 广西普通野生稻9个主要居群的地理分布
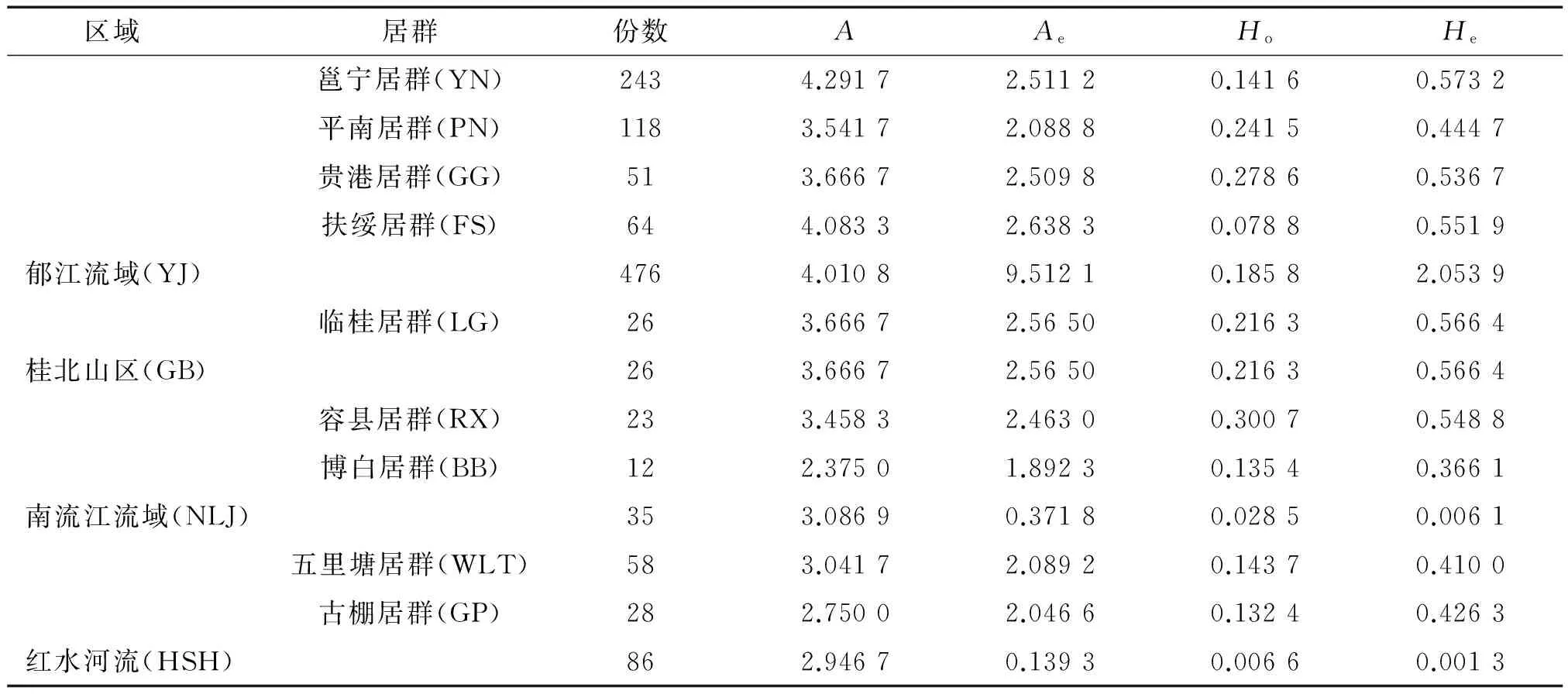
区域居群份数AAeHoHe邕宁居群(YN)2434.29172.51120.14160.5732平南居群(PN)1183.54172.08880.24150.4447贵港居群(GG)513.66672.50980.27860.5367扶绥居群(FS)644.08332.63830.07880.5519郁江流域(YJ)4764.01089.51210.18582.0539临桂居群(LG)263.66672.56500.21630.5664桂北山区(GB)263.66672.56500.21630.5664容县居群(RX)233.45832.46300.30070.5488博白居群(BB)122.37501.89230.13540.3661南流江流域(NLJ)353.08690.37180.02850.0061五里塘居群(WLT)583.04172.08920.14370.4100古棚居群(GP)282.75002.04660.13240.4263红水河流(HSH)862.94670.13930.00660.0013
1)A:平均等位基因数;Ae:有效等位基因数;Ho:实际观察杂合度;He:平均期望杂合度。
2.3广西普通野生稻核心种质初步构建
采取逐步聚类的方法,构建了10%和5%的核心样本,其遗传多样结果见表4、聚类分析结果见图2和图3。从表4和图2可以看出,10%的核心样本材料间遗传距离较近,为0.38,其A能保持原始样本的92%以上,Ae、I、He和平均多态信息含量(PIC)等4个参数比原始样本的分别高3.70%、2.86%、3.22%和2.46%。图3表明,5%核心样本的材料间遗传距离较远,达到了0.54,其A保持原始样本的89%以上,Ae和I比10%的核心样本有较大的下降,但仍分别比原始样本高2.12%和1.79%,He比原始样本高3.40%,比10%的核心样本也有所提高(表4)。说明5%的核心样本所取材料间的位点重复性小,但仍能保持原始样本极高的遗传多样性。
表4不同核心样本的遗传多样性比较1)
Tab.4Comparisons of genetic diversities of different core samples

样本AAeIHePIC10%核心样本4.37503.11121.21390.65940.65405%核心样本4.25003.06381.20120.66050.6498总样本4.75003.00011.18010.63880.6383
1)A:平均等位基因数;Ae:有效等位基因数;I:Shannon信息指数;He:平均期望杂合度;PIC:平均多态信息含量。
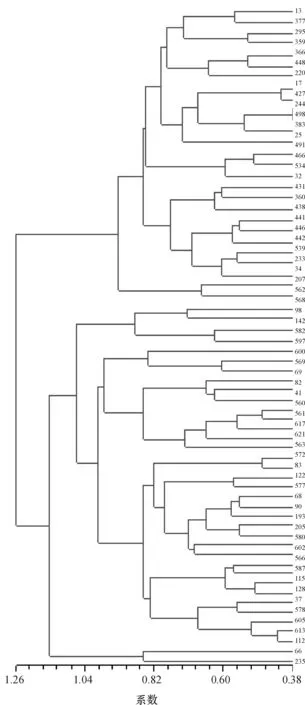
图2 广西普通野生稻10%核心样本的UPGMA聚类图
Fig.2UPGMA dendrograms of 10% core collections of common wild rice in Guangxi

图3 广西普通野生稻5%核心样本的UPGMA聚类图
Fig.3UPGMA dendrograms of 5% core collections of common wild rice in Guangxi
从表5可以得出:在10%的核心样本中,邕宁居群的核心样本比例是本居群分析样本的10.29%,与全体材料的总平均数一致,表明邕宁居群的核心样本数多的原因是该群供试样本数占总样本数的比例大;扶绥居群的核心样本比例是本居群分析样本的31.25%,远高于总平均数,表明扶绥居群的核心样本数多的原因是该群供试样本的遗传多样性特别丰富。5%的核心样本构成的变化趋势也一样。相比之下,博白居群、容县居群和五里塘居群占核心样本数均极低,表明其遗传多样性差。
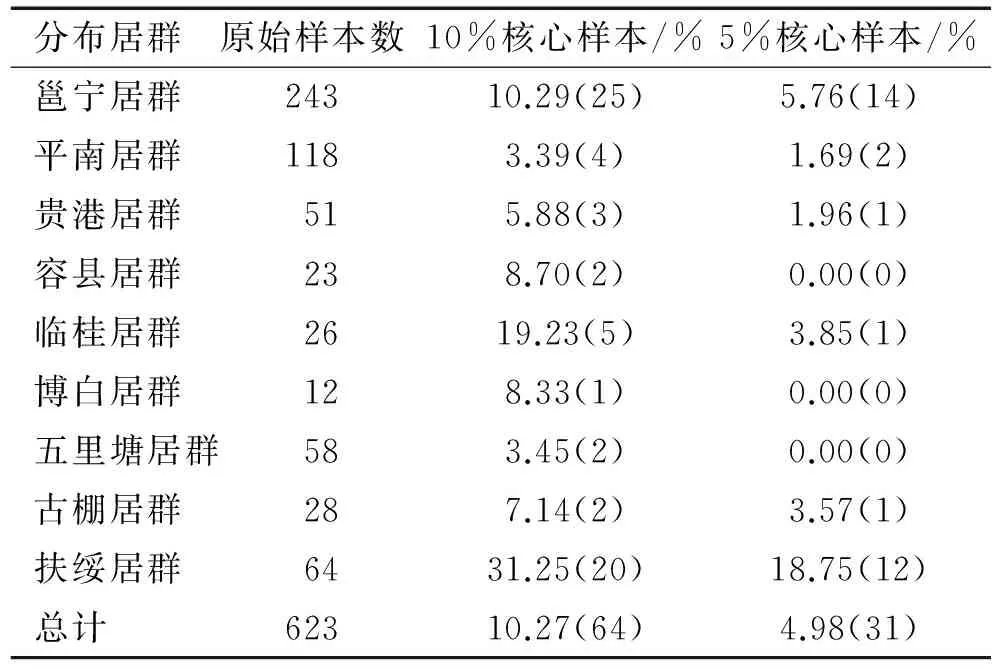
表5 各居群占核心样本的比例1)
1)括号内为样本数。
3讨论与结论
3.1广西普通野生稻的遗传多样性评价
本研究对郁江流域,红水河流域,南流江流域和桂北山区623份普通野生稻材料的遗传多样性分析结果(A=4.75、Ae=3.000 1、He=0.638 8)高于前人[5-9]对江西、湖南、福建、海南、云南等省区普通野生稻遗传多样性的估值,表明广西普通野生稻具有丰富的遗传多样性。本研究结果与任民等[14]对桂东南地区普通野生稻的研究和盖红梅等[13]对武宣濠江流域普通野生稻的遗传多样性研究的结果基本相符。但低于余萍等[11]和黄娟等[12]的研究结果,主要原因可以归纳为3点;一是由于所研究群体的规模、分布及材料代表性等的不同。余萍等[11]研究的材料是中国普通野生稻初级核心种质中的材料,因此遗传多样性相对较高。黄娟等[12]研究的材料主要来自玉林博白区和福棉区,北海银海区、合浦县和铁山港区,防城区,贺州钟山县,来宾上林、武宣及桂林雁山区等区域。均与本研究的取材地域不同。二是由于检测位点不同。不同检测位点对多样性的影响不同,在供试个体中分配的均匀性和多样性也不一样。余萍等[11]使用34对引物,黄娟等[12]使用36对引物但未平均分配在12条染色体上,因此遗传多样性相对较高。本研究使用平均分配于12条染色体的24对SSR引物,试验结果能更客观地反映广西普通野生稻的遗传多样性。三是取材时间不同。近年来,普通野生稻及其栖息地遭受严重破坏。据调查,广西野生稻分布点有31%已经完全毁灭,覆盖面积也急剧减少[21],这也可能造成遗传多样性的降低。
3.2广西普通野生稻遗传多样性中心的确定
广西是我国普通野生稻遗传多样性丰富的省区之一。2012年 Huang等[18]比较了世界各国和我国各省的普通野生稻资源,认为广西是我国普通野生稻遗传多样性中心和栽培稻的起源中心。黄娟等[12]将广西普通野生稻分布分为桂北山区、百色区、红水河-浔江流域和南流江流域4大区域。但广西的普通野生稻多样性中心究竟在哪里没有跟踪报道。本研究对郁江流域、桂北山区、南流江流域和红水河流域的9个居群623份材料进行遗传多样性分析,9个居群遗传多样性指数大小为:邕宁居群(YN)>临桂居群(LG)>扶绥居群(FS)>容县居群(RX)>贵港居群(GG)>平南居群(PN)>古棚居群(GP)>五里塘居群(WLT)>博白居群(BB)。区域的多样性指数大小为:郁江流域(YJ)>桂北山区(GB)> 南流江流域(NLJ)> 红水河流域(HSH)。表明普通野生稻遗传多样性异常丰富的郁江流域是广西普通野生稻的多样性中心。
3.3广西普通野生稻核心种质构建与资源分布、保护和利用
邓国富等[21]和徐志健等[22]年对广西野生稻自然资源开展第2次全面调查发现,由于原生地消失、原生地环境恶化和外来物种的入侵,广西野生稻自然资源濒危现状严重,31%的原生态分布点已经完全毁灭,覆盖面积也急剧减少。因此,核心种质构建的工作需要进一步加强。本研究使用逐步聚类法构建了10%和5%的核心样本。其平均等位基因数(A)能保持原始样本的92%和89%以上。邕宁居群和扶绥居群是核心样本的主要组成来源。邕宁居群的核心样本数多的原因主要是由于收集资源量多和遗传多样性较丰富,而扶绥居群的收集资源量相对较少但遗传多样性异常丰富,也表明扶绥居群是普通野生稻遗传多样性的特殊地区。郁江流域作为广西普通野生稻遗传多样的新中心,其分布分散、范围广,这使野生稻的保存及保护困难更大,且目前的野生稻保护区域都未涉及到这些地方,应当引起重视。
本研究构建了较完整和较有代表性的普通野生稻遗传多样性核心种质,普通野生稻(AA基因组)是栽培稻的近缘祖先,在长期的逆境生长中具有优异的育种基因资源,对水稻基础研究和水稻育种有巨大的贡献。Zhang等[23]和章琦等[24]通过抗谱评价、抗性类型比较以及遣传分析,鉴定出一个来自普通野生稻的抗白叶枯病新基因Xa-23,并将其进行了精细定位。Huang等[25]利用广西普通野生稻GX2183挖掘出抗褐飞虱基因Bph27。Wang等[26]利用广西普通野生稻转育后代RBPH54精细定位并克隆了抗褐飞虱基因Bph29。广西的2个优质 “三系”杂交水稻恢复系“桂99”和“测253”都是利用野生稻资源培育出来的[27-28]。利用广西普通野生稻培育基于优良栽培稻遗传背景的染色体片段代换系也获得丰富的特异类型[29-30]。由此可以肯定,广西普通野生稻核心种质的构建将进一步加速普通野生稻遗传多样性在水稻育种上的利用。
参考文献:
[1]FULLER D Q, SATO Y I, CASTILLO C,et al. Consilience of genetics and archaeobotany in the entangled history of rice[J]. Archaeol Anthropol Sci,2010,2(2):115-131.[2]杨庆文,黄娟.中国普通野生稻遗传多样性研究进展[J].作物学报,2013,39(4): 580-588.
[3]王美兴,张洪亮,张冬玲,等.中国普通野生稻(O.rufipogonGriff.)的地理多样性与分化[J]. 科学通报,2008,53(22):2768-2775.
[4]韩东飞. 中国普通野生稻(OryzarufipogonGriff.)的遗传多样性研究及栽培稻起源探讨[D].北京:中国农业科学院,2006.
[5]ZHOU H F, XIE Z W,GE S. Microsatelite analysis of genetic diversity and population genetic structure of a wild rice(0ryzarufipogonGril1.)in China[J]. Theor Appl Genet,2003,107(2):332-339.
[6]SONG Z P, XU X, WANG B, et al.Genetic diversity in the northernmost 0ryzarufipogonpopulations estimated by SSR markers[J]. Theor Appl Genet,2003,107(8):1492-1499.
[7]陈玲,殷富有,李维蛟,等.云南三种野生稻遗传多样性和系统进化研究[J].中国稻米,2014,20(3):30-35.
[8]齐兰,王效宁,张吉贞,等.利用SRAP标记研究海南野生稻的遗传多样性与遗传分化[J].植物遗传资源学报,2015,14(3):402-406.
[9]李杜娟,陈雨,潘大建,等.粤东地区普通野生稻表型多样性分析[J].广东农业科学,2012(2):13-17.
[10]孙希平,杨庆文,李润植,等. 海南三种野生稻遗传多样性的比较研究[J].作物学报,2007,33(7):1100-1107.
[11]余萍,李白超,张洪亮,等.广西普通野生稻(OryzarufipogonGril1.)表型性状和SSR多样性研究[J].遗传学报,2004,3l(9):934-940.
[12]黄娟,杨庆文,陈成斌,等.广西普通野生稻的遗传多样性及分布特征[J].中国农业科学,2009,42(8):2633-2642.
[13]盖红梅,陈成斌,沈法富,等.广西武宣濠江流域普通野生稻居群遗传多样性及保护研究[J].植物遗传资源学报,2005,6(2):156-162.
[14]任民,陈成斌,荣廷昭,等.桂东南地区普通野生稻遗传多样性研究[J].植物遗传资源学报,2005,6(1):31-36.
[15]黄金艳,陈淼,梁燕理,等.广西来宾市五里塘普通野生稻(OryzarufipogonGriff.)居群遗传多样性与核心种质研究[J].西南农业学报,2008,21(2):245-249.
[16]梁燕理,陈淼,刘驰,等.广西邕宁普通野生稻(OryzarufipogonGriff.) 种群遗传多样性及核心种质构建研究[J].安徽农业科学,2008, 36(11) : 4439- 4441.
[17]李亚非,陈成斌,张万霞,等.我国北回归线区域普通野生稻遗传多样性和遗传结构研究[J].植物遗传资源学报,2007,8(3):280-284.
[18]HUANG X H, NORI KURATA,WEI X H, et al.A map of rice genome variation reveals the origin of cultivated rice[J].Nature,2012,490(7421):497-501.
[19]CHEN D H, RONALD P C. A rapid DNA minipreparation method suitable for AFLP and other PCR applications[J].Plant Mol Biol Rep,1999,17(1):53-57.
[20]HU J, ZHU J, XU H M. Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J]. Theor Appl Genet,2000, 101(1/2): 264-268.
[21]邓国富,张宗琼,李丹婷,等.广西野生稻资源保护现状及育种应用研究进展[J].南方农业学报,2012,43(9):1425-1428.
[22]徐志健,陈成斌,梁世春,等.广西野生稻自然资源濒危现状评估报告[J].广西农业科学,2010,41(3):281-285.
[23]ZHANG J W,XIE J K,WAN Y. et al. Research progresson the protection of wild rice resources in China[J].Agr Sci Technol,2010,11(2):95-97.
[24]章琦,赵炳宇,赵开军,等.普通野生稻的抗水稻白叶枯病(Xanthomonasoryzaepv.oryzae)新基因Xa-23(t)的鉴定和分子标记定位[J].作物学报,2000,26(5):535-542.
[25]HUANG D,QIU Y,ZHANG Y, et al. Fine mapping and characterization ofBPH27, a brownplanthopper resistance gene from wild rice(OryzarufipogonGriff)[J].Theor Appl Genet,2013,126(1):219-229.
[26]WANG Y, CAO L M, ZHANG Y X. et al. Map-based cloning and characterization ofBPH29, a B3 domain-containing recessive gene conferring brownplanthopper resistance in rice[J]. J Exp Bot,2015,66(19):6035-6045.
[27]覃惜阴,韦仕邦,黄英美,等.杂交水稻恢复系桂99的选育与应用[J].杂交水稻,1994(2):1-3.
[28]莫永生,韦政,黎志方,等.杂交水稻强优广谱恢复系测253的选育与应用[J].作物杂志,2003(6):46-47.
[29]张月雄,颜群,黄大辉,等.利用单片段代换系鉴定水稻稻瘟病抗性座位[J].西南农业学报,2014,27(4):1478-1482.
[30]覃宝祥,刘立龙,韩飞怡,等.普通野生稻染色体片段代换系的孕穗期耐冷性研究[J].基因组学与应用生物学,2015,34(6):1283-1289.
【责任编辑周志红】
收稿日期:2016- 01- 12优先出版时间:2016- 07- 05
作者简介:薛艳霞(1982—), 女,博士研究生,E-mail:xueyanxia820824@163.com;通信作者:李容柏(1957—), 男,研究员,博士, E-mail:lirongbai@126.com
基金项目:广西科学技术与开发项目(桂科重14121001-1-5);广西研究生教育创新计划项目 (T3310098601);广西大学科研基金(XDZ110082)
中图分类号:S324
文献标志码:A
文章编号:1001- 411X(2016)05- 0024- 07
Center of genetic diversity and core collection of common wild rice,Oryza rufipogon Griff.,in Guangxi
XUE Yanxia, LIANG Yanli, FENG Xuan, HUANG Jinyan, LIU Fang, QIN Baoxiang, QIU Yongfu, LI Rongbai
(College of Agriculture, Guangxi University/State Key Laboratory for Conservation Utilization of Subtropical Agro-bioresources, Nanning 530004, China)
Abstract:【Objective】 To confirm genetic diversity distribution center of common wild rice,Oryza rufipogon Griff., in Guangxi, and construct the core collection which will provide a reference for protecting and utilizing wild rice resources in Guangxi. 【Method】 A total number of 623 common wild rice resources from Yujiang River Basin,Hongshui River Basin,Nanliujiang River Basin and Guibei Mountain were used to study the genetic diversity with 24 SSR markers. The stepwise cluster analysis was used to construct 10% and 5% core collections of common wild rice in Guangxi. 【Result】There were 114 alleles to be detected at 24 SSR loci. The number of alleles per locus (A) was 4.75, the average number of effective alleles (Ae) was 3.000 1, the Shannon-weaver information index (I) was 1.180 1 and the average expected heterozygosity (He) was 0.638 8. The order of genetic diversity of 9 regional populations was Yongning (YN) > Lingui (LG) > Fusui (FS) > Rongxian (RX) > Guigang (GG) > Pingnan (PN) > Gupeng (GP) > Wulitang (WLT) > Bobai (BB). The order of genetic diversity of four regions was Yujiang River Basin>Guibei Mountain > Nanliujiang River Basin > Hongshui River Basin. There were 31 copies belong to 5% core collection,including 14 copies of Yongning population and 12 copies of Fusui population which accounted for 5.76% and 18.75% of analyzed samples respectively. 【Conclusion】The genetic diversity center of Guangxi common wild rice is Yujiang River Basin. Wild rice of Yongning is widely distributed and presents various types. Wild rice of Fusui shows great rich genetic diversity. Yongning and Fusui regional populations,which are main sources of the core collection samples,are considered to be the most important populations of Guangxi common wild rice,and should be paid particular attention to their protection and exploitation.
Key words:Oryza rufipogon; SSR; genetic diversity; core collection; Guangxi
优先出版网址:http://www.cnki.net/kcms/detail/44.1110.s.20160705.1200.054.html
薛艳霞, 梁燕理, 冯璇,等.广西普通野生稻遗传多样性中心的确定与核心种质构建[J].华南农业大学学报,2016,37(5):24- 30.
