菊花MADS-box基因的克隆与表达载体构建
2016-07-28许申平蒋素华袁秀云
梁 芳,许申平,蒋素华,袁秀云,崔 波,张 静
(1.郑州师范学院生物工程研究所,河南郑州 450044;2.皂市高级中学,湖北天门 431703)
菊花MADS-box基因的克隆与表达载体构建
梁 芳1,许申平1,蒋素华1,袁秀云1,崔 波1,张 静2
(1.郑州师范学院生物工程研究所,河南郑州 450044;2.皂市高级中学,湖北天门 431703)
为了给菊花CmFUL基因功能鉴定与遗传改良奠定基础,采用RT-PCR法从菊花叶片中克隆出1个MADS-box A类基因的编码区序列,命名为CmFUL(GenBank登录号KT894379)。CmFUL基因编码区ORF片段长度为741 bp,编码246个氨基酸。CmFUL蛋白属于亲水性蛋白,预测该蛋白内含12个磷酸化位点和2个潜在的N-糖基化位点。氨基酸序列比对和系统进化树分析表明,Cm FUL蛋白与CMD41蛋白的相似性为95.6%,同属于AP1/FUL亚家族的FUL-like进化枝。实时荧光定量PCR分析表明,CmFUL基因在花期不同组织中均有表达,但表达丰度不一。在花蕾中表达量最高,其次是管状花,根和舌状花中痕量表达;同一朵花中管状花的表达量约为舌状花中的12倍。分析认为,CmFUL可能在促进开花及子房建成中起重要作用。将CmFUL基因连接到pCAMBIA1300载体上,成功构建了高效植物表达载体。
菊花;FUL-like;基因克隆;表达分析;生物信息学
花发育的第1步是花序分生组织向花分生组织的转变。花分生组织的诱导及花器官的形成受许多基因的联合调控,参与花发育的绝大多数基因均属于MADS-box基因。MADS-box基因是一个数量庞大且序列特异的调控基因家族,其编码的蛋白为转录因子,与靶基因的顺式作用元件结合,从而调控靶基因以特定的强度在特定的时间和空间表达。MADS-box基因广泛存在于植物中,在动物及真菌中也有发现。MADS-box基因在某些序列具有高度保守性,所有已知功能的MADS-box基因都属于TypeⅡ型,其编码的蛋白由保守程度不一的M(MADS)、I(Intervening)、K(Keratin-like)和C(C-teminal)4个不同的结构域组成[1]。其中MADS盒是由约60个氨基酸组成的高度保守结构域,主要参与DNA的结合;K盒由约70个氨基酸组成,是植物MADS-box蛋白所特有的中度保守结构域。大量研究表明,MADS-box蛋白在植物绝大多数发育过程中发挥着重要作用,包括控制营养生长到生殖生长的转变,从而控制开花时间、决定分生组织的分化[2]、决定花器官的形成、控制胚的发育[3]、促进根的形成[4]以及种子和果实的发育与成熟等[5-6];此外,还与细胞的衰老和植物的冬眠有关[7-9]。
在解释花器官发育的ABCDE模型中,控制花发育的同源异型基因绝大部分属于MADS-box基因,APETALA1(AP1)、CAULIFLOWER(CAL)和FRUITFULL(FUL)同属于MADS-box的AP1/FUL亚家族的A类基因,且三者在控制花分生组织特异性发育的功能方面具有冗余性[10]。AP1是花序分生组织特征基因,调控花序分生组织向花分生组织的转变,同时也是花器官特征基因,是花瓣和萼片的正常发育所必须的[11]。CAL是AP1的旁系同源基因,只在花发育过程中促进花分生组织的形成[12]。相比AP1和CAL,FUL基因具有非常广泛的功能。FUL基因是开花途径中的下游基因之一。在模式植物拟南芥中,FUL基因也被称为AGL8,在花序、茎生叶及心皮发育中发挥重要的作用[13]。FUL不仅与SOC1一起参与控制开花时间,而且对于维持花序分生组织继续发育成花,抑制其逆转为营养分生组织起到重要作用[14-15];此外,FUL还能影响叶片的发育[16]、角果的伸长[17]及裂片细胞的发育[5]。Xu等[18]将桃的FUL同源基因PpMADS6转入拟南芥中过表达,转基因植物表现出提前开花及花瓣、雄蕊和心皮数目增加的现象,表明FUL同源基因可能还具有调节花器官数目的功能。GhMADS22在棉花的茎尖、苞片和萼片中高表达,过表达该基因将延迟花器官的衰老与脱落[19]。Jia等[20]的研究表明大豆中Gm FULa的表达受短日照的诱导及长日照的抑制,并推测在根冠中表达量的比值决定了营养生长是否向生殖生长转变。
菊花的MADS-box基因已有部分研究。Shchennikova等[21]从菊花中分离出4个MADS-box基因,序列比对分析表明CDM111属于AP1类基因,CDM8和CDM41属于FUL类基因,CDM44属于SEP3类基因。将CDM41转入烟草,转基因烟草提前20 d开花,与野生型相比茎长度减小36.7 cm,叶片数减少16片[22]。Wang等[23]从菊花中克隆了一个MADS-box基因CIM8,该基因在拟南芥中过表达则表现提早开花及花序结构异常现象。近年来,利用基因工程手段培育植物新品种已成为现代遗传育种的重要途径。本试验以菊花(Chrysanthemum morifolium)黄色晚花品种狮子头为材料,利用RTPCR技术克隆了MADS-box基因的同源基因,并对该基因进行生物信息学分析;采用实时荧光定量PCR(RT-qPCR)法对花期不同组织器官中该基因的表达量进行了定量分析;构建了高效植物表达载体。旨在为后续通过基因工程技术改变花的外观形态及进行花期调控奠定理论基础。
1 材料和方法
1.1 试验材料
试验材料为菊花黄色大花品种狮子头。在盛花期,分别取健康植株的叶片、根、茎、花蕾、管状花及舌状花,迅速用液氮冷冻,置于-80℃超低温冰箱保存。
1.2 试验方法
1.2.1 总RNA的提取及cDNA第1链的合成 将菊花不同组织在液氮中充分研磨成粉状,用TRIzol(Invitrogen公司)试剂分别提取总RNA。用1%琼脂糖凝胶电泳检测所提RNA的完整性,并利用Quawell Q5000微量分光光度计测定其所提RNA的A260/280及A260/230值,计算RNA的浓度及纯度。反转录参照RevertAid TM First strand cDNA synthesis Kit试剂盒说明书合成普通PCR的cDNA第1链,作为PCR模板进行目的基因的克隆;用PrimeScriptRT reagent Kitwith gDNA Eraser试剂盒反转录成cDNA,用于RT-qPCR检测。
1.2.2 引物设计与合成 根据NCBI上已登陆的菊花AP1/FUL亚家族(GenBank:AY173055)基因序列,运用Primer Premier 5.0和DNA MAN软件分别设计1对引物扩增目的基因编码区序列。为构建真核表达载体,在引物的5′端分别加上酶切位点。FULF:5′-TCTAGAATGGGTAGAGGAAGAGTTCAGAT-3′,FUL-R:5′-CCCGGGATTGGTTAAGGTGGCGAATC-3′。下划线部分分别为XbaⅠ和SmaⅠ酶切位点。
1.2.3 目的基因的克隆 以叶片的cDNA为模板,利用设计合成的引物进行PCR扩增反应。各个反应体系为20.0μL:10×PCR Buffer 2.0μL、dNTP 150.0μmol/L、引物各0.5μmol/L、cDNA 1 000~2 000 ng、r Taq DNA聚合酶1.0 U。PCR扩增程序为:95℃预变性5 min;95℃变性35 s,55℃退火35 s,72℃延伸35 s,进行35个循环;最后72℃延伸10 min。PCR产物用1%的琼脂糖凝胶检测及回收。将回收的目的片段连接到pGEM-T Easy载体上,转入大肠杆菌DH5α感受态细胞中,经抗性筛选及菌落PCR鉴定,阳性克隆菌送出测序,测序由上海英骏生物技术公司完成。
1.2.4 在不同组织中的表达分析 根据克隆测序获得的菊花FUL基因序列,重新设计实时荧光定量PCR引物:qRT-FUL-F:5′-CATTAAGCCTTAGGGA TCTTCAG-3′和qRT-FUL-R:5′-CATTAAGCCTTAGG GATCTTCAG-3′。以菊花肌动蛋白基因Actin作为内参基因,引物为:Actin-F:5′-ACTCAGCACCTTCC AACAGA-3′和Actin-R:5′-CTCTGGCAACAATCGA CAA-3′。
采用SYBR Prem ix Ex TaqTMⅡkit(TaKaRa)进行RT-qPCR,反应体系为25μL,反应条件为:95℃15 s,58℃15 s,72℃15 s(40个循环)。反应在Eppendorf Mastercycler荧光定量PCR仪上进行,每个样品重复3次,同时做阴性对照。通过溶解曲线和扩增曲线确定引物的特异性。目的基因相对表达量Rel.Exp=2-ΔΔCt,其中ΔCt=Ct(Cm FUL)-Ct(ACTIN),ΔΔCt=(各组织ΔCt)-(根ΔCt)。
1.2.5 高效植物表达载体的构建 将测序正确的菌液扩摇并进行PCR扩增,提取阳性质粒pGEMFUL,分别经限制性内切酶XbaⅠ和SmaⅠ消化后,用1%琼脂糖凝胶电泳分离回收目的基因,通过T4连接酶16℃过夜处理,将目的片段定向连接到经同样处理的植物表达载体pCAMBIA1300中。连接产物转化感受态细胞DH5α,在含有卡那霉素(Kan 100 mg/L)的LB平板上筛选阳性菌落,重组质粒命名为pCAMBIA1300-FUL,提取质粒后进行PCR检测及酶切鉴定,均为阳性者再进行测序鉴定。
1.2.6 基因的生物信息学分析 利用ProtParam工具对蛋白质的理化性质进行分析,SMART分析蛋白的结构功能域,运用在线分析软件TargetP 1.1 Server对蛋白进行亚细胞定位预测。NetPhos 2.0 Server和NetNGlyc 1.0 Server分别分析磷酸化位点和N-糖基化位点。蛋白质二级结构预测采用ExPASy网站上的GOR Protein secondary structure prediction方法,氨基酸同源序列的多重比对利用DNAMAN软件生成比对结果,系统进化树使用MEGA 5.0软件中的NJ法建成。
2 结果与分析
2.1 菊花FUL基因的ORF克隆
采用带有不同酶切位点的引物,进行PCR扩增菊花FUL基因的编码区序列,得到1个约750 bp的片段(图1)。将回收纯化的克隆片段连接到pGEMT Easy克隆载体上,经过菌落PCR筛选和双酶切鉴定,获得阳性重组质粒pGEM-FUL。测序结果表明FUL基因长为741 bp,编码246个氨基酸,经过BlastN和BlastX在线分析,该基因与已登录的菊花MADS-box基因AP1/FUL亚家族的CDM41基因同源性最高为95.6%,与CsAFT(AB839770)基因同源性为77.8%,确定克隆目的基因成功,该基因命名为Cm FUL(GenBank登录号KT894379)。

图1 菊花Cm FUL基因的PCR扩增产物检测Fig.1 Agarose gel electrophoresis of PCR am p licons of Cm FUL gene in Chrysanthem um m orifolium
2.2 氨基酸的基本性质分析及二级结构预测
通过对Cm FUL基因的氨基酸序列进行分析(图2),表明该氨基酸序列属于植物特有的MIKC型MADS-box基因家族序列。SMART结果也表明,Cm FUL蛋白在1~60位是个高度保守的MADS结构功能域,即MADS家族成员共有的典型结构域,在81~173位是个中度保守的结构功能域K-box。
经蛋白质亲疏水性分析表明,该CmFUL蛋白属于亲水性蛋白。利用ProtParam工具对蛋白质的理化性质进行分析,结果表明:Cm FUL的分子质量约为28.49 kDa、理论等电点p I为9.49,分子式为C1242H2020N368O379S10;负电荷残基总数(Asp+Glu)为32,正电荷残基总数(Arg+Lys)为40。在组成Cm-FUL蛋白的20种氨基酸中,谷氨酸(Glu)和亮氨酸(Leu)所占的比例最高,均为10.6%,半胱氨酸(Cys)所占比例最低,为0.4%。该蛋白的不稳定指数为60.69,脂肪指数为73.78,根据Guruprasad方法表明Cm FUL蛋白不稳定。运用在线分析软件TargetP 1.1 Server对该蛋白进行亚细胞定位分析发现,该蛋白有可能定位于线粒体,预测剪切位点序列 为99个氨基酸。

图2 菊花Cm FUL蛋白保守结构域Fig.2 Cm FUL conservative protein dom ain structure
NetPhos 2.0 Server预测表明,CmFUL蛋白有9个Ser磷酸化位点(Ser22、Ser61、Ser62、Ser74、Ser121、Ser167、Ser 169、Ser179、Ser211),2个Thr磷酸化位点(Thr141、Thr160)和1个Tyr磷酸化位点(Tyr203)。NetNGlyc 1.0 Server预测表明CmFUL蛋白存在2个潜在的N-糖基化位点:Asn85和 Asn148。此结果表明蛋白翻译后修饰对CmFUL蛋白功能的实现起着重要的作用。
由GOR4进行二级结构预测结果表明,该蛋白二级结构由 α螺旋、伸展链和无规则卷曲组成,其中α螺旋(Hh)占61.79%,伸展链(Ee)占9.35%,无规则卷曲(Cc)占28.86%(图3)。

图3 Cm FUL蛋白序列的二级结构预测Fig.3 Pu tative resu lt of Cm FUL p rotein secondary structure from Ch rysanthem um m orifolium
2.3 氨基酸序列比对与系统发育分析

图4 氨基酸序列比对Fig.4 Com parison of the am ino acid sequence of Cm FUL
序列比对分析表明(图4),CmFUL的氨基酸序列与AY173055(CDM41)的同源性为95.6%,与AB839770(CsAFT)的同源性为75.9%,三者具有共同的eu FUL基序。为进一步分析CmFUL基因与菊科其他MADS-box基因的关系,将NCBI上已登陆的菊科MADS-box基因进行分析,构建了菊科控制花发育的MADS-box基因的系统进化树。从图5可以看出,菊科MADS-box基因分为4个大进化枝,分属于花发育模型中的A、B、C、E类基因。其中,第1个大进化枝为AP1/FUL亚家族的A类基因,CmFUL基因(KT894379)与AY173055和AB839770关系最近,同属于MADS-box的FUL-like进化枝,与A类其他基因进化关系较远。第2个进化枝为SEPALLATA亚家族基因,属于花发育模型中的E类基因;第3个进化枝为AGAMOUS亚家族基因,属于C类基因;第4个进化枝为AP3亚家族基因,属于B类基因。
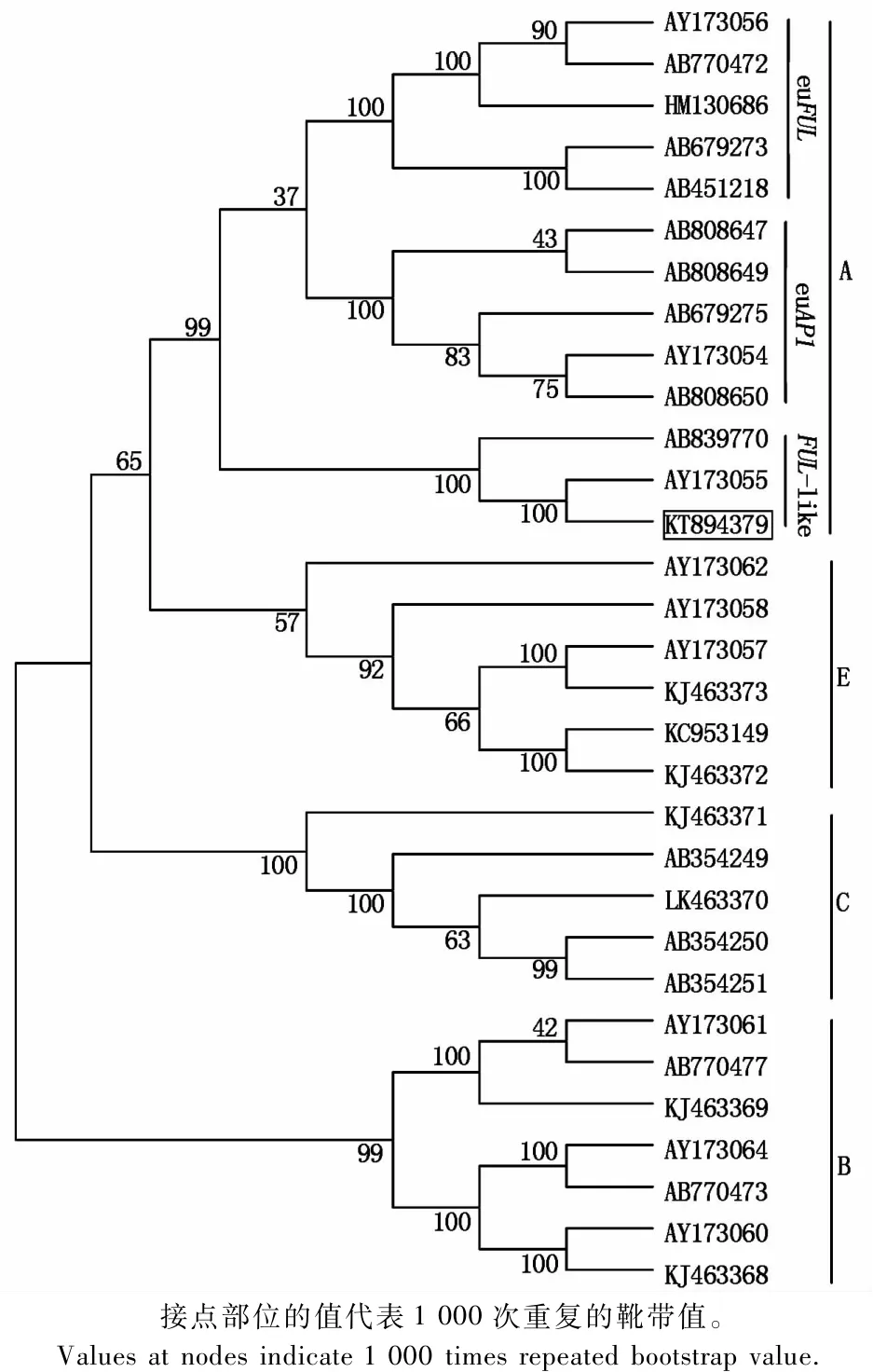
图5 Cm FUL氨基酸的系统进化树分析Fig.5 Phylogenetic tree of Cm FUL am ino acid sequence
2.4 Cm FUL基因的表达分析
利用实时荧光定量PCR(RT-qPCR)技术对菊花花期的根、茎、叶、花蕾、管状花和舌状花的Cm-FUL基因的表达量进行了分析。结果表明,CmFUL基因在各个组织中均有表达,但表达丰度不一。在花蕾中表达量最高,其次是管状花,在根和舌状花中痕量表达。从图6可以看出,同一朵花中间部位的管状花和外缘的舌状花Cm FUL的表达量差异很大,管状花中的表达量约为舌状花中的12倍。在花蕾中的表达量约为花瓣中表达量的3倍。表明CmFUL基因可能在促进开花及子房发育中发挥重要作用。

图6 Cm FUL的表达特性分析Fig.6 Analysis on the expression level of Cm FUL gene
2.5 植物表达载体的构建
将测序成功的质粒pGEM-FUL经XbaⅠ和SmaⅠ限制性内切酶酶切消化后,得到2个目的片段,分别约为750,3 000 bp(图7),切胶回收小片段;质粒pCAMBIA1300经XbaⅠ和SmaⅠ酶切消化后,切胶回收大片段。将回收的pCAMBIA1300大片段与pGEM-FUL的小片段用T4DNA连接酶连接,然后转入DH5α感受态细胞。阳性转化子经质粒酶切鉴定,得到了1条约750 bp的小片段和1条约10 000 bp的pCAMBIA1300载体片段(图8),测序验证结果表明,成功构建了植物表达载体pCAMBIA1300-FUL。

图7 pGEM-FUL的双酶切鉴定Fig.7 Restriction enzym e digestion of pGEM-FUL

图8 pCAMBIA1300-FUL的双酶切鉴定Fig.8 Restriction enzym e digestion of pCAM BIA1300-FUL
3 结论与讨论
本试验通过RT-PCR的方法从菊花中克隆了1个MADS-box基因,命名为CmFUL,该基因编码246个氨基酸,通过Blast分析具有典型的MADS-box蛋白超家族的MADS-MEF2-like结构域和K-box结构域。氨基酸序列比对分析表明,该基因属于APETALA1/FRUITFULL(AP1/FUL)亚家族,与CDM41(AY173055)基因具有95.6%的同源性,与CsAFT(AB839770)基因具有77.8%的同源性。
大量的系统进化研究表明,随着核心双子叶植物的进化,AP1/FUL-like基因至少经历了2次基因复制事件,产生了3个大的进化枝:eu AP1、eu FUL和FUL-like(AGL79)。与此相比,在基部被子植物和单子叶植物的A类基因则只有FUL-like进化枝[24-25]。eu AP1类基因编码的氨基酸序列在C末端具有典型的eu AP1基序,常以CFAA结尾;eu FUL和FUL-like类则具有相同的paleo AP1(eu FUL)基序,常具有L/MPPWML结构。本试验通过系统发育分析表明,已登陆的菊花A类基因可分为3类,分属于3个大的进化枝,Cm FUL基因与AY173055和AB839770基因进化关系最近,属于MADS-box基因AP1/FUL亚家族中的FUL-like进化枝,与其他菊花A类基因进化关系较远,反映了菊花Cm FUL基因可能属于较为原始的类型。
本试验利用荧光定量PCR技术对菊花花期的各组织器官进行了CmFUL表达分析,结果表明Cm-FUL在花蕾中高表达,反映了A类基因在促进开花方面的功能,在茎、叶和管状花中少量表达。此结果与CDM41的功能不完全一致[21],表明相似的基因序列不一定代表具有相似的基因功能。本试验首次对菊花盛花期的管状花和舌状花中CmFUL基因的表达量进行了比较分析,发现同一朵花中管状花中的表达量远远高于舌状花。菊花具有典型的头状花序,中间是两性的管状花,外缘是雌性的舌状花。现有的栽培品种经过数代杂交后,花器官的形态及功能发生了很大的变化,基因的多次重组也使得基因功能更加复杂化。管状花中CmFUL基因的高表达而在舌状花中的痕量表达,表明Cm FUL基因功能较为复杂。为了进一步研究CmFUL的功能,构建了高效植物表达载体,拟转入模式植物拟南芥中。
[1] Alvarez-Buylla E R,Pelaz S,Liljegren S J,et al.An ancestral MADS-box gene duplication occurred before the divergence of plants and animals[J].Proceedings of National Academy Sciences,2000,97(10):5328-5333.
[2] Chi Y,Huang F,Liu H C,et al.An APETALA1-like gene of soybean regulates flowering time and specifies floral organs[J].Journal of Plant Physiology,2011,168(18):2251-2259.
[3] Galimba K D,Di Stilio V S.Sub-functionalization to ovule development following duplication of a floral organ identity gene[J].Development Biology,2015,405(1):158-172.
[4] Yu C,Liu Y,Zhang A,et al.MADS-box transcription factor OsMADS25 regulates root development through affection of nitrate accumulation in rice[J].PLoS One,2015,10(8):e0135196.
[5] Ferrándiz C,Liljegren S J,Yanofsky M F.Negative regulation of the SHATTERPROOF genes by FRUITFFULL during Arabidopsis fruit development[J].Science,2000,289(5478):436-438.
[6] Shima Y,Kitagawa M,Fujisawa M,et al.Tomato FRUITFULL homologues act in fruit ripening via forming MADS-box transcription factor complexes with RIN[J].Plant Molecular Biology,2013,82(4-5):427-438.
[7] Voogd C,Wang T,Varkonyi-Gasic E.Functional and expression analyses of kiwifruit SOC1-like genes suggest that they may not have a role in the transition to flowering but may affect the duration of dormancy[J].Journal of Experimental Botany,2015,66(15):4699-4710.
[8] Xie Q,Hu Z,Zhu Z,et al.Overexpression of a novel MADS-box gene SlFYFL delays senescence,fruit ripening and abscission in tomato[J].Scientific Reports,2014,4:4367.
[9] Hoenicka H,Nowitzki O,Hanelt D,et al.Heterologous overexpression of the birch FRUITFULL-like MADS-box gene BpMADS4 prevents normal senescence and winter dormancy in Populua tremula L.[J].Planta,2008,227(5):1001-1011.
[10] Ferrándiz C,Gu Q,Martienssen R,et al.Redundant regulation of meristem identity and p lant architecture by FRUITFULL,APETALA1 and CAULIFLOWER[J].Development,2000,127(4):725-734.
[11] Mandel M A,Gustafson-Brown C,Savidge B,et al.Molecular characterization of the Arabidopsis floral homeoticgene,APETALA1[J].Nature,1992,360(6401):273-277.
[12] Kempin S A,Savidge B,Yanofsky M F,et al.Molecular basis of the cauliflower phenotype in Arabidopsis[J]. Science,1995,267(5197):522-25.
[13] Mandel M A,Yanofsky F M.The Arabidopsis AGL8 MADS box gene is expressed in inflorescence meristems and is negatively regulated by APETALA1[J].The Plant Cell,1995,7(11):1763-1771.
[14] Torti S,Fornara F.AGL24 acts in concertwith SOC1 and FUL during Arabidopsis floral transition[J].Plant Signaling and Behavior,2012,7(10):1251-1254.
[15] Melzer S,Lens F,Gennen J,et al.Flowering-time genes modulate meristem determ inacy and growth form in Arabidopsis thaliana[J].Nature Genetics,2008,4(12):1489-1492.
[16] Pabón-Mora N,Sharma B,Holappa L D,et al.The Aquilegia FRUITFULL-like genes play key roles in leafmorphogenesis and inflorescence development[J].Plant Journal,2013,74(2):197-212.
[17] Gu Q,Ferr ndiz C,Yanofsky M F,et al.The FRUITFULL MADS-box gene cell differentiation during Arabidopsis fruit development[J].Development,1998,125(8):1509-1517.
[18] Xu Y,Zhang L,Ma R C.Functional characterization and mapping of two MADS box genes from peach(Prunus persica)[J].Chinese Science Bulletin,2008,53(6):853-859.
[19] Zhang W X,Fan S L,Pang C Y,et al.Molecular cloning and function analysis of two SQUAMOSA-like MADS-box genes from Gossypium hirsutum L.[J].Journal of Intergrative Plant Biology,2013,55(7):597-607.
[20] Jia Z,Jiang B J,Gao X W,et al.GmFULa,a FRUITFULL homolog,functions in the flowering and maturation of soybean[J].Plant Cell Reports,2015,34(1):121-132.
[21] Shchennikova V A,Shugla A O,Immink R,et al.Identification and characterization of four Chrysanthemum MADS-Box genes,belonging to the APETALA1/FRUITFULL and SEPALLATA3 subfam ilies[J].Plant Physiology,2004,134(4):1632-1641.
[22] Goloveshkina N E,Shchennikova V A,Kamionshaya M A,et al.Influence of ectopic expression of Asteraceae MADS box genes on plant ontogeny in tobacco[J].Plant Cell Tissue and Organ Culture,2012,109(1):61-71.
[23] Wang Y,Ma Y P,Fu JX,et al.Isolation and functional analysis of the ClM 8-FRUITFULL-like MADS-box gene from Chrysanthemum lavandulifolium[J].Scientia Horticulturae,2013,161(2):125-133.
[24] Litt A,Irish F V.Duplication and diversification in the APETALA1/FRUITFULL floral homeotic gene lineage:implications for the evolution of floral development[J]. Genetics,2003,165(2):821-833.
[25] Shan H Y,Zhang N,Liu C J,et al.Patterns of gene duplication and functional diversification during the evolution of the AP1/SQUA subfamily of plant MADS-box genes[J].Molecular Phylogenetics and Evolution,2007,44(1):26-41.
Cloning,Exp ression and Vector Construction of a MADS-box Gene in Chrysanthemum morifolium
LIANG Fang1,XU Shenping1,JIANG Suhua1,YUAN Xiuyun1,CUIBo1,ZHANG Jing2
(1.Institute of Bioengineering,Zhengzhou Normal University,Zhengzhou 450044,China;2.Zaoshi High School of Tianmen City,Tianmen 431703,China)
In order to research the identification of gene function and genetic improvement in Chrysanthemum,a MADS-box fam ily class A gene was isolated from the leaf of Chrysanthemum morifolium by using RT-PCR method,which was designated CmFUL gene(The accession number in GenBank:KT894379).The coding region of this gene(ORF)was 741 bp and encoded 246 putative am ino acid residues.Encoding product of Cm FUL gene was a kind of hydrophilic protein and predicted that the protein contained 12 phosphorylation sites and 2 potential N-Glycosylation sites.Sequence and phylogenic analyses revealed that Cm FUL was close to CDM41 with 95.6%sequence similarity which both belonged to the FUL-like clade of AP1/FUL subfamily.The expression analyses of CmFUL by Real-time fluorescent quantitative PCR indicated that this gene was expressed in different parts in bloom stage,but the expression abundance was varied widely.The highest amount of expression was observed in buds,followed by in tubiform florets.The expression level in roots and lingulate florets were lowest.It was worth noting that the level expressed in tubiform florets was 12 times of that in lingulate florets between a same flower.The results implied that CmFUL m ight play an important role in promoting flowering and ovary development.CmFUL was linked to pCAMBIA1300 vector and the plant expression vector was constructed successfully.
Chrysanthemum morifolium;FUL-like;Gene cloning;Expression analysis;Bioinformatics
Q949.71+8.43 文献标识码:A 文章编号:1000-7091(2016)03-0025-07
10.7668/hbnxb.2016.03.004
2015-12-03
郑州市重大科技专项(141PZDGG189);河南省科技攻关项目(524050005)
梁 芳(1982-),女,湖北郧西人,讲师,博士,主要从事植物生物技术研究。
崔 波(1962-),男,河南泌阳人,教授,博士,主要从事植物分子生物学研究。
