基于线粒体DNA D–loop序列的黄郎鸡遗传多样性与品种起源研究
2016-07-28黄勋和翁茁先李威娜陈洁波钟福生唐寿桂赵宇旗
黄勋和,翁茁先,李威娜,陈洁波,钟福生*, 唐寿桂,赵宇旗
(1.嘉应学院生命科学学院,广东 梅州 514015;2.湖南农业大学动物科技学院,湖南 长沙 410128;3.衡山县畜牧水产局,湖南 衡阳 421300;4.衡山湘旗农牧有限责任公司,湖南 衡阳 421341)
基于线粒体DNA D–loop序列的黄郎鸡遗传多样性与品种起源研究
黄勋和1,翁茁先2,李威娜1,陈洁波1,钟福生1*, 唐寿桂3,赵宇旗4
(1.嘉应学院生命科学学院,广东 梅州 514015;2.湖南农业大学动物科技学院,湖南 长沙 410128;3.衡山县畜牧水产局,湖南 衡阳 421300;4.衡山湘旗农牧有限责任公司,湖南 衡阳 421341)
摘 要:采用PCR产物直接测序法对黄郎鸡50个个体的线粒体DNA D–loop序列进行分析,统计黄郎鸡周边省份地方鸡的进化支地理分布特征。结果表明,黄郎鸡具有较高的线粒体遗传变异水平,检测到 26个变异位点,核苷酸多样性为0.012 54±0.000 94,核苷酸差异均数为6.569,单倍型多态性为0.886±0.035。在定义的23种单倍型中,13种单倍型为首次发现,单倍型落在A、B、C、E共4个进化支,B为优势单倍群。系统进化分析和进化支的地理分布特征表明,黄郎鸡起源于中国北方和西南地区,同时受邻省地方鸡基因交流的影响。
关 键 词:黄郎鸡;D–loop序列;遗传多样性;单倍型;地理分布
投稿网址:http://xb.ijournal.cn
地方家禽遗传资源是生物多样性的重要组成部分,是经长期进化而形成的宝贵资源,是未来家禽品种改良和适应生产条件变化、保持生产长期持续发展的遗传基础,即使是低产量的品种,也可能因其对人们感兴趣的性状有所贡献而成为未来育种的宝贵遗传资源[1–2]。黄郎鸡又称湘黄鸡,属兼用型地方品种,原产地为湖南省衡南县的泉溪、双林、花桥,中心产区为衡阳市[3]。黄郎鸡具有遗传性能稳定、适应性强、耐粗饲、肉质鲜嫩等特点,深受当地人的喜爱。黄郎鸡生长较慢,饲料转化比
线粒体DNA(mtDNA)因具有结构简单、含量丰富、进化快、严格的母系遗传特点而被广泛用于动物群体遗传多样性、群体遗传结构、系统进化等研究[4–8]。本研究中应用线粒体DNA D–loop序列对黄郎鸡进行遗传多样性分析,评估其品种遗传资源现状及保护潜力,探讨其品种的起源分化,旨在为黄郎鸡的科学保护和开发利用提供参考依据。
1 材料与方法
1.1 材料
黄郎鸡共50只,其中30只来源于衡山湘旗农牧有限责任公司,20只来源于衡阳市畜牧局黄郎鸡原种场。翅静脉取黄郎鸡血0.2~0.5 mL,2% EDTA抗凝–70 ℃保存。其余中国地区家鸡及原鸡的D–loop序列下载于 GenBank(http://www.ncbi.nlm. nih.gov/nuccore/)。使用试剂盒 HiPure Blood DNA Mini Kit(美基生物,广州)提取样品DNA,-20 ℃保存,备用。
1.2 方法
1.2.1 PCR扩增与序列测定
D–loop扩增引物序列为 L16750(5'–AGGACT ACGGCTTGAAAAGC–3'[9])和H522(5'–ATGTGCC TGACCGAGGAACCAG–3'[10])。PCR反应体系30 µL,含10×PCR Buffer 3 µL,dNTP mixture(2.5 mmol/L) 2.4 µL,引物各0.3 µL(20 µmol/L),Taq DNA聚合酶(宝生物工程有限公司,大连)1.5 U,DNA模板100 ng。扩增条件为94 ℃预变性4 min,35个循环(94 ℃变性30 s,63 ℃退火1 min,72 ℃延伸50 s),最后72 ℃延伸10 min。PCR产物于1.5%琼脂糖凝胶电泳检测后送上海立菲生物技术有限公司(广州)双向测序。
1.2.2 序列分析
用BioEdit 7.2.5[11]软件读取测序序列,对每个序列进行人工检查校对。将所获序列用Clustal X软件[12]进行比对排列后,以 NC_007235为参考序列,用DnaSP 5.0软件[13]定义单倍型,并计算单倍型多样性(haplotype diversity)、核苷酸多样性(nucleotide diversity)和核苷酸差异均数。统计湖南省和周边省份已发表D–loop数据的家鸡个体数及其在该省份的唯一单倍型数。通过NETWORK 4.6.1.3软件[14]构建中值连接网络图(Median-joining networks),颠换/转化的权重比设为 3∶1[15]。用 MEGA 6.0 软件[16]中的Kimura 2–parameter (K2P)模型计算遗传距离,采用邻接法(neighbor–joining,NJ)构建分子系统树,1 000步自检举(bootstrap)检验系统发育树各节点的置信值。
2 结果与分析
2.1 遗传多样性分析
表1列出了黄郎鸡线粒体DNA D loop的单倍型及其在不同地区的共享单倍型数量。

表1 黄郎鸡线粒体DNA D–loop的单倍型及其在不同地区的共享单倍型数量Table 1 Variable sites and shared haplotypes among the different sub-region in China
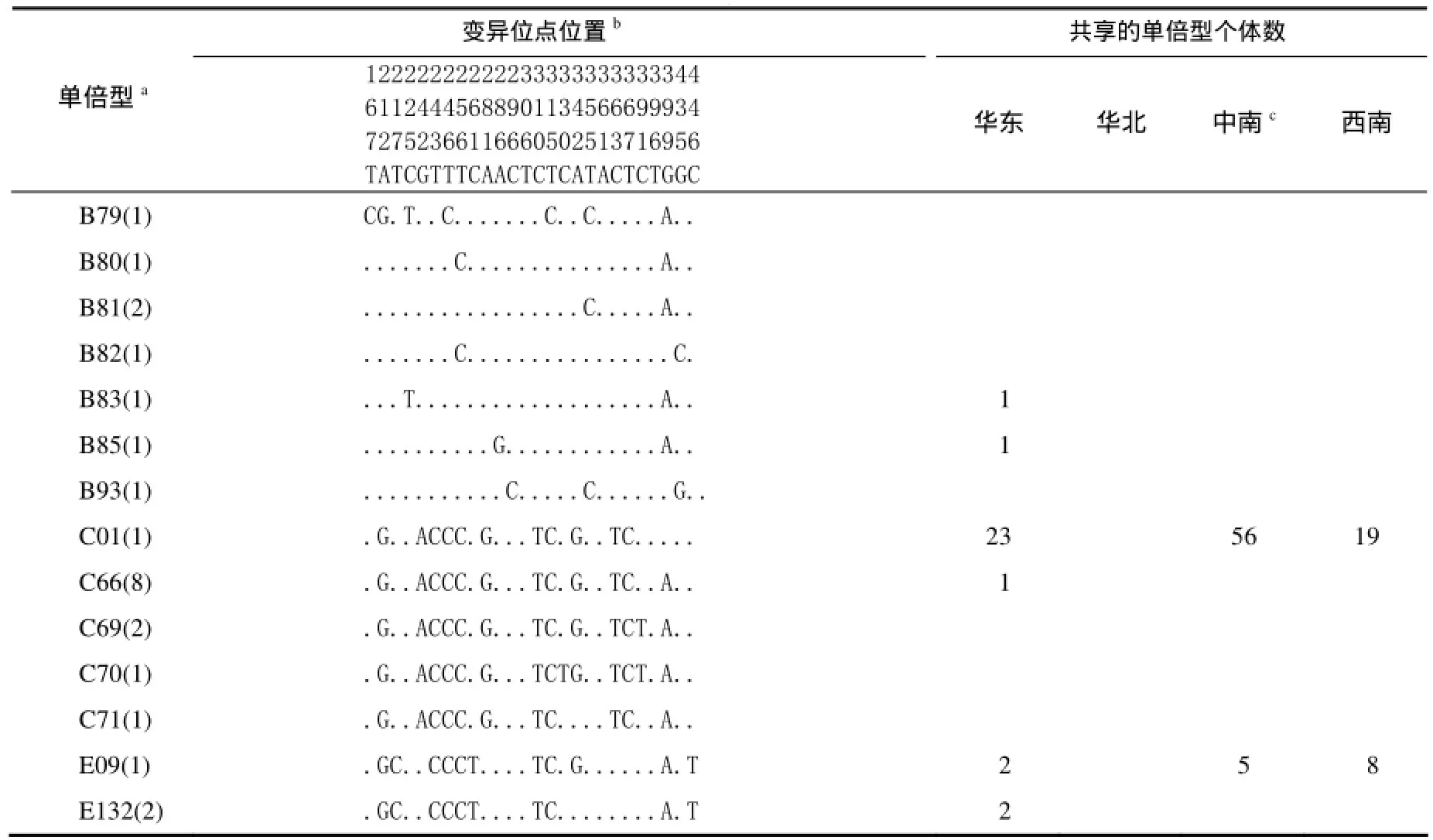
表1(续)
除去 PCR引物序列,共获得 524 bp mtDNA D–loop序列。50 个个体线粒体D–loop中共检测到26个变异位点(24个转换,2个颠换),占分析位点的 4.96%,其中单一位点突变(singleton variable sites)7个,简约信息位点(parsimony informative sites)19个(表1)。T、C、A、G的平均含量分别为30.1%、30.0%、27.5%、12.4%。A+T 的含量为57.6%,高于 G+C 的含量(42.4%)。核苷酸多样性为 0.012 54±0.000 94,核苷酸差异均数为6.569。在26个变异位点中定义了 23种单倍型,单倍型多态性为0.886±0.035,其中 13种单倍型为首次发现,B20为优势单倍型,其次是C66。
2.2 系统进化分析
从GenBank选择代表主要单倍群的390种单倍型与黄郎鸡的单倍型构建无根邻接树(unrooted NJ tree)。无根邻接树分为13枝分化明显的分支(A–I,W–Z),黄郎鸡主要落在进化支A、B、C和E,其分布频率分别为5/50、29/50、13/50、3/50,B为优势单倍群(表1、图1)。在2 658个中国家鸡中,与黄郎鸡单倍群A共享单倍型的有315个,与B单倍群共享的有490个,与C单倍群共享的有99个,与E单倍群共享的有17个(表1)。将与湖南省及周边地区进一步划为华东、华北、中南、西南4个地区[17],与黄郎鸡共享单倍群最多的为西南地区(347个,27.52%),其次是中南地区(311个,43.02%),第三是华东地区(244个,40.46%),最后是华北地区(19个,26.76%)。
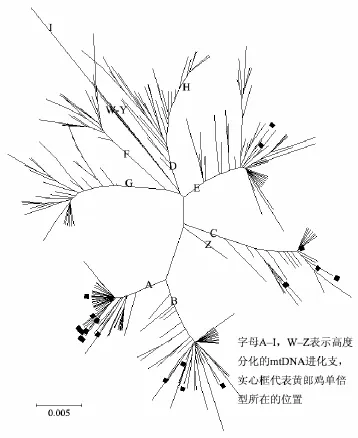
图1 390种从GenBank下载的家鸡和红原鸡单倍型和23种黄郎鸡单倍型构建的无根邻接树Fig. 1 Unrooted neighbor–joining (NJ) tree of 390 haplotypes in domestic chickens and red jungle fowls downloaded from GenBank and 23 haplotypes in Huanglang
2.3 进化支的地理分布
中国家鸡主要进化支的网络图和地理分布情况分别见图2和表2。
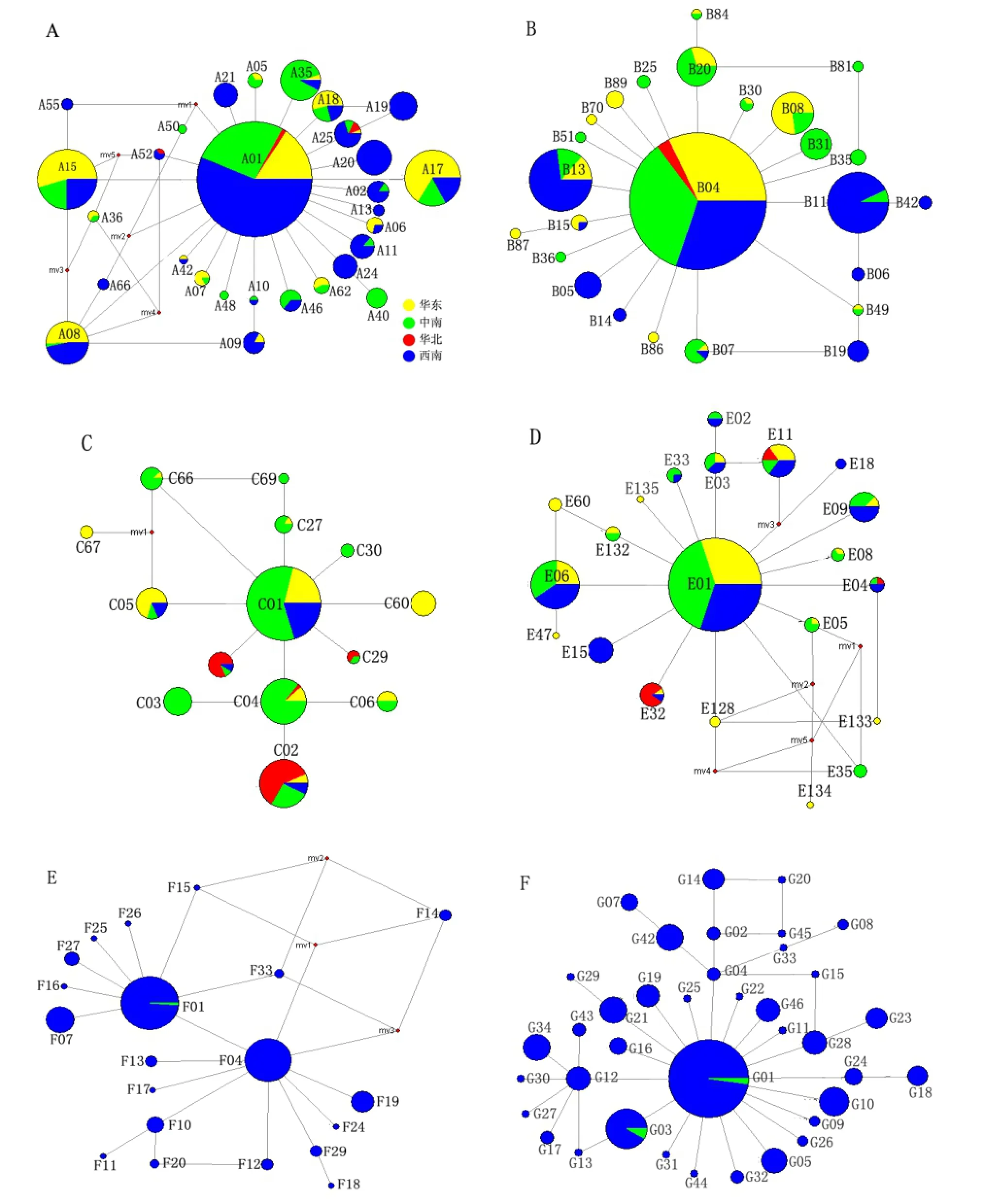
图2 主要进化支的网络图Fig.2 Network profiles of the major clades
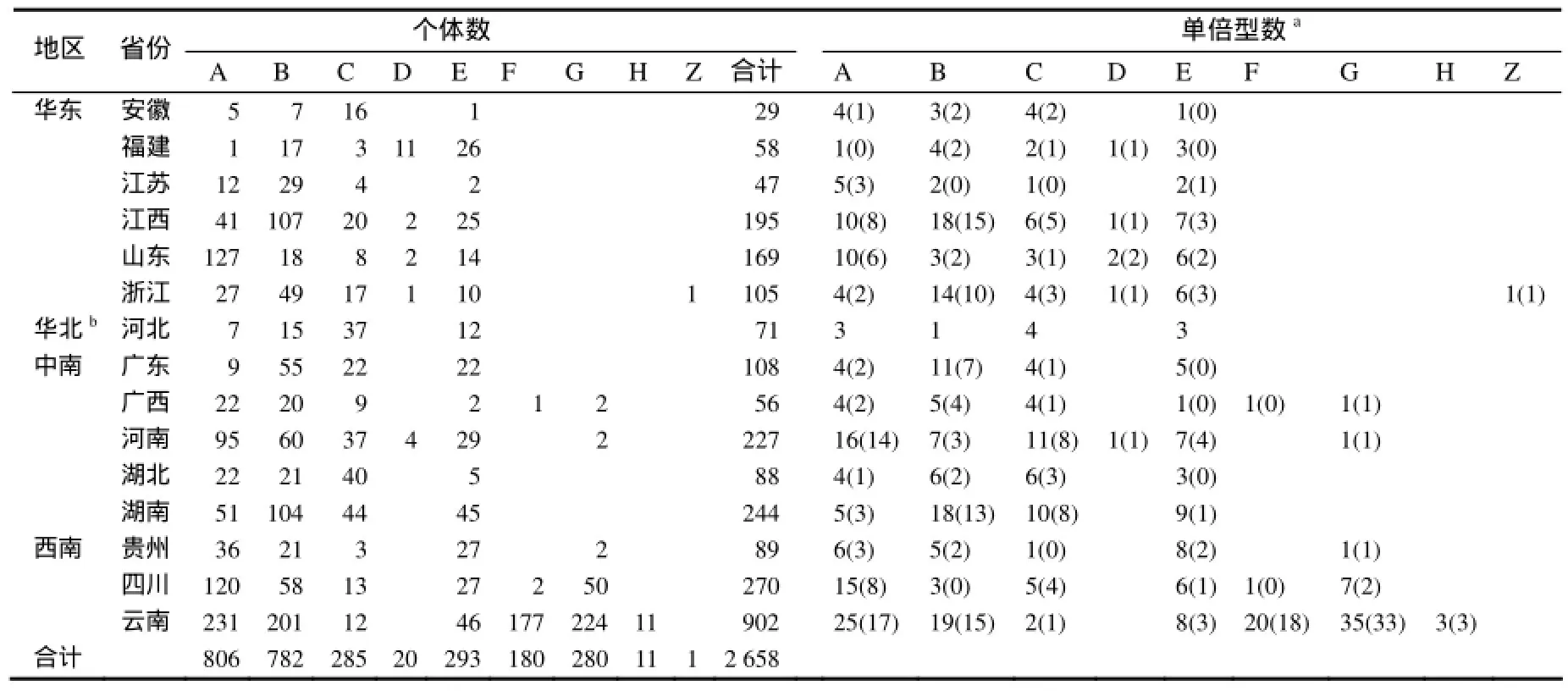
表2 中国家鸡主要进化支的地理分布Table 2 Geographical distribution of the major clades in domestic chickens of China
在统计的2 658只家鸡的单倍型中,进化支A、B、C和E在所有省份均有分布,其中A和B为优势单倍群,占统计的59.74%,而D、F、G、H、Z只在部分省份分布(表2)。进化支D只在河南省有分布(文献[4]曾报道其在云南省有分布,但校正结果表明在云南分布的为单倍群H[17]),F只分布在四川贵州和广西,G只分布在广西和河南,H只分布在云南。可见,与一些家养动物[18]相似,家鸡表现出一定的地理分布特征[4]。有趣的是,在华东地区的浙江省发现了罕见的进化支Z,该个体为江山乌骨鸡。由于地理毗邻,所以许多省份的单倍群组成相似,如云南、四川、贵州的与广西的相似,福建、江西、浙江的与山东和河南的相似。
与中国其他地区相比,西南地区家鸡遗传资源更为丰富(这与西南地区为家鸡的起源地之一[4,17]和生物多样性理论[19]是相一致的)。单倍群A与B在中国华东、华北、中南、西南地区分布较为均匀(占所在地区分布数量的比例最低为9.86%,最高为37.65%,平均为27.54%),单倍群F与G大部分分布在西南地区,极少部分分布在中南地区。虽然华北地区家鸡数目较少,但部分单倍型所占比重较大,如C02、C25、C29和E32(图2)。河北地方鸡的单倍群C的比例占该地区单倍型达52.11%,与其接近的有安徽(55.17%)和湖北(45.45%),而西南地区单倍型C的比例(云南1.33%、四川4.81%、贵州3.37%)远低于其他地区。
3 结论与讨论
本研究中黄郎鸡的遗传多样性高于宁都黄鸡、万载黄鸡[20]、固始鸡和白耳黄鸡[21]等地方鸡;并且在23种单倍型中,13种为首次发现,表明黄郎鸡具有较高的遗传保护潜力和开发利用价值。
黄郎鸡A、B、C和E这4个单倍群在中国家鸡群体中为优势单倍群,在华东、华北、中南、西南的所有省份均有分布。此外,没有发现品种特异的母系分支,说明过去频繁的人类迁徙扩散,经贸往来在家鸡的驯化与品种形成中起着重要的作用,家鸡遗传组成逐渐融合,品种间差异逐渐减小[4,22]。黄郎鸡单倍群B占总数的50%以上,与湖南省除研究区域之外的其他地方的单倍型组成相似,也与江西省、江苏省、浙江省的单倍型组成接近。由地理毗邻、频繁的人类迁徙和贸易等使地方鸡之间发生基因交流,这些地区地方鸡对黄郎鸡品种的形成具有一定的遗传贡献。
现代家鸡驯化的多起源学说已得到广泛的认可[4,5,7,17,23–24]。Xiang等[21]认为中国北方地区是现代家鸡的重要起源地和最早驯化地。在本研究中统计的中国家鸡的单倍型中,河北地方鸡的单倍群C占该地区单倍型的50%以上,与其接近的有安徽、湖北、湖南、河南、浙江、广西,而西南地区单倍型C的比例远低于其他地区,表明河北等中国北方地区可能也是家鸡的驯化地之一,这些地区对中国现代家鸡的驯化和品种多样性的形成有重要贡献。本研究结果支持了Xiang等[25]的观点。
综上所述,黄郎鸡起源于中国西南和华北地区,同时受邻近地区家鸡的影响,是在人工选育和特殊地理和人文环境中逐渐形成的具有地方特性的家鸡品种。
感谢湖南环境生物职业技术学院李丽平副教授和许美解教授在采集样品时提供了帮助。
参考文献:
[1] Bruford M W,Bradley D G,Luikart G.DNA markers reveal the complexity of livestock domestication [J].Nature Reviews Genetics,2003(4):900–910.
[2] Toro M, Caballero A.Characterization and conservation of genetic diversity in subdivided populations [J]. Philosophical Transactions of the Royal Society of London:Series B(Biological Sciences),2005,360:1367–1378.
[3] 国家畜禽遗传资源委员会.中国畜禽遗传资源志:家禽志[M].北京:中国农业出版社,2011.
[4] Liu Y P,Wu G S,Yao Y G,et al.Multiple maternal origins of chickens:out of the Asian jungles[J]. Molecular Phylogenetics Evolution,2006,38(1):12–19.
[5] Berthouly–Salazar C,Rognon X,Nhu Van T,et al. Vietnamese chickens:a gate towards Asian genetic diversity [J].BMC Genet,2010,11:53.
[6] 郑将臣,万全,程起群,等.基于16S rRNA序列探讨龟鳖类的遗传分化和系统发生[J].湖南农业大学学报(自然科学版),2011,37(2):199–205.
[7] Langford S M,Kraitsek S,Baskerville B,et al.Australian and Pacific contributions to the genetic diversity of Norfolk Island feral chickens[J].BMC Genet,2013,14:91.
[8] Wu Y P,Huo J H,Xie J F,et al.Phylogeography and origin of Chinese domestic chicken[J].Mitochondrial DNA,2014,25(2):126–130.
[9] Fumihito A,Miyake T,Sumi S,et al.One subspecies of the red jungle fowl (Gallus gallus gallus) suffices as the matriarchic ancestor of all domestic breeds[J]. Proc Natl Acad Sci USA,1994,91(26):12505–12509.
[10] 傅衍,牛冬,罗静,等.中国家鸡的起源探讨[J].遗传学报,2001,28(5):411–417.
[11] Hall T A.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT [J].Nucl Acids Symp Ser,1999,41:95–98.
[12] Thompson J D,Gibson T J,Plewniak F,et al.The Clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876–4882.
[13] Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data [J].Bioinformatics,2009,25(11):1451–1452.
[14] Bandelt H J,Forster P,Rohl A.Median-joining networks for inferring intraspecific phylogenies[J].Molecular Biology and Evolution,1999,16(1):37–48.
[15] Amills M,Ramırez O,Tomas A,et al.Mitochondrial DNA diversity and origins of south and central american goats [J].Anim Genet,2009,40(3):315–322.
[16] Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,2013,30(12):2725–2729.
[17] Miao Y W,Peng M S,Wu G S,et al.Chicken domestication: an updated perspective based on mitochondrial genomes[J].Heredity,2012,110(3):277–282.
[18] Bigi1 D,Perrotta G,Zambonelli P.Genetic analysis of seven Italian horse breeds based on mitochondrial DNA D–loop variation[J].Anim Genet,2014,45(4):593–595.
[19] Huo J L,Wu G S,Chen T,et al.Genetic diversity of local Yunnan chicken breeds and their relationships with Red Junglefowl[J].Genetics and Molecular Research,2014,13(2):3371–3383.
[20] 武艳平,霍俊宏,刘林秀,等.江西地方鸡的系统进化及遗传多样性研究[J].江西农业大学学报,2011,33(6):1160–1163.
[21] 包文斌,束婧婷,王存波,等.中国家鸡和红色原鸡mtDNA 控制区遗传多态性及系统进化分析[J].畜牧兽医学报,2008,39(11):1449– 1459.
[22] Mwacharo J M,Bjornstad G,Mobegi V,et al. Mitochondrial DNA reveals multiple introductions of domestic chicken in East Africa[J].Mol Phylogenet Evol 2011,58(2):374–382.
[23] Nishibori M,Shimogiri T,Hayashi T,et al.Molecular evidence for hybridization of species in the genus Gallus except for Gallus varius[J].Anim Genet,2005,36(5):367–375.
[24] Gongora J,Rawlence N J,Mobegi V A,et al. Indo-European and Asian origins for Chilean and Pacific chickens revealed by mtDNA[J].Proc Natl Acad Sci USA,2008,105(30):10308–10313.
[25] Xiang H,Gao J Q,Yu B Q,et al.Early Holocene chicken domestication in northern China[J].Proc Natl Acad Sci USA,2014,111(49):11564–11569.
责任编辑:王赛群
英文编辑:王 库
中图分类号:S 831.2
文献标志码:A
文章编号:1007-1032(2016)01-0075-06
收稿日期:2015–08–05 修回日期:2016–01–05
基金项目:广东省自然科学基金项目(2014A030307018);嘉应学院“创新强校工程”项目(CQX019);广东省公益研究与能力建设项目(2015A020208020);国家星火计划项目(2013GA780066);嘉应学院重点科技计划项目(2015KJM03)
作者简介:黄勋和(1982—),男,广东河源人,博士,主要从事中国地方鸡遗传多样性与分子进化研究;*通信作者,钟福生,博士,教授,主要从事动物生产与环境科学研究,zfs@jyu.edu.cn低,个体间差异较大,外来品种的大量引进及规模化养殖也导致其品质降低和遗传多样性下降,因此,探索黄郎鸡的遗传多样性,对黄郎鸡的品种起源和遗传资源评价及种质资源保护与利用都有重要的参考价值。
Genetic diversity and breed origin of Huanglang chicken inferred from mitochondrial DNA D–loop sequence
Huang Xunhe1, Weng Zhuoxian2, Li Weina1, Chen Jiebo1, Zhong Fusheng1*, Tang Shougui3, Zhao Yuqi4
(1.School of Life Sciences, Jiaying University, Meizhou, Guangdong 514015, China; 2.College of Animal Science & Technology, Hunan Agricultural University, Changsha 410128, China; 3.Bureau of Animal Husbandry and Fisheries of Hengshan County, Hengyang, Hunan 421300, China; 4. Hengshan Xiangqi Agriculture and Animal Husbandry Co., Ltd., Hengyang, Hunan 421341, China)
Abstract:Fifty Huanglang chickens were selected to directly sequencing using the PCR products. Geographic distribution of the clades of chickens from China was analysis. The results showed that Huanglang had high levels of genetic diversity, with nucleotide diversity, nucleotide differences and haplotype diversity were 0.012 54±0.000 94, 6.569 and 0.886±0.035, respectively. In the 26 variable sites, 23 haplotypes were defined, 13 of which was new. These haplotypes were belonging to clade A, B, C and E, respectively, and E was dominated haplogroup. Phylogenetic analysis and geographic distribution of the clades suggested that Huanglang originated from north and southwest China, and the gene flow with neighbor chickens was contributed significantly to this breed formation.
Keywords:Huanglang chicken; DNA D–loop sequence; genetic diversity; phyletic evolution; geographic distribution
