利用高密度SNP遗传图谱定位小麦穗部性状基因
2016-07-14邓志英李青芳孙彩铃田纪春陈建省山东农业大学农学院小麦品质育种研究室作物生物学国家重点实验室山东泰安271018
刘 凯 邓志英 李青芳 张 莹 孙彩铃 田纪春陈建省山东农业大学农学院小麦品质育种研究室 / 作物生物学国家重点实验室,山东泰安271018
利用高密度SNP遗传图谱定位小麦穗部性状基因
刘 凯 邓志英 李青芳 张 莹 孙彩铃 田纪春*陈建省*
山东农业大学农学院小麦品质育种研究室 / 作物生物学国家重点实验室,山东泰安271018
摘 要:小麦穗部性状之间相关性密切,其中穗粒数和千粒重是重要的产量构成要素,挖掘与穗部性状相关联的基因位点对分子标记辅助育种及解释基因效应具有重要意义。本研究以RIL群体(山农01-35×藁城9411) 173个F8∶9株系为材料,利用90 k小麦SNP基因芯片、DArT芯片技术及传统的分子标记技术构建的高密度遗传图谱,在5个环境下进行穗部相关性状QTL定位。检测到位于1B、4B、5B、6A染色体上7个控制千粒重的加性QTL,解释表型变异率 6.00%~36.30%,加性效应均来自大粒母本山农 01-35;检测到 8个控制穗长的加性 QTL,解释表型变异率14.34%~25.44%;3个控制穗粒数的加性QTL;5个控制可育小穗数的加性QTL;3个控制不育小穗数的加性QTL,贡献率为8.70%~37.70%;4个控制总小穗数的加性QTL;6个控制小穗密度的加性QTL。通过基因型与环境互作分析,检测到32个加性QTL,解释表型变异率0.05%~1.05%。在4B染色体区段EX_C101685-RAC875_C27536检测到控制粒重、穗长、穗粒数、可育小穗数、不育小穗数、总小穗数的一因多效 QTL,其贡献率为 5.40%~37.70%,该位点在多个环境中被检测到,是稳定主效QTL。在6A染色体wPt-0959-TaGw2-CAPS区间上检测到控制粒重、总小穗数的QTL。研究结果为穗部性状的分子标记开发、基因精细定位和功能基因克隆奠定了基础。
关键词:普通小麦;90 k基因芯片;QTL定位;穗部;SNP
本研究由山东省自然科学基金项目(2015ZRB01179,ZR2013CM004)和山东省种质资源创制课题资助。
This study was supported by the Natural Science Foundation of Shandong Province,China (2015ZRB01179 and ZR2013CM004) and the Project of Germplasm Resource Enhancement in Shandong province.
第一作者联系方式∶ E-mail∶ liukaiyouxiang@163.com
URL∶ http∶//www.cnki.net/kcms/detail/11.1809.S.20160321.1056.004.html
高产是小麦育种的永恒主题。穗粒数和千粒重等穗部性状是产量构成的重要因素,且与穗长、有效穗数等存在密切关系。在基因水平解析穗部相关性状的遗传机制对利用分子标记辅助选择和分子设计育种具有重要的意义。
已报道的小麦QTL大多是用RFLP和SSR等标记定位,连锁图谱平均图距10~20 cM,标记密度偏低,存在许多无多态性标记的大间隙,而且自动化程度低、操作过程较繁琐[1]。随着SNP研究的迅猛发展,以及基因芯片技术和测序技术的发展,自动化程度更高的第3代SNP标记已迅速应用[2],在模式动植物和重要农作物中发挥着越来越重要的作用。SNP标记具有密度高、代表性强、遗传稳定性好和易于实现自动化分析检测等优点,现已广泛应用于植物遗传连锁图谱构建、生物物种起源与亲缘关系以及生物多态性的研究等方面[3]。
自Paterson等[4]1988年利用分子标记定位QTL的技术以来,已经报道了 200多个控制小麦穗部相关性状的QTL,遍布在小麦21条染色体上[5-10]。其中,控制小麦粒重的QTL有100多个,解释表型变异超过10%的有50多个,主要分布于4DS、6AL、6D、7DS等染色体上[11-15];还至少有80个穗长相关QTL,主要分布于1B、5A等染色体上[9,14-21];然而,对不育小穗数QTL的定位研究较少。Cui等[9]利用潍麦8号×济麦20和潍麦8号×烟农19构建的2个F8∶9RIL群体对小麦穗部性状进行QTL的定位。同时在两个群体3D、4A、7B 染色体上检测到控制基部不育小穗数的 QTL,并且发现控制总小穗数的 3个主效基因分布在5A、2D和3D上[22-26]。穗部性状QTL常在染色体上成簇分布,Song等[19]利用RIL群体将总小穗数、穗粒数定位到1B、5B、5D染色体的同一区段;Ma等[27]利用望水白和南大2419构建的RIL群体和永久F2群体将穗粒数、总小穗数、可育小穗数、穗长定位到 7D 染色体的 Xcfd46-Xwmc702标记区间;Wang等[28]利用 F2∶3群体在4BL、5A、6A染色体上相同的标记区间定位了穗粒数、可育小穗数、穗长、总小穗数QTL;Deng等[7]利用F2群体将小麦穗粒数、总小穗数、穗长性状定位到4B染色体的同一区段。
穗部性状与产量关系密切,性状间关系错综复杂,都是受多基因控制的数量性状,受环境影响较大[29]。前人大多研究使用的遗传图谱标记密度小,虽然获得了大量的控制穗部性状的 QTL,但因置信区间大,效应值不高,加之遗传背景等因素的影响,可应用于遗传育种的位点及其分子标记非常少。本研究利用构建的高密度遗传图谱在5个环境下进行的穗部相关性状的基因定位,目的是获得稳定的主效位点,为进一步分子标记开发应用及功能基因的挖掘奠定基础。
1 材料与方法
1.1 试验材料
选用主要农艺和产量性状差异大的两个普通小麦品系配制杂交组合,其中母本山农 01-35为大粒品种,但成穗率低,父本藁城 9411表现为小粒、分蘖成穗率高。其杂交F1经一粒传法自交9代,得到含173个家系的RIL群体(F8∶9)。
1.2 田间设计及表型数据分析
RIL群体及其亲本于2009年10月至2013年6月连续4个生长季种植于山东农业大学试验站(E1、E2、E3和E5),2010—2011年度种植于安徽宿州(E4)。泰安点试验地为棕壤,宿州点试验地为黑姜沙土。采取随机区组设计,小区行长2.00 m,行距0.21 m,2次重复,2008—2009年度泰安点为3行区,其余均为4行区。按当地正常栽培方法进行田间管理,生长期内没有发生严重病虫害和倒伏,成熟时按小区机械收获各株系。
从每个株系随机选20株,考察穗长、穗粒数、总小穗数、可育小穗数、不育小穗数;小穗密度 =(总小穗数/穗长) × 100;每株系数500粒称重,2次重复取平均值,换算成千粒重。2次生物学重复测定结果取平均值,作为该试点的表型数据,若重复间误差超过6%,则重新测定。
利用SPSS 19.0对表型数据统计分析。
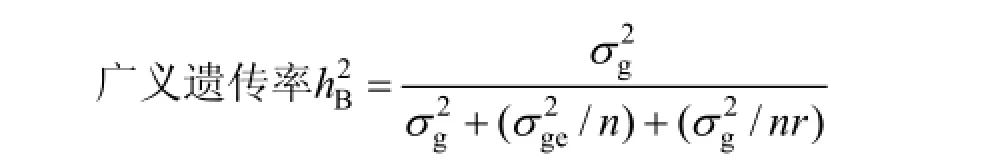
式中,σ2g为遗传方差,σ2ge为遗传与环境互作方差,σ2e为环境方差,n为环境数目,r为实验重复数。
1.3 分子标记检测
DArT标记的检测及序列信息由澳大利亚Triticarte Pty. Ltd. (http∶//www.triticarte.com.au/)完成。SNP分析由美国加利福尼亚大学戴维斯分校植物科学系生物技术检测中心利用 Illumina SNP Genotyping技术测试平台,使用微珠芯片技术(BeadArray)完成,用Genomestudio v1.0软件分析其多态性。SSR引物的序列等信息通过 GrainGene2.0 (http∶//wheat.pw.usda.gov/GG2/index.shtml)获得,引物由上海桑尼生物科技有限公司合成。首先利用两亲本进行多态性筛选,再用筛选出的多态性引物检测RIL群体所有株系。
1.4 遗传图谱及QTL定位
利用陈建省等[30]构建的高密度遗传图谱,该图谱覆盖小麦21条染色体,有6244个标记,其中SNP标记6001个,DArT标记216个,SSR标记27个。染色体总长度为 4895.29 cM,两标记间平均距离为0.78 cM。
用MAPMAKER/EXP3.0b软件和Mapchart 2.1构建分子标记连锁图。利用基于混合线性模型的QTLNetwork 2.0软件,以单个环境中重复的平均值及5个环境平均值分别单独进行QTL定位。以P = 0.05 为统计检测阈值,即当标记的P值小于统计检测阈值时,认为该标记处存在一个与性状有关的QTL。以穗部相关性状英文简写和所对应的染色体序号及在染色体上的位置命名检测到的 QTL,如QGW4B.4-17表示位于4B染色体上第17个标记区间的QTL。最后,将检测到的所有QTL及其上位性互作整合到遗传图谱上。
2 结果与分析
2.1 穗部性状表型分析
5个环境中总体表现,母本山农01-35千粒重57.95 g,明显大于藁城9411的26.80 g,差异达31.15 g;RIL群体没有出现超高亲分离个体,但出现超低亲现象(表1和图1),说明育种中如果要以提高千粒重为目标需有高千粒重亲本;穗长、穗粒数、可育小穗数、不育小穗数和小穗密度出现超高和超低亲分离个体(表1和图1),平均值介于两亲本之间;RIL群体千粒重、可育小穗数偏度和峰度绝对值都小于1,其他性状偏度和峰度绝对值大部分小于1,符合正态分布;千粒重、穗长、不育小穗数、小穗密度变异系数都大于10,说明千粒重、穗长、不育小穗数、小穗密度具有较大的改良潜力,其中不育小穗数改良潜力最大;穗粒数、不育小穗数、小穗密度变幅较大(表1和图1),说明穗粒数、不育小穗数、小穗密度也有较大改良潜力。总小穗数在5个环境中变幅为18.33~20.94,平均19.46;母本总小穗数为17.33~20.50,平均18.76,两亲本在不育小穗数上差异不大。5个环境中RIL群体总小穗数出现超高、超低分离个体。方差分析表明,不育小穗数的基因型与环境互作效应差异不显著,而其他穗部性状基因型与环境互作效应差异极显著(表2)。穗部性状的广义遗传率为62.00%~82.10% (表2),其中可育小穗数广义遗传率最小为62.00%,表明可育小穗数受环境影响较大;小穗密度广义遗传率最大,为82.10%,表明小穗密度主要由基因型控制,受环境影响相对较小。

表1 小麦RIL群体穗部性状在不同环境下表型值Table 1 Phenotypic values about spike related traits for RIL population in different environments
2.2 穗部性状QTL定位
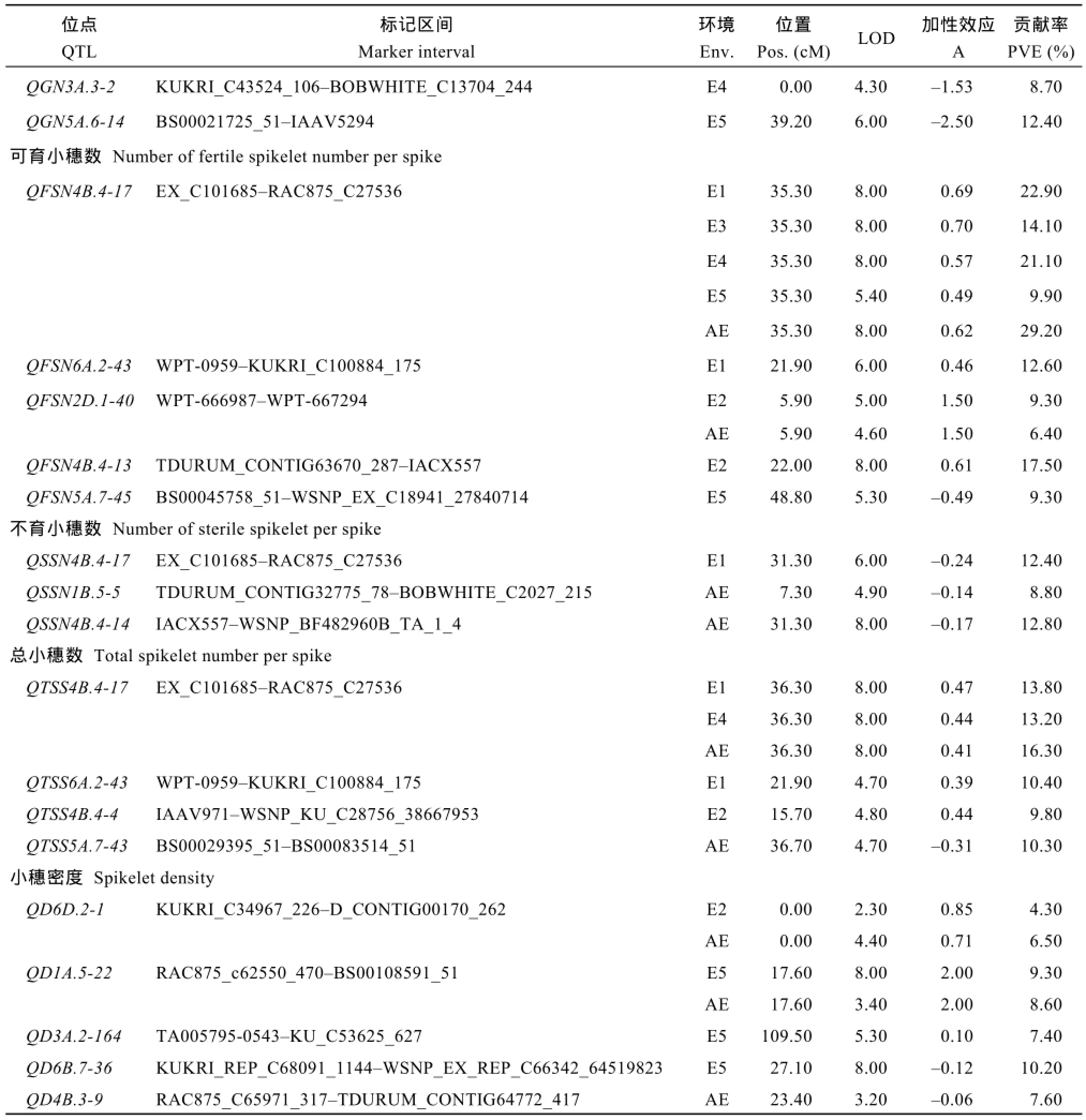
利用单个环境中表型数据及5个环境平均值分别进行QTL分析,共检测到37个穗部性状相关QTL,其中18个位点的贡献率超过10%,为主效QTL(表3)。

(续表1)
检测到位于1B、4B、5B、6A染色体上的7个控制千粒重加性QTL,解释表型变异率6.0%~36.3%,加性效应均来自大粒母本山农 01-35。其中QGW4B.4-17和 QGW4B.4-2贡献率大于 10.0%,尤其是QGW4B.4-17在E3、E4和AE下都被检测到,其贡献率分别为23.3%、36.3%和22.5%,可分别增加千粒重2.56、3.06和2.19 g (表3和图2)。
检测到8个控制穗长的加性QTL,解释表型变异率14.34%~25.44%。位于4B、5A、6B和7B染色体上,其中 QL4B.3-8、QL4B.4-17、QL6B.7-21和QL6B.7-3的贡献率超过10%,为主效QTL (表3和图2)。
3个控制穗粒数的加性QTL分别位于4B、3A 和5A染色体上,单个QTL可增加穗粒数3.22~4.88。其中 QGN4B.4-17在所有环境中均被检测到,贡献率均大于10% (表3和图2)。
5个控制可育小穗数的加性QTL,位于4B、6A、2D、5A上;3个控制不育小穗数的加性QTL位于1B和 4B上,贡献率为 8.7%~37.7%,其加性效应值全部来自父本藁城 9411;还检测到控制总小穗数的 4个加性QTL和控制小穗密度的6个加性QTL (表3和图2)。
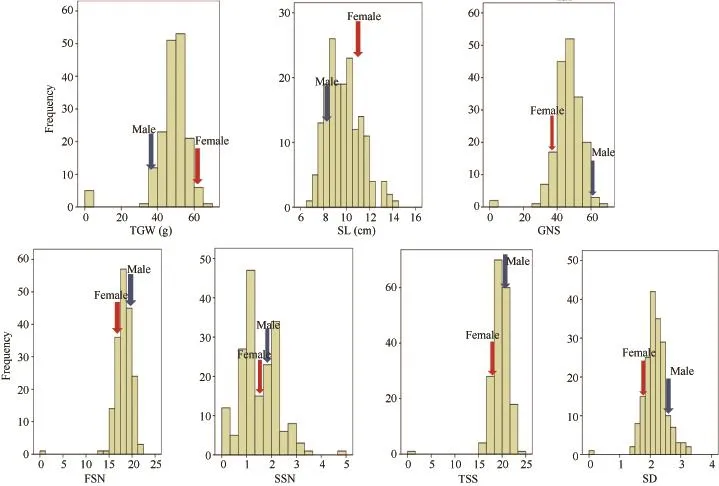
图1 穗部相关性状的变异分布Fig. 1 Frequency distribution of panicle traits
在4B染色体标记 EX_C101685至 RAC875_ C27536区段,存在一个同时控制粒重、穗长、穗粒数、可育小穗数、不育小穗数和总小穗数的位点,且在至少3个环境中检测到,是一个稳定的穗部性状QTL。在6A染色体wPt-0959至TaGw2-CAPS区间,检测到控制粒重和总小穗数的5个 QTL,但该位点只在一个环境中被检测到(表3和图2)。
2.3 环境互作效应分析
5个环境中共检测到32个与环境互作的 QTL,其中5个控制千粒重,10个控制穗长,4个控制穗粒数,2个控制可育小穗数,1个控制不育小穗数,3个控制总小穗数,7个控制小穗密度;这些QTL的贡献率在0.05%~1.05%之间,以位于 EX_C101685-RAC875_ C27536之间的QGN4B.4-17贡献率最大。有7个QTL与单一环境下检测到的加性QTL位置相同,分别位于1A、4B、5A、6B和6D上。千粒重加性效应值都为正值,表现为遗传正效应,可增加千粒重 0.04 g。在 4B染色体EX_C101685至 RAC875_C27536区段,多次检测到 QGW4B.4-17的位点,与单一环境下检测结果基本一致(表4)。
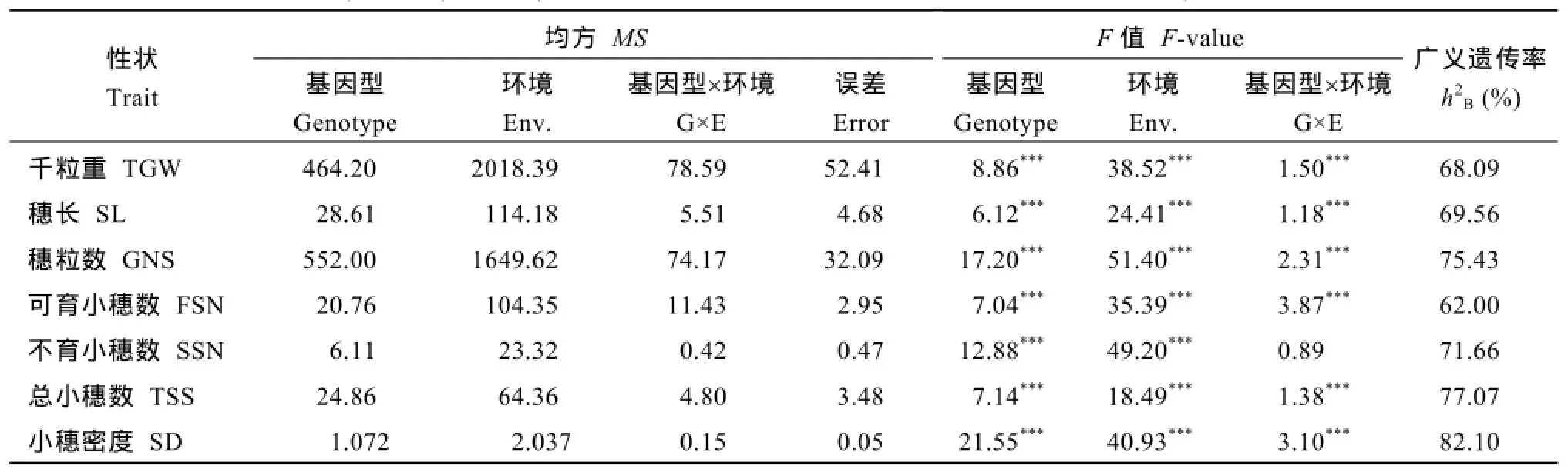
表2 5个环境下穗部性状广义遗传率及方差分析Table 2 Analysis of variance (ANOVA) and broad-sense heritability (h2B) for the spike related traits in five environments
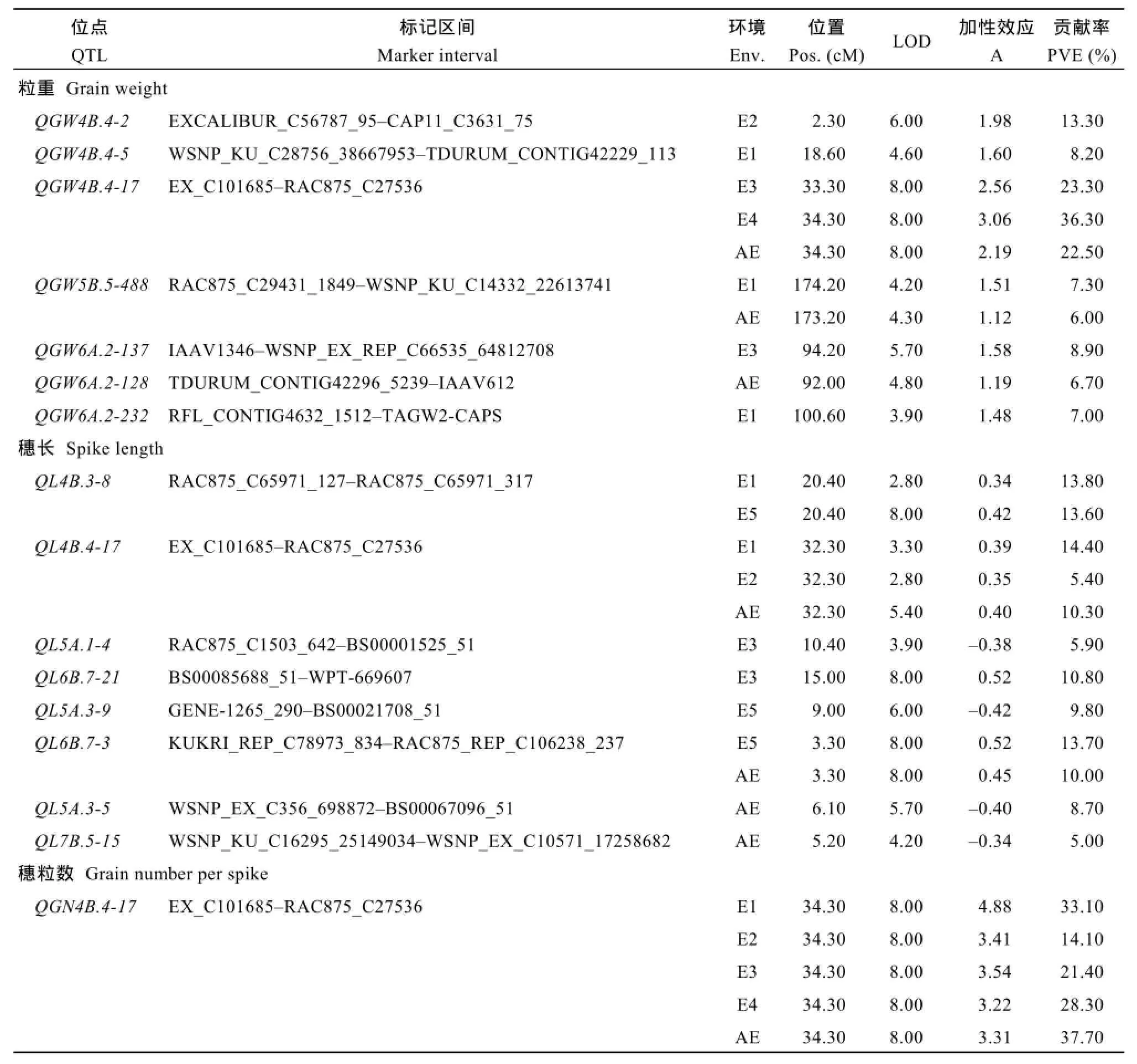
表3 穗部性状的QTLTable 3 Additive QTLs for panicle traits
(续表2)
3 讨论
3.1 群体亲本的选择和遗传图谱的利用

图2 穗部性状的QTL在遗传图谱上的分布Fig. 2 Distribution of identified QTLs for panicle traits on genetic linkage maps

表4 5个环境下小麦穗部性状环境互作效应分析Table 4 Environment interaction of panicle traits in wheat across five environments
本研究的作图群体包含173个RIL株系,其亲本山农01-35和藁城9411在产量构成三要素上差别较大,其中山农 01-35是本实验室创制的大粒特异种质,目前已成为重要的小麦育种亲本。本研究旨在获得能够高效应用于分子辅助育种的有效标记,避免以往研究中仅仅主要利用 2个单一性状极端的远缘亲本构建群体的一些弊端,即获得的分子标记虽然效应值很高,但经常出现与产量性状紧密连锁的不利性状而不能有效被利用。与SSR等传统分子标记相比,SNP具有三个方面的优势,即标记数量多、分布广,在基因组中密度更高和分布更均匀;已实现SNP基因分型的高通量、快速和自动化分析[31];更易实现数据整合比较。本研究中利用最近开发的小麦90 k基因芯片、DArT芯片技术及传统的分子标记技术构建的高密度遗传图谱。此图谱标记间平均距离0.78 cM,包含SSR、DArt和SNP多种类型标记,是当前利用 RIL群体构建的最精密图谱之一[30],某些区段在一定程度上达到传统意义的精细程度。利用该图谱获得的穗部性状分子标记,置信区间小,假阳性低,有助于进一步分子标记开发,并有效应用于精细定位或功能基因克隆。
3.2 QTL定位结果比较
本研究首次报道在 4B染色体标记区间EXCALIBUR_C56787_95-RAC875_C27536检测到控制粒重、穗长、穗粒数、可育小穗数、不育小穗数、总小穗数的8个位置接近的QTL簇(表3和图2),贡献率为5.4%~37.7%。在检测到的37个穗部性状 QTL中,11个 (QGW4B.4-17、QGW5B.5-488、QL4B.3-8、QL4B.4-17、QL6B.7-3、QGN4B.4-17、QFSN4B.4-17、 QFSN2D.1-40、 QTSS4B.4-17、QD6D.2-1和 QD1A.5-22)在多个环境中被检测到,是稳定QTL,而其他26个只在单一环境中被检测到,可能由于穗部性状多数受环境影响大,受多基因控制,遗传率偏低;穗部性状之间相互影响,存在此消彼长现象,因此有的穗部性状QTL在多环境检测中表现不稳定性或环境特异表达特性。
与吴秋红等[18]的检测结果相似,本研究也在5A染色体上检测到控制穗长的 QTL;在该染色体已报道了两个控制穗长 QTL,即位于 4AS的 QSl.cau-4A.1[15,37]和4AL的QSl.cau-4A.2[9,12,36]。此外,4B 和6B上也检测到控制穗长QTL[14]。本研究也在多个环境下发现 4B和 6B上存在控制穗长的 QTL (QL4B.3-8和QL6B.7-3),进一步说明在4B和6B上可能存在控制穗长的重要基因位点。
本研究在3A、4B和5A上检测到控制小穗数的3个稳定 QTL,贡献率为 8.7%~37.7%。Shah等[34]利用3A染色体上的30个RFLP标记和1个形态标记,定位了3个控制穗粒数的QTL。此外,卢翔等[32]和吴秋红等[18]都在 1A染色体上检测到控制穗粒数的QTL。
本研究在5A和6A上检测到控制小穗数的稳定QTL,其贡献率都超过 10%。丁安明等[12]利用 2个群体定位产量相关性状 QTL,也曾报道 2个群体分别在6A和5A上存在控制小穗数的QTL。目前,对穗部不育小穗数相关基因的研究较少,Cui等[9]利用由潍麦8号×济麦20和潍麦8号×烟农19构建的2 个RIL群体(F8∶9),同时在3D、4A和7B染色体上检测到控制基部不育小穗数的 QTL,同时在 6B 染色体上检测到控制可育小穗数的QTL。本研究在多个环境下也在4B、5A和6A上检测到控制不育小穗数和可育小穗数的QTL,贡献率为6.4%~29.2%。
不同的遗传群体和试验地点所定位的同一性状的 QTL 有很大的差异,但也有一些标记区间相同或相近且遗传效应大的 QTL,这些稳定表达的位点对QTL的精细定位、图位克隆和分子标记辅助选择具有很重要的应用价值[33]。
3.3 QTL的一因多效性
穗部相关性状间的关系复杂,同时又往往受到多种因素的影响,在检测小麦籽粒产量及穗部相关性状的QTL过程中,表现出一因多效或紧密连锁效应,即高度相关的性状间有一些共同的位点[19,32]。通过不同性状的QTL共定位可以在一定程度上解释复杂性状及其构成要素或相关性状之间的关系,也在一定程度上证明了该位点的稳定性。
本研究定位到2个穗部性状的QTL富集区,位于4B和6A上,其中4B染色体上EX_C101685至RAC875_C27536的标记区间存在控制粒重、穗长、穗粒数、可育小穗数、不育小穗数、总小穗数的QTL,表明这些QTL可能是一因多效位点,这在以往未曾报道过。丁安明等[14]利用2个定位群体,在6A上检测到控制千粒重、穗粒数和小穗数的QTL簇;本研究在也在6A上检测到控制粒重、总小穗数的QTL,位于标记 wPt-0959-TaGw2-CAPS区间,贡献率为6.7%~12.6%,因此,该染色体区段可能存在穗部性状的一因多效位点。根据本试验结果和前人报道,初步认为在4B、5A和6A上可能存在控制穗部性状的基因,是以后的研究重点。
4 结论
利用基因芯片构建的小麦高密度的遗传图谱,检测到控制穗部相关性状的多个加性效应位点。在4B染色体EX_C101685至RAC875_C27536区段检测到控制粒重、穗长、穗粒数、可育小穗、不育小穗和总小穗数的 QTL,在 6A染色体 wPt-0959至TaGw2-CAPS区段检测到控制粒重和总小穗数的QTL。这些位点的遗传贡献率较大,为穗部性状的分子标记开发、基因精细定位、功能基因克隆奠定了基础。
References
[1] 郑德波,杨小红,李建生,严建兵,张士龙,贺正华,黄益勤.基于 SNP标记的玉米株高及穗位高 QTL定位. 作物学报,2013,39∶ 549-556 Zheng D B,Yang X H,Li J S,Yan J B,Zhang S L,He Z H,Huang Y Q. QTL identification for plant height and ear height based on SNP mapping in maize (Zea mays L.). Acta Agron Sin,2013,39∶ 549-556 (in Chinese with English abstract)
[2] 关强,张月学,徐香玲,孙德全,林红,潘丽艳,马延华. DNA分子标记的研究进展及几种新型分子标记技术. 黑龙江农业科学,2008,(1)∶ 102-104 Guan Q,Zhang Y X,Xu X L,Sun D Q,Li S Y,Lin H,Pan L Y,Ma Y H. Development of DNA molecular marker and several new types of molecular markers. Heilongjiang Agric Sci,2008,(1)∶ 102-104 (in Chinese with English abstract)
[3] Zou Y P,Ge S. A novel molecular marker-SNPs and its application. Biodiversity Sci,2003,11∶ 370-382
[4] Paterson A H,Lander E S,Hewitt J D,Peterson S,Lincoln S E,Tanksley S D. Resolution of quantitative traits into Mendelian factors by using a complete linkage map of restriction fragment length polymorphisms. Nature,1988,335∶ 721-726
[5] Huang X Q,Kempf H,Canal M W,Roder M S. Advanced backcross QTL analysis in progenies derived from a cross between a German elite winter wheat variety and a synthetic wheat (Triticum aestivum L.). Theor Appl Genet,2004,109∶ 933-943
[6] Ma Z Q,Zhao D M,Zhang C Q,Zhang Z Z,Xue S L,Lin F,Kong Z X,Tian D G,Luo Q Y. Molecular genetic analysis of five spike-related traits in wheat using RIL and immortalized F2populations. Mol Genet Genomics,2007,277∶ 31-42
[7] Deng S,Wu X,Wu Y,Zhou R.,Wang H,Jia J,Liu S. Characterization and precise mapping of a QTL increasing spike number with pleiotropic effects in wheat. Theor Appl Genet,2011,122∶281-289
[8] Cui F,Zhao C H,Ding A M,Li J,Wang L,Li X F,Bao Y G,Li J M,Wang H G. Construction of an integrative linkage map and QTL mapping of grain yield-related traits using three related wheat RIL populations. Theor Appl Genet,2014,127∶ 659-675
[9] Cui F,Ding A M,Li J,Zhao C H,Wang L,Wang X Q,Qi X L,Li X F,Li G Y,Gao J R,Wang H G. QTL detection of seven spike-related traits and their genetic correlations in wheat using two related RIL populations. Euphytica,2012,186∶ 177-192
[10] Jia H,Wan H,Yang S,Zhang Z,Kong Z,Xue S,Zhang L,Ma Z. Genetic dissection of yield-related traits in a recombinant inbred line population created using a key breeding parent in China's wheat breeding. Theor Appl Genet,2013,126∶ 2123-2139
[11] Huang X Q,Cloutier S,Lycar L,Radovanovic N,Humphreys D G,Noll J S,Somers D J,Brown P D. Molecular detection of QTLs for agronomic and quality traits in a doubled haploid population derived from two Canadian wheat (Triticum aestivum L.). Theor Appl Genet,2006,113∶ 753-766
[12] 丁安明,李君,崔法,赵春华,马航运,王洪刚. 利用小麦关联 RIL群体定位产量相关性状 QTL. 作物学报,2011,37∶1511-1524 Ding A M,Li J,Cui F,Zhao C H,Ma H Y,Wang H G. QTL mapping for yield related traits using two associated RIL populations of wheat. Acta Agron Sin,2011,37∶ 1511-1524 (in Chinese with English abstract)
[13] 王瑞霞,张秀英,伍玲,王瑞,海林,游光霞,闫长生,肖世和.不同生态环境下冬小麦籽粒大小相关性状的QTL分析. 中国农业科学,2009,42∶ 398-407 Wang R X,Zhang X Y,Wu L,Wang R,Hai L,You G X,Yan C S,Xiao S H. QTL analysis of grain size and related traits in winter wheat under different ecological environments. Sci Agric Sin,2009,42∶ 398-407 (in Chinese with English abstract)
[14] 姚琴,周荣华,潘昱名,傅体华,贾继增. 小麦品种偃展 1号与品系早穗30重组自交系群体遗传连锁图谱构建及重要农艺性状的QTL分析. 中国农业科学,2010,43∶ 4130-4139 Yao Q,Zhou R H,Pan Y M,Fu T H,Jia J Z. Construction of genetic linkage map and QTL analysis of agronomic important traits based on a RIL population derived from common wheat variety Yanzhan 1 and Zaosui 30. Sci Agric Sin,2010,43∶ 4130-4139 (in Chinese with English abstract)
[15] Börner A,Schumann E,Fürste A,Cöster H,Leithold B,Röder M S,Weber W E. Mapping of quantitative trait loci determining agronomic important characters in hexaploid wheat (Triticum aestivum L.). Theor Appl Genet,2002,105∶ 921-936
[16] Sourdille P,Tixier M H,Charmet G,Gay G,Cadalen T,Bernard S,Bernard M. Location of genes involved in ear compactness in wheat (Triticum aestivum) by means of molecular markers. Mol Breed,2000,6∶ 247-255
[17] 张坤普,徐宪斌,田纪春. 小麦籽粒产量及穗部相关性状的QTL定位. 作物学报,2009,35∶ 270-278 Zhang K P,Xu X B,Tian J C. QTL mapping for grain yield and spike related traits in common wheat. Acta Agron Sin,2009,35∶270-278 (in Chinese with English abstract)
[18] 吴秋红,陈娇娇,陈永兴,周升辉,张德云,王国鑫,王振忠,王立新,袁成国,尤明山,刘志勇. 燕大1817/北农6号重组自交系群体穗部性状的QTL定位. 作物学报,2015,41∶349-358 Wu Q H,Chen J J,Chen Y X,Zhou S H,Zhang D Y,Wang G X,Wang Z Z,Wang L X,Yuan C G,You M S,Liu Z Y. Mapping quantitative trait loci related to spike traits using a RILs population of Yanda 1817 × Beinong 6 in wheat (Triticum aestivum L.). Acta Agron Sin,2015,41∶ 349-358 (in Chinese with English abstract)
[19] 宋彦霞,景蕊莲,霍纳新,任正隆,贾继增. 普通小麦(Triticum aestivum L.)不同作图群体抽穗期QTL分析. 中国农业科学,2006,39∶ 2186-2193 Song Y X,Jing R L,Huo N X,Ren Z L,Jia J Z. Detection of QTL for heading in common wheat (Triticum aestivum L.) using different populations. Sci Agric Sin,2006,39∶ 2186-2193 (inChinese with English abstract)
[20] 李文才,李涛,赵逢涛,李兴峰,王洪刚. 小麦 D基因组产量性状QTL定位. 华北农学报,2005,20(1)∶ 23-26 Li W C,Li T,Zhao F T,Li X F,Wang H G. QTL of wheat yield traits in D genome. Acta Agric Boreali-Sin,2005,20(1)∶ 23-26 (in Chinese with English abstract)
[21] Patil R M,Tamhankar S A,Oak M D. Mapping of QTL for agronomic traits and kernel charactersin durum wheat (Triticum durum Desf.). Euphytica,2013,190∶ 117-129
[22] Sears E R. The aneuploids of common wheat. Univ Miss Res Bull,1954,572∶ 1-58
[23] Rao M V P. Mapping of the sphaerococcum gene “S” on chromosome 3D of wheat. Cereal Res Commun,1977,5∶ 15-17
[24] Kato K,Miura H,Sawada S. QTL mapping of genes controlling ear emergence time and plant height on chromosome 5A of wheat. Theor Appl Genet,1999,98∶ 472-477
[25] Paillard S,Schnurbusch T,Winzeler M,Messmer M,Sourdille P,Abderhalden O,Keller B,Schachermayr G. An integrative genetic linkage map of winter wheat (Triticum aestivum L.). Theor Appl Genet,2003,107∶ 1235-1242
[26] Johnson E B,Nalam V J,Zemetra R S,Riera-Lizarazu O. Mapping the compactum locus in wheat (Triticum aestivum L.) and its relationship to other spike morphology genes of the Triticeae. Euphytica,2008,163∶ 193-201
[27] Ma Z Q,Zhao D M,Zhang C Q,Zhang Z Z,Xue S L,Lin F,Kong Z X,Tian D G,Luo Q Y. Molecular genetic analysis of five spike-related traits in wheat using RIL and immortalized F2populations. Mol Genet Genomics,2007,277∶ 31-42
[28] Wang J S,Liu W H,Wang H,Li L H,Wu J,Yang X M,Li X Q,Gao A N. QTL mapping of yield-related traits in the wheat germplasm 3228. Euphytica,2011,177∶ 277-292
[29] Kearsey M J,Pooni H S. The Genetical Analysis of Quantitative Traits. Garland Science (Publishers) Ltd.,Chapman and Hall,London,2004. pp 65-66
[30] 陈建省,陈广凤,李青芳,张晗,师翠兰,孙彩铃,邓志英,刘凯,谷植群,田纪春. 利用基因芯片技术进行小麦遗传图谱构建及粒重QTL分析. 中国农业科学,2014,47∶ 4769-4779 Chen J S,Chen G F,Li Q F,Zhang H,Shi C L,Sun C L,Deng Z Y,Liu K,Gu Z Q,Tian J C. Construction of genetic map using genotyping chips and QTL analysis of grain weight. Sci Agric Sin,2014,47∶ 4769-4779 (in Chinese with English abstract)
[31] 唐立群,肖层林,王伟平. SNP分子标记的研究及其应用进展.中国农学通报,2012,28(12)∶ 154-158 Tang L Q,Xiao C L,Wang W P. Research and application progress of SNP markers. Chin Agric Sci Bull,2012,28(12)∶154-158 (in Chinese with English abstract)
[32] 卢翔,张锦鹏,王化俊,杨欣明,李秀全,李立会. 小麦-冰草衍生后代3558-2穗部相关性状的遗传分析和QTL定位. 植物遗传资源学报,2011,12∶ 86-91 Lu X,Zhang J P,Wang H J,Yang X M,Li X Q,Li L H. Genetic Analysis and QTL mapping of wheat spike traits in a derivative line 3558-2 from wheat × Agropyron cristatum offspring. J Plant Genet Resour,2011,12∶ 86-91 (in Chinese with English abstract)
[33] Cavanagh C R,Chao S,Wang S,Huang B E,Stephen S,Kiani S,Forrest K,Saintenac C,Brown-Guedira G L,Akhunova A. Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars. Proc Natl Acad Sci USA,2013,110∶ 8057-8062
[34] Shah M M,Gill K S,Baenziger P S,Yen Y,Kaeppler S M,Ariyarathne H M. Molecular mapping of loci for agronomic traits on chromosome 3A of bread wheat. Crop Sci,1999,39∶ 1728-1732
[35] Li Q F,Zhang Y,Liu T T,Wang F F,Liu K,Chen J S,Tian J C. Genetic analysis of kernel weight and kernel size in wheat (Triticum aestivum L.) using unconditional and conditional QTL mapping. Mol Breed,2015,35∶ 194
[36] Jantasuriyarat C,Vales M I,Waston C J W,Riera-Lizarazu O. Identification and mapping of genetic loci affecting the free-threshing habit and spike compactness in wheat (Triticum aestivum L.). Theor Appl Genet,2004,108∶ 261-273
[37] 王瑾,廖祥政,杨学举,周荣华,贾继增. 人工合成小麦Am3大穗多粒 QTL的发掘与利用. 植物遗传资源学报,2008,9∶ 277-282 Wang J,Liao X Z,Yang X J,Zhou R H,Jia J Z. Mapping of large-spike and much-kernel QTL by using a synthetic wheat Am3 as donor. J Plant Genet Resour,2008,9∶ 277-282 (in Chinese with English abstract)
Mapping QTLs For Wheat Panicle Traits with High Density SNP Genetic Map
LIU Kai,DENG Zhi-Ying,LI Qing-Fang,ZHANG Ying,SUN Cai-Ling,TIAN Ji-Chun*,and CHEN Jian-Sheng*
Group of Wheat Quality Breeding,College of Agronomy,Shandong Agricultural University / State Key Laboratory of Crop Biology,Tai'an 271018,China
Abstract:Panicle traits of wheat are closely correlated between each other,of them grain number per spike and 1000-grain weight are important components of grain yield. In this study,we mapped quantitative trait loci (QTLs) associated with wheat spike traits using a recombinant inbred line (RIL) population (173 lines of F8∶9) derived from a cross of Shannong 01-35 × Gaocheng 9411. The phenotypic data were collected in five environments and the high density genetic map was constructed using 90 k SNP array,DArT technology and traditional molecular markers. In a combination analysis of five environments,many additive QTLs were detected including seven for 1000-grain weight,eight for spike length,three for grain number per spike,five for fertile spikelet number per spike,three for sterile spikelet number per spike,four for spikelet number per spike,and six for spike density. Some QTLs showed high rates of phenotypic variation explained (PVE). For example,the PVE of QTLs for 1000-grain weight on 1B,4B,5B,and 6A ranged from 6.00% to 36.30%,with the favorable alleles from the large-grain parent Shannong 01-35;the PVE of QTLs for spike length ranged from 14.34% to 25.44%,and that for sterile spikelet number per spike from 8.70% to 37.70%. In addition to additive loci,32 pairs of epistatic QTLs were detected,which explained 0.05-1.05% of the phenotypic variations. The marker interval between EX_C101685 and RAC875_C27536 on chromosome 4B showed pleiotropic effects in 1000-grain weight,spike length,grain number per spike,fertile spike number,sterile spikelet number,and spikelet number per spike,with the PVE ranging from 5.40% to 37.70%. There stable main QTLs were detected in multiple environments. Besides,marker interval between wPt-0959 and TaGw2-CAPS on 6A had a locus controlling both 1000-grain weight and spikelet number per spike. Theseresults are valuable in developing molecular markers,fine mapping and cloning genes for spike traits in wheat.
Keywords:Common wheat;90 k array;QTL mapping;Panicle;SNP
DOI:10.3724/SP.J.1006.2016.00820
*通讯作者(
Corresponding authors)∶ 陈建省,E-mail∶ jshch@sdau.edu.cn,Tel∶ 0538-8249236;田纪春,E-mail∶ jctian@sdau.edu.cn
收稿日期Received()∶ 2015-11-09;Accepted(接受日期)∶ 2016-03-14;Published online(网络出版日期)∶ 2016-03-21.
