籼粳稻基因组295个InDel标记的开发
2016-07-14初志战郭海滨曾栋昌刘耀光华南农业大学生命科学学院亚热带农业生物资源保护与利用重点实验室广东广州5064华南农业大学公共基础课实验教学中心广东广州5064
初志战郭海滨曾栋昌刘耀光,*华南农业大学生命科学学院 / 亚热带农业生物资源保护与利用重点实验室,广东广州 5064;华南农业大学公共基础课实验教学中心,广东广州 5064
籼粳稻基因组295个InDel标记的开发
初志战1郭海滨2曾栋昌1刘耀光1,*1华南农业大学生命科学学院 / 亚热带农业生物资源保护与利用重点实验室,广东广州 510642;2华南农业大学公共基础课实验教学中心,广东广州 510642
摘 要:插入/缺失(InDel)分子标记具有使用简单,结果清晰可靠的优点。本研究通过比对粳稻品种日本晴和籼稻品种93-11的基因组序列,在全基因组范围内设计了634对InDel候选标记,通过PCR检测比较2种粳稻(日本晴和台中65)和2种籼稻(93-11和黄华占)的多态性,发现295对标记在2种籼稻间及2种粳稻间均带型一致,而在籼、粳亚种间有多态性,因此这套295对标记可以在涉及籼粳亚种的基因定位和分子育种中应用。
关键词:InDel标记;水稻;基因定位
本研究由亚热带农业生物资源保护与利用国家重点实验室开放课题(SKL-CUSAb-2013-04)和广东省自然科学基金-博士启动项目(2015A030310485)资助。
This study was supported by the Open Fund Project of State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources (SKL-CUSAb-2013-04) and the Natural Science Fund of Guangdong Province-the Launch Program for Doctor (2015A030310485).
第一作者联系方式∶ E-mail∶ chuben@scau.edu.cn
URL∶ http∶//www.cnki.net/kcms/detail/11.1809.S.20160314.1444.004.html
自2002年完成水稻基因组测序后,水稻基因组的研究重心开始转向功能基因[1]的鉴定。目前大多水稻基因功能的研究还是以正向遗传学为主∶ 先通过物理或化学方法诱导基因突变,再经过构建定位群体,利用分子标记定位和筛选突变基因。分子标记是继形态标记、细胞标记和生化标记后发展起来的一种更理想的遗传标记。随着分子标记技术的不断发展,到目前为止己经建立20多种,例如RFLP (restriction fragment length polymorphisms)标记、RAPD (random amplified polymorphic DNAs)标记、AFLP (amplified fragment length polymorphisms)标记等,其中被广泛用于基因定位的有 SSR (simple sequence repeat)标记、InDel (Insertion/Deletion)标记和SNP (single nucleotide polymorphisms)标记。栽培稻在长期的栽培驯化过程中,形成籼稻和粳稻2个主要的亚种,由于这2个亚种的农艺性状、生理生化特性及基因组序列存在明显差异,常被用来作为构建定位群体的双亲。双亲间DNA序列的多态性是发展分子标记的基础。Shen等[2]比对粳稻(japonica)品种日本晴和籼稻(indica)品种93-11的全基因组序列,发现平均每268 bp有1个SNP,每953 bp有1个InDel,这2种标记均有很高的密度,尤其 SNP分子标记在理论上具有极大的潜力,但是目前由于检测方法的限制,并未广泛采用SNP分子标记于基因定位尤其是初步定位。SSR标记技术由Moore等于1991年创立[3],GRAMENE网站提供了2240个水稻SSR标记(http∶//archive.gramene.org/markers/)。在水稻基因组中平均每157 kb有1个SSR标记[4-5],而平均每10.36 kb有1个基因[6],因此SSR分子标记密度还远未达到图位克隆的要求。冯芳君等[7]通过测验20对InDel标记和53对SSR标记在46份粳稻和47份籼稻的遗传多样性实验中发现,InDel标记的PCR扩增稳定性比SSR标记好,扩增产物非特异条带少,特异性更好,因此分离效果明显好过 SSR标记。我们的大量应用实践也表明,InDel分子标记具有使用简单和电泳结果比 SSR标记清晰可靠的优点。
本研究通过比对日本晴和93-11的全基因组序列,以缺失5~20 bp为标准设计634对InDel候选标记,以期找出覆盖全基因组的具有籼粳共同差异的高通用性的InDel标记。
1 材料与方法
1.1 水稻材料
包括2份籼稻(93-11和黄华占)和2份粳稻(日本晴和台中65)。
1.2 InDel标记的设计
以每隔 1~2 Mb的距离从 RGP网站(http∶//rgp.dna. affrc.go.jp/)或者 GRAMENE网站(http∶//www.gramene.org/)下载对应的 BAC克隆序列,然后在 NCBI网站(http∶//www.ncbi.nlm.nih.gov/blast/)的 BLAST网页,将此BAC序列与93-11基因组序列进行BLAST比较,以中间缺失 5~20个碱基,两边高度保守的区域为理想的 InDel设计区。为了后续检测方便,InDel设计长度一般为100~160 bp,尽量不超过200 bp,引物的Tm值为55~60℃。
1.3 DNA提取方法
在三至四叶期,从4种材料中各选1个单株采集新鲜叶片,以略加修改的SDS法[8]提取叶片基因组总DNA。
1.4 PCR扩增及电泳检测
20 μL的PCR体系中包含模板DNA 0.5 μL、10 mmol L-1dNTPs 0.3 μL、10 μmol L-1正反引物各0.5 μL、10 × PCR buffer 2 μL和Taq DNA聚合酶0.5~0.6 U,ddH2O 17 μL。
PCR程序为94℃预变性3 min;94℃ 30 s,55℃ 30 s,72℃ 25 s,32个循环;72℃延伸3 min。
1.5 电泳检测及记录方法
20 μL PCR产物加4 μL 6×loading buffer于52孔电泳槽,每孔上样4 μL,8.8%聚丙烯酰胺凝胶180V稳压电泳70 min,用银染法[9]染色。
2 结果与分析
2.1 InDel标记的筛选结果
对供试的634对候选InDel标记进行PCR检测,选出在2种籼稻和2种粳稻之间显示共同多态的标记(图1),共获得 295对籼-粳差异 InDel标记。这些标记在每条染色体上的数目为14~35个,平均间隔为0.76~1.60 Mb (表1)。这套标记的引物信息列在表2。
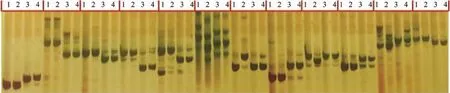
图1 InDel候选标记在4个品种的PCR检测Fig. 1 Detection of candidate InDel markers among four rice cultivars by PCR amplification
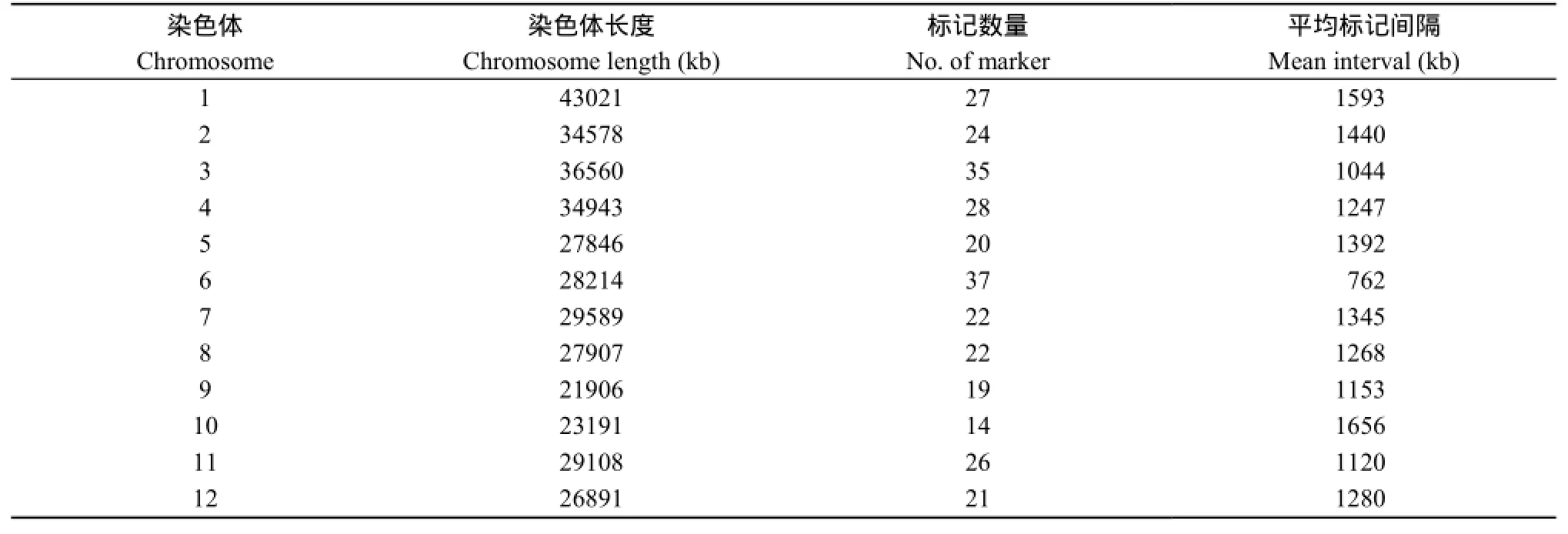
表1 各染色体的InDel标记分布Table 1 Distribution of InDel markers on the chromosomes

表2 籼粳间有共同多态性的295对InDel标记引物Table 2 Primer sets of 295 InDel markers polymorphic between indica and japonica rice

(续表2)

(续表2)

(续表2)

(续表2)

(续表2)
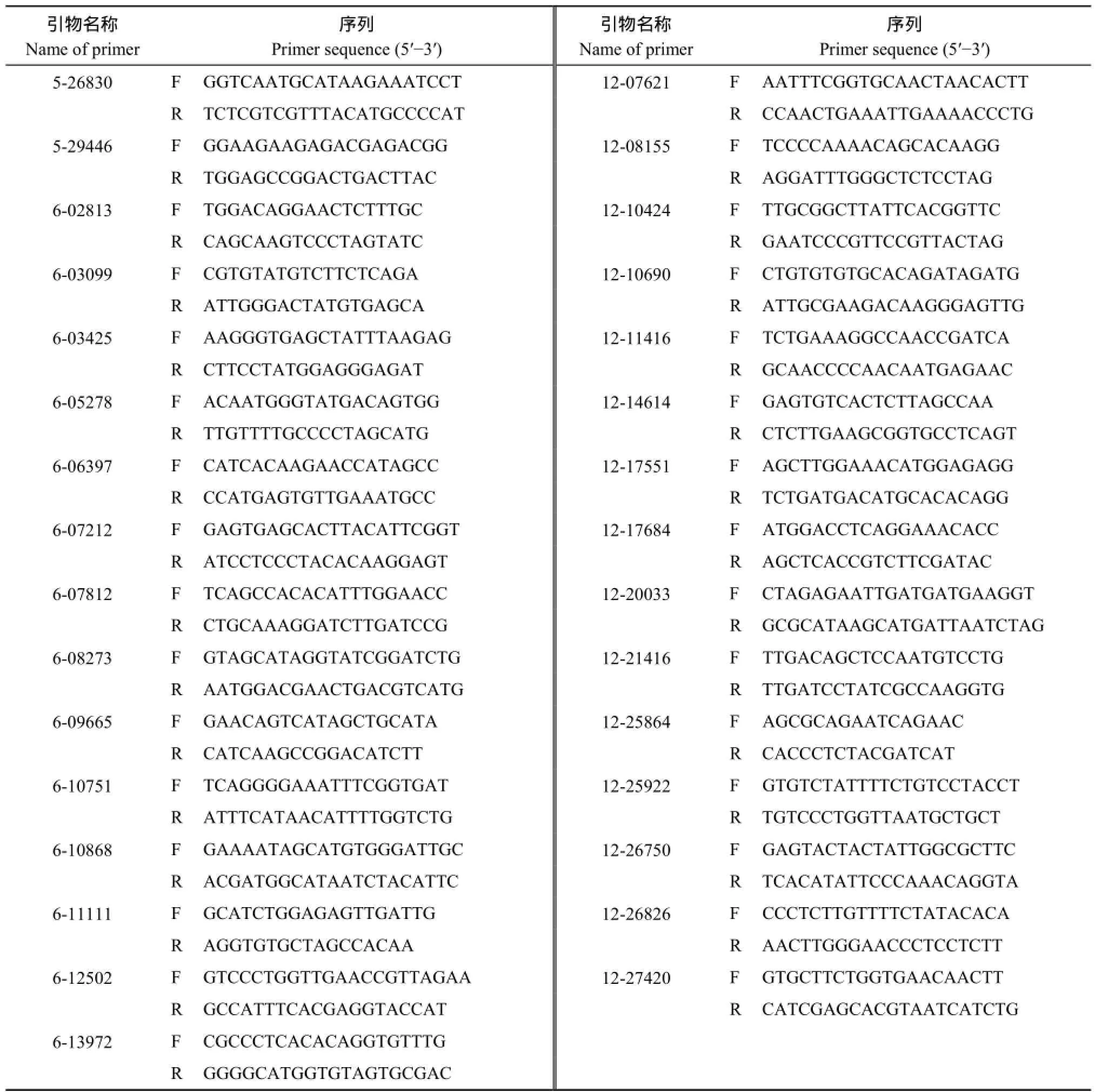
(续表2)
3 讨论
水稻不仅是世界上最主要的粮食作物之一,也是遗传学和基因组研究的模式植物。随着分子标记种类的不断开发,标记数量的不断增加,分子标记的应用领域也在不断拓展。目前分子标记技术除了被广泛应用于基因定位外,还大量应用于水稻籼、粳分类[10-12],作物育种[13],物种进化关系研究[14],作物种子纯度鉴定及材料的品种鉴定[15-16]等诸多领域。在水稻基因定位方面,由于InDel分子标记比SSR标记密度更大,因此应用前景更好。本研究获得295对InDel标记可以作为以籼、粳(尤其是这4种材料)为亲本的图位克隆分子标记。每条染色体上的标记间隔在0.7~1.6 Mb之间,相当于遗传距离约4~10 cM,基本上可以满足初定位的需要。
致谢: 本文中有些标记由本实验室多位研究生提供,在此一并致谢。
References
[1] Jeon J S,An G. Gene tagging in rice∶ a high throughput systemfor functional genomics. Plant Sci,2001,161∶ 211-219
[2] Shen Y J,Jiang H,Jin J P,Zhang Z B,Xi B,He Y Y,Wang G,Qian L L,Li X,Yu Q B,Liu H J,Chen D H,Gao J H,Huang H,Shi T L,Yang Z N. Development of genome-wide DNA polymorphism database for map-based cloning of rice genes. Plant Physiol,2004,135∶ 1198-1205
[3] 王林友,张礼霞,勾晓霞,范宏环,金庆生,王建军. 利用InDel标记鉴定浙优系列杂交稻籼粳属性和预测杂种优势. 中国农业科学,2014,47∶ 1243-1255 Wang L Y,Zhang L X,Gou X X,Fan H H,Jin Q S,Wang J J. Identification of indica-japonica attribute and prediction of heterosis of Zheyou hybrids rice using indel molecular markers. Sci Agric Sin,2014,47∶ 1243-1255 (in Chinese with English abstract)
[4] Temnykh S,DeClerck G,Lukashova A,Lipovich L,Cartinhour S,McCouch S. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.)∶ frequency,length variation,transposon associations,and genetic marker potential. Genome Res,2001,11∶ 1441-1452
[5] McCouch S R,Teytelman L,Xu Y,Lobos K B,Clare K,Walton M,Fu B Y,Maghirang R,Li Z K,Xing Y Z,Zhang Q F,Kono L,Yano M,Fjellstrom R,DeClerck G,Schnbeider D,Cartinhour S,Ware D,Stein L. Development and mapping of 2240 new SSR markers for rice (Oryza sativa L.). DNA Res,2002,9∶ 199-207
[6] International Rice Genome Sequencing Project. The map-based sequence of the rice genome. Nature,2005,436∶ 793-800
[7] 冯芳君,罗利军,李荧,周立国,徐小艳,吴金,陈宏伟,陈亮,梅捍卫. 水稻InDel和SSR标记多态性的比较分析. 分子植物育种,2005,3∶ 725-730 Feng F J,Luo L J,Li Y,Zhou L G,Xu X Y,Wu J,Chen H W,Chen L,Mei H W. Comparative analysis of polymorphism of In-Del and SSR markers in rice. Mol Plant Breed,2005,3∶ 725-730 (in Chinese with English abstract)
[8] Guillemaut P,Mardchal-Drouard L. Isolation of plant DNA∶ a fast,inexpensive,and reliable method. Plant Mol Biol Rep,1992,10∶ 60-65
[9] Bassam B J,Caetano-Anollés G,Gresshoff P M. Fast and sensitive silver staining of DNA in polyacrylamide gels. Anal Biochem,1991,196∶ 80-83
[10] 王明军,王云月,陆春明,杨慧,王云涛. 利用籼粳稻特异InDe标记分析云南糯稻品种的籼粳特性. 云南农业大学学报(自然科学版),2010,25∶ 333-337 Wang M J,Wang Y Y,Lu C M,Yang H,Wang Y T. Characteristic of Yunnan Glutinous rice varieties revealed by the indica-japonica specified Insertion/Deletion (InDel) molecular markers. J Yunnan Agric Univ (Nat Sci),2010,25∶ 333-337 (in Chinese with English abstract)
[11] 姚国新,黄文超. 利用水稻籼粳分化InDel标记鉴定育种材料的籼粳属性. 杂交水稻,2013,28(3)∶ 53-57 Yao G,Huang W. Identifying indica-japonica characteristic of rice breeding materials with indica-japonica differentiation InDel markers. Hybrid Rice,2013,28(3)∶ 53-57 (in Chinese with English abstract)
[12] Qian H R,Zhuang J Y,Lin H X,Lu J,Zheng K L. Identification of a set of RFLP probes for subspecies differentiation in Oryza sativa L. Theor Appl Genet,1995,90∶ 878-884
[13] Dudley J W,Maroof M A,Rufener G K. Molecular markers and grouping of parents in maize breeding programs. Crop Sci,1991,31∶ 718-723
[14] Joshi S P,Gupta V S,Aggarwal R K,Ranjekar P K,Brar D S. Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR) polymorphism in the genus Oryza. Theor Appl Genet,2000,100∶ 1311-1320
[15] Moreno S,Martín J P,Ortiz J M. Inter-simple sequence repeats PCR for characterization of closely related grapevine germplasm. Euphytica,1998,101∶ 117-125
[16] Prevost A,Wilkinson M J. A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars. Theor Appl Genet,1999,98∶ 107-112
Development of 295 InDel Markers for Indica and Japanica Rice
CHU Zhi-Zhan1,GUO Hai-Bin2,ZENG Dong-Chang1,and LIU Yao-Guang1,*1College of Life Sciences,South China Agricultural University / State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources,Guangzhou 510642,China;2Center of Experimental Teaching for Common Basic Course,South China Agricultural University,Guangzhou 510642,China
Abstract:Insertion/Deletion (InDel) markers have advantages of simplicity and reliability for genotyping. We selected 634 candidate InDel markers distributing throughout the 12 chromosomes of rice by comparing genome sequences between the japonica cultivar Nipponbare and the indica cultivar 93-11. PCR results of these candidate markers between two japonica (Nipponbare and Taizhong 65) and two indica (93-11 and Huanghuazhan) cultivars revealed that 295 InDel markers displayed common polymorphisms between the japonica and indica cultivars which are useful for gene mapping and molecular breeding involving in indica and japonica subspecies.
Keywords:InDel marker;Rice;Gene mapping
DOI:10.3724/SP.J.1006.2016.00932
*通讯作者(
Corresponding author)∶ 刘耀光,E-mail∶ ygliu@scau.edu.cn
收稿日期Received()∶ 2015-11-09;Accepted(接受日期)∶ 2016-01-11;Published online(网络出版日期)∶ 2016-03-14.
