小麦全基因组NBS类R基因分析及2AL染色体NBS-SSR特异标记开发
2016-07-14乔麟轶常建忠郭慧娟高建刚郑军畅志坚1山西大学生命科学学院山西太原00006山西省农业科学院作物科学研究所作物遗传与分子改良山西省重点实验室山西太原0001山西省农业科学院小麦研究所山西临汾01000山西省农业科学院旱地农业研究中心山西太原0001北京市农林科学院杂交小麦工程技术研究中心北京100097
乔麟轶常建忠郭慧娟高建刚郑 军畅志坚,*1山西大学生命科学学院,山西太原 00006;山西省农业科学院作物科学研究所 / 作物遗传与分子改良山西省重点实验室,山西太原0001;山西省农业科学院小麦研究所,山西临汾 01000;山西省农业科学院旱地农业研究中心,山西太原0001;北京市农林科学院杂交小麦工程技术研究中心,北京 100097
小麦全基因组NBS类R基因分析及2AL染色体NBS-SSR特异标记开发
乔麟轶1,2常建忠4郭慧娟2高建刚5郑 军3,*畅志坚1,2,*1山西大学生命科学学院,山西太原 030006;2山西省农业科学院作物科学研究所 / 作物遗传与分子改良山西省重点实验室,山西太原030031;3山西省农业科学院小麦研究所,山西临汾 041000;4山西省农业科学院旱地农业研究中心,山西太原030031;5北京市农林科学院杂交小麦工程技术研究中心,北京 100097
摘 要:NBS (nucleotide binding site)类基因是植物界中最重要的一类抗病基因。用信息学方法从普通小麦(Triticum aestivum L.)全基因组中分离出2406条含有NBS结构的完整蛋白序列,每条包含48~2272个氨基酸残基。根据NBS结构域两端是否连接CC或LRR结构域,将TaNBS分为N、CN、NL和CNL 4类。对TaNBS所在scaffold序列的SSR位点进行诊断,从1203条scaffold序列上发现2177个SSR位点,以二碱基重复位点最多,占73.5%。针对小麦2AL染色体上的51个SSR位点开发标记,缺体-四体和双端体验证结果表明,有39个标记(76.5%)为2AL特异标记,其中24个特异标记在抗白粉病材料Khapli (2AL上携带Pm4a)和感病材料Chancellor间存在多态性。利用近等基因系Khapli/8*Cc筛选出3个可能与Pm4a连锁的NBS-SSR标记,分别是Sxaas_2AL22、Sxaas_2AL39和Sxaas_2AL46。本研究开发的与抗病序列紧密连锁的特异SSR标记可用于2AL染色体上抗病新基因的检测以及已有抗病基因的候选序列筛选。
关键词:小麦;抗病;NBS类基因;SSR标记;2AL染色体
本研究由国家自然科学基金项目(31171839,31401385),山西省青年基金项目(2015021145),山西省农业攻关项目(20150311001-1,20150311001-5),山西省农业科学院攻关项目(15YGG01)和北京市农林科学院青年基金项目(QNJJ201428)资助。
This study was supported by the National Natural Science Foundation of China (31171839,31401385),Shanxi Province Science Foundation for Youths (2015021145),Shanxi Province Technologies R&D Program (20150311001-1,20150311001-5),the Technologies R&D Program of the Shanxi Academy of Agricultural Sciences (15YGG01),and the Youth Foundation of Beijing Academy of Agricultural and Forestry Sciences (QNJJ201428).
第一作者联系方式∶ E-mail∶ qiaoly1988@126.com
URL∶ http∶//www.cnki.net/kcms/detail/11.1809.S.20160321.1056.002.html
小麦是世界的主要口粮作物之一,是世界第二大粮食作物。近40年来,由于白粉病、锈病等病害的大面积流行,小麦生产不断遭到严重威胁。以白粉病为例,由于小麦种植密度加大、水肥条件改善和品种抗源单一等原因,小麦白粉病从我国西南和东南沿海局部地区迅速蔓延至全国所有麦区,发病面积从1980年的100万公顷扩展到2015年的570万公顷(http∶//cb.natesc.gov.cn/)。1990年和 1991年全国白粉病大爆发分别造成当年1.44亿千克和0.77亿千克小麦产量的损失[1]。抗病品种是有效控制小麦主要病害的最经济和有效的方法[2],也是小麦育种的重要目标。但是,随着病原菌的变异和新类型不断产生,原有抗病品种逐渐丧失抗性,使小麦生产面临新的挑战。因此,广泛挖掘抗病基因是一项持续而紧迫的工作,是选育小麦抗病品种的重要前提。
分子标记选择技术极大地促进了抗病基因的鉴定,尤其是简单重复序列(SSR)标记,因其大量分布于基因岛和重组热点区域,且变异常与临近基因的交换重组密切相关[3],从而被广泛应用于小麦抗病基因的定位。目前,正式命名并定位到小麦分子图谱上的抗病基因包括54个抗白粉病基因、74个抗条锈病基因、73个抗叶锈病基因和57个抗秆锈病基因,其中有13个基因被成功克隆[4-14]。在已克隆基因中,Pm3b[4]、Pm3c[5]、Pm3f[6]、Pm3g[5]、Yr10[7]、Lr1[8]、Lr10[9]、Lr21[10]、Sr33[11]、Sr35[12]10个基因均为NBS类抗病基因。NBS基因是植物界最重要、家族成员最多的一类抗病基因,其编码的NBS结构域包含 Kinase1、Kinase2、Kinase3、HD残基等保守基序[15],可通过结合ATP或GTP来直接或间接引导宿主细胞产生识别病原体效应,并参与抗病信号的传导过程[16]。在单子叶植物中,一些NBS结构域的N端有时连接有一个与蛋白互作有关的CC (coiled-coil)结构,而 C端通常连接有一个与病原菌识别相关的LRR (leucine-rich repeat)结构域[17]。
史静东等[18]曾利用巢式PCR技术获得4个小麦NBS类抗病基因片段,并通过群体检测,证实其中一个片段与Yr26共分离。随着小麦测序草图的公布,在全基因组范围内对小麦NBS基因家族的研究成为可能。Bouktila等[19]利用普通小麦品种中国春的454测序数据[20],分离了包含NBS结构的小麦序列,然而这些序列在染色体上的具体位置尚不得知。本研究利用基于染色体臂分离测序技术的小麦基因组数据[21],从全基因组范围内分离了小麦NBS序列,诊断了序列邻近的SSR位点,并在2AL染色体上针对NBS-SSR位点开发分子标记,筛选出染色体臂特异标记,同时利用小麦抗、感材料,对新开发的标记进行了多态性分析和抗病基因连锁性检测。本研究结果丰富了小麦抗病基因的分子标记,可用于抗病基因的标记辅助检测以及候选序列筛选。
1 材料与方法
1.1 植物材料
小麦品种Khapli (2AL染色体上携带Pm4a)由中国农业科学院作物科学研究所贾继增研究员惠赠,Pm4a的近等基因系 Khapli/8*Cc及其轮回亲本Chancellor (Cc)由中国农业科学院植物保护研究所周益林研究员惠赠。用于分子标记检测的中国春缺体-四体材料和双端体材料由美国农业部种质研究中心惠赠。
1.2 家族蛋白序列的分离
从URGI数据库(http∶//wheat-urgi.versailles.inra. fr/)下载小麦全基因组数据建立 BLAST蛋白数据库和核酸数据库。从Pfam数据库(http∶//pfam.sanger.ac. uk/)中下载NBS家族(Pfam登录号为PF00931)的隐马尔可夫模型文件,用HMM 3.0软件检索本地蛋白数据库得到目标氨基酸序列,设置E≤1e-5。
1.3 结构域分析和蛋白序列特征分析
利用SMART服务器(http∶//smart.embl-hei-delberg. de/)对检索结果进行 NBS蛋白保守结构域的检查,参数为默认值。筛选得到的序列递交NCBI的CDD数据库(http∶//www.ncbi.nlm.nih.gov/Structure/cdd),进一步分析与NBS关联的CC、LRR等其他结构域的特征。
用DNAstar软件中的Editseq工具获得TaNBS蛋白的长度信息。用Clustal X软件进行蛋白序列多重比对,参数为默认值,用Jalview输出比对结果。
1.4 染色体定位
利用小麦NBS蛋白序列编号在本地核酸数据库中提取相应的CDS序列和基因组(scaffold)序列。将scaffold序列返回本地核酸数据库检索,获得其在小麦基因组中的位置信息,从而将 TaNBS排列到染色体上。
1.5 NBS-SSR位点诊断和引物设计
利用SSRhurnter软件查找TaNBS所在scaffold序列的SSR位点。设定每重复单元碱基数为2~5,重复次数≥5。利用Primer5软件对SSR位点设计引物,由生工生物工程(上海)有限公司合成(表1)。
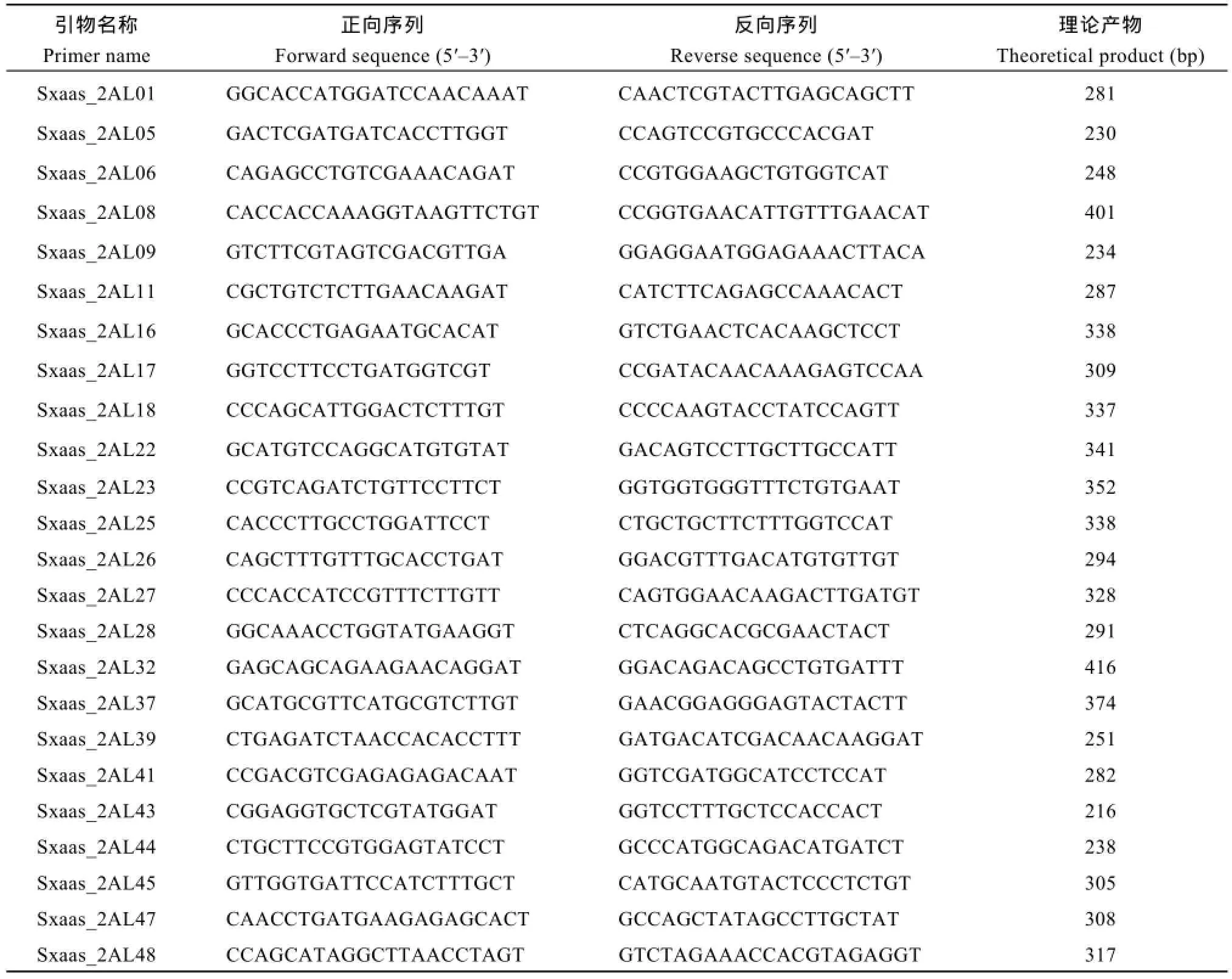
表1 24对多态性的小麦2AL染色体特异标记Table 1 Twenty-four polymorphic markers specific to wheat chromosome 2AL
1.6 DNA提取和SSR标记检测
采用SDS法提取基因组总DNA。PCR总体系为15 μL,含1.5 μL 10× buffer (TaKaRa)、0.2 mmol L-1dNTP、1 U Taq酶(TaKaRa)、0.25 μmol L-1引物和100 ng模板DNA。反应扩增程序为94℃变性5 min;94℃变性45 s,58℃复性45 s,72℃延伸 1 min,36个循环;72℃延伸10 min。扩增产物经8%非变性聚丙烯酰胺凝胶(Acr与 Bis质量比为 29∶1)电泳,硝酸银染色后观察照相。
2 结果与分析
2.1 小麦 NBS家族基因的染色体分布和结构分类
利用NBS隐马模型文件在小麦本地蛋白数据库检索,并检测检索结果,获得2406条含有NBS结构的完整蛋白序列,分布于小麦基因组21条染色体上,以 B基因组比率最高(37.4%),其次是 A基因组(34.3%),而D基因组最低(28.3%);从单个染色体看,在4A染色体上分布最多,达248条,而在4D染色体上分布最少,仅24条(图1)。

图1 小麦NBS序列的染色体分布Fig. 1 Distribution of NBS sequences on chromosomes in wheat
分离出的TaNBS序列长度差异较大,最短的序列Ta1asLoc007418只有48个氨基酸残基,最长的为Ta7blLoc002272,有2272个氨基酸残基。根据是否包含CC和LRR结构域,将2406个TaNBS序列分为4类,分别是CC-NBS-LRR、NBS-LRR、CC-NBS 和NBS,其中只具有NBS结构的TaNBS最多,有1209条,占总数的50.2% (图2)。
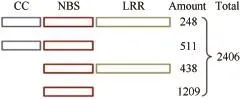
图2 小麦TaNBS序列的结构分类Fig. 2 Structural classification of TaNBS protein sequences
2.2 小麦NBS-SSR位点诊断
从本地核酸数据库中提取TaNBS的CDS序列和基因组(scaffold)序列,查找所在 scaffold序列的SSR位点,在其中的1203条scaffold序列上共检测到2177个SSR位点,A、B、D基因组分别占33%、40%和27% (图3-a)。其中二碱基重复位点最多,占73.5%;五碱基重复位点最少,仅0.3% (图3-b)。
2.3 2AL染色体上TaNBS序列特征分析
将位于2AL上的56个TaNBS蛋白序列多重比对的结果显示,一些TaNBS序列与已克隆抗白粉病基因的NBS结构域有高度相似的基序(图4),如用于结合 ATP/GTP的磷酸结合环P-loop (GGV/LGKTT),以及Kinase2 (VLDDVW)、Kinase3 (GSxxLA/VTTR)和末端HD残基。推测这些TaNBS蛋白也可能具有类似的抗病作用。2.4 2AL染色体NBS-SSR标记开发及检测
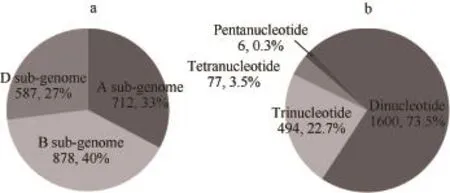
图3 小麦全基因组NBS-SSR位点诊断Fig. 3 Genome-wide diagnosis of NBS-related SSR loci in wheat
在小麦2AL染色体上56个TaNBS所在的基因组序列片段中,共有25个scaffold存在NBS-SSR位点,通过比对 2AL基因组数据获得的位置信息,将这25个scaffold在2AL染色体臂上线性排列(表2)。在这些基因组片段中共检测到52个SSR位点,除了位于 Sca1697100起始端口的重复位点(TG)5以外,对其余51个SSR位点的侧翼序列分别设计引物,依次命名为Sxaas_2AL01~Sxaas_2AL51。
对51个2AL-NBS-SSR标记进行特异性筛选,有39个标记在缺体-四体N2BT2A、N2BT2D、N2DT2A、N2DT2B和中国春中均扩增出目标带,而在N2AT2B、N2AT2D和双端体 Dt2AS中没有目标带,表明这些NBS-SSR标记为2A染色体长臂特异标记。
利用抗病品种Khapli及其 Pm4a-NIL群体、感病轮回亲本 Chancellor,对39个2AL-NBS-SSR特异标记检测发现,24个标记(表1)在 Khapli和Chancellor中的扩增产物存在差异,表明这些特异标记在抗、感材料间具有较好的多态性,其中Sxaas_2AL22、Sxaas_2AL39和 Sxaas_2AL46在Pm4a-NILs中的扩增条带与Khapli中的一致,表明这3个标记很可能与Pm4a连锁(图5)。

图4 小麦2AL上部分TaNBS结构域多重序列比对Fig. 4 Multiple alignment of some TaNBS sequences on chromosome 2AL

表2 小麦2AL染色体上52个NBS-SSR位点Table 2 Fifty-two NBS-SSR loci on chromosome 2AL of wheat

图5 Sxaas_2AL22、Sxaas_2AL39和Sxaas_2AL46的特异性及与Pm4a的连锁性检测Fig. 5 Detection of chromosome specificity and the genetic
3 讨论
3.1 小麦NBS家族概况
NBS类基因在苔藓植物、石松植物、裸子植物和被子植物中均有相关研究报道[17],是植物中数量最多的抗病基因家族。而小麦作为世界上最重要、种植最广的农作物,常年遭受白粉菌、条锈菌、叶锈菌、秆锈菌及赤霉菌等病原菌的侵染,深入研究小麦NBS类基因对于减少小麦产量损失显得尤为重要。
本研究从小麦基因组中分离出2406个完整TaNBS序列,数目远高于粳稻的 653个[22]、籼稻的553个[22]、大豆的319个[23]、高粱的211个[24]以及玉米的109个[25]。我们还分别从乌拉尔图小麦和粗山羊草的基因组中分离出681条和946条NBS序列(结果未列出),由此初步推测,普通小麦TaNBS家族是由其3个亚基因组供体物种的NBS家族通过两次多倍体化事件整合而来。此外,在TaNBS中还存在多个NBS基因位于一个scaffold上的现象,仅2AL染色体上就有8个这样的基因簇(部分见表2),推断TaNBS家族可能还经历了串联重复事件,最终造成小麦基因组中NBS庞大的成员数目。然而同一同源群中各染色体上的NBS数目并不相近,如4A染色体上有248个TaNBS基因,而4B和4D上分别只具有 40个和 24个,这可能由于六倍体小麦基因组复制而造成大量基因的功能冗余,因此冗余的部分TaNBS基因将会变成假基因或者逐渐丢失,最终形成各基因组间基因数目的不同现象。
TaNBS蛋白包含NBS的4个特征基序,根据两端是否连接CC或LRR结构域,可将其分为N、CN、NL和CNL四类,已克隆的10个小麦NBS类抗病基因均为CNL结构[4-12]。一些TaNBS还包含其他类型的结构域,如 Ta1blLoc007763、Ta2bsLoc001037 和 Ta3alLoc007069等包含 STK (serine/threonine kinase)结构;Ta2bsLoc003709和Ta2bsLoc014737等包含 ABC (ATP-binding cassette)转运蛋白结构;Ta2alLoc020734、Ta2dlLoc021209和Ta6bsLoc009346等包含Jacalin结构;Ta2alLoc012596、Ta2blLoc002392、Ta5dlLoc013799和 Ta5dlLoc017355等包含锌指结构。其中 STK结构域和 ABC转运结构域分别是Pm21和Lr34的功能域[13-14],Jacalin结构域和锌指结构域也被证实与小麦抗病性有关[26-27]。这些结构域通常与NBS结构域互相作用,共同抵抗病原菌的入侵。类似研究已有报道,如拟南芥中 RRS1蛋白的NBS域C端连接一个WRKY结构域,该WRKY结构域在拟南芥对青枯菌的响应通路中起抑制作用,与NBS结构域一起形成独特的抗病机制[28]。因此下一步有必要对这些包含其他类型结构域的TaNBS深入研究。
3.2 NBS-SSR特异标记在基因定位及分子育种中的应用价值
广泛发掘、鉴定抗病基因是小麦抗病育种的基础,在抗病基因定位过程中应用最广的分子标记是SSR标记。然而普通小麦包含A、B、D 3个基因组和高比例的重复序列(80%)[21],常规SSR标记在小麦染色体间存在多位点现象,很可能造成定位结果的偏差。本研究以小麦 2A染色体长臂为例,对TaNBS基因所在scaffold上的51个SSR位点设计引物,通过缺体四体和双端体材料筛选,有 39个(76.5%) SSR标记具有染色体特异性,且具有良好的多态性。目前我们正在用作图群体将这些NBS-SSR特异标记定位到小麦遗传图谱,而小麦2AL上存在抗白粉病基因Pm4a[29]、Pm4b[30]、Pm4c[31]、Pm4d[32]、Pm50[33],抗条锈病基因Yr1[34]、Yr32[35],抗叶锈病基因Lr38[36]和抗秆锈病基因Sr21[37]等多个抗病基因位点,其中Sr21所在区段更是与短柄草和水稻染色体上的 NBS基因簇同源[37],因此 2AL-NBS-SSR特异标记的开发对上述基因的精细定位以至克隆有重要的参考价值。另外本研究从小麦全基因组中共诊断出2177个 NBS-SSR位点,通过特异性筛选和群体遗传定位,预计可开发出1000余对位置明确、染色体特异的NBS-SSR标记,这将显著减少小麦抗病基因定位过程中存在的误差。
目前,在小麦中被正式命名的各类抗病基因将近 300个左右,其中只有少数几个被用于生产,大部分抗病基因在未用于育种之前就有可能因病原菌变异而面临失去抗性的危险。真正利用分子标记辅助选择(marker-assisted selection,MAS)育出的小麦抗病品种鲜有报道,一个很重要原因就是分子标记与目标基因距离较远,造成MAS过程中分子标记与目标基因重组,导致标记跟踪信息丢失。虽然基于小麦基因组测序数据已开发出高密度、高精度的SNP芯片,但费用较高,暂时不适合用于育种过程中的大群体筛选。本研究从提高常规分子标记的精准度入手,开发的每个SSR标记均与小麦NBS序列位于同一条 scaffold序列上,当某个 TaNBS被确定与抗病性相关时,其最近的NBS-SSR标记与之紧密连锁,几乎不发生重组,可以作为共分离标记使用,将提高育种过程中MAS的效率。
4 结论
从普通小麦基因组中分离出分布于各染色体的2406条含有NBS结构的完整蛋白序列,按结构分为N、CN、NL和CNL共4类,每条序列包含48~2272个氨基酸残基。在TaNBS所在基因组序列中的1203 条scaffold序列上诊断出2177个SSR位点,其中二碱基重复位点占73.5%。针对小麦2AL染色体上的51个NBS-SSR位点开发标记,筛选出39个2AL染色体特异标记,包括24个在抗白粉病材料Khapli和感病材料 Chancellor间存在多态性,其中 Sxaas_ 2AL22、Sxaas_2AL39和Sxaas_2AL46可能与Pm4a连锁。
致谢: 北京安贞医院/北京市心肺血管疾病研究所李小燕博士在信息学分析中给予了宝贵指导,谨致谢忱。
References
[1] Li H J,Wang X M. Thinopyrum ponticum and Th. intermedium∶the promising source of resistance to fungal and viral diseases of wheat. J Genet Genomics,2009,36∶ 557-565
[2] Line R F,Chen X M. Success in breeding for and managing durable resistance to wheat rusts. Plant Dis,1995,79∶ 1254−1255
[3] Ott A,Trautschold B,Sandhu D. Using microsatellites to understand the physical distribution of recombination on soybean chromosomes. PLoS One,2011,6∶ e22306
[4] Yahiaoui N,Srichumpa P,Dudler R,Keller B. Genome analysis at different ploidy levels allows cloning of the powdery mildew resistance gene Pm3b from hexaploid wheat. Plant J,2004,37∶528-538
[5] Tommasini L,Yahiaoui N,Srichumpa P,Keller B. Development of functional markers specific for seven Pm3 resistance alleles and their validation in the bread wheat gene pool. Theor Appl Genet,2006,114∶ 165-175
[6] Srichumpa P,Brunner S,Keller B,Yahiaoui N. Allelic series of four powdery mildew resistance genes at the Pm3 locus in hexaploid bread wheat. Plant Physiol,2005,139∶ 885-895
[7] Liu W,Frick M,Huel R,Nykiforuk C L,Wang X,Gaudet D A,Eudes F,Conner R L,Kuzyk A,Chen Q,Kang Z,Laroche A. The stripe rust resistance gene Yr10 encodes an evolutionaryconserved and unique CC-NBS-LRR sequence in wheat. Mol Plant,2014,7∶ 1740-1755
[8] Cloutier S,McCallum B D,Loutre C,Banks T W,Wicker T,Feuillet C,Keller B,Jordan M C. Leaf rust resistance gene Lr1,isolated from bread wheat (Triticum aestivum L.) is a member of the large psr567 gene family. Plant Mol Biol,2007,65∶ 93-106
[9] Sela H,Spiridon L N,Petrescu A J,Akerman M,Mandel-Gutfreund Y,Nevo E,Loutre C,Keller B,Schulman A H,Fahima T. Ancient diversity of splicing motifs and protein surfaces in the wild emmer wheat (Triticum dicoccoides) LR10 coiled coil (CC)and leucine-rich repeat (LRR) domains. Mol Plant Pathol,2012,13∶ 276-287
[10] Huang L,Brooks S A,Li W,Fellers J P,Trick H N,Gill B S. Map-based cloning of leaf rust resistance gene Lr21 from the large and polyploid genome of bread wheat. Genetics,2003,164∶655-664
[11] Periyannan S,Moore J,Ayliffe M,Bansal U,Wang X,Huang L,Deal K,Luo M,Kong X,Bariana H,Mago R,McIntosh R,Dodds P,Dvorak J,Lagudah E. The gene Sr33,an ortholog of barley Mla genes,encodes resistance to wheat stem rust race Ug99. Science,2013,341∶ 786-788
[12] Saintenac C,Zhang W,Salcedo A,Rouse M N,Trick H N,Akhunov E,Dubcovsky J. Identification of wheat gene Sr35 that confers resistance to Ug99 stem rust race group. Science,2013,341∶ 783-786
[13] Cao A,Xing L,Wang X,Yang X,Wang W,Sun Y,Qian C,Ni J,Chen Y,Liu D,Wang X,Chen P. Serine/threonine kinase gene Stpk-V,a key member of powdery mildew resistance gene Pm21,confers powdery mildew resistance in wheat. Proc Natl Acad Sci USA,2011,108∶ 7727-7732
[14] Krattinger S G,Lagudah E S,Spielmeyer W,Singh R P,Espino J H,McFadden H,Bossolini E,Selter L L,Keller B. A putative ABC transporter confers durable resistance to multiple fungal pathogens in wheat. Science,2009,323∶ 1360-1363
[15] Meyers B C,Kozik A,Griego A,Kuang H,Michelmore R W. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis. Plant Cell,2003,15∶ 809-834
[16] Bourne H R,Sanders D,McCormick F. The GTPase superfamily∶conserved structure and molecular mechanism. Nature,1991,349∶117-127
[17] Belkhadir Y,Subramaniam R,Dangl J L. Plant disease resistance protein signaling∶ NBS-LRR proteins and their partners. Curr Opin Plant Biol,2004,7∶ 391-399
[18] 史静东,张小娟,黄丽丽,韩德俊,康振生. 小麦 NBS-LRR类抗病基因类似片段分离和定位. 中国农业科学,2013,46∶2022-2031 Shi J D,Zhang X J,Huang L L,Han D J,Kang Z S. Isolation and mapping of NBS-LRR resistance gene homology sequences from wheat. Sci Agric Sin,2013,46∶ 2022-2031 (in Chinese with English abstract)
[19] Rachel B,Manuel S,Matthias P,Gary L A B,Rosalinda D A,Alexandra M A,Neil M,Melissa K,Arnaud K,Dan B,Suzanne K,Darren W,Martin T,Ian B,Gu Y,Huo N X,Luo M C,Sunish S,Bikram G,Sharyar K,Olin A,Paul K,Jan D,Richard M,Anthony H,Klaus F M,Keith J E,Michael W B,Hall N. Analysis of the bread wheat genome using whole-genome shotgun sequencing. Nature,2012,491∶ 705-710
[20] Bouktila D,Khalfallah Y,Habachi Y,Mezghani M,Makni M,Makni H. Full-genome identification and characterization of NBS-encoding disease resistance genes in wheat. Mol Genet Genomics,2015,290∶ 257-271
[21] International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome. Science,2014,345∶ 1251788
[22] Shang J,Tao Y,Chen X,Zou Y,Lei C,Wang J,Li X,Zhao X,Zhang M,Lu Z,Xu J,Cheng Z,Wan J,Zhu L. Identification of a new rice blast resistance gene,Pid3,by genome wide comparison of paired nucleotide-binding site leucine-rich repeat genes and their pseudogene alleles between the two sequenced rice genomes. Genetics,2009,182∶ 1303-1311
[23] Kang Y J,Kim K H,Shim S,Yoon M Y,Sun S,Kim M Y,Van K,Lee S H. Genome-wide mapping of NBS-LRR genes and their association with disease resistance in soybean. BMC Plant Biol,2012,12∶ 139
[24] Paterson A H,Bowers J E,Bruggmann R,Dubchak I,Grimwood J,Gundlach H,Haberer G,Hellsten U,Mitros T,Poliakov A,Schmutz J,Spannagl M,Tang H,Wang X,Wicker T,Bharti A K,Chapman J,Feltus F A,Gowik U,Grigoriev I V,Lyons E,Maher C A,Martis M,Narechania A,Otillar R P,Penning B W,Salamov A A,Wang Y,Zhang L,Carpita N C,Freeling M,Gingle A R,Hash C T,Keller B,Klein P,Kresovich S,McCann M C,Ming R,Peterson D G,Mehboob-ur-Rahman,Ware D,Westhoff P,Mayer K F,Messing J,Rokhsar D S. The Sorghum bicolor genome and the diversifcation of grasses. Nature,2009,457∶ 551-556
[25] Cheng Y,Li X,Jiang H,Ma W,Miao W,Yamada T,Zhang M. Systematic analysis and comparison of nucleotide-binding site disease resistance genes in maize. FEBS J,2012,279∶ 2431-2443
[26] Ma Q H,Zhen W B,Liu Y C. Jacalin domain in wheat jasmonate-regulated protein Ta-JA1 confers agglutinating activity and pathogen resistance. Biochimie,2013,95∶ 359-365
[27] Guo J,Bai P,Yang Q,Liu F,Wang X,Huang L,Kang Z. Wheat zinc finger protein TaLSD1,a negative regulator of programmed cell death,is involved in wheat resistance against stripe rust fungus. Plant Physiol Biochem,2013,71∶ 164-172
[28] Deslandes L,Olivier J,Theulieres F,Hirsch J,Feng D X,Bittner-Eddy P D,Beynon J,Marco Y. Resistance to Ralstonias olanacearum in Arabidopsis thaliana is conferred by the recessive RRS1-R gene,a member of a novel family of resistance genes. Proc Natl Acad Sci USA,2002,99∶ 2404-2409
[29] Ma Z Q,Wei J B,Cheng S H. PCR-based markers for the powdery mildew resistance gene Pm4a in wheat. Theor Appl Genet,2004,109∶ 140-145
[30] Yi Y J,Liu H Y,Huang X Q,An L Z,Wang F,Wang X L. Development of molecular markers linked to the wheat powdery mildew resistance gene Pm4b and marker validation for molecular breeding. Plant Breed,2008,127∶ 116-120
[31] Hao Y,Liu A,Wang Y,Feng D,Gao J,Li X,Liu S,Wang H. Pm23∶ a new allele of Pm4 located on chromosome 2AL in wheat. Theor Appl Genet,2008,117∶ 1205-1212
[32] Michael Schmolke M,Mohler V,Hartl L,Zeller F J,Hsam S K. A new powdery mildew resistance allele at the Pm4 wheat locus transferred from einkorn (Triticum monococcum). Mol Breed,2012,29∶ 449-456
[33] Mohler V,Bauer C,Schweizer G,Kempf H,Hartl L. Pm50∶ a new powdery mildew resistance gene in common wheat derived from cultivated emmer. J Appl Genet,2013,54∶ 259-263
[34] Bariana H S,McIntosh R A. Cytogenetic studies in wheat∶ XV. Location of rust resistance genes in VPM1 and their genetic linkage with other disease resistance genes in chromosome 2A. Genome,1993,36∶ 476-482
[35] Eriksen L,Afshari F,Christiansen M J,McIntosh R A,Jahoor A,Wellings C R. Yr32 for resistance to stripe (yellow) rust present in the wheat cultivar Carstens V. Theor Appl Genet,2004,108∶567-575
[36] Friebe B,Zeller F J,Mukai Y,Forster B P,Bartos P,McIntosh R A. Characterization of rust-resistant wheat-Agropyron intermedium derivatives by C-banding,in situ hybridization and isozyme analysis. Theor Appl Genet,1992,83∶ 775-782
[37] Chen S,Rouse M N,Zhang W,Jin Y,Akhunov E,Wei Y,Dubcovsky J. Fine mapping and characterization of Sr21,a temperature-sensitive diploid wheat resistance gene effective against the Puccinia graminis f. sp. tritici Ug99 race group. Theor Appl Genet,2015,128∶ 645-656
Genome-Wide Analysis of TaNBS Resistance Genes and Development of Chromosome 2AL-specific NBS-SSR Markers in Wheat
QIAO Lin-Yi1,2,CHANG Jian-Zhong4,GUO Hui-Juan2,GAO Jian-Gang5,ZHENG Jun3,*,and CHANG Zhi-Jian1,2,*1College of Life Science,Shanxi University,Taiyuan 030006,China;2Institute of Crop Science,Shanxi Academy of Agricultural Sciences / Shanxi Key Laboratory of Crop Genetics and Molecular Improvement,Taiyuan 030031,China;3Institute of Wheat Research,Shanxi Academy of Agricultural Sciences,Linfen 041000,China;4Institute of Dryland Farming,Shanxi Academy of Agricultural Sciences,Taiyuan 030031,China;5Hybrid Technology Engineering Research Center of Wheat,Beijing Academy of Agriculture and Forestry Sciences,Beijing 100097,China
Abstract:Nucleotide binding site (NBS)-encoding genes are the most important resistance genes (R genes) in plant kingdom. In this study,2406 full-length NBS protein sequences,of which single protein varies from 48 aa to 2272 aa,were identified from wheat (Triticum aestivum L.) genome using bioinformatic method. These TaNBSs were divided into four categories,including N,CN,NL,and CNL,according to whether the NBS domains connect CC or LRR domains at both ends. Diagnosis results showed that 1203 of all the scaffolds with TaNBS contained 2177 simple sequence repeats (SSR) loci,73.5% of which were dinucleotide repeat sites. Based on the NBS-SSR loci on chromosome 2AL in wheat,we developed 51 molecular markers,and 39 of them (76.5%) were confirmed as chromosome specific markers using Chinese Spring nulli-tetrasomic and ditelosomic lines. Further-more,24 2AL-specific NBS-SSR markers showed polymorphism between resistant material Khapli carried Pm4a on chromosome 2AL and susceptible material Chancellor. Finally,three 2AL-specific NBS-SSR markers,Sxaas_2AL22,Sxaas_2AL39,and Sxaas_2AL46,were probably linked to Pm4a gene based on genetic linkage test using Pm4a-NILs (Khapli/8*Cc). These chromosome specific 2AL-NBS-SSR markers can be used to locate novel R genes or screen the candidate sequences for known R genes on chromosome 2AL.
Keywords:Wheat;Disease resistance;NBS-encoding genes;SSR markers;Chromosome 2AL
DOI:10.3724/SP.J.1006.2016.00795
*通讯作者(
Corresponding authors)∶ 畅志坚,E-mail∶ wrczj@126.com;郑军,E-mail∶ zhengjsxaas@126.com
收稿日期Received()∶ 2015-10-20;Accepted(接受日期)∶ 2016-03-14;Published online(网络出版日期)∶ 2016-03-21.
