绵羊不同部位脂肪组织microRNA高通量测序及生物信息学分析
2016-07-14胡子乔景炅婕潘洋洋乔利英李宝钧刘建华刘文忠
张 方,胡子乔,景炅婕,潘洋洋,乔利英,李宝钧,刘建华,刘文忠
(山西农业大学动物科技学院,太谷 030801)
绵羊不同部位脂肪组织microRNA高通量测序及生物信息学分析
张方#,胡子乔#,景炅婕,潘洋洋,乔利英,李宝钧,刘建华,刘文忠*
(山西农业大学动物科技学院,太谷 030801)
本研究旨在挖掘绵羊不同脂肪组织中特异表达的miRNAs。构建肾周(PEF)、皮下(SUF)和尾部(TAF)脂肪组织的3个小RNA文库,利用Solexa测序技术进行测序,用Stem-loop qRT-PCR技术和生物信息学方法对测序结果进行验证和分析。结果表明,8个随机选择的miRNAs中有7个miRNAs的表达与测序结果一致。在肾周和皮下脂肪组织的sRNA文库中分别检测到128和112个保守miRNAs,分别发现198和196个新miRNAs。结合前期在尾脂中检测到miRNAs的分析结果,肾周脂肪中特异表达的有12个保守的和66个新miRNAs;皮下脂肪中特异表达的保守和新miRNAs分别为1个和30个;尾脂中特异表达的保守和新miRNAs分别为1个和34个。上述结果将为进一步研究miRNA调控绵羊的脂肪代谢提供科学依据。
绵羊;脂肪组织;microRNA;Solexa测序;生物信息学方法
miRNA是一类广泛存在于动植物中,具有全局调控作用,长约22 nt的内源性非编码的单链小RNA分子。它们通过调控基因表达,广泛参与细胞的分化、增殖与凋亡、个体发育与器官形成等众多生物学过程,所以被称为多细胞生物中最主要的基因表达调控因子[1-2]。然而,其调控基因表达的主要方式是通过与靶基因mRNA 3′非翻译区的特异性碱基互补配对,进而引起靶mRNA降解、稳定性下降或者翻译抑制[3-4]。研究绵羊脂肪组织miRNA表达谱和特征对于进一步揭示其在绵羊脂肪细胞增殖、分化和凋亡中的作用及其机理具有重要意义。
脂肪是人和动物贮存能量最主要的形式。然而对于肉用动物来说,脂肪的过量或过少沉积均会影响人和动物健康,或者影响肉的品质和风味。所以,对肉用动物脂肪代谢的研究至关重要。miRNA在脂肪代谢中的作用首先在果蝇中报道。miR-14的缺失可引起果蝇细胞凋亡和表型特征的改变,脂肪细胞的脂滴增大,三酰甘油和二酰甘油的水平升高[5]。随后的研究表明,miR-335在脂质加工过程中表达上调,且在肥胖小鼠的脂肪组织中高度表达[6]。miR-143是最早发现的调节脂肪细胞分化的miRNA。有人研究发现,miR-143抑制剂可以抑制人类脂肪细胞的分化并下调PPARγ2、FABP和GLUT4基因的表达[7]。此外,体外研究表明,miR-143可促进牛的成纤维样前体脂肪细胞分化为成熟的脂肪细胞[8]。此外,还有很多miRNAs在脂类代谢中都发挥着重要的调控作用[9-10]。这表明miRNA对脂肪代谢具有重要调控作用。
目前,miRNA在小鼠、人、猪、牛等动物的脂肪代谢过程中的研究都有大量报道,但在绵羊脂肪代谢中的研究仅见少量文献报道。本研究拟通过对绵羊脂肪组织小RNA进行Solexa测序,并进行Stem-loop RT-PCR验证和生物信息学分析,挖掘在脂肪组织中特异表达的miRNAs,为进一步研究miRNA调控绵羊脂肪代谢过程提供科学依据。
1 材料与方法
1.1材料
试验羊为8只饲养管理条件一致的10月龄广灵大尾羊,公母各半。对其进行屠宰,宰后迅速采集皮下(SUF)、尾部(TAF)和肾周脂肪(PEF)样品,分别装入无RNase的离心管中,立即置于液氮中保存,用于总RNA的提取。本试验中屠宰程序和饲养管理按照中华人民共和国国家标准(GB13078-2001和GB/T17237-1998)和农业行业标准(NY 5148-2002)进行。
1.2方法
1.2.1总RNA提取及Solexa测序样品在液氮中研磨至粉末状,然后按照Trizol(Invitrogen,Gaithersburg,MD,USA)法提取绵羊脂肪组织总RNA。将4只公羊的总RNA放入泡沫盒中用干冰作为保护剂密封送往深圳华大基因科技有限公司完成文库的构建:先检测所送样品的质量及浓度,合格的miRNA 利用15%变性聚丙烯酰胺凝胶电泳技术(PAGE)从总RNA中分离小片段(15~30 nt)RNA,纯化后对分离的小片段RNA连接3′和5′接头。由此构建皮下、尾部和肾周脂肪3个小RNA(sRNA)文库。再运用SuperScriptⅡ反转录酶将连接有5′和3′接头的RNA进行反转录,合成cDNA。然后,进行PCR扩增,扩增程序:98 ℃预变性30 s;98 ℃变性10 s,60 ℃退火30 s,72 ℃延伸15 s,14个循环;72 ℃延伸8 min;4 ℃保存。最后,扩增产物用Solexa高通量测序方法测序。
1.2.2生物信息学分析
1.2.2.1绵羊保守miRNAs的鉴定:对上述测序获得的原始序列数据进行去接头、去低质量、去污染等过程的处理,然后统计序列总数(total)、种类(unique)和长度分布,最后得到的全部有效序列用于后续分析。将有效序列用SOAP软件与NCBI的绵羊基因组进行比对注释,分析sRNA在基因组上的表达与分布情况。与GenBank和Rfam(12.0)数据库进行比对,注释测序后的sRNA序列(这两个数据库间的优先级为GenBank>Rfam),尽可能发现并去除rRNA、tRNA、scRNA、snoRNA、snRNA等非编码小RNAs。用tag2repeat软件将sRNA与重复序列进行比对,得到repeat associate sRNA。将sRNA与mRNA的外显子和内含子序列进行比对,找出来自mRNA的降解片段。这些降解片段就是sRNA。与miRBase 21.0中已知的绵羊miRNA的成熟序列或前体序列进行比对并注释,得到物种内保守的miRNAs及其含量,并统计这些miRNAs各位点的碱基分布。本研究中,仅分析PEF和SUF两个文库的测序结果,关于TAF的测序分析结果参见文献[11]。
1.2.2.2统计不同部位脂肪组织中特异表达的miRNA:根据测序结果分别统计3个不同部位脂肪组织中有表达的保守miRNAs和新miRNAs,用Venny2.0.2(http://bioinfogp.cnb.csic.es/tools/venny/)软件做韦恩图,分析统计特异表达的miRNAs。
1.2.3绵羊新miRNAs的预测由于miRNA前体带有的标志性发夹结构能够用来预测新的miRNA,所以对在GenBank和Rfam(12.1)数据库中没有比对上任何注释信息的sRNA,根据绵羊基因组数据,利用商用软件Mireap(http://sourceforge.net/projects/mireap/)分析其发夹结构、Dicer酶切位点信息及能量等特征。预测标准:首先,筛选miRNA的成熟体必须位于茎环结构的茎干部位,且序列长度介于20~24 nt;其次,Drosha和Dicer酶的剪切位点至少在3个碱基以上;最后,允许候选miRNA前体最小自由能不能大于-75.24 kJ·mol-1[12]。 1.2.4Stem-loop qRT-PCR验证 用于测序结果验证的样本为8只广灵大尾羊。从测序结果中随机选取8个miRNAs,以U6 snRNA作为内参基因[13],根据这8个miRNAs的成熟序列设计反转录及荧光定量PCR引物。利用SYBR PrimeScriptTMmiRNA RT-PCR Kit反转录试剂盒和SYBR®Premix Ex TaqⅡ定量试剂盒,对目标miRNAs在绵羊脂肪组织中的表达进行检测。检测严格参照试剂盒说明书进行,相对表达量结果采用2-ΔΔCt法进行计算[14-15]。miRNA引物及内参基因序列见表1。
表1miRNAs PCR引物序列及U6引物序列
Table 1PCR primer sequences for miRNAs and U6 primer sequence

miRNAs和内参基因miRNAsandreferencegene引物序列(5'-3')PrimersequencemiR-103F:ACACTCCAGCTGGGAGCAGCATTGTACAGGR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCATAGCCCmiR-143F:ACACTCCAGCTGGGTGAGATGAAGCACTGR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGAGCTACAmiR-191F:ACACTCCAGCTGGGCAACGGAATCCCAAAAR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAGCTGCTTmiR-23aF:ACACTCCAGCTGGGATCACATTGCCAGGGAR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGGAAATCmiR-25F:ACACTCCAGCTGGGATTGCACTTGTCTCGR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTCAGACCGmiR-30a-5pF:ACACTCCAGCTGGGTGTAAACATCCTCGACTR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGCTTCCAGmiR-3958-3pF:ACACTCCAGCTGGGAGATATTGCACGGTTGR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAGAGATCAmiR-432F:ACACTCCAGCTGGGTCTTGGAGTAGGTCATTR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCCACCCAAU6F:CTCGCTTCGGCAGCACAR:AACGCTTCACGAATTTGCGT统一反向引物UnifiedreverseprimerTGGTGTCGTGGAGTCG
F.上游引物;R.下游引物
F.Forward;R.Reverse
2 结 果
2.1Solexa测序结果
经Solexa测序和生物信息学分析,去除接头,去除污染及低质量序列后,PEF和SUF两文库分别得到了13 479 569和11 589 758条清洁序列,结果见表2。
表2小RNA测序质量
Table 2Summary of cleaning of reads produced by sRNA sequencing
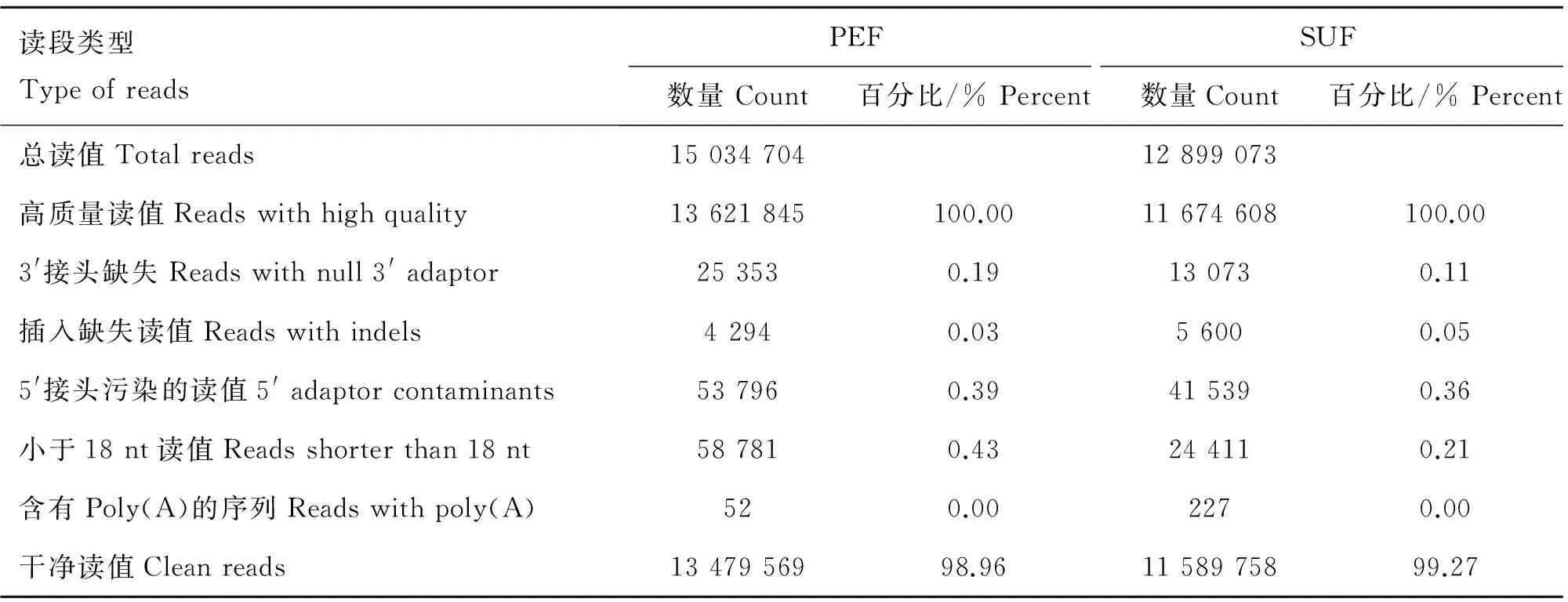
读段类型TypeofreadsPEFSUF数量Count百分比/%Percent数量Count百分比/%Percent总读值Totalreads1503470412899073高质量读值Readswithhighquality13621845100.0011674608100.003'接头缺失Readswithnull3'adaptor253530.19130730.11插入缺失读值Readswithindels42940.0356000.055'接头污染的读值5'adaptorcontaminants537960.39415390.36小于18nt读值Readsshorterthan18nt587810.43244110.21含有Poly(A)的序列Readswithpoly(A)520.002270.00干净读值Cleanreads1347956998.961158975899.27
统计2个sRNA文库中所有sRNA序列的长度分布,大部分序列长度都集中在21~24 nt(图1),其中,长度为22 nt的序列频率最高,在PEF和SUF文库中分别占60.35%和56.74%;其次是23 nt,频率分别为20.67%和21.90%。

图1 皮下和肾周脂肪组织中sRNA的长度分布Fig.1 Length distribution of sRNA in subcutaneous and perirenal adipose tissues
2.2小RNA的分类注释结果
将测序得到的所有序列数据与GenBank、Rfam、intron、exon和重复序列数据库进行比对,将比对到数据库中的sRNA进行注释,没有比对上任何注释信息的用unann表示。随后分别统计PEF和SUF文库中小RNA的种类及数量,结果见图2。可见,PEF和SUF文库都以miRNA的数量最多,所占的比例分别为64.54%和63.24%;无注释信息的sRNA分别占29.58%和30.10%;其他种类的RNAs含量较低。
将2个文库与绵羊基因组进行比对,结果见表3。其中,Unique sRNAs表示clean reads中所有sRNA的种数及比对上基因组的sRNA的种数;Total sRNAs表示clean reads中所有sRNA的总数及比对上基因组的sRNA的总数。可见,肾周和皮下脂肪组织文库中比对上基因组的sRNA种数所占的比例分别为18.13%和14.64%,而比对上基因组的sRNA总数所占的比例分别为73.4%和71.73%。

图2 广灵大尾羊肾周(A)和皮下(B)脂肪组织文库中小RNA的分类注释Fig.2 Annotation of small RNAs in perirenal (A) and subcutaneous (B) adipose tissues of Guangling Large-tailed sheep
表32个文库中小RNA基因组比对分析
Table 3Genomic alignment of sRNAs from 2 libraries

组织Tissue项目ItemsRNA种数UniquesRNAs百分比/%PercentsRNA总数TotalsRNAs百分比/%Percent肾周脂肪PEF小RNA总量436880100.0013479569100.00比对上基因组部分7918618.13989337073.40皮下脂肪SUF小RNA总量538565100.0011589758100.00比对上基因组部分7885014.64831337971.73
2.3保守miRNA鉴定
将有效去除rRNA、tRNA、snoRNA、snRNA和repeat等非编码小RNAs后的剩余序列比对到miRBase21.0中绵羊已知miRNA的成熟序列或前体序列(默认G-U配对,允许1~2个碱基错配),从而得到绵羊基因组中保守的miRNAs,并分析已知miRNAs各位点的碱基偏向性,结果见图3。PEF和 SUF两个sRNA文库中首碱基都偏向U,频率分别为95.42%和95.92%;其次为A,频率分别为3.94%和3.47%;C和G出现的频率较低。其他各位点的碱基分布基本一致,并且2个文库中的第1、9和末端位点处富含U碱基。
统计绵羊保守miRNAs,结果见表4。可见,miRBase21.0中绵羊有153个保守miRNAs成熟序列和105个miRNA前体序列。
广灵大尾羊PEF文库中获得128个保守miRNAs,其中有51个直接匹配上目标序列的miRNAs,33个由前体miRNA的5′端剪切而来的miRNA-5p和44个由前体miRNA的3′端而来的miRNA-3p;SUF文库中获得112个保守miRNAs,其有50个直接匹配上目标序列的miRNAs,28个由前体miRNA的5′端剪切而来的miRNA-5p和34个由前体miRNA 3′剪切而来的miRNA-3p组成。
表42个小RNA文库中的已知miRNA和发夹数
Table 4Number of conserved miRNAs and their hairpins from 2 libraries
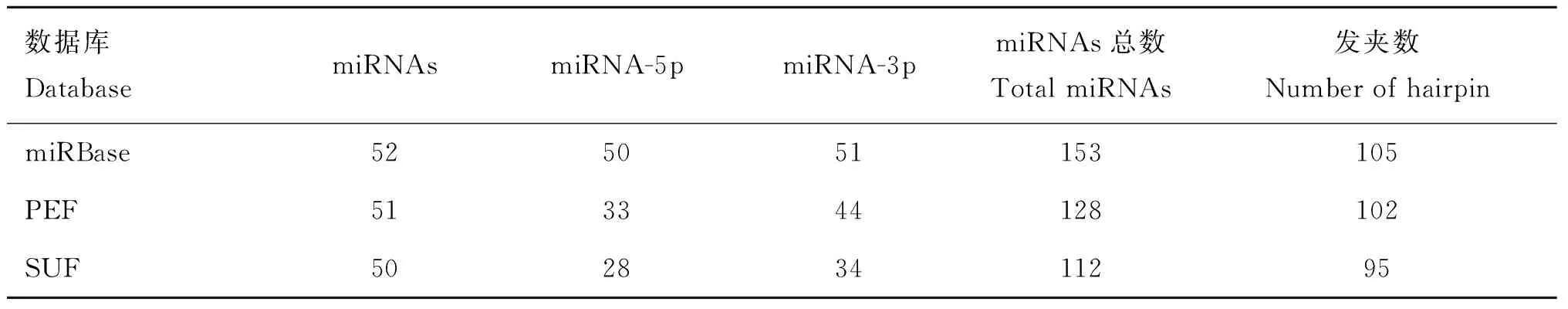
数据库DatabasemiRNAsmiRNA-5pmiRNA-3pmiRNAs总数TotalmiRNAs发夹数NumberofhairpinmiRBase525051153105PEF513344128102SUF50283411295

图3 广灵大尾羊肾周(A)和皮下(B)脂肪组织文库中miRNAs片段各位点碱基的偏向性Fig.3 Nucleotide bias at each position of miRNAs in perirenal (A) and subcutaneous (B) adipose tissues of Guangling Large-tailed sheep
表4中还依次列出了PEF和SUF文库中匹配上已知miRNA发夹结构上的sRNA数量,分别为102和95。进一步分析表明,这2个文库中保守的miRNAs都属于50个miRNA家族,而且这些家族在多个物种中都表现出了保守性。如let-7、miR-8、miR-10、miR-25和 miR-133家族分别在75、68、76、73 和 69个物种中被发现。
对3个sRNA文库中的保守miRNA进行比较发现,在PEF中特异表达的有12个miRNAs,分别是miR-1197-3p、miR-370-3p、miR-376a-3p、miR-377-3p、miR-381-3p、miR-3956-3p、miR-3957-5p、miR-3959-3p、miR-412-5p、miR-431、miR-433-5p、miR-487a-5p;SUF中特异表达的只有1个miRNA(miR-329b-5p),TAF中特异表达的也只有1个miRNA(miR-411b-5p)(图4A)。
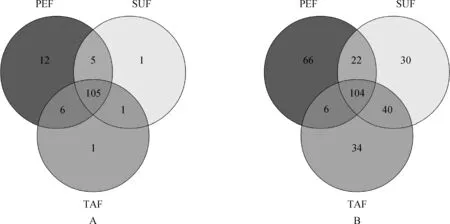
图4 广灵大尾羊肾周(PEF)、皮下(SUF)和尾部脂肪(TAF)中特异表达的保守miRNAs(A)和新miRNAs(B)Fig.4 Conservative (A) and novel (B) miRNAs specifically expressed in perirenal (PEF),subcutaneous (SUF) and tail fat (TAF) of Guangling Large-tailed sheep
2.4预测的绵羊新miRNAs
将与miRBase 21.0中绵羊的保守miRNAs没有比对上,但利用miReap软件筛选出具有发夹结构的序列作为绵羊新miRNA的候选序列。PEF和SUF文库中均有220个候选序列。进一步与miRBase 21.0中所有物种的保守miRNAs比对,2个文库中依次得到198和196个新miRNAs。随后,分析这些新miRNAs在3种不同脂肪组织中的特异性表达情况,结果见图4B。可见,在PEF、SUF和TAF中特异表达的新miRNAs分别有66、30和34个。并且,这些新miRNAs的长度为20~24 nt,最小自由能(MFE)为-75.24~-294.69 kJ·mol-1。
2.5Stem-loop RT-PCR验证结果
随机选择的8个miRNAs在绵羊3种脂肪组织中的相对表达量列于图5A。这些miRNAs在绵羊脂肪组织中都有表达,且除miR-23a外,组织间的差异表达趋势与Solexa测序的结果(图5B)一致。肾周脂肪中,miR-23a在验证时的高表达和测序时的低表达存在着明显的不同。

图5 随机选取的8个miRNAs在广灵大尾羊3种脂肪组织中的定量表达量(A)和测序表达量(B)Fig.5 Expressions by quantification (A) and sequencing (B) of 8 randomly selected miRNAs in 3 adipose tissues of Guangling Large-tailed sheep
3 讨 论
目前,高通量测序技术已广泛应用于家畜小RNA组学研究[16-17]。利用高通量测序,可以发现新的miRNAs,也可以鉴别已知的miRNAs,并研究其功能机制。本研究采用Solexa测序技术,在广灵大尾羊的肾周和皮下脂肪组织中分别发现了128和112个已知的miRNAs以及198和196个新miRNAs。本课题组前期在广灵大尾羊尾脂中发现了113个保守miRNAs和184个新miRNAs[11],这些结果将为深入研究绵羊脂肪组织中miRNA的作用机制提供试验依据。
对高通量测序结果进行定量PCR验证是基因组学研究的重要步骤之一。本研究随机选取在3种脂肪组织中都有表达的8个miRNAs,用Stem-loop qRT-PCR技术检测测序结果的准确性。其中的7个miRNAs的表达趋势与Solexa测序结果一致。这表明测序结果具有一定的准确性和可靠性。但miR-23a验证时在肾周脂肪中高表达,但测序时却发生低表达。这种不一致的情况在其他研究中也有报道。如在猪的背部脂肪组织中,miR-1a实时定量PCR时的表达量与测序中的表达量不一致[18]。这些不一致的结果可能与测定方法有关[18]。
miRNA碱基的分布可能与miRNA的作用机制,如对靶基因的调控有关。本研究中,2个sRNA文库中已知miRNA的首位碱基都偏向U。这与对广灵大尾羊TAF文库的分析结果[11]一致。研究表明,5′端碱基为U有利于RNA诱导沉默复合体中miRNA与Agol蛋白的结合,进而有利于该复合体对靶mRNA的特异性切割[19]。2个文库中的第1、9和末端位点处富含U碱基,这与B.Zhang等[20]报道的结果一致。第2~8位点各碱基分布的趋势是一致的,因为成熟miRNA的第2~8位点被称作种子序列,这一区域具有高度的保守性,miRNA的靶标基因可能会随着这个区域碱基的改变而不同[16]。
在鉴别出的保守miRNAs中,有12个特异表达于肾周脂肪,而只有miR-329b-5p和miR-411b-5p分别特异表达于皮下和尾部脂肪。其中,部分miRNAs的作用已有研究报道。miR-433和miR-431由PEG11as基因编码[21],该基因是一个印记基因,在Callipyge绵羊骨骼肌中以一个全长蛋白的形式表达[22],促进肌肉发育,同时上调miR-433和miR-431的表达,进而抑制脂肪的生成。miR-487b能直接靶向胰岛素受体底物1(Insulin receptor substrate1,IRS1)的3′UTR区,下调IRS1表达[23]。而IRS1能增强胰岛素的功能[24]。由此,miR-487b减弱胰岛素的作用,从而抑制脂肪生成。miR-376a和miR-376c抑制胰岛素样生长因子1受体(Insulin-like growth factor 1 receptor,IGF1R)的表达[25],而IGF1R则促进脂肪组织的生成[26],由此推断,miR-376a/c也可抑制脂肪组织的生成。此外,miR-370直接靶向肉碱棕榈酰转移酶1α(Carnitine palmitoyl transferase 1α,Cpt1α)的3′UTR区,且下调Cpt1α的表达[27]。而Cpt1α促进脂肪酸β氧化[28],所以得出,miR-370能抑制脂肪酸β氧化。
分别在广灵大尾羊皮下和尾部脂肪中特异表达的miR-329b-5p和miR-411b-5p的作用主要与脂肪生成有关。miR-329可以促进过氧化物酶增殖物激活受体γ(Peroxisome proliferator-activated receptor,PPARγ)表达[29],而PPARγ可促进脂肪细胞分化[30],因此,miR-329对脂肪细胞的分化有一定促进作用。过表达miR-411能降低肌球蛋白重链1(MYH1)的表达[31],说明,miR-411与MYH1的编码基因存在靶标关系。而MYH1能提高人类脂肪源间充质干细胞向肌源性细胞分化潜能[32]。所以,推断miR-411可抑制人类脂肪源间充质细胞向肌源性细胞分化。
基于上述研究,推测广灵大尾羊3个部位脂肪组织中特异表达的这些miRNAs在绵羊脂肪调控中发挥着不同作用。由于肾周脂肪组织属于深层脂肪组织,脂肪代谢比较活跃,其特异表达的这些miRNAs的功能多为抑制脂肪生成,少数抑制脂肪酸氧化;而皮下和尾部是绵羊脂肪组织的主要储积场所,这些脂肪组织属浅层脂肪组织,其特异表达的2个miRNAs的功能均为促进脂肪生成。
在发现的新miRNAs中,有66个在肾周脂肪中特异表达,30个在皮下脂肪中特异表达,34个在尾部脂肪中特异表达。这为挖掘与绵羊脂肪代谢的相关miRNAs提供了科学依据。但目前还没有文献报道这些新miRNAs在脂肪代谢中的具体作用,其功能尚需进一步研究。
4 结 论
通过高通量测序和生物信息学分析,发现了一些在肾周、皮下和尾部脂肪中特异表达的保守miRNAs和新miRNAs。这些miRNAs在不同部位脂肪组织中发挥着不同作用,在肾周脂肪组织中特异表达的miRNA的功能多为抑制脂肪生成,而在皮下和尾部脂肪组织中特异表达的2个miRNAs的功能均为促进脂肪生成。
[1]BARTEL D P.MicroRNAs:genomics,biogenesis,mechanism and function [J].Cell,2004,116(2):281-297.
[2]TUFEKCI K U,MEUWISSEN R L,GENEC S.The role of microRNAs in biological processes [J].MethodsMolBiol,2014,110(7):15-31.
[3]BALEY J,LI J.MicroRNAs and ovarian function [J].JOvarianRes,2012,5:8.
[4]SHUKLA G C,SINGH J,BARIK S.MicroRNAs:Processing,maturation,target recognition and regulatory functions [J].MolCellPharmacol,2011,3(3):83-92.
[5]XU P,VERNOOY S Y,GUO M,et al.The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism [J].CurrBiol,2003,13(9):790-795.
[6]NAKANISHI N,NAKAGAWA Y,TOKUSHIGE N,et al.The up-regulation of microRNA-335 is associated with lipid metabolism in liver and white adipose tissue of genetically obese mice [J].BiochemBiophysResCommun,2009,385(4):492-496.
[7]ESAU C,KANG X,PERALTA E,et al.MicroRNA-143 regulates adipocyte differentiation [J].BiolChem,2004,279(50):52361-52365.
[8]LI H,ZHANG Z,ZHOU X,et al.Effects of microRNA-143 in the differentiation and proliferation of bovine intramuscular preadipocytes [J].MolBiolRep,2011,38(7):4273-4280.
[9]VINNIKOV I A,HAJDUKIEWICZ K,REYMANN J,et al.Hypothalamic miR-103 protects from hyperphagic obesity in mice [J].Neurosci,2014,34(32):10659-10674.
[10]RAYNER K J,SUAREZ Y,DAVALOS A,et al.MiR-33 contributes to the regulation of cholesterol homeostasis [J].Science,2010,328(5985):1570-1573.
[11]贾夏丽.绵羊尾脂的全基因组microRNA表达谱分析及其与性状的关联研究[D].太谷:山西农业大学,2014.
JIA X L.Whole genome profiling of microRNAs expressed in tail fat and their associations with traits in sheep [D].Taigu:ShanxiAgriculturalUniversity,2014.(in Chinese)
[12]凌英会,张晓东,王丽娟,等.山羊肌肉组织microRNA Solexa测序与生物信息学分析 [J].畜牧兽医学报,2013,44(3):481-487.
LING Y H,ZHANG X D,WANG L J,et al.Solexa sequencing and bioinformatics analysis on microRNA from the goat muscle [J].ActaVeterinariaetZootechnicaSinica,2013,44(3):481-487.(in Chinese)
[13]HAN H S,JO Y N,LEE J Y,et al.Identification of suitable reference genes for the relative quantification of microRNAs in pleural effusion [J].OncolLett,2014,8(4):1889-1895.
[14]DEO A,CARLSSON J,LINDLOF A.How to choose a normalization strategy for miRNA quantitative real-time (qPCR) arrays [J].BioinformComputBiol,2011,9(6):795-812.
[15]习欠云,周莲莲,李虹仪,等.不同品种猪肌肉组织miR-1和miR-133基因的表达分析 [J].畜牧兽医学报,2012,43(6):843-848.
XI Q Y,ZHOU L L,LI H Y,et al.The expression analysis of miR-1 and miR-133 inlongissimusdorsimuscle from different pig breeds [J].ActaVeterinariaetZootechnicaSinica,2012,43(6):843-848.(in Chinese)
[16]SUN J,LI M,LI Z,et al.Identification and profiling of conserved and novel microRNAs from Chinese Qinchuan bovine longissimus thoracis [J].BMCGenomics,2013,14:42.
[17]LI G,LI Y,LI X,et al.MicroRNA identity and abundance in developing swine adipose tissue as determined by Solexa sequencing [J].CellBiochem, 2011,112(5):1318-1328.
[18]陈晨,柴进,蒋思文.Solexa测序鉴定大白猪和梅山猪背部脂肪组织中microRNA [C].扬州:第十六次全国动物遗传育种学术讨论会,2011:35-40.
CHEN C,CAI J,JIANG S W.Identification of microRNA in back fat tissue of Large white and Meishan swine [C].Yangzhou:The 16thnational conference on animal genetics and breeding,2011:35-40.(in Chinese)
[19]MI S,CAI T,HU Y,et al.Sorting of small RNAs into Arabidopsis argonaute complexes is directed by the 5' terminal nucleotide [J].Cell,2008,133(1):116-127.
[20]ZHANG B,STELLWAG E J,PAN X.Large-scale genome analysis reveals unique features of microRNAs [J].Gene,2009,443(1-2):100-109.
[21]DAVIS E,CAIMENT F,TORDOIR X,et al.RNAi-mediated allelic trans-interaction at the imprinted Rtl1/Peg11 locus [J].CurrBiol,2005,15(8):743-749.
[22]BYRNE K,COLGRAVE M L,VUOCOLO T,et al.The imprinted retrotransposon-like gene PEG11 (RTL1) is expressed as a full-length protein in skeletal muscle from Callipyge sheep [J].PLoSOne,2010,5(1):e8638.
[23]NOSSENT A Y,ESKILDSEN T V,ANDERSEN L B,et al.The 14q32 microRNA-487b targets the anti-apoptotic insulin receptor substrate 1 in hypertension-induced remodeling of the aorta [J].AnnSurg,2013,258(5):743-751.
[24]LIU Y,LIU C,LU M L,et al.Vibration exercise decreases insulin resistance and modulates the insulin signaling pathway in a type 2 diabetic rat model [J].IntJClinExpMed,2015,8(8):13136-13144.
[25]ZEHAVI L,AVRAHAM R,BARZILAI A,et al.Silencing of a large microRNA cluster on human chromosome 14q32 in melanoma:biological effects of mir-376a and mir-376c on insulin growth factor 1 receptor [J].MolCance,2012,11:44.
[26]HOLZENBERGER M,HAMARD G,ZAOUI R,et al.Experimental IGF-I receptor deficiency generates a sexually dimorphic pattern of organ-specific growth deficits in mice,affecting fat tissue in particular [J].Endocrinology,2001,142(10):4469-4478.
[27]ILIOPOULOS D,DROSATOS K,HIYAMA Y,et al.MicroRNA-370 controls the expression of microRNA-122 and Cpt1alpha and affects lipid metabolism [J].LipidRes,2010,51(6):1513-1523.
[28]MOORE K J,RAYNER K J,SUAREZ Y,et al.microRNAs and cholesterol metabolism [J].TrendsEndocrinolMetab,2010,21(12):699-706.
[29]DHARAP A,POKRZYWA C,MURALI S,et al.Mutual induction of transcription factor PPARγ and microRNAs miR-145 and miR-329 [J].Neurochem,2015,35(1):139-146.
[30]HE Y F,LIU F Y,ZHANG W X.Tangeritin inhibits adipogenesis by down-regulating C/EBPα,C/EBPβ,and PPARγ expression in 3T3-L1 fat cells [J].GenetMolRes,2015,14(4):13642-13648.
[31]HARAFUJI N,SCHNEIDERAT P,WALTER M C,et al.MiR-411 is up-regulated in FSHD myoblasts and suppresses myogenic factors [J].OrphanetJRareDis,2013,8(55):1172-1186.
[32]STERN-STRAETER J,BONATERRA G A,JURITZ S,et al.Evaluation of the effects of different culture media on the myogenic differentiation potential of adipose tissue- or bone marrow-derived human mesenchymal stem cells [J].IntJMolMed,2014,33(1):160-170.
(编辑郭云雁)
High-throughput Sequencing and Bioinformatics Analysis on microRNAs Expressed in Adipose Tissues of Sheep
ZHANG Fang#,HU Zi-qiao#,JING Jiong-jie,PAN Yang-yang,QIAO Li-ying,LI Bao-jun,LIU Jian-hua,LIU Wen-zhong*
(CollegeofAnimalScienceandVeterinaryMedicine,ShanxiAgriculturalUniversity,Taigu030801,China)
This study aimed to find tissue-specifically expressed miRNAs in ovine adipose tissues.Three small RNA (sRNA) libraries from perirenal,subcutaneous and tail adipose tissues were built for Solexa sequencing.The sequencing results were validated by Stem-loop qRT-PCR technology and further analyzed by bioinformatics methods.The PCR results shown that seven in eight randomly selected miRNAs were consistent with the sequencing results.In perirenal and subcutaneous adipose tissues,128 and 112 conserved miRNAs were obtained,198 and 196 novel miRNAs were found,respectively.Combined with the previous analysis results of miRNAs detected in tail adipose tissue,12 conserved and 66 novel miRNAs were specifically expressed in perirenal adipose tissue;one conserved and 30 novel miRNAs were specifically expressed in subcutaneous adipose tissue;and one conserved and 34 novel miRNAs were specifically expressed in tail adipose tissue.These results provide scientific bases for further research on the regulatory role of miRNA in fat metabolism.
sheep;adipose tissues;microRNA;Solexa sequencing;bioinformatics methods
10.11843/j.issn.0366-6964.2016.06.003
2015-12-23
国家自然科学基金(31372292);山西省研究生优秀创新项目(2015SY28)
张方(1989-),女,河南商丘人,硕士生,主要从事肉用绵羊的分子遗传育种研究,E-mail:zhangfycyz@163.com;胡子乔(1991-),女,黑龙江伊春人,硕士生,主要从事肉用绵羊分子遗传育种研究,E-mail:ziqiaohu@163.con。二者并列为第一作者
刘文忠,教授,E-mail:tglwzyc@163.com
S813.3;S826
A
0366-6964(2016)06-1093-09
