近红外高光谱成像技术用于转基因大豆快速无损鉴别研究
2016-07-12王海龙杨向东郭东全鲍一丹
王海龙,杨向东,张 初,郭东全,鲍一丹*,何 勇, 刘 飞
1. 浙江大学生物系统工程与食品科学学院,浙江 杭州 310058 2. 吉林省农业科学院农业生物技术研究所,吉林 长春 130033
近红外高光谱成像技术用于转基因大豆快速无损鉴别研究
王海龙1,杨向东2,张 初1,郭东全2,鲍一丹1*,何 勇1, 刘 飞1
1. 浙江大学生物系统工程与食品科学学院,浙江 杭州 310058 2. 吉林省农业科学院农业生物技术研究所,吉林 长春 130033
以近红外高光谱成像技术,结合化学计量学方法,研究了转基因大豆的快速、无损检测方法。实验以3种不同非转基因亲本(HC6, JACK, TL1)及其转基因大豆作为研究对象。采用高光谱成像系统采集874~1 734 nm波长范围的256个波段范围的高光谱图像,提取大豆的光谱信息,剔除明显噪声部分后,采用Moving Average(MA)平滑预处理的941~1 646 nm范围光谱数据进行分析。采用偏最小二乘判别分析算法(partial least squares-discriminant analysis, PLS-DA),对3种非转基因亲本大豆建立模型进行判别分析,其相应的建模集和预测集的判别正确率分别为97.50%和100%,100%和100%,96.25%和92.50%,结果表明,高光谱成像技术可用于非转基因大豆的识别。对非转基因亲本及其转基因大豆进行判别分析,基于全谱,3种的建模集和预测集的判别正确率分别为99.17%和99.17%,87.19%和81.25%,99.17%和98.33%; 以x-loading weights提取非转基因亲本及其转基因大豆判别分析的特征波长并建立PLS-DA模型,3种的建模集和预测集的判别正确率分别为72.50%和80%,80.63%和79.38%,85%和85%,该结果表明非转基因亲本与转基因品种的判别分析是可行的,特征波长的选择也可用于非转基因亲本与转基因品种的判别分析。研究表明采用近红外高光谱成像技术对非转基因大豆、非转基因亲本及其转基因大豆进行鉴别是可行的,为转基因大豆的快速无损准确鉴别提供了一种新方法。
近红外高光谱成像; 转基因大豆; PLS-DA;x-loading weights
引 言
由于粮食需求的增加,转基因技术在作物种植中的应用更加快速增长[1]。从1996年开始商业化后,转基因作物的种植面积一直持续增长。1.752亿公顷,是2013年转基因作物在全球范围的种植面积,相比前一年的统计增长了3%,并且是最初商业化时种植面积(170万公顷)的100多倍[2]。大豆是最主要的转基因作物来源,大豆、玉米、棉花和油菜分别占全球转基因作物面积的47%,32%,15%和5%[3]。转基因作物的产量一般都远高于非转基因作物,大大增加了全球粮食产量,有利于减轻贫困和饥饿[2]。
转基因在世界范围内存在很大的争议。中国是世界上最大的大豆进口国,尽管对转基因作物的监管十分严格,常有转基因大豆通过非法途径进入中国销售。基于转基因大豆及其他转基因作物存在的安全隐患和争议等现实问题,研究转基因与非转基因的检测和鉴别方法具有重要意义。
传统的转基因与非转基因的检测和鉴别方法,主要有蛋白质检测方法包括酶联免疫分析法(enzyme linked immunoassay,ELISA)、侧向流动型免疫试纸条法(lateral flow strip)、核酸检测方法[定性PCR法(qualitative PCR)、实时荧光PCR法(real-time fluorescent PCR)]、基因芯片检测技术和高效液相色谱法等[4-5]。这些方法会一定程度的破坏蛋白质或基因片段,且费时费力,程序复杂,成本较高,非专业人员难以胜任,不适用于转基因与非转基因的实时在线快速检测鉴别。
光谱与光谱成像技术具有快速、无损、准确等优良特点,近年来在农作物鉴别及品质分析的检测中得到广泛应用[6-8]。目前,国内外已对近红外光谱技术在转基因农作物的应用进行了研究[9-14],但近红外光谱技术只能获取检测对象部分区域的光谱信息,缺少对检测对象的空间信息的研究,预测集的信息相比建模集可能会存在较大差异。高光谱成像技术融合了图像与光谱信息,能够将研究对象的光谱信息与空间信息同时采集,对样本的内外部信息可以更大范围的获取[15-16]。因此,在鉴别农产品品种和无损检测其品质的研究中高光谱成像技术的应用越来越多[15, 17-19],但国内尚无采用近红外高光谱成像技术对非转基因亲本及其转基因大豆的品种鉴别的研究。
本实验的主要目的是研究基于高光谱成像技术的转基因大豆的快速、无损检测与鉴别的方法。具体的目的为: (1)非转基因大豆(亲本)的品种鉴别研究; (2)非转基因亲本与其转基因品种的品种鉴别研究。
1 实验部分
1.1 样本
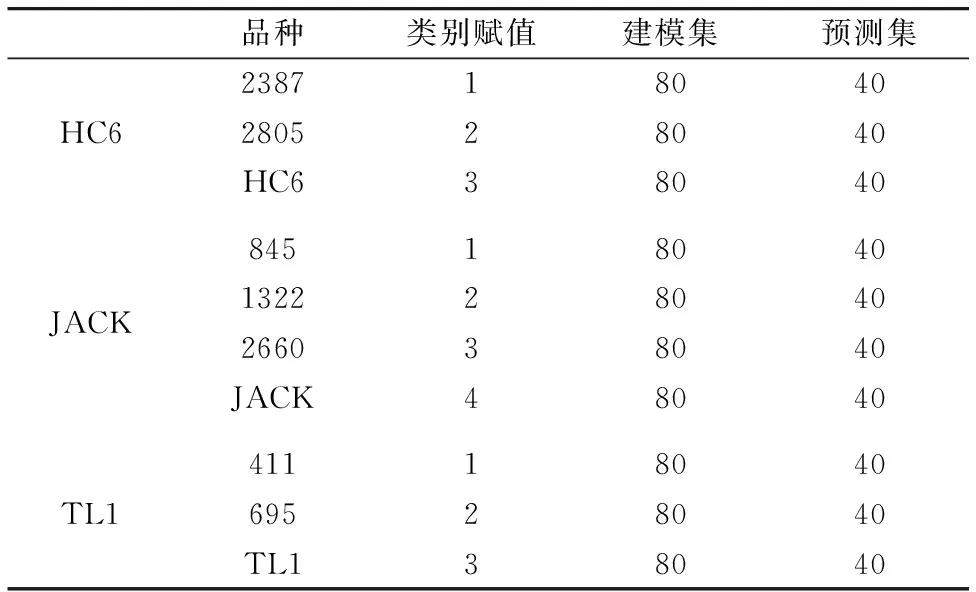
试验用的非转基因亲本大豆及其转基因大豆均由吉林省农业科学院提供,有HC6,JACK和TL1等3种不同非转基因亲本大豆及其转基因大豆共10个品种,在外观上均无明显差异。其中HC6大类下,3个品种包括有HC6(非转基因亲本)、2805(转基因)和2387(转基因); JACK大类下,4个品种包括有JACK(非转基因亲本)、1322(转基因)、845(转基因)和2660(转基因); TL1大类下,3个品种包括有TL1(非转基因亲本)、411(转基因)和695(转基因)。试验中选用的转基因大豆与对照非转基因大豆相比仅在目标性状(抗病、抗虫、高油酸)方面有显著差异,在其他表型性状方面则差异不显著。
1.2 高光谱成像系统
实验采用高光谱成像系统实现对转基因大豆及其亲本的光谱图像采集。
系统主要包括: 成像光谱仪(N17E-QE, Spectral Imaging Ltd., Oulu, Finland),其光谱范围为874~1 734 nm,在光谱范围内共有256个波段,配有镜头(OLES22, Specim, Spectral Imaging Ltd., Oulu, Finland)。系统配有两个150 W卤钨灯的Fiber-Lite DC950线光源(Dolan Jenner Industries Inc., USA),可驱动载有样本的传送带的IRCP0076型电控移位平台(Isuzu Optics Corp, 中国台湾),用来控制系统运行的计算机以及中国台湾五铃光学公司提供的高光谱成像系统采集软件。系统的光谱分辨率为5 nm,图像分辨率为320×256像素点。
1.3 高光谱图像采集及校正
采集高光谱图像之前,先对系统进行校正,主要通过调节光强、图像清晰度和图像的失真来实现,而平台移动速度、相机曝光时间和物距等参数是影响图像清晰度和是否失真的主要因素。由于这些参数之间会彼此影响,为了使采集到的图像达到不变形、不失真、更清晰等目的,需进行最优参数设置。多次尝试后,分别设置参数为: 样品到镜头边缘的距离为18 cm,曝光时间为4 ms,平台移动速度为18 mm·sec-1。
对采集到的光谱图像处理之前进行校正,图像校正公式如式(1)
(1)
式(1)中,R,Iraw,Iwhite和Idark分别为经校正后的图像、原始采集的图像、白板图像、黑板图像。从采集的大豆样本高光谱图像中选取单粒大豆为感兴区域,感兴区域内每一点都有一条光谱,计算感兴区域内的所有像素点光谱的平均值则是该样本的光谱,从而进行分析。
1.4 数据处理
1.4.1 判别分析方法
采用偏最小二乘判别分析(partial least squares-discriminant analysis, PLS-DA)进行判别分析。在光谱数据分析中,PLS算法[20]是用较多的一种回归分析算法,新的变量组合(LVs)可以由光谱数据通过线性变换得到,一般前几个LVs包含绝大多数信息,用于预测分析。PLS-DA以代表类别的整数值代替化学值进行回归分析,根据得到的预测结果进行判别分析。为了对样本的类别进行判定,也因为预测结果中预测值不是代表类别的整数而是实际数值,故需设定判别阈值。在本研究中,设定判别阈值为0.5,即当实际值与预测值的差的绝对值大于0.5时,则判别错误,反之则视为判别正确。
1.4.2x-loading weights选择特征波长
通过特定方法挑选出的特征波长或波长区间,用少数带有最多有用信息的波长代替全谱,将不相关或者非线性变量剔除,模型的计算量和复杂度都得到降低,从而使模型具有预测能力强、稳健性好的特点,同时可以为研发基于特征波长的仪器提供支持。
应用x-loading weights选择特征波长。将PLS-DA用于建模分析,各波长对应的载荷系数(loading weights,LW)均可以从每个隐含变量(latent variable,LV)中得到,而该波长对所建模型预测性能的影响则可以通过载荷系数绝对值的大小来说明,因此特征波长的选取可以将某一隐含变量下各波长对应的载荷系数绝对值的大小作为依据[21]。
高光谱图像分析采用分析软件ENVI 4.7(ITT, Visual Information Solutions),采用Matlab R2012b(The Math Works, Natick, USA)及The Unscrambler X 10.1(CAMO AS, Oslo, Norway)做数据分析。
2 结果与讨论
2.1 大豆的光谱特征曲线
实验中采集的高光谱数据,波长范围是874~1 734 nm,共有256个波段。由于光谱数据前端和后端在采集时均明显受到噪声的影响,研究时应去掉前端和后端中有明显噪声的部分,故采用了波段21到波段230共210个波段,即波长范围在941~1 646 nm之间的光谱来分析。光谱预处理时用了7点移动平均平滑法[22](moving average,MA),图1为其平均光谱图。由图1可知,大豆光谱曲线基本相似,无法将转基因与非转基因大豆从原始光谱直接区分开来。

图1 MA预处理后大豆平均光谱图
2.2 非转基因亲本大豆的判别分析
将所有非转基因亲本大豆进行类别赋值并通过Kennard-Stone算法[23]按照2∶1的比例将各个样本划分为建模集和预测集,分别包含80个与40个样本。将亲本HC6类别赋值为1,亲本JACK赋值为2,亲本TL1赋值为3。以该样本划分的数据为输入,表1为基于PLS-DA模型的判别分析结果。

表1 非转基因亲本大豆的PLS-DA判别分析结果
由表1可知,对亲本大豆HC6,JACK以及TL1的判别正确率均较高,建模集和预测集判别正确率均达到了90%以上,且JACK亲本大豆建模集与预测集判别正确率均为100%,可能是因为JACK非转基因亲本大豆与其他两类亲本大豆之间光谱特性具有较大的差异。结果表明本实验用不同非转基因亲本大豆之间能相互鉴别,即高光谱成像技术用于非转基因大豆品种鉴别是可行的。
2.3 非转基因亲本大豆及其转基因大豆的品种鉴别研究
对非转基因亲本大豆及其转基因大豆品种进行鉴别研究,不同的非转基因亲本大豆及其转基因大豆品种的赋值及样本划分如表2所示。
2.3.1 基于全波段光谱的PLS-DA判别模型
以经MA预处理后得到的全谱光谱数据作为输入,建立PLS-DA的判别分析模型,其判别结果如图2及表3所示。
由表3可知,HC6亲本及其转基因品种整体的建模集和预测集的判别效果非常好,建模集240个样本,判断对238个,判别正确率达到了99.17%,而预测集120个样本,判断对119个,判别正确率也达到了99.17%。JACK亲本及其转基因品种整体的建模集和预测集的判别正确率略低,分别为87.19%与81.25%。原因可能是JACK非转基因亲本及其转基因品种之间差异较小。TL1亲本及其转基因品种整体的判别效果较好,建模集和预测集的判别正确率分别为99.17%以及98.33%。
表2 不同非转基因亲本及其转基因大豆品种赋值与建模集和预测集样本划分
Table 2 Class value assignment and dataset split of different non-GMO parent and transgenic soybeans
品种类别赋值建模集预测集HC6238718040280528040HC638040JACK84518040132228040266038040JACK48040TL14111804069528040TL138040

图2 基于全谱的不同非转基因亲本大豆及其转基因大豆品种判别结果
Fig.2 Specific discriminant results of different non-GMO parent and transgenic soybeans on full spectra
表3 基于全谱的不同的非转基因亲本大豆及其转基因大豆品种判别结果
Table 3 Total discriminant results of different non-GMO parent and transgenic soybeans based on full spectra

建模集预测集识别数判别正确率/%识别数判别正确率/%HC623899.1711999.17JACK27987.1913081.25TL123899.1711898.33
由图2可知各品种的判别正确率均较高,而JACK亲本的判别正确率较低,预测集判别正确率低于60%,可能是JACK非转基因亲本及其转基因品种之间差异较小,共有特性较多所导致。从图3也可以看出对亲本的判别正确率并未高于对应的转基因品种的判别正确率,原因可能是亲本与其不同的转基因品种之间各自共有的特性较多,导致无法正确判别。
综上所述,基于全谱的HC6,JACK和TL1亲本及其转基因大豆的判别识别率都是比较高的,说明了以全谱光谱数据建立PLS-DA判别模型用于转基因大豆与非转基因大豆的品种识别是可行的。
2.3.2 基于x-loading weights的特征波长选择
以全谱数据作为输入而建立的PLS-DA模型,取得了比较好的结果。但大量的数据,会增加模型的复杂度,降低计算速度。同时,在全谱数据信息中,由于大量的冗余和共线性数据的存在,会影响模型的效果。
研究中基于x-loading weights进行特征波长的选择,提取光谱中有效特征信息建立品种识别模型。因此,品种HC6选出的特征波长数为9(961,1 002,1 119,1 204,1 311,1 402,1 446,1 598,1 622 nm),品种JACK选出的特征波长数为11(999,1 113,1 156,1 197,1 234,1 291,1 342,1 399,1 446,1 480,1 554 nm),品种TL1选出的特征波长数为13(965,999,1 019,1 096,1 126,1 177,1 217,1 254,1 328,1 386,1 450,1 480,1 619 nm)。对不同样本数据选择的特征波长相近,并且选出来的部分特征波长完全相同。

图3 基于特征波长不同的非转基因亲本大豆及其转基因大豆品种判别结果
Fig.3 Specific discriminant results of different non-GMO parent and transgenic soybeans on sensitive wavelengths
2.3.3 基于特征波长的PLS-DA判别模型
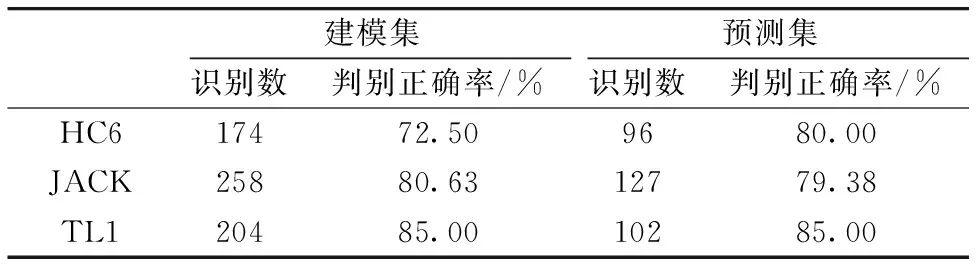
为了验证选出的特征波长对品种鉴别的效果,以选出的特征波长的光谱数据作为输入,建立PLS-DA判别模型,其判别分析结果如图3及表4。
表4 基于特征波长不同的非转基因亲本大豆及其转基因大豆品种总体判别结果
Table 4 Total discriminant results of different non-GMO parent and transgenic soybeans based on sensitive wavelengths
建模集预测集识别数判别正确率/%识别数判别正确率/%HC617472.509680.00JACK25880.6312779.38TL120485.0010285.00
由表4可知,基于特征波长建立的PLS-DA模型中,HC6亲本及其转基因品种整体的建模集和预测集的判别正确率分别达到了72.50%和80%,JACK亲本及其转基因品种整体的建模集和预测集的判别正确率分别达到了80.63%和79.38%,TL1亲本及其转基因品种整体的建模集和预测集的判别正确率分别达到了85%和85%。由图3可知,整体而言,尽管整体判别正确率低于基于全谱的模型,各非转基因亲本及其转基因品种的均取得了较好的判别正确率,但需要进一步的提高。与图2基于全谱的模型效果相似,亲本大豆的判别正确率略低,可能是因为亲本与其转基因品种之间的共有特性较多。
3 结 论
基于近红外高光谱成像技术,结合判别分析算法,对非转基因大豆与转基因大豆进行快速、无损检测研究。分别研究了非转基因亲本大豆品种鉴别,转基因大豆及其亲本大豆的品种鉴别等。通过研究其光谱数据之间的差异,建立了鉴别分析模型,其中对非转基因亲本品种鉴别以及非转基因亲本及其转基因品种的鉴别,取得了较好的判别正确率。同时通过提取特征波长进行重新建模,进一步对转基因大豆与非转基因大豆进行品种分类研究,也取得了比较好的效果。
本研究为转基因与非转基因大豆的快速、无损检测,提供了理论依据和一个新的方法,为进一步开发转基因大豆的便携式快速识别检测仪器与系统提供了方法依据。
[1] Alishahi A, Farahmand H, Prieto N, et al. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2010, 75(1): 1.
[2] James C. Chinese Journal of Biomedical Engineering(中国生物工程杂志), 2014, 34(1): 1.
[3] James C. Chinese Journal of Biomedical Engineering(中国生物工程杂志), 2012, 32(1): 1.
[4] WANG Guang-yin, FAN Wen-xiu, CHEN Bi-hua, et al(王广印, 范文秀, 陈碧华, 等). Food Science(食品科学), 2008, 29(10): 698.
[5] HE Yan, ZHENG Wen-jie, LIU Yuan, et al(贺 艳, 郑文杰, 刘 垣, 等). Food Research and Development(食品研究与开发), 2009, 30(3): 170.
[6] Font R, del Río-Celestino M, de Haro-Bailón A. Industrial Crops and Products, 2006, 24(3): 307.
[7] Zhang X L, Liu F, He Y, et al. Sensors, 2012, 12(12): 17234.
[8] ZHANG Chu, LIU Fei, KONG Wen-wen, et al(张 初, 刘 飞, 孔汶汶, 等). Transactions of the Chinese Society of Agricultural Engineering(农业工程学报), 2013, 29(20): 270.
[9] Roussel S A, Hardy C L, Hurburgh C R, et al. Applied Spectroscopy, 2001, 55(10): 1425.
[10] Yamada T, Yeh T F, Chang H M, et al. Holzforschung, 2006, 60(1): 24.
[11] XIE Li-juan, YING Yi-bin(谢丽娟, 应义斌). Journal of Jiangsu University(江苏大学学报·自然科学版), 2012, 33(5): 538.
[12] Hurgurgh C R, Heithoff C, Rippke G R. US Patent, 071993, 2000.
[13] Munck L, MΦller B, Jacobsen S. Journal of Cereal Science, 2004, 40: 213.
[14] Luna A S, da Silva A P, Pinho J S A, et al. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2013, 100: 115.
[15] Kamruzzaman M, ElMasry G, Sun D W, et al. Innovative Food Science & Emerging Technologies, 2012, 16: 218.
[16] Huang M, Wan X, Zhang M, et al. Journal of Food Engineering, 2013, 116(1): 45.
[17] Kong W W, Zhang C, Liu F, et al. Sensors, 2013, 13(7): 8916.
[18] ZHANG Chu, LIU Fei, ZHANG Hai-liang, et al(张 初, 刘 飞, 章海亮, 等). Spectroscopy and Spectral Analysis(光谱学与光谱分析), 2014, 34(3): 746.
[19] Mahesh S, Manickavasagan A, Jayas D S, et al. Biosystems Engineering, 2008, 101(1): 50.
[20] Guo W L, Du Y P, Zhou Y C, et al. World Journal of Microbiology and Biotechnology, 2012, 28(3): 993.
[21] Liu F, He Y, Wang L. Analytica Chimica Acta, 2008, 615(1): 10.
[22] CHU Xiao-li, YUAN Hong-fu, LU Wan-zhen(褚小立, 袁洪福, 陆婉珍). Progress in Chemistry(化学进展), 2004, 16(4): 528.
[23] TANG Yu-lian, LIANG Yi, ZENG Fan-wei, et al(唐玉莲, 梁 逸, 曾范伟, 等). Infrared(红外), 2010,(1): 30.
*Corresponding author
(Received Mar. 28, 2015; accepted Jul. 19, 2015)
Fast Identification of Transgenic Soybean Varieties Based Near Infrared Hyperspectral Imaging Technology
WANG Hai-long1, YANG Xiang-dong2, ZHANG Chu1, GUO Dong-quan2, BAO Yi-dan1*, HE Yong1, LIU Fei1
1. College of Biosystems Engineering and Food Science, Zhejiang University, Hangzhou 310058, China 2. Agriculture Biotechnology Research Center, Jilin Academy of Agricultural Sciences, Changchun 130033, China
Near-infrared hyperspectral imaging technology combined with chemometrics was applied for rapid and non-invasive transgenic soybeans variety identification. Three different non-GMO parent soybeans(HC6, JACK, TL1)and their transgenic soybeans were chosen as the research object. The developed hyperspectral imaging system was used to acquire the hyperspectral images in the spectral range of 874~1 734 nm with 256 bands of soybeans, and the reflectance spectra were extracted from the region of interest (ROI) in the images. After eliminating the obvious noises, the moving average(MA)was applied as smooth pretreatment, and the wavelengths from 941~1 646 nm were used for later analysis. Partial least squares-discriminant analysis (PLS-DA)was employed as pattern recognition method to class the three different non-GMO parent soybeans. The classification accuracy of both the calibration set and the prediction set were 97.50% and 100% for the HC6, 100% and 100% for the JACK, 96.25% and 92.50% for the TL1, which indicated that hyperspectral imaging technology could identify the varieties of the non-GMO parent soybeans. Then PLS-DA was applied to classify non-GMO parent soybean and its transgenic soybean cultivars for building discriminant models. For the full spectra, the classification accuracy of both the calibration set and the prediction set were 99.17% and 99.17% for the HC6 and its transgenic soybean cultivars, 87.19% and 81.25% for the JACK and its transgenic soybean cultivars, 99.17% and 98.33% for the TL1 and its transgenic soybean cultivars, respectively. The sensitive wavelengths were selected byx-loading weights, and the classification accuracy of the calibration set and prediction set of PLS-DA models based on sensitive wavelengths were 72.50% and 80% for the HC6 and its transgenic soybean cultivars, 80.63% and 79.38% for the JACK and its transgenic soybean cultivars, 85% and 85% for the TL1 and its transgenic soybean cultivars, respectively. These results showed that the pattern recognition for non-GMO parent soybean and their transgenic soybeans was feasible, and the selected sensitive wavelengths could be used for the pattern recognition of non-GMO parent soybeans and transgenic soybeans. The overall results indicated that it was feasible to use near-infrared hyperspectral imaging technology for the pattern recognition of the non-GMO parent soybeans varieties, non-GMO parent soybean and its transgenic soybeans. This study also provided a new alternative for rapid and non-destructive accurate identification of transgenic soybean.
Near-infrared hyperspectral imaging; Transgenic soybean; PLS-DA;x-loading weights
2015-03-28,
2015-07-19
国家自然科学基金项目(31471417),国家(863)计划项目(2012AA101903),国家转基因生物新品种培育重大专项(2014ZX08004-004)和吉林省科技发展计划项目(20150204011NY)资助
王海龙,1989年生,浙江大学生物系统工程与食品科学学院硕士研究生 e-mail: hl_wang@zju.edu.cn *通讯联系人 e-mail: ydbao@zju.edu.cn
O433.4; S529
A
10.3964/j.issn.1000-0593(2016)06-1843-05
猜你喜欢
杂志排行
光谱学与光谱分析的其它文章
- 基于光声光谱联合主成分回归法的血糖浓度无损检测研究
- HP-β-CD降低油田地层水中SDBS胶束对其检测光谱的干扰
- Sensitivity Enhancement in Uranium Determination by UV-Visible Spectroscopy Using Ion Imprinted Polymer
- 采用小波分析方法降低可调谐半导体激光吸收光谱技术测量下限的实验研究
- 钠钾替代条件下不同基因型棉花叶片的FTIR光谱研究
- Structural, Morphological and Optical Properties of Well-Ordered CdO Nanostructures Synthesized by Easy-Economical Chemical Bath Deposition Technique
