海带磷酸甘露糖变位酶(PMM)基因的克隆与表达分析
2016-07-11张朋艳姚建亭段德麟
张朋艳,于 雪,姚建亭,段德麟
海带磷酸甘露糖变位酶(PMM)基因的克隆与表达分析
张朋艳1,2,3,于雪1,2,3,姚建亭1,2,段德麟1,2
(1.中国科学院海洋研究所实验海洋生物学重点实验室,山东 青岛 266071; 2.青岛海洋科学与技术国家实验室实验海洋生物学与生物技术实验室,山东 青岛 266071; 3.中国科学院大学,北京 100049)
摘要:磷酸甘露糖变位酶(PMM)是褐藻胶和岩藻聚糖合成过程中的关键酶之一。本研究利用cDNA末端快速克隆(RACE)技术,获得2条海带PMM基因(Sjpmm1,Sjpmm2)序列。其中,Sjpmm1的开放阅读框(ORF)长 759 bp,其编码的SjPMM1为卤酸脱卤酶(HAD)超家族成员,含 252个氨基酸,分子量约为28.51 kDa; 而Sjpmm2的 ORF长1866 bp,其编码的SjPMM2属于磷酸己糖变位酶超家族的成员,含621个氨基酸,分子量约为66.49 kDa。海带PMM的二级结构均以α-螺旋为主。进化分析表明,Sjpmm1来自于原始真核生物,而 Sjpmm2来源于质体的第一次内共生作用。实时定量PCR分析发现,海带受到高温或低温胁迫时,Sjpmm1和 Sjpmm2转录水平上升,以合成岩藻聚糖抵抗环境影响。此外,利用pMAL-c5X载体对SjPMM1进行体外表达,得到高浓度的可溶性融合蛋白,为后续的SjPMM功能分析提供基础。
关键词:海带; 磷酸甘露糖变位酶; 岩藻聚糖; 褐藻胶; 实时定量PCR分析
[Foundation: National Key Technology Research and Development Program(2013BAB01B01); Ocean Public Welfare Scientific Research Project(201405040)]
海带(Saccharina japonica)是一种冷温水性潮下带褐藻,原产于日本海沿岸,自 1927年引入中国,通过不断地驯化与适应,逐渐适应了中国的浅海环境,目前,已成为中国海藻养殖最主要品种[1]。中国海带年产量约为490万吨(鲜质量),约占世界总产量86%(FAO,2012)。海带含有褐藻胶、岩藻聚糖等代谢产物,可广泛应用于食品、医药、化工等领域[2]。目前,有关海带褐藻胶、岩藻聚糖的代谢机制尚未得到全面了解,因此,解析其褐藻胶、岩藻聚糖合成途径中关键酶具有重要意义。
磷酸甘露糖变位酶(phosphomannomutase,PMM)是海带碳代谢过程中的一种关键酶,在Mg2+及甘露糖-1,6-二磷酸作用下,可催化甘露糖-6-磷酸向甘露糖-1-磷酸方向转化。其催化产物可被进一步催化生成 GDP-甘露糖,参与褐藻胶和岩藻聚糖的合成,维持细胞结构的完整性并参与细胞对外界环境的应答[3]。在细菌及各类真核生物中,PMM还参与细胞壁多糖[4]、抗坏血酸[5]以及糖基化蛋白和脂质的寡糖中心的合成[6]。
PMM 基因在某些真核生物中对温度诱导的适应性调节起重要作用[7]。酵母PMM突变株能够增强酵母对温度的敏感性,使其无法在 37℃条件下生存[8]。Hoeberichts等[9]也发现拟南芥中PMM功能的缺失突变株能够增强其在高温下对氧化胁迫的敏感性,并提高死亡率。虽然藻类领域对PMM基因的研究有报道[3,10],但对PMM基因数目、来源及其对环境的响应与适应依然缺乏全面的了解。而人类、细菌等的PMM晶体结构的解析为分析海带中PMM酶的结构提供了依据[11-12]。
本研究针对海带中褐藻胶和岩藻聚糖合成过程中的关键酶之一 ——PMM进行了克隆与分析,通过对序列的结构、进化分析,以及温度诱导下转录水平的差异分析验证该基因的特征与功能,为解析海带褐藻胶、岩藻聚糖的合成途径提供理论依据。
1 材料与方法
1.1实验材料
海带幼孢子体(中科2号,长15~20 cm)采自于山东荣成高绿水产养殖公司的海带养殖筏架。将采集的海带用无菌海水冲洗3遍,然后于培养箱黑暗条件下培养过夜,培养温度为10℃。次日将其置于光照为50 μmol/(m2·s)条件下培养6 h后,再分别置于不同温度(5、10、15、20、25、30℃)下培养2 h,分别取出3棵海带,液氮冷冻保存,用于后续的RNA制备。
RNA提取试剂盒(RNeasy Plant Mini Kit )购自Qiagen公司; cDNA合成试剂盒(PrimeScript™ II 1st Strand cDNA Synthesis Kit)、3′- cDNA末端快速克隆(3′-RACE)试剂盒(3′-Full RACE Core Set Ver.2.0)、pMD19-T Simple Vector 购自 TaKaRa公司; 5′-cDNA末端快速克隆(5′-RACE)试剂盒(SMARTer RACE cDNA Amplification Kit)购自Clontech公司;pMAL-c5X 质粒、克隆感受态细胞(DH5α)、NEB 表达用大肠杆菌感受态细胞均由实验室保存。
1.2实验方法
1.2.1cDNA的合成
按照Qiagen试剂说明提取海带总RNA,通过分光光度计检测其质量,选取高纯度的 RNA(OD260/280为 1.8~2.1)用于后续 cDNA的合成。全长扩增所用cDNA按照TaKaRa单链cDNA合成说明书方法进行。5′-RACE及3′-RACE所用cDNA也按照相应的试剂盒说明书要求分别合成。
1.2.2海带 PMM1基因(Sjpmm1)和 PMM2基因(Sjpmm2)的扩增
根据本研究组前期完成的海带转录数据信息(注册号GSE33853)[13],设计3′-RACE特异引物(PMM1-3O、PMM1-3I、PMM2-3O、PMM2-3I)和 5′-RACE特异引物(PMM1-5O、PMM1-5I、PMM2-5O1、PMM2-5I1、PMM2-5O2、PMM2-5I2)(表 1)。按照RACE 扩增体系及要求,采用LA Taq DNA聚合酶,进行3′-RACE套式PCR反应,同时采用HiFi高保真DNA聚合酶进行5′-RACE巢式PCR反应。将所获得的片段进行回收,连接于 pMD19-T载体上,转化至大肠杆菌(DH5α)中,挑选PCR检测为阳性的克隆送至上海生物工程有限公司进行测序。
1.2.3序列分析及比对
通过BLAST比对分析,将RACE得到的序列与NCBI数据库相关序列进行比对,选择候选海带PMM基因片段序列,利用 BioEdit软件将其与已知序列进行拼接。使用 ORF finder软件预测 Sjpmm1 和Sjpmm2的cDNA序列的开放阅读框(ORF),并将其 ORF序列翻译为氨基酸序列。利用 ProtParam[14]预测其分子量和等电点(PI)。利用SOPMA[15]对海带PMM1和PMM2(SjPMM1和SjPMM2)的二级结构进行预测,并利用DNAman 6.0软件将两个SjPMM分别与GenBank数据库中已知的3种PMM的氨基酸序列进行多重序列比对,预测这2种SjPMM的结构与功能。

表1 海带PMM基因扩增及实时定量分析所用引物信息Tab.1 Primer sequences used in the amplification and real-time PCR analysis of Sjpmm
为了比较与其他物种 PMM 的进化关系,在NCBI数据库中获得 21个其他物种的 PMM氨基酸序列,使用MEGA 6.0软件将其与海带的PMM氨基酸序列进行比对,选用邻接法(NJ)设置重复1000次,构建系统进化树。
1.2.4实时定量PCR分析
为了研究温度刺激对Sjpmm1和Sjpmm2转录情况的影响。选择β-Actin为内参基因,以qPMM1-F、qPMM1-R和qPMM2-F、qPMM2-R为引物(表1),对Sjpmm进行定量分析。所用的聚合酶为SYBR Premix Ex Taq II酶,扩增程序为: 94℃预变性30 s; 94℃变性5 s,55℃复性10 s,72℃延伸20 s,45个循环。用2-ΔΔCt法计算相对定量结果,并利用 SPSS 19.0软件对数据进行统计学分析。
1.2.5SjPMM1的体外原核重组表达
1.2.5.1Sjpmm1的ORF序列扩增
通过WatCut分析软件,对Sjpmm1的ORF进行分析,选择NdeI和EcoRI作为双酶切位点,设计全长扩增引物PMM1-F和 PMM1-R(表1)。以总cDNA为模板,LA Taq DNA聚合酶扩增Sjpmm1的ORF序列。反应条件:94℃预变性10 min; 94℃变性30 s,58℃复性30 s,72℃延伸1 min,35个循环; 72℃延伸7 min。将纯化的目的序列连接至 pMD19-T载体中并转化大肠杆菌(DH5α),对PCR检测阳性的克隆进行测序验证。
1.2.5.2表达载体的构建
使用NdeI和EcoRI两种酶对pMAL-c5X载体和提取的重组质粒pMD19-T/SjPMM1进行双酶切,电泳检测酶切产物并回收pMAL-c5X载体和SjPMM1基因。用T4-DNA连接酶连接回收产物并转化NEB表达用大肠杆菌感受态细胞,挑选阳性克隆,经PCR检测、测序验证后用于SjPMM1的体外表达。
1.2.5.3SjPMM1的诱导与表达
取适量菌液移入含有氨苄青霉素的LB液体(4 mL)培养过夜,分别取2 mL菌液至含200 mL 高营养培养基(含葡萄糖和氨苄青霉素)中,在 37℃水浴中培养,160 r/min震荡培养至OD600约为0.6时,分别加入 0、0.3 mmol/L的异丙基-β-D-硫代半乳糖苷(IPTG),诱导表达2 h。同时以0、0.3 mmol/L IPTG诱导只含pMAL-c5X载体的NEB大肠杆菌表达细胞中,其编码1个约42.5 kDa的MBP标签蛋白。诱导结束取适量菌液于4000 r/mim离心20 min,收集沉淀,用于后续SDS-PAGE检测分析。

图1 RACE扩增产物电泳检测Fig.1 Electrophoresis of RACE products
A: Sjpmm1检测结果,其中1和2分别代表5′和3′-RACE扩增结果; B: Sjpmm2扩增结果,1~3分别表示第一轮5′-RACE结果、第二轮5′-RACE结果、3′-RACE结果
A: 1~2,5′-RACE and 3′-RACE products for Sjpmm1;B: Detection of Sjpmm2; 1~3 represent the products of the first 5′-RACE and the second 5′- and 3′-RACE,respectively
2 实验结果
2.1Sjpmm1和Sjpmm2的获得
取5 μL的Sjpmm1的5′-RACE和3′-RACE扩增产物,进行凝胶电泳检测均显示明显条带(图 1A),测序分别得到458 bp和1391 bp的序列。对Sjpmm2扩增也分别得到424 bp和1698 bp序列,在该序列信息的基础上,对Sjpmm2进行再次5′-RACE扩增,得到189 bp的5′端序列(图1B)。其中3′-RACE扩增得到的序列3′末端含有明显的AATAA加尾信号序列和polyA结构。
2.2序列分析
通过序列拼接得到2条Sjpmm的cDNA序列。其中Sjpmm1(GenBank: AIW04131.1)的cDNA全长1559 bp,5′-非翻译区(5′-UTR)长47 bp,ORF长759 bp,3′-非翻译区(3′-UTR)长753 bp。经预测,其编码一个含252个氨基酸,分子量约为28.51 kDa,等电点为4.89。BLAST比对分析表明其与已知PMM序列对比,SjPMM1与假微型海链藻CCMP1335(GenBank:EED92893.1)、 小 麦 属(GenBank: AFV92896.1、ACV41082.1 等)、毛果杨(GenBank: ERP53127.1)的PMM 氨基酸序列同源区域的相似性达到 83%~85%,而与长囊水云的一个未注释蛋白(GenBank: CBJ32201.1)相似性达到 95%,初步推测也可能属于长囊水云的PMM 候选基因。Sjpmm2(GenBank: KP772272)的cDNA全长2639 bp,5′-UTR长25 bp,ORF长1866 bp,3′-UTR长748 bp,其编码621个氨基酸,分子量约为66.49 kDa,等电点为 5.32。同源比对分析表明SjPMM2与长囊水云(GenBank: CBJ29496.1)、假鱼腥藻(GenBank: AFY68962.1)、层生管孢藻(GenBank:AFY96193.1)的PMM氨基酸序列同源区域的相似性分别为77%、64%、64%。
蛋白的二级结构预测结果表明,SjPMM1的二级结构包括α-螺旋、无规则卷曲、β-折叠、β-转角,它们的比例分别为41.67%、28.57%、22.22%、7.54%;SjPMM2的二级结构包括 α-螺旋、无规则卷曲、β-折叠、β-转角,其比例分别为 40.74%、28.18%、19.81%、11.27%。
SjPMM1与长囊水云(GenBank: CBJ32201.1)、一粒小麦(GenBank: AFW03829.1)、人类(GenBank:NP_002667.2)的同源序列比对分析表明,其一致性为73.39%,且都含有HAD超家族的3个保守的motif: motif I,DXDXTLX; motif II,XXSX; motif III,K-[G/S][D/S]XXX[D/N](图2)。SjPMM2与长囊 水云(GenBank: CBJ29496.1)、 假 鱼 腥 藻(GenBank: AFY68962.1)、绿脓杆菌(GenBank:AAG08707.1)的序列一致性为 46.02%,都含有 α-磷酸己糖变位酶超家族的4个保守motif: motif I,TXSHXP; motif II,DXDXDR; motif III,XEXS;motif IV,XRXS(图3)。
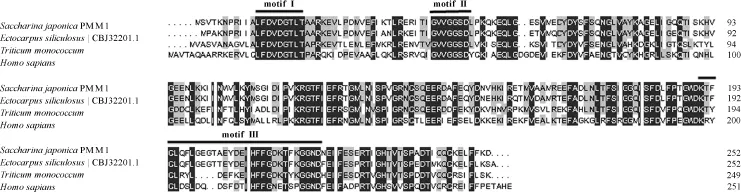
图2 SjPMM1多序列比对Fig.2 The alignment of SjPMM1 with PMM from other species
2.3系统进化分析
基于所获得的PMM序列,作者构建了系统进化树(图4)。从中可看到,PMM序列可明显地分为2个分支,而SjPMM1和SjPMM2分别位于不同的分支中。其中SjPMM1所在分支包含高等动植物、真菌以及各种藻类,覆盖了大部分物种,而SjPMM2所在分支仅包括蓝藻、细菌、古菌、红藻及褐藻,这说明,这 2种SjPMM基因可能具有不同的起源。
2.4温度对海带pmm转录的影响
在10℃处理条件下,海带Sjpmm1和Sjpmm2转录水平最低,随温度升高或者降低,其转录水平都表现逐步上升(图5)。与10℃时相比,Sjpmm1的转录水平在 5、15、20和 25℃时均显著升高(P<0.05),Sjpmm2的转录水平在 5、20和 25℃时均显著升高(P<0.05)。

图3 SjPMM2多序列比对Fig.3 The alignment of SjPMM2 with PMM from other species

图4 基于PMM氨基酸序列的系统进化树Fig.4 Polygenetic tree based on the amino acid sequence of PMM
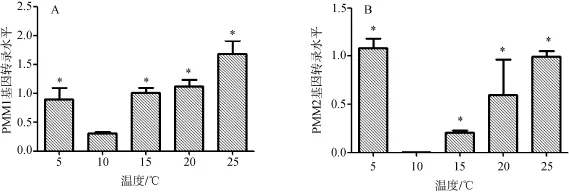
图5 温度对Sjpmm转录的影响Fig.5 The effects of temperature on the transcription of Sjpmm
A: Sjpmm1转录水平; B: Sjpmm2转录水平
A: Transcription of Sjpmm1; B: Transcription of Sjpmm2
2.5SjPMM1的体外原核表达
琼脂糖凝胶电泳检测发现对Sjpmm1 ORF的扩增得到单一条带,其大小与预期的 759 bp一致(图6A)。对pMD19-T/SjPMM1和pMAL-c5X载体的双酶切也成功得到了带黏性末端的 Sjpmm1和线性pMAL序列(图6B)。体外表达表明,其与未诱导组相比,诱导的细胞能成功表达出约为70 kDa的融合蛋白MBP/SjPMM1,这与预期的分子量71.01 kDa相吻合(图7)。
3 讨论
海带中可能存在2个长度与结构不同的PMM基因。其中,Sjpmm1编码的蛋白含252个氨基酸,与大部分真核生物中的PMM长度相似,属于HAD超家族的成员。HAD超家族主要包括葡萄糖磷酸变位酶、磷酸酯酶、磷酸丝氨酸磷酸酶等,均含有4个保守的motif,其中motif I中的第一个天冬氨酸可作为亲核剂进攻底物的磷酸基团[16],motif II为活性位点,其中的丝氨酸残基通过氢键结合到底物的磷酸基团[17],motif III参与ASP亲核剂对底物磷酸基团的定向,并结合Mg2+离子[11]。而Sjpmm2编码的蛋白长621个氨基酸,该长度的PMM仅存在于少数物种中,属于α-磷酸己糖变位酶超家族的成员,该家族中保守的motif I中的丝氨酸残基为活性中心的磷酸化位点,motif II则作为金属结合环连接Mg2+离子,motif III为糖结合位点,决定着酶对底物的特异性结合,motif IV中以精氨酸为关键位点对维持磷酸化后构想的稳定起重要作用[12,18]。虽然Sjpmm1和Sjpmm2隶属于不同的超家族,但它们同时具有丝氨酸催化中心以及金属离子结合位点等磷酸变位酶特征性结构域,反映出它们都为海带PMM基因。

图6 Sjpmm1 ORF扩增及双酶切电泳检测Fig.6 Electrophoresis analysis of Sjpmm1 amplification and digestion

图7 融合蛋白MBP/SjPMM1的SDS-PAGE分析Fig.7 SDS-PAGE of the fusion protein MBP/SjPMM1
系统进化分析表明,海带中存在的两个 PMM基因在进化上具有不同的地位,其中,Sjpmm1所在分支包含物种广泛,可能来自于最古老的真核生物; 而Sjpmm2所在分支仅包含少数物种,其中蓝藻与褐藻亲缘关系较近,作者推测其可能来自于质体的一次内共生作用,即最原始的真核生物吞噬蓝藻获得。分析还发现,较原始的单细胞红藻 Cyanidioschyzon merolae中也存在2种PMM基因,其分别位于这两个进化支上,这从另一方面支持了作者对海带PMM起源的推测。
自然条件下,海带适宜生长温度为10~15℃,当温度过高或过低,均会对海带产生生理胁迫,引起海带体内物质含量的变化[19]。本研究发现海带中的PMM基因在10℃时表达量最低,而温度升高或降低均会诱导其大量表达,因为岩藻聚糖具有抗氧化、抗菌等活性[20],作者推测海带可以通过增加 PMM 等酶的表达来加速岩藻聚糖的合成,减少环境胁迫对藻体的损伤。
大部分PMM是双功能的变位酶,兼具葡萄糖磷酸变位酶活性,且 2种活性差别明显。为了对海带PMM的功能进行解析,作者还对SjPMM1进行了体外融合性表达,将其连接入 pMAL-c5X载体中,成功得到了可溶性的 MBP/SjPMM1蛋白,浓度较高,适于进行后续的功能验证。但由于尚未获得纯化的重组蛋白,暂时未对其酶学特征进行深入分析,有待后续深入探讨,完善海带中PMM动力学特性分析。
参考文献:
[1]Tseng Chengkui.Algal biotechnology industries and research activities in China[J].Journal of Applied Phycology,2001,13(4): 375-380.
[2]魏玉西,张虹,牛锡珍.巨藻生产褐藻胶工艺改进方
法的探讨[J].海洋科学,2002,26(6): 10-12.
Wei Yuxi,Zhang Hong,Niu Xizhen.A study on the improving methods of the alginate production technology from macrocystis pyrifera[J].Marine Sciences,2002,26(6): 10-12.
[3]Michel G,Tonon T,Scornet D,et al.The cell wall polysaccharide metabolism of the brown alga Ectocarpus siliculosus.Insights into the evolution of extracellular matrix polysaccharides in Eukaryotes[J].New Phytologist,2010,188(1): 82-97.
[4]Seifert G J.Nucleotide sugar interconversions and cell
wall biosynthesis: how to bring the inside to the outside [J].
Current Opinion in Plant Biology,2004,7(3): 277-284.[5]Smirnoff N,Conklin P L,Loewus F A.Biosynthesis of ascorbic acid in plants: A renaissance[J].Annual Review of Plant Physiology and Plant Molecular Biology,2001,52: 437-467.
[6]Spiro R G.Protein glycosylation: nature,distribution,enzymatic formation,and disease implications of glycopeptide bonds[J].Glycobiology,2002,12(4): 43R-56R.
[7]Yu Chunmei,Li Yiwen,Li Bin,et al.Molecular analysis of phosphomannomutase(PMM)genes reveals a unique PMM duplication event in diverse Triticeae species and the main PMM isozymes in bread wheat tissues[J].BMC Plant Biology,2010,10: 214.
[8]Kepes F,Schekman R.The yeast SEC53 gene encodes phosphomannomutase[J].Journal of Biological Chemistry,1988,263(19): 9155-9161.
[9]Hoeberichts F A,Vaeck E,Kiddle G,et al.A Temperature-sensitive mutation in the Arabidopsis thaliana phosphomannomutase gene disrupts protein glycosylation and triggers cell death[J].Journal of Biological Chemistry,2008,283(9): 5708-5718.
[10]Feng Yanjing,Chi Shan,Liu Cui,et al.The discoveryof archaea origin phosphomannomutase in algae based on the algal transcriptome[J].Acta Oceanologica Sinica,2014,33(2): 108-113.
[11]Silvaggi N R,Zhang C C,Lu Z B,et al.The X-ray crystal structures of human alpha-phosphomannomutase 1 reveal the structural basis of congenital disorder of glycosylation type 1a[J].Journal of Biological Chemistry,2006,281(21): 14918-14926.
[12]Regni C,Tipton P A,Beamer L J.Crystal structure of PMM/PGM: An enzyme in the biosynthetic pathway of P.aeruginosa virulence factors[J].Structure,2002,10(2): 269-279.
[13]Deng Yunyan,Yao Jianting,Wang Xiuliang,et al.Transcriptome sequencing and comparative analysis of Saccharina japonica(Laminariales,Phaeophyceae)under blue light induction[J].PloS one,2012,7(6): e39704.
[14]Gasteiger E,Hoogland C,Gattiker A,et al.Protein Identification and Analysis Tools on the ExPASy Server [M]//WALKER J.The Proteomics Protocols Handbook.Humana Press,2005: 571-607.
[15]Geourjon C,Deleage G.SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Computer Applications in the Biosciences: CABIOS,1995,11(6): 681-684.
[16]Collet J F,Stroobant V,Pirard M,et al.A new class of phosphotransferases phosphorylated on an aspartate residue in an amino-terminal DXDX(T/V)motif [J].Journal of Biological Chemistry,1998,273(23): 14107-14112.
[17]Wang W R,Kim R,Jancarik J,et al.Crystal structure of phosphoserine phosphatase from Methanococcus jannaschii,a hyperthermophile,at 1.8 angstrom resolution[J].Structure,2001,9(1): 65-71.
[18]Videira P A,Cortes L L,Fialho A M,et al.Identification of the pgmG gene,encoding a bifunctional protein with phosphoglucomutase and phosphomannomutase activities,in the gellan gum-producing strain Sphingomonas paucimobilis ATCC 31461[J].Applied and Environmental Microbiology,2000,66(5): 2252- 2258.
[19]吴海一,王翔宇,朱安成.脆嫩-厚成期海带物质成分变化分析研究[J].海洋科学,2015,39(8): 35-38.Wu Haiyi,Wang Xiangyu,Zhu Ancheng.Analysis of component changes of Saccharina japonica at mushroom-adult stage[J].Marine Sciences,2015,39(8): 35-38.[20]Jiao Guangling,Yu Guangli,Zhang Junzeng,et al.Chemical structures and bioactivities of sulfated polysaccharides from marine algae[J].Marine Drugs,2011,9(2): 196-223.
ZHANG Peng-yan1,2,3,YU Xue1,2,3,YAO Jian-ting1,2,DUAN De-lin1,2
(1.Key Laboratory of Experimental Marine Biology,Institute of Oceanology,Chinese Academy of Sciences,Qingdao 266071,China; 2.Laboratory for Marine Biology and Biotechnology,Qingdao National Laboratory for Marine Science and Technology,Qingdao 266071,China; 3.University of Chinese Academy of Sciences,Beijing 100049,China)
Received: Jan.13,2015
(本文编辑: 张培新)
Cloning and expression of phosphomannomutase from Saccharina japonica(Laminariales,Phaeophyceae)
Key words:Saccharina japonica; phosphomannomutase; fucoidan; alginate; real-time PCR analysis
Abstract:Phosphomannomutase(PMM)is an important enzyme involved in the synthesis of fucoidan and alginate.Two PMM genes of Saccharina japonica(Sjpmm1 and Sjpmm2)were cloned by rapid-amplification of cDNA ends(RACE).The open reading frame(ORF)length of Sjpmm1 is 759 bp,encoding 252 amino acids(SjPMM1)with a molecular weight(MW)of 28.51 kDa which belongs to the haloacid dehalogenase(HAD)superfamily.While the ORF length of Sjpmm2 is 1866 bp,encoding 621 amino acids(SjPMM2)with a MW of 66.49 kDa which belongs to the phosphohexomutase superfamily.The α-helix is the major secondary structure for both SjPMM1 and SjPMM2.The phylogenetic analysis showed that Sjpmm1 evolved from ancient eukaryotes,while Sjpmm2 originated from primary endosymbiosis.In addition,real-time PCR analysis indicated that temperature could increase the transcriptional level of Sjpmm,which may lead to the upregulation of fucoidan.Furthermore,a high concentration of the SjPMM1 fusion protein was expressed with the pMAL-c5X vector,contributing to the further function studies.
中图分类号:X55
文献标识码:A
文章编号:1000-3096(2016)03-0032-08
doi:10.11759/hykx20150113001
收稿日期:2015-01-13; 修回日期: 2015-03-10
基金项目:国家科技支撑计划项目(2013BAB01B01); 国家海洋公益性行业科研专项(201405040)
作者简介:张朋艳(1989-),女,山东临沂人,硕士研究生,主要从事海洋生物学研究,电话,0532-82898554,E-mail: zhangpengyan3249@ 126.com; 段德麟,通信作者,研究员,电话,0532-82898556,E-mail:dlduan@qdio.ac.cn
