骨骼肌发育调控相关lncRNAs研究进展
2016-07-11占思远王林杰张红平
占思远,李 利,王林杰,仲 涛,张红平
(四川农业大学畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130)
骨骼肌发育调控相关lncRNAs研究进展
占思远,李利,王林杰,仲涛,张红平*
(四川农业大学畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130)
摘要:长链非编码RNA(Long non-coding RNA,lncRNA)是一类长度大于200 个核苷酸、无蛋白质编码功能的RNA。相对于研究较多的短链非编码RNA,lncRNA的种类繁多,数量占哺乳动物基因组的绝大部分,功能目前尚不完全清楚。近年来研究发现,lncRNA的功能涉及表观遗传、转录调控、细胞分化、胚胎发育以及疾病发生等诸多方面。本文总结了lncRNA的分类、作用机制及其在骨骼肌发育调控和家养动物中的研究进展。
关键词:长链非编码RNA;作用机制;骨骼肌发育;调控
骨骼肌的发育是由多种因素参与并受严格调控的复杂生物学过程,对哺乳动物的生长发育有着重要的影响。骨骼肌细胞的增殖与分化是骨骼肌发育研究的核心问题,以往的研究主要集中于各类成肌调控因子的时序表达及其对骨骼肌细胞增殖分化的调控机理,包括成肌调节因子家族(Myf5、MyoD、MyoG 和 Myf6)、肌细胞增强因子-2(Myocyte enhancer factor-2,MEF-2)家族、含有配对结构域的转录调控因子Pax家族成员Pax3和Pax7等。随着对哺乳动物中RNA转录本日渐深入的研究,哺乳动物基因组上大部分的非编码RNA被证实具有重要的基础代谢以及调控功能,例如rRNA、tRNA、miRNA、siRNA以及Piwi蛋白互作RNA(piRNA)等。其中短链ncRNA(miRNA、siRNA以及piRNA)被重点关注并被证实在哺乳动物生长、发育和繁殖等生理代谢过程中具有广泛的调控作用[1]。然而长链非编码RNA(Long non-coding RNA,lncRNA)是一类在真核生物体内主要由RNA聚合酶Ⅱ指导转录而成并且长度大于200 nt的ncRNA[2],由于已知功能甚少,其对应的基因组DNA序列甚至被认为是“垃圾”DNA。近年的研究表明,lncRNA能在表观遗传、转录及转录后水平上等多个层次调控基因的表达,并参与了X染色体沉默、基因组印记、胚胎发育、脂肪代谢调控、骨骼肌生长发育以及染色质修饰等重要的调控过程。本文将概述lncRNA的分类、作用机制及其在骨骼肌发育调控方面的研究进展。
1lncRNA的分类与作用机制
lncRNA是一类长度大于200个核苷酸,无蛋白质编码潜能的RNA。lncRNA通常较长,具有mRNA样结构和启动子结构,有些具有poly(A)尾巴,有些没有poly(A)尾巴,与编码基因相比,lncRNA表达量更低。许多lncRNA都具有保守的二级结构,一定的剪切形式以及亚细胞定位;同时lncRNA的表达也具有时空特异性。这种保守性和特异性表明它们是具有功能的。根据它们在基因组上相对于蛋白质编码基因的位置,将其分为5类,包括反义型lncRNA、增强子型lncRNA、发散型lncRNA、内含子型lncRNA和基因间型lncRNA(lincRNA)[3]。依据lncRNA的功能,可以将其划分为信号分子(Signal molecule)、诱骗分子(Decoy molecule)、引导分子(Guide molecule)以及骨架分子(Scaffold molecule)等4类分子类型[4]。目前相关科研人员已经建立了多个lncRNA的数据库(表1)。这些数据库所收录的lncRNA数据来自于NCBI和已发表的论文,包括人、小鼠、猪、鸡和牛等物种的lncRNA信息。
目前的研究表明,lncRNA发挥生物学功能的主要作用机制有基因印记、染色质重塑、组蛋白修饰、转录和转录后水平调控、细胞凋亡和细胞周期调控等。已有研究证实,H19[5]、X染色体特异性失活转录本[6](X inactivation-specific transcription,Xist)等多种lncRNAs参与了基因组印记。lncRNA调控机制中很重要的一部分就是导致染色质重塑,有作者研究了干细胞中lncRNA与组蛋白甲基化的调控关系。研究人员利用定制的lncRNA芯片筛选不同类型样本(小鼠胚胎干细胞、谱系限制性神经前体细胞和终端分化成纤维细胞)中lncRNA表达差异,分析其表达变化及启动子区域H3K4和H3K27甲基化程度,发现在不同类型细胞中lncRNA表达模式存在差异。在ES细胞中,lncRNA表达下调导致启动子区域H3K4甲基化程度变高,从而提出Ezh2介导H3K27甲基化的lncRNA沉默机制[7]。另外,lncRNA还可以在转录水平上直接与靶基因结合,促进或抑制靶基因的表达。已经有研究表明,lncRNA不仅可以招募转录抑制复合物来沉默基因的表达,还可以作为辅助因子招募转录因子,参与基因表达的调控[8]。例如细胞周期蛋白D1(cyclin D1)在人类细胞系中的转录抑制机制[9],DNA损伤信号可以诱导cyclin D1基因启动子区域的lncRNA表达,这些lncRNA可以将TLS(RNA 结合蛋白)招募到cyclin D1基因启动子区域。随后,TLS通过抑制CREB结合蛋白和p300的组蛋白乙酰转移酶活性,进而抑制cyclin D1基因的表达。此外,还有研究表明,lncRNA也具有调控细胞生长的作用,这主要是通过调控细胞凋亡和细胞周期来实现的。生长阻滞特异转录物5(Growth arrest-specific transcript 5,Gas5)是哺乳动物细胞生长和凋亡的关键性调控因子,它通过模拟糖皮质激素应答元件来结合糖皮质激素受体的 DNA结合域,阻止糖皮质激素受体与其他糖皮质激素应答元件之间的相互作用,从而抑制下游基因的表达,促进细胞凋亡的发生[10]。
2lncRNA调控骨骼肌发育的作用机制
近年来,越来越多的研究证实非编码RNA(ncRNAs)也是肌肉调控网络中重要的成员。到目前为止,miRNAs是研究最广泛的一种非编码RNA,许多肌肉特异性和非肌肉特异性表达的miRNAs被发现,提示了骨骼肌发育调控的复杂性。而作为非编码RNA中另一个重要成员,lncRNA广泛存在于多种生物体内,其功能涉及到细胞生命活动和个体发育与疾病发生的各个方面。目前关于lncRNA的研究主要集中在筛选、鉴定存在于不同物种的不同组织或调控机体生命活动过程的lncRNAs[28-36]。随着lncRNAs研究的深入,在人和小鼠等模式动物上,lncRNAs的功能机制研究日益增多。因此,越来越多的研究发现骨骼肌的发育过程也会受到各种lncRNAs的调控(表2)。
2.1lncRNA通过竞争性结合miRNA调控骨骼肌的生长发育
M.Cesana等[37]的研究证实了一种在成肌细胞分化过程中特异性表达的长链非编码RNA-linc-MD1可发挥ceRNA(Competing endogenous RNA,竞争性内源RNA)活性,通过miR-133 和miR-135实现对肌肉特异性转录调控因子MAML1和MEF2C的表达调控。在未分化的肌细胞中,miR-133和miR-135可分别结合MAML-1和MEF2C基因的mRNA,并抑制其表达。linc-MD-1含有这两个miRNA的结合位点,分化过程中高表达的linc-MD-1竞争性结合细胞中的miR-133和miR-135,降低了这两种miRNA的浓度,从而解除了对肌肉分化相关蛋白质因子的抑制作用,促进了细胞的分化。另外,linc-MD1的表达抑制可导致成肌细胞分化延迟,与杜氏肌营养不良症的发病密切相关[37]。近年来,关于肺腺癌转移相关转录本Malat1(Metastasis-associated lung adenocarcinoma transcript 1)在肿瘤方面的研究已经很清楚,但最近的研究发现Malat1还能调节肌细胞的生长。X.Han等[38]在C2C12细胞中研究发现,Malat1通过竞争性的结合miR-133,调控其靶基因肌细胞分化关键因子SRF(Serum response factor)的表达,从而影响肌细胞的分化。研究表明,Malat1被沉默时,与Malat1结合的miR-133被释放出来作用于SRF的3′UTR区,导致SRF的表达抑制从而抑制了肌细胞的分化。
表1lncRNA相关数据库
Table 1lncRNA databases

数据库Database网址Website参考文献ReferenceLNCipediahttp://www.lncipedia.org[11]ncRNAdatabasehttp://biobases.ibch.poznan.pl/ncRNA[12]NONCODEv4.0http://www.noncode.org[13]lncRNAdbhttp://www.lncrnadb.org[14]NREDhttp://jsm-research.imb.uq.edu.au/NRED[15]ncFANshttp://www.noncode.org/ncFANs[16]fRNAdbhttp://www.ncrna.org/frnadb[17]LncRNADiseasehttp://cmbi.bjmu.edu.cn/lncrnadisease[18]ChIPBasehttp://deepbase.sysu.edu.cn/chipbase[19]DIANA-LncBasehttp://www.microrna.gr/LncBase[20]starBasev2.0http://starbase.sysu.edu.cn[21]FunctionallncRNADatabasehttp://www.valadkhanlab.org/database[22]lnCeDBhttp://gyanxet-beta.com/lncedb[23]ALDBhttp://res.xaut.edu.cn/aldb/index.jsp[24]LncRNA2Targethttp://www.lncrna2target.org[25]LncRNASNPhttp://bioinfo.life.hust.edu.cn/lncRNASNP[26]LncRNAWikihttp://lncrna.big.ac.cn/index.php/Main_Page[27]
2.2lncRNA通过调节靶mRNA的表达和降解影响骨骼肌的生长发育
Malat1是一种高度保守的约8.7 kb的非编码转录本,在癌细胞中大量表达,同时也是癌细胞转移的重要信号分子[39]。有试验研究发现重组了肌肉抑制素(Myostatin)的小鼠腓肠肌中,Malat1的表达量显著下降[40];同时也发现在小鼠成肌细胞向肌管分化的过程中,Malat1表达量上升,敲除了Malat1的成肌细胞的增殖被抑制。这些结果表明,Malat1作为一种新的myostatin的下游靶标,在肌细胞的生成过程中起着重要的调控作用。J.Wang等[41]研究表明,鼠类含有SINE(Short interspersed elements)的非编码长链RNA能够控制肌肉生成。比如1/2-sbsRNA通过与RNA结合蛋白STAU1或STAU2的结合引起SMD(Staufen1-mediated mRNA decay)对mRNA的降解从而正向调控小鼠C2C12细胞的成肌分化过程。
2.3lncRNA通过顺式作用影响骨骼肌的成肌分化
L.Lu等[42]研究发现了一种肌肉相关lincRNA—Yam-1,它能够被转录因子Yin Yang 1(YY1)正向调节,同时Yam-1也是肌细胞生成的抑制因子,它通过顺式调控miR-715,影响其靶基因Wnt7b的表达,从而达到抑制成肌细胞分化的目的。另外有研究也发现了lncRNA的非顺式调控作用模式,例如MUNC(MyoD Upstream Non-Coding),通常被认为是一种DRR(Distal Regulatory Region)增强子RNA(DRReRNA),在骨骼肌组织中特异性地表达[43]。试验结果表明,MUNC的作用机制并不像传统的eRNA那样,通过刺激邻近位点的MyoD基因的表达来达到顺式调控的目的,而更像一种前肌源lncRNA,通过直接或间接地作用于多个启动子区域增加生肌调节因子基因的表达,从而促进骨骼肌细胞的成肌分化过程[43]。
2.4lncRNA作为印记基因在骨骼肌生长发育中发挥重要作用
B.K.Dey等[44]研究发现,作为参与基因组印记的典型lncRNA—H19,它在胚胎组织中大量转录,呈母系表达,但出生后表达被显著抑制,唯独在骨骼肌中仍维持显著的转录水平,进一步研究发现,H19敲低的小鼠成肌细胞和H19敲除小鼠的骨骼肌卫星细胞中,成肌分化显著降低。H19的外显子1编码两个保守miRNAs前体,分别为miR-675-3p和miR-675-5p,H19缺陷小鼠在受伤后表现为畸形的骨骼肌再生,当外源引入miR-675-3p和miR-675-5p后可以被调整,它们能直接靶定和下调抗分化转录因子Smad和DNA复制起始因子Cdc6的表达,从而促进骨骼肌的成肌分化和肌肉再生。另有研究结果表明,let-7的过度表达会引起过早的肌肉分化,而H19可以充当分子海绵来调节let-7的活性,进而阻止肌肉分化的过早发育[5]。
2.5lncRNA调控肌源性前体细胞的分化决定
M.Ballarino等[45]研究新发现了一种调控肌肉生成的长链非编码RNA—lnc-31,它和miR-31来自相同的核内miR-31前体,但产生途径却独立于miR-31。小鼠lnc-31与它的人类同源序列hsa-lnc-31被证实在维持细胞增殖和抑制分化中扮演了重要的作用。同样地,小鼠lnc-31和hsa-lnc-31被发现在mdx(DMD动物模型)小鼠肌肉和Duchenne型肌营养不良(DMD)的病人成肌细胞中呈高表达,伴随着大量的肌源性前体细胞的激活。这些结果表明,lnc-31在调控肌源性前体细胞的分化决定方面起着关键性的作用。
2.6lncRNA编码微肽调控骨骼肌细胞的生理学过程
最近,有研究者发现了一个保守微肽,命名为肌调素(Myoregulin,MLN),它由骨骼肌特异性RNA编码,这个RNA被注释为假定的长链非编码RNA[46]。MLN与肌浆网受磷蛋白(Phospholamban,PLN)和肌脂蛋白(Sarcolipin,SLN)结构和功能相似,并抑制心肌肌浆网Ca2+-ATP酶(SERCA),SERCA通过调节钙离子摄取到肌质网(SR)控制肌肉松弛。MLN直接与SERCA相互作用并阻碍钙吸收到SR。PLN和SLN在小鼠心和慢速骨骼肌中表达,MLN积极的表达于所有骨骼肌。在小鼠中的MLN基因缺失,增强钙离子在骨骼肌中的处置能力并提高运动能力[46]。这些发现表明了MLN是骨骼肌生理的一个重要调节因子,最大的亮点是在许多被注释为非编码的RNA可能编码“微肽(Micropeptides)”。
2.7lncRNA通过调节DNA甲基化影响骨骼肌成肌分化
L.Wang等[47]在骨骼肌成肌细胞中鉴定出一个lncRNA—Dum(多能发育相关基因2(Dppa2)上游结合肌肉lncRNA),体内外研究发现,Dum的表达量在肌细胞生成过程中呈动态变化,它能被上游的MyoD转录诱导从而影响成肌细胞分化;进一步研究表明,Dum能够促进成肌细胞的分化和受损肌肉的再生,作用机制在于在染色体上Dum基因座与Dppa2基因的启动子区域是并排的,Dum通过招募多种DNA甲基化转移酶—Dnmts(Dnmt1,Dnmt3a和Dnmt3b)到Dppa2的启动子区域,从而导致CpG位点的高甲基化和Dppa2基因的沉默。这些研究结果表明,MyoD-Dum-Dppa2调节轴在成肌分化和肌肉再生中发挥了重要的功能。
2.8lncRNA调控骨骼肌发育的其他机制
目前虽然有很多lncRNAs被发现,但是大部分的功能尚未清楚,比如人类的ε/β lncRNAs(Men ε/β lncRNAs),它有ε和β两种亚型,在患有I型多发性内分泌瘤的病人多个组织中表达,包括肌肉组织[48],在C2C12细胞分化过程中,Men ε/β lncRNAs的表达量显著上升,但是具体的生物学机制仍不清楚;另有研究发现,类固醇受体RNA激活因子(Steroid receptor RNA activator,SRA)RNA具有两种特异的存在形式,分别是非编码和编码的RNA(Yielding SRA ncRNA and protein SRAP),其中SRA ncRNA作为MyoD的共同激活因子,在肌细胞分化过程中高度表达[49-50]。最近有研究还发现了一种lncRNA—细胞核因子非编码抑制因子(Non-coding repressor of NFAT,NRON),它本身具有组织特异性的表达模式,在胎盘、肌肉和淋巴组织中高度表达[51]。以上的这些研究都表明,在骨骼肌的生长发育过程中,长链非编码RNA起着重要的调控作用,并且作用机制具有多元化特点,需要更深入的功能机制研究。
表2骨骼肌发育调控相关lncRNAs
Table 2lncRNAs in the regulation of skeletal muscle development
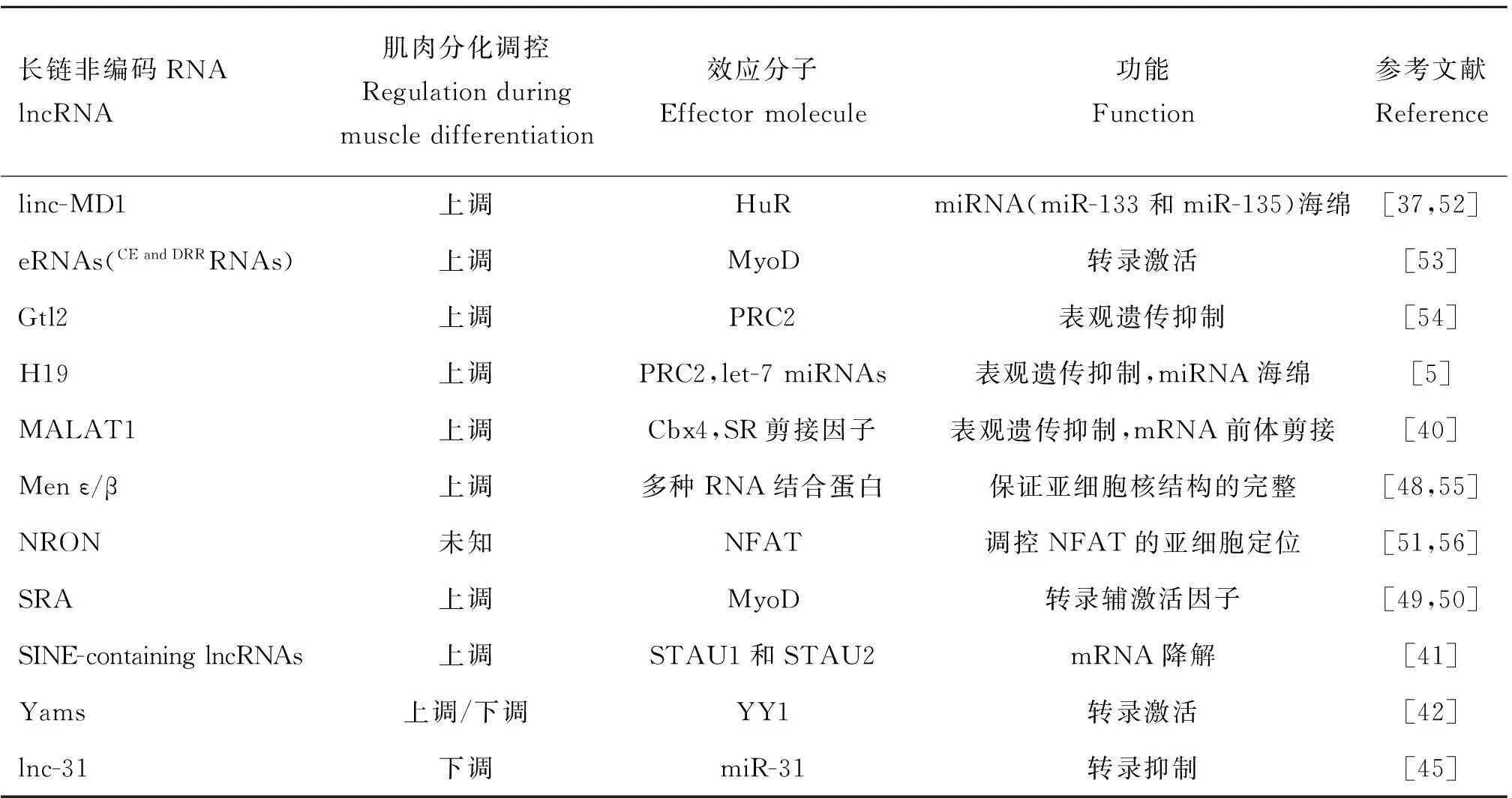
长链非编码RNAlncRNA肌肉分化调控Regulationduringmuscledifferentiation效应分子Effectormolecule功能Function参考文献Referencelinc-MD1上调HuRmiRNA(miR-133和miR-135)海绵[37,52]eRNAs(CEandDRRRNAs)上调MyoD转录激活[53]Gtl2上调PRC2表观遗传抑制[54]H19上调PRC2,let-7miRNAs表观遗传抑制,miRNA海绵[5]MALAT1上调Cbx4,SR剪接因子表观遗传抑制,mRNA前体剪接[40]Menε/β上调多种RNA结合蛋白保证亚细胞核结构的完整[48,55]NRON未知NFAT调控NFAT的亚细胞定位[51,56]SRA上调MyoD转录辅激活因子[49,50]SINE-containinglncRNAs上调STAU1和STAU2mRNA降解[41]Yams上调/下调YY1转录激活[42]lnc-31下调miR-31转录抑制[45]
3家养动物骨骼肌相关lncRNAs的研究进展
目前,国内外lncRNA的研究还处于初步阶段,lncRNA的鉴定以及功能研究主要集中于人以及模式动物中(如小鼠、果蝇和线虫等),其他物种的lncRNA研究相对滞后,特别是在家养动物领域。至今只有较少的文章对鸡、牛和猪的lncRNA进行了研究报道,其他家养动物中lncRNA的相关研究仍然是空白。
T.Li等[36]在2012年报道了鸡骨骼肌中lncRNA的鉴定研究结果。该研究组利用RNA-seq 技术对3个发育阶段的鸡骨骼肌进行了转录组测序,并利用自行开发的生物信息学流程鉴定出281个鸡的lncRNAs。并在进一步分析中发现,相比蛋白编码基因,鸡lncRNA的保守性更低。其后,J.Luo[57]对马莱克氏病抵抗型和易感型的鸡CD4 细胞系进行了转录组测序和比对,共鉴定出2 626个鸡的lncRNAs,其中1 177个特异性表达于马莱克氏病抵抗型细胞,另有1 048个特异性表达于马莱克氏病易感型细胞中。
W.Huang等[58]对公共的牛特异性表达序列标签(Expressed sequence tags,EST)数据进行了重新组装,并利用支持向量机算法对RNA转录本进行了编码蛋白质潜能筛选,共鉴定出405个基因间lncRNAs(lincRNAs)。这些牛lncRNAs普遍具有组织特异性表达特征,它们的GC含量比随机选择的基因间序列的要高,但比蛋白质编码基因的低,并且它们在哺乳动物之间中度保守。C.Billerey等[35]利用RNA-seq技术对9头利木赞牛的胸腰最长肌进行了转录组测序,共鉴定出了418个lincRNAs,并对其基因组特征进行了分析。此后2014年12月,J.Caballero等[59]利用转录组芯片技术对奶牛早期胚胎发育不同阶段细胞进行测序分析,发现了3个从卵母细胞到8细胞期差异表达的lncRNAs,进一步研究表明,这些lncRNAs是通过调节靶基因mRNA的翻译水平,从而调控奶牛早期胚胎的发育过程。
Z.Y.Zhou等[60]利用已知的猪RNA-seq信息和ESTs数据,鉴定出6 621个lincRNAs,发现其中一些lincRNA具有同线性和序列保守性,如linc-sscg2561,在基因组上与其邻近的是与情绪行为相关联的Dnmt3a(DNA methyltransferase 3a)基因。研究还发现linc-sscg2561和Dnmt3a在家猪和野猪的额叶皮质中表达差异显著,提示linc-sscg2561在家猪的驯化过程中可能发挥了重要的作用。
4展望
尽管长度小于50个碱基的非编码 RNA(如 miRNA 和 piRNA 等)的研究已取得突破性进展,但长度大于200个碱基的非编码RNA还有很多的功能和作用机制尚未阐明。lncRNA不仅数量巨大,且其作用机制具有多样性和复杂性,而随着lncRNA研究技术和手段的不断发展,lncRNA的功能机制研究也会越来越多元化。通过对骨骼肌发育相关lncRNAs的研究,将进一步深化对哺乳动物肌肉生成分子机制的认识,并可能为肌肉发育相关疾病的治疗提供新的思路和应对策略。
参考文献(References):
[1]STEFANI G,SLACK F J.Small non-coding RNAs in animal development[J].NatRevMolCellBiol,2008,9(3):219-230.
[2]WILUSZ J E,SUNWOO H,SPECTOR D L.Long noncoding RNAs:functional surprises from the RNA world[J].GenesDev,2009,23(13):1494-1504.
[3]KNOLL M,LODISH H F,SUN L.Long non-coding RNAs as regulators of the endocrine system[J].NatRevEndocrinol,2015,11(3):151-160.
[4]WANG K C,CHANG H Y.Molecular mechanisms of long noncoding RNAs[J].MolCell,2011,43(6):904-914.
[5]KALLEN A N,ZHOU X B,XU J,et al.The imprinted H19 lncRNA antagonizes let-7 microRNAs[J].MolCell,2013,52(1):101-112.
[6]ENGREITZ J M,PANDYA-JONES A,MCDONEL P,et al.The Xist lncRNA exploits three-dimensional genome architecture to spread across the X chromosome[J].Science,2013,341(6147):1237973.
[7]WU S C,KALLIN E M,ZHANG Y.Role of H3K27 methylation in the regulation of lncRNA expression[J].CellRes,2010,20(10):1109-1116.
[8]CHEN L L,CARMICHAEL G G.Decoding the function of nuclear long non-coding RNAs[J].CurrOpinCellBiol,2010,22(3):357-364.
[9]WANG X,ARAI S,SONG X,et al.Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription[J].Nature,2008,454(7200):126-130.
[10]KINO T,HURT D E,ICHIJO T,et al.Noncoding RNA gas5 is a growth arrest-and starvation-associated repressor of the glucocorticoid receptor[J].SciSignal,2010,3(107):ra8.
[11]VOLDERS P J,HELSENS K,WANG X,et al.LNCipedia:a database for annotated human lncRNA transcript sequences and structures[J].NucleicAcidsRes,2013,41(Database issue):D246-D251.
[12]ERDMANN V A,SZYMANSKI M,HOCHBERG A,et al.Non-coding,mRNA-like RNAs database Y2K[J].NucleicAcidsRes,2000,28(1):197-200.
[13]XIE C,YUAN J,LI H,et al.NONCODEv4:exploring the world of long non-coding RNA genes[J].NucleicAcidsRes,2014,42(Database issue):D98-D103.
[14]AMARAL P P,CLARK M B,GASCOIGNE D K,et al.lncRNAdb:a reference database for long noncoding RNAs[J].NucleicAcidsRes,2011,39(Database issue):D146-D151.
[15]DINGER M E,PANG K C,MERCER T R,et al.NRED:a database of long noncoding RNA expression[J].NucleicAcidsRes,2009,37(Database issue):D122-D126.
[16]LIAO Q,XIAO H,BU D,et al.ncFANs:a web server for functional annotation of long non-coding RNAs[J].NucleicAcidsRes,2011,39(Web Server issue):W118-W124.
[17]MITUYAMA T,YAMADA K,HATTORI E,et al.The functional RNA database 3.0:databases to support mining and annotation of functional RNAs[J].NucleicAcidsRes,2009,37(Database issue):D89-D92.
[18]CHEN G,WANG Z,WANG D,et al.LncRNADisease:a database for long-non-coding RNA-associated diseases[J].NucleicAcidsRes,2013,41(Database issue):D983-D986.
[19]YANG J H,LI J H,JIANG S,et al.ChIPBase:a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data[J].NucleicAcidsRes,2013,41(Database issue):D177-D187.
[20]PARASKEVOPOULOU M D,GEORGAKILAS G,KOSTOULAS N,et al.DIANA-LncBase:experimentally verified and computationally predicted microRNA targets on long non-coding RNAs[J].NucleicAcidsRes,2013,41(Database issue):D239-D245.
[21]LI J H,LIU S,ZHOU H,et al.StarBase v2.0:decoding miRNA-ceRNA,miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J].NucleicAcidsRes,2014,42(Database issue):D92-D97.
[22]NIAZI F,VALADKHAN S.Computational analysis of functional long noncoding RNAs reveals lack of peptide-coding capacity and parallels with 3′UTRs[J].RNA,2012,18(4):825-843.
[23]DAS S,GHOSAL S,SEN R,et al.lnCeDB:database of human long noncoding RNA acting as competing endogenous RNA[J].PLoSOne,2014,9(6):e98965.
[24]LI A,ZHANG J,ZHOU Z,et al.ALDB:a domestic-animal long noncoding RNA database[J].PLoSOne,2015,10(4):e0124003.
[25]JIANG Q,WANG J,WU X,et al.LncRNA2Target:a database for differentially expressed genes after lncRNA knockdown or overexpression[J].NucleicAcidsRes,2015,43(Database issue):D193-D196.
[26]GONG J,LIU W,ZHANG J,et al.lncRNASNP:a database of SNPs in lncRNAs and their potential functions in human and mouse[J].NucleicAcidsRes,2015,43(Database issue):D181-D186.
[27]MA L,LI A,ZOU D,et al.LncRNAWiki:harnessing community knowledge in collaborative curation of human long non-coding RNAs[J].NucleicAcidsRes,2015,43(Database issue):D187-D192.
[28]ZHU J G,SHEN Y H,LIU H L,et al.Long noncoding RNAs expression profile of the developing mouse heart[J].JCellBiochem,2014,115(5):910-918.
[29]MATKOVICH S J,EDWARDS J R,GROSSENHEIDER T C,et al.Epigenetic coordination of embryonic heart transcription by dynamically regulated long noncoding RNAs[J].ProcNatlAcadSciUSA,2014,111(33):12264-12269.
[30]MALOUF G G,ZHANG J,YUAN Y,et al.Characterization of long non-coding RNA transcriptome in clear-cell renal cell carcinoma by next-generation deep sequencing[J].MolOncol,2015,9(1):32-43.
[31]LV J,HUANG Z,LIU H,et al.Identification and characterization of long intergenic non-coding RNAs related to mouse liver development[J].MolGenetGenomics,2014,289(6):1225-1235.
[32]LIPOVICH L,TARCA A L,CAI J,et al.Developmental changes in the transcriptome of human cerebral cortex tissue:long noncoding RNA transcripts[J].CerebCortex,2014,24(6):1451-1459.
[33]LIANG M,LI W,TIAN H,et al.Sequential expression of long noncoding RNA as mRNA gene expression in specific stages of mouse spermatogenesis[J].SciRep,2014,4:5966.
[34]LV J,CUI W,LIU H,et al.Identification and characterization of long non-coding RNAs related to mouse embryonic brain development from available transcriptomic data[J].PLoSOne,2013,8(8):e71152.
[35]BILLEREY C,BOUSSAHA M,ESQUERRÉ D,et al.Identification of large intergenic non-coding RNAs in bovine muscle using next-generation transcriptomic sequencing[J].BMCGenomics,2014,15:499.
[36]LI T,WANG S,WU R,et al.Identification of long non-protein coding RNAs in chicken skeletal muscle using next generation sequencing[J].Genomics,2012,99(5):292-298.
[37]CESANA M,CACCHIARELLI D,LEGNINI I,et al.A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J].Cell,2011,147(2):358-369.
[38]HAN X,YANG F,CAO H,et al.Malat1 regulates serum response factor through miR-133 as a competing endogenous RNA in myogenesis[J].FASEBJ,2015,29(7):3054-3064.
[39]SCHMIDT L H,SPIEKER T,KOSCHMIEDER S,et al.The long noncoding MALAT-1 RNA indicates a poor prognosis in non-small cell lung cancer and induces migration and tumor growth[J].JThoracOncol,2011,6(12):1984-1992.
[40]WATTS R,JOHNSEN V L,SHEARER J,et al.Myostatin-induced inhibition of the long noncoding RNA Malat1 is associated with decreased myogenesis[J].AmJPhysiolCellPhysiol,2013,304(10):C995-C1001.
[41]WANG J,GONG C,MAQUAT L E.Control of myogenesis by rodent SINE-containing lncRNAs[J].GenesDev,2013,27(7):793-804.
[42]LU L,SUN K,CHEN X,et al.Genome-wide survey by ChIP-seq reveals YY1 regulation of lincRNAs in skeletal myogenesis[J].EMBOJ,2013,32(19):2575-2588.
[43]MUELLER A C,CICHEWICZ M A,DEY B K,et al.MUNC,a long noncoding RNA that facilitates the function of MyoD in skeletal myogenesis[J].MolCellBiol,2015,35(3):498-513.
[44]DEY B K,PFEIFER K,DUTTA A.The H19 long noncoding RNA gives rise to microRNAs miR-675-3p and miR-675-5p to promote skeletal muscle differentiation and regeneration[J].GenesDev,2014,28(5):491-501.
[45]BALLARINO M,CAZZELLA V,D’ANDREA D,et al.Novel long noncoding RNAs (lncRNAs) in myogenesis:a miR-31 overlapping lncRNA transcript controls myoblast differentiation[J].MolCellBiol,2015,35(4):728-736.
[46]ANDERSON D M,ANDERSON K M,CHANG C L,et al.A micropeptide encoded by a putative long noncoding RNA regulates muscle performance[J].Cell,2015,160(4):595-606.
[47]WANG L,ZHAO Y,BAO X,et al.LncRNA Dum interacts with Dnmts to regulate Dppa2 expression during myogenic differentiation and muscle regeneration[J].CellRes,2015,25(3):335-350.
[48]SUNWOO H,DINGER M E,WILUSZ J E,et al.MEN epsilon/beta nuclear-retained non-coding RNAs are up-regulated upon muscle differentiation and are essential components of paraspeckles[J].GenomeRes,2009,19(3):347-359.
[49]HUBÉ F,VELASCO G,ROLLIN J,et al.Steroid receptor RNA activator protein binds to and counteracts SRA RNA-mediated activation of MyoD and muscle differentiation[J].NucleicAcidsRes,2011,39(2):513-525.
[50]CARETTI G,SCHITZ R L,DILWORTH F J,et al.The RNA helicases p68/p72 and the noncoding RNA SRA are coregulators of MyoD and skeletal muscle differentiation[J].DevCell,2006,11(4):547-560.
[51]WILLINGHAM A T,ORTH A P,BATALOV S,et al.A strategy for probing the function of noncoding RNAs finds a repressor of NFAT[J].Science,2005,309(5740):1570-1573.
[52]LEGNINI I,MORLANDO M,MANGIAVACCHI A,et al.A feedforward regulatory loop between HuR and the long noncoding RNA linc-MD1 controls early phases of myogenesis[J].MolCell,2014,53(3):506-514.
[53]MOUSAVI K,ZARE H,DELL’ORSO S,et al.eRNAs promote transcription by establishing chromatin accessibility at defined genomic loci[J].MolCell,2013,51(5):606-617.
[54]SCHUSTER-GOSSLER K,BILINSKI P,SADO T,et al.The mouse Gtl2 gene is differentially expressed during embryonic development,encodes multiple alternatively spliced transcripts,and may act as an RNA[J].DevDyn,1998,212(2):214-228.
[55]CLEMSON C M,HUTCHINSON J N,SARA S A,et al.An architectural role for a nuclear noncoding RNA:NEAT1 RNA is essential for the structure of paraspeckles[J].MolCell,2009,33(6):717-726.
[56]SHARMA S,FINDLAY G M,BANDUKWALA H S,et al.Dephosphorylation of the nuclear factor of activated T cells (NFAT) transcription factor is regulated by an RNA-protein scaffold complex[J].ProcNatlAcadSciUSA,2011,108(28):11381-11386.
[57]LUO J.Identification of specific long noncoding RNA profiles in chicken with different susceptibility to Marek’s disease[C].in Plant and Animal Genome XXI Conference.2013.Plant and Animal Genome.
[58]HUANG W,LONG N,KHATIB H.Genome-wide identification and initial characterization of bovine long non-coding RNAs from EST data[J].AnimGenet,2012,43(6):674-682.
[59]CABALLERO J,GILBERT I,FOURNIER E,et al.Exploring the function of long non-coding RNA in the development of bovine early embryos[J].ReprodFertilDev,2014,27(1):40-52.
[60]ZHOU Z Y,LI A M,ADEOLA A C,et al.Genome-wide identification of long intergenic noncoding RNA genes and their potential association with domestication in pigs[J].GenomeBiolEvol,2014,6(6):1387-1392.
(编辑郭云雁)
Research Progress of Long Noncoding RNAs in the Regulation of Skeletal Muscle Development
ZHAN Si-yuan,LI Li,WANG Lin-jie,ZHONG Tao,ZHANG Hong-ping*
(KeyLaboratoryofFarmAnimalGeneticResourcesExplorationandInnovationofSichuanProvince,SichuanAgriculturalUniversity,Chengdu611130,China)
Abstract:Long non-coding RNAs(lncRNAs)are a novel class of RNAs,which are longer than 200 nucleotides and have no protein-coding potential.In comparison with short noncoding RNAs which was studied extensively,a variety of long noncoding RNAs account for the majority of the transcripts in mammalian genome and the functions of lncRNAs are little understood.The recent studies have indicated that lncRNAs’ function involved in epigenetic modification,transcriptional regulation,cell differentiation,embryonic development and disease occurrence.In this review,we summarized the classification and molecular mechanisms of lncRNA,and research advances in the regulation of skeletal muscle development and domestic animal.
Key words:long non-coding RNAs;mechanism of action;skeletal muscle development;regulation
doi:10.11843/j.issn.0366-6964.2016.04.001
收稿日期:2015-06-24
基金项目:四川省科技支撑计划(2014NZ0077;2015NZ0112)
作者简介:占思远(1987-),男,湖北红安人,博士生,主要从事动物遗传育种与繁殖研究,E-mail:siyuan_zhan@163.com *通信作者:张红平,博士,教授,主要从事动物遗传育种与繁殖研究,E-mail:zhp@sicau.edu.cn
中图分类号:S827.2
文献标志码:A
文章编号:0366-6964(2016)04-0637-08
