利用表面增强拉曼光谱技术分析温度及pH值对H1N1亚型流感病毒增殖的影响
2016-07-04贾潇潇李芸范文辉孙清岚周铁忠刘文军李晶
贾潇潇,李芸,范文辉,孙清岚,周铁忠,刘文军,李晶
1 中国科学院微生物研究所 病原微生物与免疫学重点实验室,北京 100101 2 中国科学院大学,北京 100049 3 中国科学院微生物研究所 网络信息中心,北京 100101 4 辽宁医学院,辽宁 锦州 121001
利用表面增强拉曼光谱技术分析温度及pH值对H1N1亚型流感病毒增殖的影响
贾潇潇1,2,李芸1,范文辉1,孙清岚3,周铁忠4,刘文军1,2,李晶1
1 中国科学院微生物研究所 病原微生物与免疫学重点实验室,北京100101 2 中国科学院大学,北京100049 3 中国科学院微生物研究所 网络信息中心,北京100101 4 辽宁医学院,辽宁 锦州121001
贾潇潇, 李芸, 范文辉, 等. 利用表面增强拉曼光谱技术分析温度及pH值对H1N1亚型流感病毒增殖的影响. 生物工程学报, 2016, 32(4): 447–456.
Jia XX, Li Y, Fan WH, et al. Effects of temperature and pH on the growth of H1N1 subtype of influenza A virus by surface-enhanced Raman spectroscopy. Chin J Biotech, 2016, 32(4): 447–456.
摘要:表面增强拉曼光谱 (SERS) 是一种基于纳米颗粒的拉曼光谱,可以高灵敏度地检测流感病毒等重要病原微生物,鉴定不同毒株间的差异。为了建立一种快速检测流感病毒SERS的方法,本实验利用SERS技术对流感病毒H1N1亚型不同毒株在不同温度和pH值的条件下进行了病毒毒价强弱的检测,将流感病毒样品与金纳米颗粒混合静置后用拉曼共聚焦显微镜进行激光扫描。结果显示在pH为7.2、温度为37 ℃的条件下3 个H1N1亚型的毒株SERS检测结果显示均出现至少1个大于 (或等于) 3 000的峰值,该状态下病毒毒价最强,最适合病毒生长。另外,细胞生物学方法与SERS技术结果一致,检测中均表现出较好的稳定性和准确性。
关键词:表面增强拉曼光谱,流感病毒,温度,pH值
Received: August 9, 2015; Accepted: September 17, 2015
Supported by: National Basic Research Program of China (973 Program) (No. 2012CB955501), National Key Technologies Research and Development Program of China (No. 2013ZX10004-610), National Natural Science Foundation of China (No. 31100644).
国家重点基础研究发展计划 (973计划) (No. 2012CB955501),“艾滋病和病毒性肝炎等重大传染病防治”科技重大专项 (No.
流感病毒属正粘病毒科,含有单股负链RNA基因组,是在世界范围内流行并引发疾病和死亡的一种病毒。至今正粘病毒科包括:A型流感病毒[1]、B型流感病毒[2]、C型流感病毒、托高土病毒[3]和蜱媒病毒[4]。A型流感病毒(Influenza A virus,IAV) 根据其表面血凝素蛋白(HA) 和神经氨酸酶 (NA) 的变异情况分为不同的亚型,HA分16个亚型,NA分9个亚型,以HxNx方式组合,从H1N1至H15N9,目前共有135种亚型。绝大多数的A型流感病毒都分离自鸟类,历史上易引起人或禽类间大范围传播的主要是H1N1[5]、H3N2[6]等亚型,H5N1、H7N9[7]等亚型的流感病毒致死率较高。然而,目前涉及环境因子 (如温度、pH值等) 与流感病毒粒子毒价相互影响及作用关系的报道较少。
1928年,印度科学家 (Raman C.V.) 发现了由光子的非弹性碰撞产生的一种光散射现象——拉曼散射并因此命名[8]。这种非弹性碰撞的原理是在碰撞过程中光子与分子之间发生不完全碰撞,从而光子与分子之间进行能量的传递,使得光子既改变了运动方向也改变了运动的频率[9]。拉曼光谱被发现后主要应用于物质结构的测定。表面增强拉曼光谱作为拉曼光谱的一种,其主要原理是利用纳米金属颗粒与被测分子间的相互作用来提高图谱的信噪比,增强不同样品之间的峰图差异[10]。近年来研究人员利用SERS技术对一些常见病原菌进行了快速检测鉴定,并对混合菌种进行了区分[11-13],本文旨在构建表面增强拉曼光谱技术检测不同温度及pH值条件下H1N1亚型流感病毒毒价强弱的方法,并初步探究与细胞生物学方法的关联性。
1 材料与方法
1.1病毒株
流感病毒A/WSN/33 (H1N1) (以下简称为WSN)、A/PR/8 (H1N1) (以下简称为PR8)、A/California/04/2009 (以下简称为CA04),由中国科学院微生物研究所分离、鉴定和保藏。
1.2细胞株及鸡胚
犬肾上皮连续细胞系MDCK (Madin-Daby canine kidney cells) 用含10%胎牛血清的DMEM培养,由本实验室传代和保藏。SPF鸡胚 (10日龄) 购自 (北京) 梅里亚动物保健有限公司。
1.3主要试剂
DMEM培养基、胎牛血清、胰酶购自英潍捷基 (上海) 贸易有限公司;青链霉素混合液 (储存浓度107U/mL、终浓度104U/mL)、TPCK-Trypsin(储存浓度2 mg/mL、终浓度2 μg/mL) 购自赛默飞世尔科技 (中国) 有限公司;0.1 mol/L柠檬酸购自国药集团化学试剂有限公司;实验中运用的KlariteTM金纳米基底购自D3技术 (英国)有限责任公司,由国家纳米科学中心合成并惠赠。
1.4病毒样品的收集与纯化
将流感病毒毒株WSN、CA04、PR8分别接种于SPF鸡胚,72 h收集尿囊液以5 000 r/min离心20 min,取上清液;将上清液以105×g (Ti40)离心2 h,将沉淀物用1 mL 0.01 mol/L PBS (pH 7.4) 悬浮,振荡均匀;加至依次由30%、 40%、50%、60%蔗糖制成的密度梯度上,以105×g (SW40) 离心2 h,取沉淀条带;取沉淀条带加10倍量PBS悬浮、振匀。再次以105×g (Ti40) 离心2 h,取沉淀用200 μL 0.01 mol/L PBS (pH 7.4)悬浮、振匀,–80 ℃保存。
1.5拉曼光谱仪检测参数的设定
设置拉曼光谱仪的如下参数。
关键参数为:Spectral acquisition setup。
在Range界面设置如下参数:
Grating scan type: Extended; Confocality: Standard; Spectrum Range: Low 100.00; Centre Raman shit/cm–1; High 2 000.00; Configuration: Laser name 532 nm edge; Grating name 1 800 l/mm。
在Acquisition界面设置如下参数:
Exposure time/s: 20.00; AccumuLations: 1; Objective: 50; Laser power %: 100。
1.6不同毒株SERS稳定性检测
在温度条件为37 ℃,pH条件为7.2的环境下分别培养WSN、CA04、PR8三种病毒并纯化。将纯化后的流感病毒液样品滴加10 μL到载玻片中,向载玻片的样品中滴加5 μL 100 nm金纳米颗粒,静置10 s,拉曼光谱仪检测。实验结果使用Origin8.0收集峰图并进行平滑均一化处理,最后将峰图进行拟合。
1.7细胞感染及病毒滴定检测
取生长良好的细胞,运用10% FBS、1‰ PS DMEM制备细胞悬液,对细胞进行计数,调整细胞浓度为1×105个/mL的悬液,然后加入到96孔细胞培养板中,100 µL/孔,37 ℃、5% CO2培养过夜,使其铺满单层。将待测定病毒进行10倍梯度稀释,选取6–8个梯度进行试验。
96孔板中细胞长满单层后,将原培养液弃掉;在96孔板的第11、12两列每孔各加100 µL含TPCK-Trypsin (终浓度为2µg/mL) 1‰ PS 的DMEM作为阴性对照。病毒稀释液从低浓度开始每个稀释度加8个复孔,每孔100 µL,用10–1至10–10连续梯度稀释的病毒稀释液依次感染96孔板中的细胞。置于37 ℃、5% CO2条件下培养72 h,观察细胞病变孔数并记录,按Reed-Muench氏法[14]计算TCID50。
1.8不同温度条件拉曼技术检测
将在温度条件分别为37 ℃、50 ℃、52.5 ℃、55 ℃、57.5 ℃和60 ℃下培养的病毒进行纯化后,将纯化的流感病毒液样品滴加10 μL到载玻片中,随后向载玻片的样品中滴加5 μL 100 nm金纳米颗粒,静置10 s,拉曼光谱仪检测。实验结果使用Origin8.0收集峰图并进行平滑均一化处理,最后将峰图进行拟合。
1.9不同pH条件拉曼技术检测
将在pH条件分别为4.6、5.0、5.4、5.8、6.2、6.6、7.2下培养的病毒进行纯化后,将纯化的流感病毒液样品滴加10 μL至载玻片中,随后向载玻片的样品中滴加5 μL 100 nm金纳米颗粒,静置10 s,拉曼光谱仪检测。实验结果使用Origin8.0收集峰图并进行平滑均一化处理,最后将峰图进行拟合。
2 结果与分析
2.1流感病毒颗粒形态
为观察流感病毒颗粒的形态,利用电镜将PR8毒株按不同倍数放大,分别得到比例尺为50 nm、100 nm及0.5 μm的流感病毒颗粒形态图 (图1)。电镜结果显示流感病毒颗粒呈球状或丝状,表面不光滑且有突起,可见核心、包膜与基质蛋白区域。


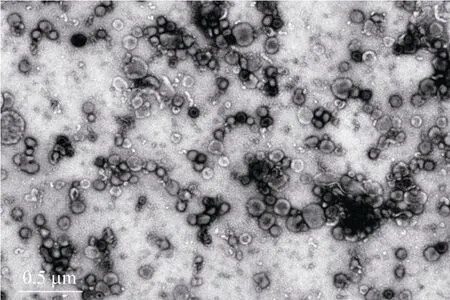
图1 电镜下的流感病毒颗粒形态Fig. 1 Influenza A virus particles under electron microscope.
2.2SERS技术稳定性的检测
为了检测SERS技术的稳定性,将WSN、PR8、CA04流感病毒毒株置于pH值为7.2、温度为37 ℃的条件下培养并制备样品分别进行多次重复实验。横坐标表示拉曼峰位移值,纵坐标表示拉曼信号相对强度,如图2峰图显示,CA04毒株的主要出峰位置在1 000 cm–1处,PR8毒株主要毒株出峰位置在950 cm–1、1 125 cm–1处,WSN毒株的主要出峰位置在950 cm–1、1 125 cm–1处。结果显示峰图重现性良好,表明该方法在此实验中具有良好的稳定性。另外,图1显示在pH值为7.2、温度为37 ℃的条件下3个毒株的SERS检测结果均出现至少1个大于 (或等于) 3 000的峰值。

图2 pH值为7.2、温度为37 ℃的条件下不同毒株SERS稳定性检测Fig. 2 Stability of influenza A virus strains tested by SERS.
图3 H1N1不同亚型毒株感染细胞后病毒滴度检测Fig. 3 Viruses growth ability tests of three influenza A virus strains.
2.3细胞感染及病毒滴度的检测
为了检测在不同环境因子条件下A型流感病毒H1N1亚型不同毒株的病毒滴度变化,我们使用细胞生物学的方法使病毒感染细胞并在相应条件下培养,检测TCID50值。图3中横坐标表示不同环境因子,纵坐标表示不同流感病毒滴度,结果显示在不同pH值条件下,CA04、WSN、PR8三株毒株在pH值为5.0及以下时基本丧失毒价,而随着pH值的上升病毒滴度逐渐升高,在pH值为7.2时复制能力最强。在环境温度改变的情况下,CA04毒株在50 ℃及以上条件下毒价基本丧失,WSN和PR8毒株在温度由37 ℃上升至50 ℃时毒价显著下降,在50 ℃–52.5 ℃时病毒滴度平稳,温度上升至55 ℃及更高后基本丧失毒价,二者下降趋势基本一致。
2.4不同温度条件下SERS检测

图4 不同温度毒株拉曼技术检测Fig. 4 Three influenza A virus strains, CA04 (Black), WSN (Red), and PR8 (Blue), were analyzed under different temperatures using SERS.
为了检测A型流感病毒在不同温度条件下病毒滴度的变化,我们将流感病毒在不同环境下培养后使用SERS技术进行快速检测。图4中横坐标表示拉曼峰位移值,纵坐标表示拉曼信号相对强度,峰图显示在50 ℃–60 ℃范围内温度变化的条件下,CA04毒株的峰图变化不显著,主要出峰点均集中在1 000–1 100 cm–1等位置,峰图的趋势及走向一致。PR8和WSN毒株的峰图在55 ℃–60 ℃范围内与50 ℃–52.5 ℃范围内发生了显著的改变,其中PR8毒株在50 ℃和52.5 ℃时1 000 cm–1位置的峰强度在55 ℃–60 ℃范围内显著变弱,WSN毒株在50 ℃和52.5 ℃时910 cm–1、1 100 cm–1位置的峰强度在55 ℃–60 ℃范围内显著变弱,以上结果显示,SERS技术与细胞生物学检测的结果基本一致。
2.5不同pH值条件下SERS检测
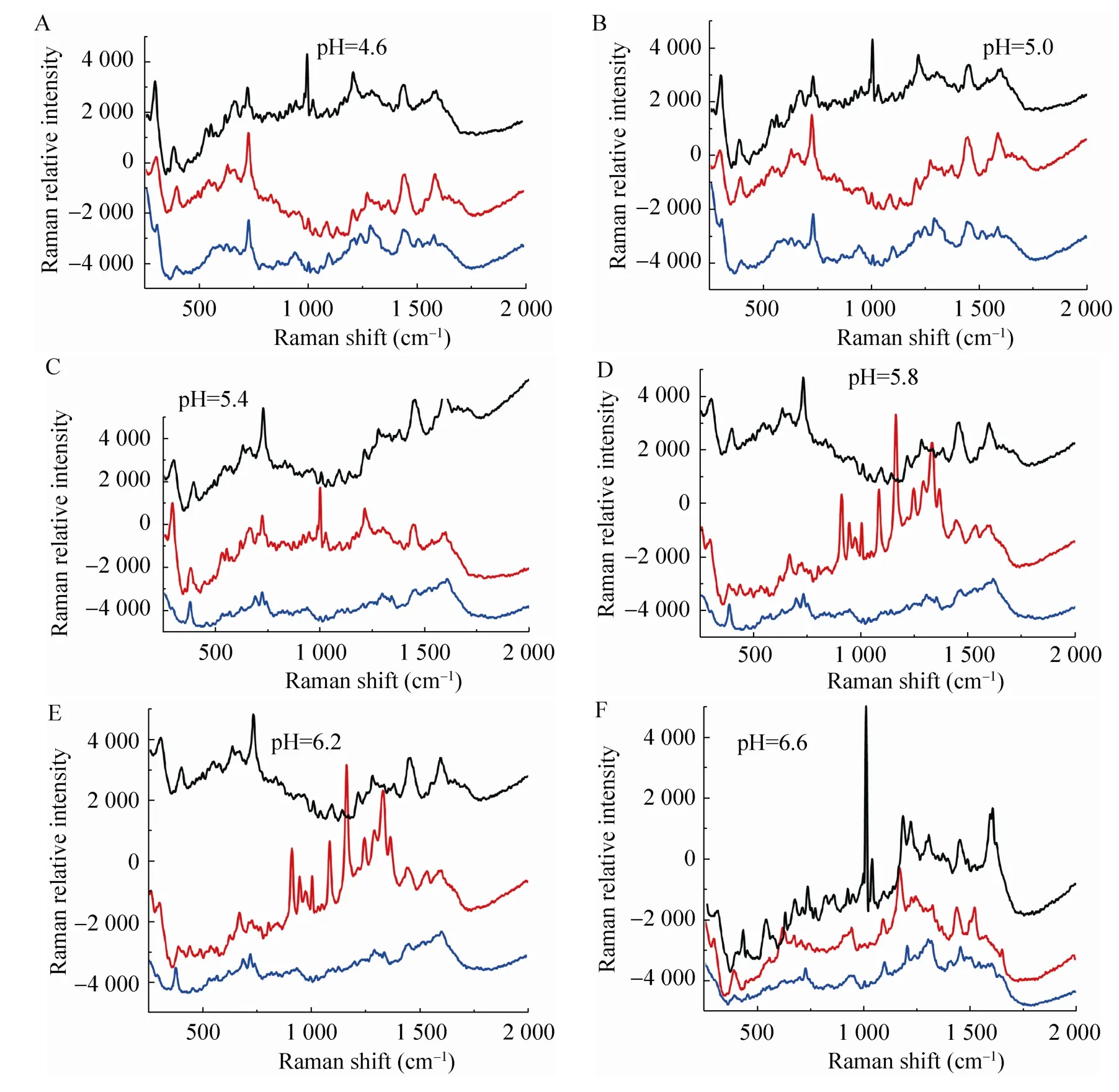
图5 不同pH值毒株拉曼技术检测Fig. 5 Three influenza A virus strains, CA04 (Black), WSN (Red), and PR8 (Blue), were analyzed under different pH using SERS.
为了检测A型流感病毒在不同pH条件下病毒滴度的变化,我们将流感病毒在不同环境下培养后使用SERS技术进行快速检测。图5中横坐标表示拉曼峰位移值,纵坐标表示拉曼信号相对强度,结果显示CA04、WSN、PR8三株毒株在pH为4.6和5.0时峰图基本一致,CA04 和PR8毒株各自的峰图在pH为5.4、5.8、6.2时基本一致,与前两个pH的峰图相比1 000 cm–1左右的峰强度减弱,而WSN毒株的结果显示pH从5.4变化至5.8后峰图发生了显著改变,1 000 cm–1左右出现了多峰现象。在pH为6.2时峰图与5.8时基本一致,在pH上升至6.6后,3株毒株的峰图均发生了显著改变。3株毒株在pH条件改变过程中峰图的变化趋势与细胞实验学检测结果一致。
3 讨论
近年来,随着应用技术的发展,对于病原菌的检测手段也从传统的分离培养、血清学检测发展到了聚合酶链式反应 (Polymerase chain reaction,PCR)、ELISA、Realtime-PCR等方法,表面增强拉曼光谱 (SERS) 的应用很大程度提高了检测的灵敏度并大幅缩短了所需时间,研究人员可以在样品不使用标签标记的情况下在几分钟之内得到准确的检测结果。表面增强拉曼光谱技术是一种高灵敏度和快速的检测方法,目前在微生物领域,我国应用较广的是对食源性大肠杆菌[15]、金黄色葡萄球菌[16]、军团菌[17]、芽孢杆菌[18]等常见致病菌的快速分离鉴定[19]。目前SERS技术在病毒检测中的应用远没有在细菌检测方面成熟和广泛。但有研究结果显示,SERS技术可以用来快速区别和鉴定流感病毒的种类,如Zhao等[20]利用银纳米颗粒作为基底,对流感病毒的3种不同毒株A/HKx31、A/WSN/33和A/PR/8/34进行了SERS的鉴定。在病毒样品体积小于5 μL的情况下拉曼光谱仪1 min内完成了收集,并且根据峰图之间的区别可以清楚地鉴别出不同种毒株,结果发现SERS可以用来鉴别同一病原菌的不同种毒株。
根据目前已知的研究结果,流感病毒M2蛋白是一种离子通道蛋白[21],病毒所处环境的pH值会影响到离子通道蛋白的功能。有研究发现在pH值升高至8.5的情况下M2蛋白中的4个组氨酸会形成一个边缘-面的π堆叠状的结构,阻止了水分子通过氢键形成长链来传导质子。在低pH值的导通状态下,组氨酸和水形成氢键,并发生环-再定向反应[22]。从本实验的结果得出,在不同酸碱度的条件下,病毒表面膜蛋白结构和功能可能会发生改变,从而影响病毒的复制能力。热休克蛋白 (HSP)是一种应激蛋白,广泛存在于真核和原核细胞内[23],一些HSP家族的蛋白,如HSP40、HSP70在抗病毒感染过程中发挥重要作用[24],能够抑制病毒在细胞内的繁殖。多项研究证明,流感病毒的M2、PB1-F2等蛋白会与宿主的热休克蛋白Hsp40、Hsp70发生相互作用[25]。本实验结果表明在温度由37 ℃开始上升后病毒滴度逐渐或迅速下降,并且SERS结果也显示病毒滴度改变的情况下峰图发生了显著变化,这一现象可能是由于在高温侵袭的状态下,宿主体内HSP迅速增加,对病毒复制的抑制增强,导致病毒滴度下降。
SERS峰图中每一位移处的拉曼峰都代表着某一种或一类原子团、化学键等物质。如720 cm–1–800 cm–1可能为C-F键,800 cm–1–950 cm–1可能为C-O-C键,910 cm–1–960 cm–1为羧酸二聚物,990 cm–1–1 100 cm–1可能为芳环,1 010 cm–1–1 095 cm–1可能为Si-O-C或Si-O-Si键,120 cm–1–1 225 cm–1可能为C=S键等[26]。本实验SERS结果中随着温度的升高或pH值的降低毒株的峰图发生显著变化,细胞生物学结果显示毒价显著下降,结合二者的结果推测可能是流感病毒中关键蛋白的构象改变导致病毒复制受到抑制或功能丧失,而具体的机制还需要更进一步分子生物学实验验证。因此,峰图变化显示的外界培养条件的改变对病毒毒价影响的更深入的分子机制探究也将是我们下一步工作的重点。
综上所述,根据本实验中的TCID50结果和SERS结果的对比,发现在细胞生物学结果显示毒价变化时,SERS图谱也相应显示了峰图的变化,表明SERS技术的检测结果是对细胞生物学实验结果的良好佐证。在pH值为7.2、温度为37 ℃的条件下不同毒株的细胞生物学及SERS检测结果说明,该状态下病毒毒价最强,最适合病毒生长,为最适的培养条件。日后,我们将收集不同毒株峰图进行汇总和归纳,建立起具有快速查阅比对功能的拉曼数据库,应用于临床样本的检测。
REFERENCES
[1] Newton HJ, Ang DKY, van Driel IR, et al. Molecular pathogenesis of infections caused by Legionella pneumophila. Clin Microbiol Rev, 2010, 23(2): 274–298.
[2] Fujinaga S, Hara T. Acute kidney injury following plastic bronchitis associated with influenza B virus in a child with nephrotic syndrome. Indian Pediatr, 2015, 52(6): 523–525.
[3] Kosoy OI, Lambert AJ, Hawkinson DJ, et al. Novel thogotovirus associated with febrile illness and death, United States, 2014. Emerg Infect Dis, 2015, 21(5): 760–764.
[4] Bílý T, Palus M, Eyer L, et al. Electron tomography analysis of Tick-Borne encephalitis virus infection in human neurons. Sci Rep, 2015, 5: 10745.
[5] Huang SSH, Lin Z, Banner D, et al. Immunity toward H1N1 influenza hemagglutinin of historical and contemporary strains suggests protection and vaccine failure. Sci Rep, 2013, 3: 1698.
[6] Bedford T, Riley S, Barr IG, et al. Global circulation patterns of seasonal influenza viruses vary with antigenic drift. Nature, 2015, 523(7559): 217–220.
[7] Taft AS, Ozawa M, Fitch A, et al. Identification of mammalian-adapting mutations in the polymerase complex of an avian H5N1 influenza virus. Nat Commun, 2015, 6: 7491.
[8] Li R, Zhou JC, Lu CF. Application of Raman scattering technology in biology. Biotech Bull, 2009(12): 62–64 (in Chinese).李睿, 周金池, 卢存福. 拉曼光谱在生物学领域的应用. 生物技术通报, 2009(12): 62–64.
[9] Zhao YC, Jackson SD. Passively Q-switched fiber laser that uses saturable Raman gain. Opt Lett, 2006, 31(6): 751–753.
[10] Jia XX, Li J, Qin T, et al. Current views on surface enhanced Raman spectroscopy in microbiology. Chin J Biotech, 2015, 31(5): 611–620 (in Chinese).贾潇潇, 李晶, 秦天, 等. 表面增强拉曼光谱技术在微生物鉴定中的应用进展. 生物工程学报, 2015, 31(5): 611–620.
[11] Cheng IF, Chang HC, Chen TY, et al. Rapid (<5 min) identification of pathogen in human blood by electrokinetic concentration and surface-enhanced Raman spectroscopy. Sci Rep, 2013, 3: 2365.
[12] Lin CC, Yang YM, Liao PH, et al. A filter-like AuNPs@MS SERS substrate for Staphylococcus aureus detection. Biosens Bioelectron, 2014, 53: 519–527.
[13] Lin HY, Huang CH, Hsieh WH, et al. On-line SERS detection of single bacterium using novel SERS nanoprobes and a microfluidic dielectrophoresis device. Small, 2014, 10(22): 4700–4710.
[14] Brown WF. Variance estimation in the Reed-Muench fifty per cent end-point determination. Am J Hyg, 1964, 79: 37–46.
[15] Fan C, Hu ZQ, Mustapha A, et al. Rapid detectionof food- and waterborne bacteria using surface-enhanced Raman spectroscopy coupled with silver nanosubstrates. Appl Microbiol Biot, 2011, 92(5): 1053–1061.
[16] Lu XN, Samuelson DR, Xu YH, et al. Detecting and tracking nosocomial methicillin-resistant Staphylococcus aureus using a microfluidic SERS biosensor. Anal Chem, 2013, 85(4): 2320–2327.
[17] Li J, Qin T, Jia XX, et al. Rapid identification of Legionella pathogenicity by surface-enhanced Raman spectroscopy. Biomed Environ Sci, 2015; 28(6): 437–444.
[18] Deng AH, Sun ZP, Zhang GQ, et al. Rapid discrimination of newly isolated Bacillales with industrial applications using Raman spectroscopy. Laser Phys Lett, 2012, 9(9): 636.
[19] Cao YC, Jin RC, Mirkin CA. Nanoparticles with Raman spectroscopic fingerprints for DNA and RNA detection. Science, 2002, 297(5586): 1536–1540.
[20] Zhao YP, Shanmukh S, Liu YJ, et al. Silver nanorod arrays can distinguish virus strains. Phys Rev Lett, 1997, 78: 1667–1670.
[21] Nguyen H, Le L. Steered molecular dynamics approach for promising drugs for influenza A virus targeting M2 channel proteins. Eur Biophys J, 2015, 44(6): 447–455.
[22] Hu FH, Luo WB, Hong M. Mechanisms of proton conduction and gating in influenza M2 proton channels from solid-state NMR. Science, 2010, 330(6003): 505–508.
[23] Finka A, Sharma SK, Goloubinoff P. Multi-layered molecular mechanisms of polypeptide holding, unfolding and disaggregation by HSP70/HSP110 chaperones. Front Mol Biosci, 2015, 2: 29.
[24] Sondermann H, Scheufler C, Schneider C, et al. Structure of a Bag/Hsc70 complex: convergent functional evolution of Hsp70 nucleotide exchange factors. Science, 2001, 291(5508): 1553–1557.
[25] Guan ZH, Liu D, Mi SF, et al. Interaction of Hsp40 with influenza virus M2 protein: implications for PKR signaling pathway. Protein Cell, 2010, 1(10): 944–955.
[26] Tammer MG. Sokrates: infrared and Raman characteristic group frequencies: tables and charts. Colloid Polym Sci, 2004, 283(2): 235.
(本文责编 郝丽芳)
工业生物技术
Effects of temperature and pH on the growth of H1N1 subtype of influenza A virus by surface-enhanced Raman spectroscopy
Xiaoxiao Jia1,2, Yun Li1, Wenhui Fan1, Qinglan Sun3, Tiezhong Zhou4, Wenjun Liu1,2, and Jing Li1
1 Key Laboratory of Pathogenic Microbiology and Immunology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China 2 University of Chinese Academy of Sciences, Beijing 100049, China 3 Network Information Center, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China 4 Liaoning Medical University, Jinzhou 121001, Liaoning, China
Abstract:Surface enhanced Raman spectroscopy technology (SERS), using gold nanoparticles as a base, was developed for rapid and sensitive detection of virus strains. SERS can be used as a rapid and reliable method to distinguish the titers of viral replication. In the present study, we characterized H1N1 subtypes of influenza A virus strains in different conditions of pH or temperatures, while we analyzed data from SERS technology using gold nanoparticles as a base and cell cultures were employed to further confirm the data from virus strains. Origin8.0 was used to collect Raman spectra, smooth and homogenize data, and to contrast spectra. Our results indicated that the peaks of different virus strains in optimal environmental conditionsreached ≥3000. This criterion was verified by subsequent virological method . The present data indicate that the established SERS protocol can be used as a rapid and reliable method to distinguish the replication rate of virus, which can be further used in clinical samples.
Keywords:SERS, influenza A virus, temperature, pH
DOI:10.13345/j.cjb.150362
Corresponding author:Jing Li. Tel/Fax: +86-10-64807503; E-mail: lj418@163.com
