植物活性长末端重复序列反转录转座子研究进展
2016-07-04梁琳琳周明兵
梁琳琳,周明兵
浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300
植物活性长末端重复序列反转录转座子研究进展
梁琳琳,周明兵
浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安311300
梁琳琳, 周明兵. 植物活性长末端重复序列反转录转座子研究进展. 生物工程学报, 2016, 32(4): 409–429.
Liang LL, Zhou MB. Plant active LTR retrotransposons: a review. Chin J Biotech, 2016, 32(4): 409–429.
摘要:长末端重复序列 (Long terminal repeat,LTR) 反转录转座子是真核生物基因组中普遍存在的一类可移动的DNA序列,它们以RNA为媒介,通过“复制粘贴”机制在基因组中不断自我复制。在高等植物中,许多活性的LTR反转录转座子已被详尽研究并应用于分子标记技术、基因标签、插入型突变及基因功能等分析。本文对植物活性LTR反转录转座子进行全面的调查,并对其结构、拷贝数和分布以及转座特性进行系统的归纳,分析了植物活性LTR反转录转座子的gag (种属特异抗原) 和pol (聚合酶) 序列特征,以及LTR序列中顺式调控元件的分布。研究发现自主有活性的LTR反转录转座子必须具备LTR区域以及编码Gag、Pr、Int、Rt 和Rh蛋白的基因区。其中两端LTR区域具有高度同源性且富含顺式调控元件;Rt蛋白必备RVT结构域;Rh蛋白必备RNase_H1_RT结构域。这些结果为后续植物活性LTR反转录转座子的鉴定和功能分析奠定了重要基础。
关键词:长末端重复序列反转录转座子,转座,LTR,gag,pol
Received: June17, 2015; Accepted: August 19, 2015
Supported b y: Natural Science Foundation of Zhejiang (No. LR12C16001), National Natural Science Foundation of China (Nos. 31270645, 31470615).
浙江省自然科学基金 (No. LR12C16001),国家自然科学基金 (Nos. 31270645, 31470615) 资助。
转座子 (Transposable elements,TEs或transposons) 是指在基因组上能从同一条染色体的一个位置转移到另一个位置或者从一条染色体转移到另一条染色体上的一段DNA序列[1-2],最早的转座子是20世纪40年代末Barbara McClintock在玉米中发现的 (Ac/Ds转座子)[3]。
转座子分为DNA转座子和反转录转座子两大类。DNA转座子在DNA介导下,通过“剪切粘贴”的方式进行转座。目前发现DNA转座子有Tc1-Mariner、hAT、Mutator、Merlin、Transib、P、PiggyBac、PIF-Harbinger和CACTA类型。反转录转座子在RNA介导下,通过“复制粘贴”的方式进行转座,根据是否含有长末端重复序列 (LTR) 又可进一步分为含有LTR的长末端重复序列(Long-terminal repeat,LTR) 和不含有LTR的非长末端重复序列(Non-long terminal repeat,non-LTR)[4]。LTR具有Ty3/Gypsy、Ty1/Copia、Bel/Pao、花椰菜花叶病毒(Caulimoviridae)、反转录病毒 (Retroviridae)、反转录转座子大片段 (Large retrotransposons derivatives,LARD) 和微型末端重复反转录转座子 (Terminal-repeat retrotransposons in miniature,TRIM) 七类。根据转座子的长度、LTR的长度和物种分布,植物中的Ty1/Copia可分为Tork、Retrofit、Oryco和Sire 4种类型;Ty3/Gypsy可分为Tat、Athila、CRM、Reina、Del和Galadriel六种类型[5]。LTR反转录转座子有自主转座子和非自主转座子两种。自主转座子结构完整,具有自主转座功能;而非自主转座子结构不完整,所以单独存在时是不能转座的,只有在自主转座子的协助下才能发生转座。
植物LTR反转录转座子长度通常在2−18 kb之间,两端各有一个长度约100−5 000 bp正向重复的长末端重复序列 (LTRs)。LTR末端为反向重复序列,通常为5′-TG…CA-3′。在5′和3′末端两侧通常具有4−6 bp的锚定重复位点(TSR)[6]。反转录转座子LTRs不编码蛋白质,但包含转录的起始信号和终止信号,内部有1−3个开放阅读框 (ORF) 编码转座所需的酶类,结构与反转录病毒十分相似。内部的编码区主要包括2个与转座有关的基因,即gag基因、pol基因。gag基因编码的蛋白质参与反转录转座子RNA的成熟与包装,使反转录转座子的RNA整合到基因组。pol基因是反转录转座子复制和转座所必需的基因,编码蛋白酶 (pr)、整合酶(int)、反转录酶 (rt) 和RNA酶 (rh)[6]。
LTR反转录转座子转座非常复杂。首先在细胞核内转录形成mRNA分子,从5′端LTR的R区起始转录,到3′端LTR的R区终止,转录本末端为poly(A)结构。该mRNA分子既需作为复制所需的模板,又要翻译为复制相关的蛋白质。反转录反应发生在细胞质中,一部分mRNA编码复制所需的蛋白质;另一部分包裹在gag编码的结构蛋白里面,形成病毒样颗粒 (Virus like particles,VLPs),作为反转录的模板,通过rt合成第1条cDNA负链,再以此为模板合成第2条正链。cDNA负链以宿主细胞内对应的tRNA作引物,mRNA5′端LTR下游的PBS位点与tRNA起始甲硫氨酸 (tRNAiMet) 的3′端序列互补形成短的RNA双链,rt利用tRNA3′端自由羟基为引物,合成互补的DNA杂交链。编码的rh水解杂交链上的RNA,释放cDNA负链,以位于3′端LTR上游的PPT作为引物,引导第2 条DNA的合成。最后,核酸内切酶使染色体上靶位点的DNA双链断开,具有反转录转座子完整结构的双链DNA被转移至细胞核内,插入靶位点,完成转座过程[7]。
LTR反转录转座子通常情况下是以静止状态存在的,其大量存在极大地影响植物基因组的稳定性,因此植物在进化过程中逐渐形成了甲基化、重组、突变以及小RNA介导的基因沉默等措施,从而抑制了反转录转座子的转座活性,起到维持基因组稳定性的作用[8-10]。即使有活性的反转录转座子也受到严格调节,还有一些转座子只转录不整合,可能转录后的修饰阻止了其发生转座。转座过程中转录、RNA加工、mRNA的输出、翻译后的修饰、插入核酸、反转录和整合[11]任何一步出错都可能限制反转录转座子的转座活性,因此转座的发生非常少。当植物受到外界条件影响,尤其是逆境时,反转录转座子会被激活并插入到靶基因中,同时,周边基因的表达也会受到影响[12]。本文对植物活性LTR反转录转座子进行全面的调查,并对活性LTR反转录转座子的结构、拷贝数和分布以及转座特性进行系统的归纳,而后进一步分析不同植物活性LTR反转录转座子gag和pol的序列特征及LTR序列的顺式作用元件分布。该研究为后续植物活性LTR反转录转座子的功能和转座特性分析奠定了基础。
1 植物活性LTR反转录转座子
目前在不同的植物中发现了活性LTR反转录转座子,达10余种以上,其中广泛应用的有水稻中的Tos17、RIRE7,烟草中的Tnt 1、Tto1,小麦属中的BARE-1、OARE-1和Ttd1a,玉米中的PREM-2、Zeon-1,拟南芥中的EVD,百脉根中的LORE1,甜橙中的CIRE1和番茄中的TLC1-1。其中Tos17和Zeon-1是非自主活性反转录转座子。各活性LTR反转录转座子的结构信息归纳在表1。
1.1水稻中的活性LTR反转录转座子
目前在水稻基因组中发现了2个活性反转录转座子Tos17、RIRE7,分别属于Ty1-copia类和Ty3-gypsy类。
Tos17是来源于水稻Oryza sativa L.基因组的活性反转录转座子。Tos17长3 986 bp,LTR 长138 bp。编码区的结构为pr-int-rt-rh,缺乏gag,故属于非自主转座子[13]。PBS序列为TGGTATCAGAGC,PPT序列为GAAGGGGGG。末端为5′-TG…CA-3′的反向重复序列。
Tos17在组织培养诱导下,拷贝数增加至5−30个,插入位点偏好于基因富集区域,可识别回文序列ANGTT-TSD-AACNT,在宿主基因组中侧翼为5 bp的重复[N(A/T)(A/T)(A/T)N]。水稻转座子Tos17和甲基化程度有关,在组织培养过程中位于日本晴Nipponbare第7染色体上的1个Tos17拷贝 (Tos17chr.7) 甲基化程度降低,其转座活性增加;而在再生植株的生长过程中,随着Tos17的甲基化程度逐渐增加,转座子的转座活性也降低或失活,证明Tos17的转座是受甲基化控制的。另外,大多数水稻材料基因组中Tos17可以被组织培养激活,但粳稻Moritawase基因组中的Tos17却不能,但是对粳稻种子用去甲基化试剂5-azaC进行处理后,Moritawase中的Tos17也可以被激活[14]。此外,在SDG714RNAi转化株或DNA糖基酶/裂解酶DNG701过表达时,H3K9me2表达降低会诱发低甲基化和Tos17的转座激活[15-16]。

表1 活性LTR 反转录转座子结构Table 1The structure of active LTR retrotransposons
利用Tos17插入到染色体的不同位点[17],产生了Osaba1 和Ostatc突变体,成功地用作基因标签克隆了脱落酸生物合成途径中的Osaba1 和Ostatc基因[18]。另外,该转座子已经应用于鉴定水稻功能基因的研究,获得了很多的插入系,在不同的染色体上鉴定到了大量的突变体,其中发芽率相关表型3 489个、生长率相关表型5 024个、叶色相关表型6 439个、叶形相关表型3 727个、秆形相关表型8 772个、抗性相关表型1 102个、分蘖期相关表型376个、抽穗期相关表型3 137个、花期相关表型719个、花序相关表型2 864个、育性相关表型16 367个、种子相关表型4 192个 (https://tos.nias.affrc. go.jp/);并分离到了OSH15[19]、MSP1[20]、GAMYB[21]、PAIR2[22]和淀粉合成酶[23]基因。其中OSH15在水稻节间发育过程中起作用[19]。
RIRE7[24]是水稻基因组中的另一个活性反转录转座子。RIRE7长7 391 bp,LTR长858 bp。编码区的结构为gag-pr-rt-rh-int。PBS序列为TGGTATCAGAGC,PPT序列为AAGAAG GGGAGGA。末端为5′-TG…CA-3′的反向重复序列。
该转座子偏好插入到染色体近着丝粒区域155 bp的串联重复序列 (Tandem repeat sequence,TrsD) 内。荧光原位杂交技术表明RIRE 7和TrsD都位于染色体的着丝粒区域,属于水稻染色体近着丝粒异染色质的保守组分[24]。
1.2烟草中的活性LTR反转录转座子
目前在烟草基因组发现了2个活性反转录转座子Tnt1、Tto1,均属于Ty1-copia类。
Tnt1[25]是来源于烟草Nicotiana tabacum基因组中的活性反转录转座子。Tnt1长5 334 bp,LTR长610 bp。只有一个ORF,编码1 328个氨基酸,编码区的结构为gag-pr-int-rt-rh。PBS序列为TGGTATCAGAGC,PPT序列为GGAGGGGGAG。末端为5′ -TG…CA- 3′的反向重复序列。
Tnt1在单倍体中拷贝数>100,插入位点偏好于基因富集区域,在宿主基因组中侧翼为5 bp的重复。Tnt1在正常情况下也有转录活性,转座活性具有组织特异性,只在根中表达[26];而烟草叶组织分离的原生质体中 Tnt1高诱导表达[26]。同时,在原生质体诱导的细胞培养再生体系中检测到 Tnt1转座到烟草的nia (Nitrate reductase) 基因内部[25]。在LTR的U3区域含有顺式调控元件控制Tnt1表达[26-27]。
将Tnt1导入拟南芥后发现了转座,之后又相继将其导入番茄和水稻中,均在新的宿主中进行了表达,并且发现宿主的内源转座子不影响异源转座子的转座,说明Tnt1反转录转座子不受植物种类差异的限制,利用该特性Tnt1可作为载体系统应用于植物进化分析[28]。另外,利用Tnt1的插入突变可作为基因标签应用于马铃薯[29]和豆类植物[30]功能基因的研究。
Tto1[31]是烟草基因组中的另一个活跃的反转录转座子。Tto1长5 300 bp,LTR长574 bp。只有一个ORF,编码1 338个氨基酸,编码区的结构为gag-pr-int-rt-rh。PBS序列为TGGTATC AGAGC,PPT序列为GGAAGGGGGAGAG。末端为5′-TG…CA-3′的反向重复序列。
Tto1拷贝数为30[27,32]。在组织培养的细胞内有转录活性,在原生质体细胞内转录增加。Tto1[33]能够通过生物因素 (如Crown rust fungus) 或非生物因素 (如机械作用、水杨酸、茉莉酮酸等) 刺激而激活转座。一个13 bp的重复区域作为顺式调控元件调控Tto1表达[33]。
Ttol在正常情况下具有转录活性[26],且其能在水稻中通过反转录进行自主转座[32],因此可以用作植物转基因的基因转移载体[31]。
1.3小麦属中的活性LTR反转录转座子
在小麦属基因组中发现了3个活性反转录转座子BARE-1、OARE-1和Ttd1a,均属于Ty1-copia类。
BARE-1[34](Barley retroelement 1) 是来源于大麦Hordeum vulgare L. 基因组中的活性反转录转座子。BARE-1长13 271 bp,5′LTR长1 829 bp,3′LTR长4 986 bp。BARE-1只有一个ORF区,依赖于TATA 1和TATA 2盒,有3个不同的转录类别[35]。编码区的结构为gag-print-rt-rh。PBS序列为TGGCATCGTGAGC TAGGTT,PPT序列为AGTGCAAGTGGGAG,末端为5′-TG…CA-3′的反向重复序列。
在单倍体基因组中拷贝数1.4×104[34,36],在宿主基因组中侧翼为4 bp的重复 (5′-GAAC-3′)。BARE-1的插入位点是染色体的远端[37]。BARE-1转座子具有组织特异性,在愈伤组织和叶诱导的原生质体中表达[11];在正常组织中无活性,在脱落酸胁迫下可被激活。
Kalendar等[38]应用反转录转座子位点间扩增多态性 (IRAP) 和反转录转座子微卫星扩增多态性 (REMAP) 标记技术检测大麦属BARE-1的多态性,均获得了所测各个种及大麦品种的DNA印迹,证明野生大麦Hordeam spontaneum种群的基因组多样性是BARE-1家族转座活性表达或缺失的结果。并运用所获得的BARE-1多态性数据,建立了能良好反映大麦属亲缘关系的进化图。
OARE-1[39](Oat retroelement 1) 是来源于燕麦Avena sativa L. 基因组中的活性反转录转座子,是BARE-1亚家族的一个成员。OARE-1长8 665 bp,5′LTR长1 714 bp,3′LTR长1 717 bp。编码区的结构为gag-pr-int-rt-rh。PBS序列为TGGTATCAGAGCTAGATCTAT,PPT序列为AAGTGGGAGA。末端为5′-TG…CA-3′的反向重复序列。
在六倍体的燕麦基因组中具有多拷贝,拷贝数在10 000以上。在宿主基因组中侧翼为5 bp的重复 (5′-GGGAC-3′)[39]。OARE-1表达可通过物理作用 (如机械作用,紫外光) 和化学作用(如茉莉酮酸和水杨酸) 胁迫诱导,模式与基因PAL (Phenylalanin ammonialyase) 相类似;此外,OARE-1 在冠锈病侵染时具有高活性。OARE-1 在非生物诱导因素 (水杨酸、茉莉酮酸)和生物 (如Crown rust fungus) 因素作用下,可诱导叶肉细胞合成植物抗毒素,导致植物的防御反应[40]。
Ttd1a是来源于硬质小麦Triticum durum L.基因组中的活性反转录转座子。Ttd1a长5 275 bp,LTR长304 bp。具有两个int区域,编码区的结构为gag-pr-int-int-rt-rh。PBS序列为TGAGGTTTAGATTGAGGGGGAG。末端为5′-TG…CA-3′的反向重复序列。插入位点位于抗性基因附近[41],转座只发生在胁迫条件生长的植株中,当转座发生在L2层细胞时,幼叶新的插入可传递给后代。在光照和盐胁迫条件下,Ttd1a可以与启动子结合进行转录。在LTR的U3区域Ttd1a启动子含有顺式调控元件,它们的序列与转录因子结合位点高度相似,控制着转座子的转录活性。
1.4玉米中的活性LTR反转录转座子
在玉米基因组发现了2个活性反转录转座子PREM-2、Zeon-1,分别属于Ty1-copia类和Ty3-gypsy类。
PREM (Pollen retroelement maize) 是来源于玉米Zea mays L. 基因组中的活性反转录转座子。PREM-2长9 439 bp,LTR长1 307 bp。具有两个gag区域,编码区的结构为gag-gag-print-rt-rh。PBS序列为TGGTATCAGAGC,PPT序列为AAAAGGGGGAGA。末端为5′-TG…CA-3′的反向重复序列。
PREM-2拷贝数大约30 000[42],在宿主基因组中侧翼为5 bp的重复 (5′-ATTAT-3′)。PREM-2反转录转座子定向插入到PREM-1LTR中,与PREM-1序列相反[43]。其整合到宿主基因组位于附近基因的上游。PREM-2在正常情况下具有转录活性,并具有组织特异性,主要在单核的小孢子中表达[42]。
Zeon-1[44](Zein retrotransposon) 是来源于玉米基因组中的另一个活性反转录转座子。Zeon-1长7 313 bp,5′LTR长649 bp,3′LTR长663 bp,该转座子只含一个gag基因相关的ORF,编码375个氨基酸,故为非自主反转录转座子。Zeon-1转座需要转录酶、核糖核酸酶H和整合酶的参与,存在PBS和PPT序列。PBS序列为TGGCGCCCA,PPT序列为AGGGGCT AC。末端为5′-TG…CA-3′的反向重复序列。
在宿主基因组中侧翼具有5 bp (5′-CCAAT-3′) 的重复。Zeon-1转座子插入到27 kDa的玉米蛋白基因的S等位基因位置上,在自交系A188未成熟的胚乳中检测到转录产物。
1.5拟南芥中的活性LTR反转录转座子
在拟南芥Arabidopsis thaliana基因组发现了活性反转录转座子EVD,属于Ty1-copia类。EVD (Evadé, French for ‘fugitive’) 长5 326 bp,LTR长403 bp。编码区的结构为gag-pr-int-rt-rh。末端为5′-TG…CA-3′的反向重复序列。
在拟南芥Col-0基因组中,EVD仅保留2个插入位点:一个是EVD本身的常染色质位点AT5G17125,另一个是近着丝粒的异染色质位点AT1G34967,被称为Attrapé (ATR)。拟南芥野生型和met1或ddm纯合子之间的杂交获得的表观重组近交系 (epiRILs) 群体中[45-46],EVD重新激活,插入到3个不相关的位点,分别是5号染色体上的LFY基因,4号染色体的BRI1 (Brassinosteriod insensitive 1) 基因和VAR2 (Variegated 2) 基因,且与5′端LTR非甲基化状态有关。EVD通过RDR6合成dsRNA,dsRNA产生siRNA诱导3′gagDNA从头甲基化,3′gag甲基化启动反转录,调节LTR的甲基化导致EVD的表观沉默。当RNA介导的DNA甲基化与H3K9me2都不存在时发生转座[47-48]。该反转录转座子能影响邻近基因的染色质状态,以发育或胁迫应答的方式通过细胞通路促进DNA甲基化和转录水平基因沉默限制基因活性。
由于该转座子具有单一拷贝、内源可逆性和灵活性等特性,因此可用于筛选稳定的和潜在的适应性性状[48]。 1.6百脉根中的活性LTR反转录转座子
LORE1 (Lotus retrotransposon 1)[49]是来源于豆科植物百脉根Lotus japonicus的Ty3-gypsy类活性反转录转座子。LORE1长5 041 bp,LTR 长225 bp。只有一个ORF,编码1 508个氨基酸,编码区的结构为gag-pr-rt-rh-int-chr。PBS序列为TTGGTATCTAGAGCCCTAAGAT T,PPT序列为AAGGGGAGGG。末端为5′-TG…CA-3′的反向重复序列。
LORE1为低拷贝转座子,拷贝数为9−10,插入无特异性,偏好于着丝粒异染色质的基因富集区域。LORE1 在完整植株中活跃,在杂交后代中可检测到新的插入位点;此外,在愈伤组织再生的植株中具有活性,认为可能其在体外组织培养极端环境条件下被激活[49]。
LORE1是插入均一、低拷贝和配子特异性[50]的反转录转座子,可诱导获得突变体[51],利用LORE1插入偏好于基因外显子区域和后代稳定性等特性用作侧翼序列标签,成功筛选到Pir1[52]、Nup133[53]、Castor、Nfr5[54]、Nin[49]以及Nin的两个等位基因共7个基因。其中Nup133、Castor、Nfr5 、Nin以及Nin的两个等位基因都与根瘤菌和根菌共生的信号转导通路有关。
1.7甜橙中的活性LTR反转录转座子
CIRE1[55]是来源于甜橙Citrus sinensis基因组的Ty1-copia类活性反转录转座子。CIRE 1长5 044 bp,LTR长461 bp。一个ORF为4 002 bp,编码区的结构为gag-pr-int-rt-rh。PBS序列为TTGGTATCTAGAGCCCTAAGATTC。末端为5′-TG…CA-3′的反向重复序列。CIRE1在单倍体基因组拷贝数平均为2 200。CIRE1转座子具有组织特异性,正常情况下仅在根中表达。经机械作用和植物激素 (萘乙酸或茉莉酸甲酯) 处理后,在叶中的转录增加,且转录产物具有同源性,认为CIRE1具有潜在活性。CIRE1的5′LTR在异源宿主中可启动报告基因的转录和瞬时表达。
1.8番茄中的活性LTR反转录转座子
TLC1-1[56]是来源于智利番茄Lycopersicon chilense基因组的Ty1-copia类活性反转录转座子。TLC1-1长5 248 bp,5′LTR长578 bp,3′LTR 长562 bp。只有单一的ORF为3 987 bp,编码区的结构为gag-pr-int-rt-rh。末端为5′-TG…CA-3′的反向重复序列。在单倍体基因组中拷贝数为900。该转座子具有组织特异性,只在叶诱导的原生质体[57]和根中表达。在机械损伤、原生质体培养和胁迫条件下,TLC1-1具有转录活性并进行表达。在LTR的U3区域有2个57 bp的串联重复序列 (TSR1、TSR2) 和8 bp的ERE (Ethylene-responsive element,ATTTCAAA) 区域,后者位于启动子区域,为乙烯诱导的顺式调控元件[56]。
2 活性LTR反转录转座子特征分析
为了获得活性LTR反转录转座子的LTR序列和各个蛋白结构域的结构特性,在NCBI上下载上述活性LTR反转录转座子的序列,根据各个LTR反转录转座子结构注释,分别获得各个活性LTR反转录转座子的LTR区域、gag基因区、pr基因区、int基因区、rt基因区和rh基因区。利用这些序列信息分析这些活性LTR反转录转座子的进化关系和结构特征,归纳出植物活性LTR反转录转座子的结构特点。
2.1活性LTR反转录转座子结构和进化分析
根据获得的各个活性LTR反转录转座子结构区域 (LTR区域、gag基因区、pr基因区、int基因区、rt基因区和rh基因区) 的长度 (碱基数),利用EXCEL条形图展示各个LTR反转录转座子的结构 (图1,右)。利用各个自主活性LTR反转录转座子的rt基因序列通过MEGA6软件中Neighbor-Joining方法构建进化树 (重复1 000次,图1,左)。

图1 活性LTR反转录转座子聚类分析图Fig. 1 Active LTR retrotransposons phylogenic analysis. Left: the evolutionary tree of active LTR retrotransposons. Right: the structure of corresponding active LTR retrotransposons. Orange represents LTR region, white represents the interval, purple represents gag gene region, gray represents pr gene region, blue represents int gene region, yellow represents rt gene region, black represents rh gene region. The line length represents the base number of each active LTR retrotransposons.
进化树结果表明,活性LTR反转录转座子主要集中于Tork类,包括有TLC1-1、Tnt1、Tto1、CIRE1、BARE-1和OARE-1,这可能与Tork类反转录转座子在植物基因组分布广泛和数量众多有关[58-59];其次是Retrofit类,包括有EVD、Ttd1a;而PREM-2为Sire类;RIRE7为CRM类。另外,Tos17、LORE1被单独分成一类。据此可知,Tos17、TLC1-1、Tnt1、Tto1、CIRE1、BARE-1、OARE-1、EVD、Ttd1a和PREM-2属于Ty1-copia类反转录转座子,而LORE1、RIRE7属于Ty3-gypsy类反转录转座子。
图中各个活性LTR反转录转座子的长度差异较大,但是结构类似,均含有两个LTR区域、gag基因区、pr基因区、int基因区、rt基因区和rh基因区。其中Ty1-copia类反转录转座子的int基因区位于rt基因区和rh基因区之前,而Ty3-gypsy类反转录转座子的int基因区位于rt基因区和rh基因区之后。各活性LTR反转录转座子的LTR区域差异很大,从138 bp到1 829 bp不等;pr基因区从222 bp到522 bp不等。Ty1-copia类反转录转座子各int基因区、rt基因区和rh基因区差异不大,Ty3-gypsy类反转录转座子各int基因区、rt基因区和rh基因区相差也不大。其中Tos17没有gag基因区,PREM-2具有两个gag基因区,Ttd1a具有两个int基因区。这些结果表明,活性自主反转录转座子必须具备编码Gag、Pr、Int、Rt、Rh蛋白的能力。
2.2活性LTR反转录转座子的结构域分析
为了进一步分析活性LTR反转录转座子gag、pr、int、rt和rh的氨基酸序列特征,利用DNAMAN软件翻译出各个自主活性LTR反转录转座子上述各基因区的氨基酸序列。利用NCBI在线Blast分析各氨基酸序列,将保守结构域利用DNAMAN进行比对,并利用WebLogo在线软件 (http://weblogo.berkeley.edu/logo.cgi)作图,分析活性LTR反转录转座子编码蛋白的特征性结构域 (图2−6)。
2.2.1Gag蛋白结构特征
Gag蛋白具有4个保守结构域 (gag-BOX1、gag-BOX2、gag-BOX3和gag-BOX4)。在Ty1-copia类反转录转座子中,gag-BOX1、 gag-BOX2和gag-BOX3结构域属于UBN2结构域,在Ty3-gypsy类反转录转座子中,属于Retrotrans_gag结构域;gag-BOX4序列是zf-CCHC结构域。UBN2结构域是LTR反转录转座子copia类gag常见的结构域,该家族存在于植物和真菌copia类gag中[60]。Retrotrans_gag结构域不但是反转录转座子gag常见的结构域,也是逆转录病毒Gag蛋白的中央结构域,但其保守性较差,与Ty1转座子的UBN2结构域相类似[61]。zf-CCHC结构域是一个由18个氨基酸残基组成的锌指结构,组成了gag蛋白壳[60-61]。图2比对结果表明gag-BOX1、gag-BOX2、gag-BOX3和gag-BOX4这4个保守结构域是活性LTR反转录转座子Gag蛋白常见结构域。其中gag-BOX1结构域由AXXXWXXL (X为任意氨基酸) 组成,第一个位置的A (丙氨酸) 是保守氨基酸;gag-BOX2结构域有4个L (亮氨酸),绝大部分活性反转录转座子相应位置还含有R(精氨酸)、M (甲硫氨酸)、E (谷氨酸)、G (甘氨酸)、F (苯丙氨酸) 和H (组氨酸);gag-BOX3结构域含有保守氨基酸L (亮氨酸);gag-BOX4结构域由CXXCXXXGHXXXXC组成,C (半胱氨酸)、G (甘氨酸) 和H (组氨酸) 是保守氨基酸。在保守结构域中,有7个位置的氨基酸在活性LTR反转录转座子中都是高度同源的 (第1个结构域中A1,第3个结构域中L8,第4个结构域中C1、C4、G8、H9和C14),暗示这些氨基酸在Gag蛋白参与的反转录转座子RNA的成熟与包装过程中具有关键作用[58,62]。
2.2.2Pr蛋白的结构特征

图2 Gag蛋白保守域分析图Fig. 2 The conserved domains of Gag protein. The height of the amino acid letters represents the frequency of occurrence of the amino acid. Red box represents conserved domain 1, green boxes represents the conserved domains 2, purple boxes represents conserved domains 3, and blue boxes represents conserved domains 4.
Pr蛋白具有3个保守结构域 (pr-BOX1、pr-BOX2和pr-BOX3),都属于RVP结构域。RVP结构域是反转录天冬氨酸蛋白酶,该蛋白酶来源于逆转录病毒、反转录转座子和Badnaviruses(植物双链DNA病毒)[63]。图3比对结果表明pr-BOX1、pr-BOX2和pr-BOX3这3个保守结构域是活性LTR反转录转座子Pr蛋白常见结构域。其中pr-BOX1结构域是WVXDT/SGXXXH结构,相应位置的D (天冬氨酸) 是保守氨基酸;pr-BOX2结构域含有5个G (甘氨酸);pr-BOX3结构域相应位置含有非极性氨基酸包括L (亮氨酸)、P (脯氨酸)、V (缬氨酸) 和I (异亮氨酸),以及极性氨基酸包括D (天冬氨酸)、G (甘氨酸)、Y (酪氨酸) 和S (丝氨酸)。在保守结构域中,pr-BOX1中D4在活性LTR反转录转座子中是高度同源的,暗示天冬氨酸对维持Pr蛋白活性具有重要作用[62]。

图3 Pr蛋白保守域分析图Fig. 3 The conserved domains of Pr protein. The height of the amino acid letters represents the frequency of occurrence of the amino acid. Red box represents conserved domain 1, green boxes represents the conserved domains 2, and purple boxes represents conserved domains 3.
2.2.3Int蛋白结构特征
Int蛋白有4个保守结构域 (int-BOX1、int-BOX2、int-BOX3和int-BOX4)。int-BOX1结构域属于gag_pre-integrs结构域;int-BOX2、int-BOX3和int-BOX4结构域属于rve结构域。gag_pre-integrs结构域是gag预整合结构域,该结构域与反转录转座子插入元件有关,位于整合酶的上游[64]。rve结构域是整合酶核心结构域,整合酶介导反转录转座子拷贝整合到宿主染色体中[64]。图4比对结果表明int-BOX1、int-BOX2、int-BOX3和int-BOX4这4个保守结构域是活性LTR反转录转座子Int蛋白常见结构域。int-BOX1结构域富含L (亮氨酸),相应位置的H (组氨酸) 是保守氨基酸;int-BOX2结构域由CXXCLXGKXXRVXF组成,相应位置的氨基酸C (半胱氨酸) 和K (赖氨酸) 是保守氨基酸;int-BOX3结构域含保守氨基酸D (天冬氨酸)、G (甘氨酸)、S (丝氨酸)、K (赖氨酸) 和F(苯丙氨酸);int-BOX4结构域含有D (天冬氨酸)、G (甘氨酸)、Q (谷氨酰胺)、I (异亮氨酸)、P (脯氨酸)、E (谷氨酸)、N (天冬酰胺) 和R (精氨酸)。在保守结构域中,有19个位置的氨基酸在活性LTR反转录转座子中都是高度同源的(第1个结构域中H6,第2个结构域中C1、C4和K8,第3个结构域中D7、G19、D28、S31、K42和F52,第4个结构域中D14、G32、I33、P43、Q44、G47、E50、N53和R54),暗示这些氨基酸对Int蛋白的整合作用具有重要意义[62]。
2.2.4Rt蛋白结构特征
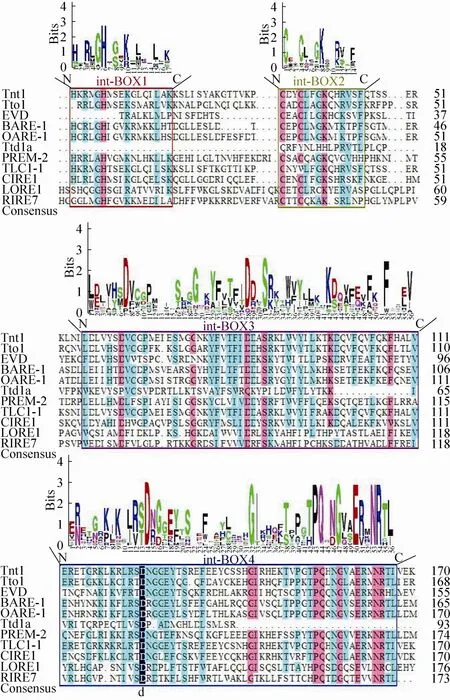
图4 Int蛋白保守域分析图Fig. 4 The conserved domains of Int protein. The height of the amino acid letters represents the frequency of occurrence of the amino acid. Red box represents conserved domain 1, green boxes represents the conserved domains 2, purple boxes represents conserved domains 3, and blue boxes represents conserved domains 4.
Rt蛋白具有4个保守结构域 (rt-BOX1、rt-BOX2、rt-BOX3和rt-BOX4),都属于RVT结构域。RVT结构域是反转录酶 (依赖RNA的DNA聚合酶),存在于反转录转座子、逆转录病毒、Ⅱ类内含子、细菌msDNAs、嗜肝DNA病毒和Caulimoviruses各种移动元件中[65]。图5比对结果表明rt-BOX1、rt-BOX2、rt-BOX3和 rt-BOX4这4个保守结构域是活性LTR反转录转座子Rt蛋白的必备结构域。rt-BOX1结构域由KWVFKXK组成,保守氨基酸是K (赖氨酸);rt-BOX2结构域由DVKXAFLXG组成;rt-BOX3结构域由YGLKQAPRXW组成,相应位置的G(甘氨酸)和L (亮氨酸) 是保守氨基酸;rt-BOX4结构域由LLLYVDDIL组成,富含L (亮氨酸),相应位置的D (天冬氨酸) 是保守氨基酸。在保守结构域中,有19个位置的氨基酸在活性LTR反转录转座子中都是高度同源的 (第1个结构域中K7,第2个结构域中D1、V2、K3、A5、F6、L7和G9,第3个结构域中Y1、G2、L3、Q5、A6、R8和W10,第4个结构域中Y4、D6、D7和L9),暗示这些氨基酸对维持Rt蛋白的反转录活性具有重要作用[65]。
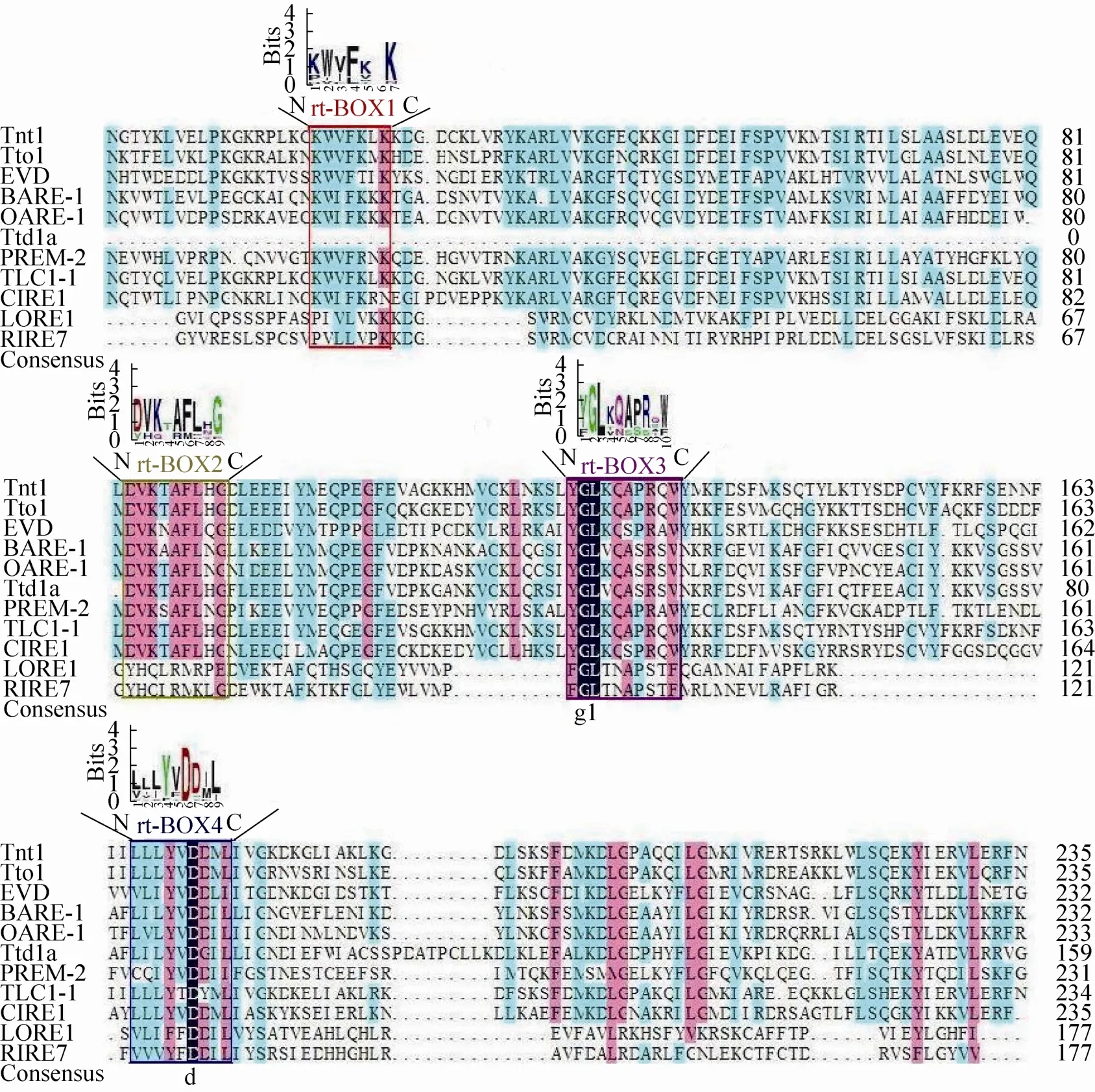
图5 Rt蛋白保守域分析图Fig. 5 The conserved domains of Rt protein. The height of the amino acid letters represents the frequency of occurrence of the amino acid. Red box represents conserved domain 1, green boxes represents the conserved domains 2, purple boxes represents conserved domains 3, and blue boxes represents conserved domains 4.
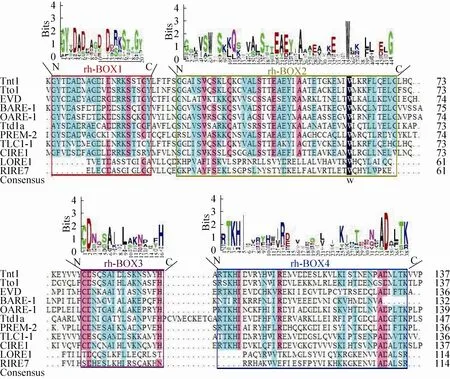
图6 Rh蛋白保守域分析图Fig. 6 The conserved domains of Rh protein. The height of the amino acid letters represents the frequency of occurrence of the amino acid. Red box represents conserved domain 1, green boxes represents the conserved domains 2, purple boxes represents conserved domains 3, and blue boxes represents conserved domains 4.
2.2.5Rh蛋白结构特征
Rh蛋白具有4个保守结构域 (rh-BOX1、rh-BOX2、rh-BOX3和rh-BOX4),都属于RNase_H1_RT结构域。RNase_H1_RT结构域是LTR反转录转座子的核糖核酸酶H1,根据氨基酸序列的相似性和生化特性,核糖核酸酶H (RNA酶H) 分为1型和2型。RNA酶H是非特异性序列内切核酸酶,切割以二价阳离子形式存在于RNA / DNA杂合链中的RNA链。RNA酶H广泛存在于细菌、古细菌和真核生物各种生物体中[66]。在LTR反转录转座子中核糖核酸酶H1可降解原始的RNA模板,产生多聚嘌呤区 (作为正链DNA合成的引物),并从新合成的正负链上最终去除RNA引物[7]。图6比对结果表明rh-BOX1、rh-BOX2、rh-BOX3和rh-BOX4 这4个保守结构域是活性LTR反转录转座子Rh蛋白必备结构域。r h - B O X 1结构域由GYXDADXAGDXDXRKSTXGY组成,含有4个D (天冬氨酸);rh-BOX2结构域富含E (谷氨酸) 和S (丝氨酸);rh-BOX3结构域由CDXXXAIXLXXXXXXH组成,相应位置的 C(半胱氨酸)、D (天冬氨酸) 和H (组氨酸) 为保守氨基酸;rh-BOX4结构域富含T (苏氨酸)。在保守结构域中,有17个位置的氨基酸在活性LTR反转录转座子中都是高度同源的 (第1个结构域中D12、S16和G19,第2个结构域中V6、S8、Q11、S17、E20、A25和W35,第3个结构域中C1、D2和H16,第4个结构域中I5、R13、A33和D34),暗示这些氨基酸对维持Rh蛋白的活性具有关键性作用[67]。
各活性LTR反转录转座子的保守结构域分析结果表明:Gag蛋白相应位置含有保守氨基酸丙氨酸、亮氨酸和半胱氨酸;Pr蛋白相应位置含有保守氨基酸天冬氨酸;Int蛋白相应位置含有保守氨基酸天冬氨酸;Rt蛋白相应位置含有保守氨基酸甘氨酸、亮氨酸和天冬氨酸;Rh蛋白相应位置含有保守氨基酸色氨酸。这些保守氨基酸可能对维持各个蛋白的功能具有重要作用,对催化反转录转座子转座和再插入起了重要的作用[62-63,67]。
2.3活性LTR反转录转座子LTR结构
由于反转录转座子在反转录过程中,LTRs序列是由同一条DNA模板合成,因此在整合进基因组时,它们在DNA序列上是相同的。但是随着时间的推移,植物基因组在进化过程中会发生适当频率的突变使得两端LTRs序列不一致[58,68]。各活性反转录转座子中Tos17、Tnt1、Tto1、EVD、LORE1和RIRE7两端LTR同源性都为100%。而其他转座子由于两端LTR的长度不同,因此同源性降低,但绝大多数转座子(PREM-2、Ttd1a、CIRE1、OARE-1、Zeon-1和 BARE-1) 同源性仍在90%以上,同源性最低的为TLC1-1,两端LTR同源性为86.38%。所有活性LTR反转录转座子两端LTR同源性平均值为97.56%,说明活性LTR转座子的LTR同源性均很高,反映了该类转座子插入事件较近,因而具有较低的积累突变频率和较高的转座活性。随着插入时间的延长,转座子在进化的过程中会逐渐丢失一些序列,导致不同区域不同程度的缺失,直至失去转座活性[36,68]。
转座子转座诱导与启动子有关,有活性的转座子LTR的U3区域都有顺式调控元件。为了系统的解析活性的LTR反转录转座子LTR区域中顺式调控元件的情况,利用PlantCARE在线软件分析了各活性LTR反转录转座子LTR区域的启动子结构 (图7)。
图7结果表明所有的活性LTR反转录转座子的LTR均含有至少一个顺式调控元件(Tos17、Ttd1a和LORE1),最多含有5个调控元件 (OARE-1),其中TCA (Tos17、Tnt1、BARE-1、OARE-1、Ttd1a、PREM-2、CIRE1、RIRE7和Zeon-1) 最丰富 (占69.2%),其次是TC-rich (Tnt1、EVD、OARE-1、PREM-2、CIRE1 和RIRE7) 占46.2%,ABRE、ARE占38.5%。这些结果表明水杨酸和脱落酸诱导可使转座子激活,胁迫或厌氧环境也可使转座子激活。
反转录转座子在植物生长发育的某些阶段或在处于压力等特定条件下可以激活。某些反转录转座子在正常情况下也具有转录活性,如烟草的反转录转座子Tnt1[26]、Tto1[26],玉米的反转录转座子PREM-2[42]。有些反转录转座子的转座活性具有组织特异性,如烟草Tntl、大麦BARE-1、玉米PREM-2、番茄TLC1-1和甜橙CIRE1主要在根部[26,55,57]、叶片[34,55]或单核小孢子中表达[42]。许多转座元件可以被多种生物和非生物的胁迫激活表达。非生物因素包括原生质体分离、组织细胞培养、机械作用、茉莉酮酸、水杨酸等;生物因素包括各种细菌、病原物侵染以及真菌、病原物提取物等[27]。反转录转座子的转座活性被激活是对寄主基因组的一种适应,通过重组获得类似于寄主相关逆境诱导基因的启动子,从而能够在逆境条件下诱导转座[69]。

图7 LTR区域启动子顺式作用元件分布图Fig. 7 The cis-acting elements in LTR sequences. Boxes indicate the different cis-acting elements, lines indicate LTR sequences.
3 总结与展望
反转录转座子激活后会导致基因突变,可用于筛选突变体和鉴定基因,如Tos17[18]、 EVD[48]和LORE1[51];也可用于植物转基因的基因转移载体,如Tto1[31]。反转录转座子广泛分布于植物的基因组中,利用此特性可开发插入标记。该标记的优势在于拷贝数丰富、高度异质性、插入多态性好[70]、插入突变稳定[71],如BARE-1[38]。然而,转座频率相对较低限制了它们在这方面的应用。
经分析,自主有活性的LTR反转录转座子必须具备LTR区域以及编码Gag、Pr、Int、Rt 和Rh蛋白的基因区。其中LTR区域具有高同源性且含有顺式调控元件;Gag蛋白含有UBN2结构域、zf-CCHC结构域 (Ty1-copia转座子) 或Retrotrans_gag结构域 (Ty3-gypsy转座子);Pr蛋白含有RVP结构域,Int蛋白含有gag_pre-integrs结构域和rve结构域;Rt蛋白含有RVT结构域;Rh蛋白含有RNase_H1_RT结构域。
本课题组致力于竹亚科LTR反转录转座子的研究,利用生物信息学方法目前已获得3个全长的LTR反转录转座子 (Ph-LTR1,PHRE1 和PHRE2),均属于Ty3-gypsy类反转录转座子,具备LTR区域以及编码Gag、Pr、Int、Rt和Rh蛋白的基因区。其中Ph-LTR1的LTR同源性为98.79%;Rt蛋白含有RVT结构域;Rh蛋白含有RNase_H1_RT结构域[72]。PHRE1的LTR区域含有顺式调控元件TCA和ABRE;Rt蛋白含有RVT结构域;Rh蛋白含有RNase_H1_RT结构域。而PHRE2的LTR同源性为98.39%,含有顺式调控元件TCA和P-box;Rt蛋白含有RVT结构域;Rh蛋白含有RNase_H1_RT结构域 (部分数据未发表)。根据以上特性,这3个全长的LTR反转录转座子可能是活性反转录转座子。这些结果为后续竹子活性LTR反转录转座子的鉴定和功能分析奠定了重要基础。
REFERENCES
[1] McClintock B. Chromosome organization and genic expression. Cold Spring Harb Symp Quant Biol, 1951, 16: 13–47.
[2] McClintock B. The significance of responses of the genome to challenge. Science, 1984, 226(4676): 792–801.
[3] McClintock B. The origin and behavior of mutable loci in maize. Proc Natl Acad Sci USA, 1950, 36(6): 344–355.
[4] Wicker T, Keller B. Genome-wide comparative analysis of copia retrotransposons in Triticeae, rice, and Arabidopsis reveals conserved ancient evolutionary lineages and distinct dynamics of individual copia families. Genome Res, 2007, 17(7): 1072–1081.
[5] Llorens C, Futami R, Covelli L, et al. The Gypsy Database (GyDB) of mobile genetic elements: release 2.0. Nucleic Acids Res, 2011, 39: D70–D74.
[6] Bowen NJ, McDonald JF. Drosophila euchromatic LTR retrotransposons are much younger than the host species in which they reside. Genome Res, 2001, 11(9): 1527–1540.
[7] Boeke JD. Transposable elements in Saccharomyces cerevisiae//: Berg DE, Howe MM, Eds. Mobile DNA. Washington D.C.: American Society for Microbiology, 1989: 335–374.
[8] Wessler SR. Plant retrotransposons: turned on by stress. Curr Biol, 1996, 6(8): 959–961.
[9] Liu B, Wendel JF. Retrotransposon activation followed by rapid repression in introgressed rice plants. Genome, 2000, 43(5): 874–880.
[10] Okamoto H, Hiroehika H. Silencing of transposable element in plants. Trends Plant Sci, 2001, 6(11): 527–534.
[11] Grandbastien MA. Activation of plant retrotransposons under stress conditions. Trends Plant Sci, 1998, 3(5): 181–187.
[12] Domingues DS, Cruz GMQ, Metcalfe CJ, et al. Analysis of plant LTR-retrotransposons at the fine-scale family level reveals individual molecular patterns. BMC Genomics, 2012, 13: 137.
[13] Sabot F. Tos17 rice element: incomplete but effective. Mob DNA, 2014, 5(1): 10.
[14] Cheng CY, Daigen M, Hirochika H. Epigenetic regulation of the rice retrotransposon Tos17. Mol Genet Genomics, 2006, 276(4): 378–390.
[15] Ding Y, Wang X, Su L, et al. SDG714, a histone H3K9 methyltransferase, is involved in Tos17 DNA methylation and transposition in rice. Plant Cell, 2007, 19(1): 9–22.
[16] La HG, Ding B, Mishra GP, et al. A 5-methylcytosine DNA glycosylase/lyase demethylates the retrotransposon Tos17 and promotes its transposition in rice. Proc Natl Acad Sci USA, 2011, 108(37): 15498–15503.
[17] Yamazaki M, Tsugawa H, Miyao A, et al. The rice retrotransposon Tos17 prefers low-copy-number sequences as integration targets. Mol Genet Genomics, 2001, 265(2): 336–344.
[18] Agrawal GK, Yamazaki M, Kobayashi M, et al. Screening of the rice viviparous mutants generated by endogenous retrotransposon Tos17 insertion. Tagging of a zeaxanthin epoxidase gene and a novel OsTATC gene. Plant Physiol, 2001, 125(3): 1248–1257.
[19] Sato Y, Swntoku N, Miura Y, et al. Loss of function mutations in the rice homeobox gene OSH15 affect the architecture of internodes resulting in dwarf plants. EMBO J, 1999, 18(4): 992–1002.
[20] Nonomura KI, Miyoshi K, Eiguchi M, et al. The MSP1 gene is necessary to restrict the number of cells entering into male and female sporogenesis and to initiate anther wall formation in rice. Plant Cell, 2003, 15(8): 1728–1739.
[21] Kazazian HH. Mobile elements: drivers of genome evolution. Science, 2004, 303(5664): 1626–1632. [22] Nonomura KI, Nakano M, Murata K, et al. An insertional mutation in the rice PAIR2 gene, the ortholog of Arabidopsis ASY1, results in a defect in homologous chromosome pairing during meiosis. Mol Genet Genomics, 2004, 271(2): 121–129.
[23] Fujita N, Yoshida M, Asakura N, et al. Function and characterization of starch synthase I using mutants in rice. Plant Physiol, 2006, 140(3): 1070–1084.
[24] Kumekawa N, Ohmido N, Fukui K, et al. A new gypsy-type retrotransposon, RIRE7: preferential insertion into the tandem repeat sequence TrsD in pericentromeric heterochromatin regions of rice chromosomes. Mol Genet Genomics, 2001, 265(3): 480–488.
[25] Grandbastien MA, Spielmann A, Caboche M. Tnt1, a mobile retroviral-like transposable element of tobacco isolated by plant cell genetics. Nature, 1989, 337(6205): 376–380.
[26] Pouteau S, Huttner E, Grandbastien MA, et al. Specific expression of the tobacco Tnt1 retrotransposon in protoplasts. EMBO J, 1991, 10(7): 1911–1918.
[27] Grandbastien MA, Lucas H, Morel JB, et al. The expression of the tobacco Tnt1 retrotransposon is linked to the plant defense responses. Genetica, 1997, 100(1/3): 241–252.
[28] Hou Y, Rajagopol J, Irwin PA, et al. Retrotransposon vectors for gene delivery in plants. Mob DNA, 2010, 1(1): 19.
[29] Duangpan S, Zhang WL, Wu YF, et al. Insertional mutagenesis using Tnt1 retrotransposon in potato. Plant Physiol, 2013, 163(1): 21–29.
[30] Cui YY, Barampuram S, Stacey MG, et al. Tnt1 retrotransposon mutagenesis: a tool for soybean functional genomics. Plant Physiol, 2013, 161(1): 36–47.
[31] Hirochika H, Otsuki H, Yoshikawa M, et al. Autonomous transposition of the tobacco retrotransposon Ttol in rice. Plant Cell, 1996, 8(4): 725–734.
[32] Hirochika H. Activation of tobacco retrotransposons during tissue culture. EMBO J, 1993, 12(6): 2521–2528.
[33] Takeda S, Sugimoto K, Otsuki H, et al. A 13-bp cis-regulatory element in the LTR promoter of the tobacco retrotransposon Tto1 is involved in responsiveness to tissue culture, wounding, methyl jasmonate and fungal eliecitors. Plant J, 1999, 18(4): 383–393.
[34] Manninen I, Schulman AH. BARE-l, a copia-like retroelement in barley (Hordeurn vulgare L.). Plant Mol Biol, 1993, 22(5): 829–846.
[35] Chang W, Jääskeläinen M, Li SP, et al. BARE retrotransposons are translated and replicated via distinct RNA pools. PLoS ONE, 2013, 8(8): e72270.
[36] Kalendar R, Tanskanen J, Immonen S, et al. Genome evolution of wild barley (Hordeum spontaneum) by BARE-1 retrotransposon dynamics in response to sharp microclimatic divergence. Proc Natl Acad Sci USA, 2000, 97(12): 6603–6607.
[37] Suoniemi A, Anamthawat-Jónsson K, Ama T, et al. Retrotransposon BARE-1 is a major, dispersed component of the barley (Hordeurn vulgare L.) genome. Plant Mol Biol, 1996, 30(6): 1321–1329.
[38] Kalendar R, Grob T, Regina M, et al. IRAP and REMAP: two new retrotransposon-based DNA fingerprinting techniques. Theor Appl Genet, 1999, 98(5): 704–711.
[39] Linares C, Serna A, Fominaya A. Chromosomal organization of a sequence related to LTR-like elements of Ty1-copia retrotransposons in Avena species. Genome, 1999, 42(4): 706–713.
[40] Kimura Y, Shimada S, Sogo R, et al. OARE-1, a Tyl-copia retrotransposon in oat activated by abiotic and biotic stresses. Plant Cell Physiol, 2001, 42(12): 1345–1354.
[41] Woodrow P, Pontecorvo G, Fantaccione S, et al. Polymorphism of a new Ty1-copia retrotransposon in durum wheat under salt and light stresses. Theor Appl Genet, 2010, 121(2): 311–322.
[42] Turcich MP, Bokhari-Riza A, Hamilton DA, et al. PREM-2, a copia-type retroelement in maize is expressed preferentially in early microspores. Sex Plant Reprod, 1996, 9(2): 65–74.
[43] Turcich MP, Mascarenhas JP. PREM-1, a putative maize retroelement has LTR (long terminal repeat) sequences that are preferentially transcribed in pollen. Sex Plant Reprod, 1994, 7(1): 2–11.
[44] Hu WM, Das OP, Messing J. Zeon-1, a member of a new maize retrotransposon family. Mol Genet Genomics, 1995, 248(4): 471–480.
[45] Reinders J, Wulff BBH, Mirouze M, et al. Compromised stability of DNA methylation and transposon immobilization in mosaic Arabidopsis epigenomes. Gene Dev, 2009, 23(8): 939–950.
[46] Johannes F, Porcher E, Teixeira FK, et al. Assessing the impact of transgenerational epigenetic variation on complex traits. PLoS Genet, 2009, 5(6): e1000530.
[47] Mirouze M, Reinders J, Bucheret E, et al. Selective epigenetic control of retrotransposition in Arabidopsis. Nature, 2009, 461(7262): 427–430.
[48] Marí-Ordóñez A, Marchais A, Etcheverry M, et al. Reconstructing de novo silencing of an active plant retrotransposon. Nat Genet, 2013, 45(9): 1029–1039.
[49] Madsen LH, Fukai E, Radutoiu S, et al. LORE1, an active low-copy-number TY3-gypsy retrotransposon family in the model legume Lotus japonicus. Plant J, 2005, 44(3): 372–381.
[50] Fukai E, Umehara Y, Sato S, et al. Derepression of the plant chromovirus LORE1 induces germline transposition in regenerated plants. PLoS Genet, 2010, 6(3): e1000868.
[51] Urbański DF, Małolepszy A, Stougaard J, et al. Genome-wide LORE1 retrotransposon mutagenesis and high-throughput insertion detection in Lotus japonicus. Plant J, 2012, 69(4): 731–741.
[52] Yano K, Shibata S, Chen WL, et al. CERBERUS, a novel U-box protein containing WD-40 repeats, is required for formation of the infection thread and nodule development in the legume-Rhizobium symbiosis. Plant J, 2009, 60(1): 168–180.
[53] Kanamori N, Madsen LH, Radutoiu S, et al. A nucleoporin is required for induction of Ca2+spiking in legume nodule development and essential for rhizobial and fungal symbiosis. Proc Natl Acad Sci USA, 2006, 103(2): 359–364.
[54] Fukai E, Soyano T, Umehara Y, et al. Establishment of a Lotus japonicus gene tagging population using the exon-targeting endogenous retrotransposon LORE1. Plant J, 2012, 69(4): 720–730.
[55] Rico-Cabanas L, Martínez-Izquierdo JA. CIRE1, a novel transcriptionally active Ty1-copia retrotransposon from Citrus sinensis. Mol Genet Genomics, 2007, 277(4): 365–377.
[56] Tapia G, Verdugo I, Yañez M, et al. Involvement of ethylene in stress-induced expression of the TLC1.1 retrotransposon from Lycopersicon chilense Dun. Plant Physiol, 2005, 138(4): 2075–2086.
[57] Yañez M, Verdugo I, Rodríguez M, et al. Highly heterogeneous families of Ty1/copia retrotransposons in the Lycopersicon chilense genome. Gene, 1998, 222(2): 223–228.
[58] Kumar A, Bennetzen JL. Plant retrotransposons. Annu Rev Genet, 1999, 33(1): 479–532.
[59] Llorens C, Muñoz-Pomer A, Bernad L, et al. Network dynamics of eukaryotic LTR retroelements beyond phylogenetic trees. Biol Direct, 2009, 4(1): 41.
[60] Chaparro C, Gayraud G, de Souza RF, et al. Terminal-repeat retrotransposons with GAG domain in plant genomes: a new testimony on the complex world of transposable elements. Genome Biol Evol, 2015, 7(2): 493–504.
[61] Nakayashiki H, Matsuo H, Chuma I, et al. Pyret, a Ty3/Gypsy retrotransposon in Magnaporthe grisea contains an extra domain between the nucleocapsid and protease domains. Nucleic Acids Res, 2001, 29(20): 4106–4113.
[62] Llorens C, Fares MA, Moya A. Relationships of gag-pol diversity between Ty3/Gypsy and Retroviridae LTR retroelements and the three kings hypothesis. BMC Evol Biol, 2008, 8: 276.
[63] Llorens C, Futami R, Renaud G, et al. Bioinformatic flowchart and database to investigate the origins and diversity of Clan AA peptidases. Biol Direct, 2009, 4(1): 3.
[64] Dyda F, Hickman AB, Jenkins TM, et al. Crystal structure of the catalytic domain of HIV-1 integrase: similarity to other polynucleotidyl transferases. Science, 1994, 266(5193): 1981–1986.
[65] Xiong Y, Eickbush TH. Origin and evolution of retroelements based upon their reverse transcriptase sequences. EMBO J, 1990, 9(10): 3353–3362.
[66] Cerritelli SM, Crouch RJ. Ribonuclease H: the enzymes in eukaryotes. FEBS J, 2009, 276(6): 1494–1505.
[67] Malik HS, Eickbush TH. Phylogenetic analysis of ribonuclease H domains suggests a late, chimeric origin of LTR retrotransposable elements and retroviruses. Genome Res, 2001, 11(7): 1187–1197.
[68] Vitte C, Panaud O, Quesneville H. LTR retransposons in rice (Oryza sativa L.): recent burst amplifications followed by rapid DNA loss. BMC Genomics, 2007, 8: 218.
[69] Flavell AJ, Dunbar E, Anderson R, et al. Ty1-copia group retrotransposons are ubiquitous and heterogeneous in higher plants. Nucleic Acids Res, 1992, 20(14): 3639–3644.
[70] Zhao HY, Zhao FK. Plants retrotransposon molecular markers and application. Bull Agric Sci Technol, 2009, (4): 95–97 (in Chinese).赵海艳, 赵福宽. 植物反转录转座子分子标记及其应用. 农业科技通讯, 2009, (4): 95–97.
[71] Lei JL, Xu ZH. Application of transposons in the plant functional genomics. Acta Agric Zhejiang, 2002, 14(5): 291–296 (in Chinese).雷娟利, 徐志豪. 转座子在植物功能基因组学中的应用. 浙江农业学报, 2002, 14(5): 291–296.
[72] Zhou M, Tang DQ, Zhou MB. Cloning, characterization and phylogenetic analysis of a typical long terminal repeat retrotransposon in Phyllostachys heterocycla cv. pubescens. J Bamb Res, 2014, 33(3): 1–10, 21 (in Chinese).周敏, 汤定钦, 周明兵. 一个毛竹典型LTR转座子的克隆、鉴定及进化分析. 竹子研究汇刊, 2014, 33(3): 1–10, 21.
(本文责编陈宏宇)
Plant active LTR retrotransposons: a review
Linlin Liang, and Mingbing Zhou
The Nurturing Station for the State Key Laboratory of Subtropical Sil-viculture, Zhejiang A & F University, Lin’an 311300, Zhejiang, China
Abstract:Long terminal repeat (LTR) retrotransposons are mobile DNA sequences that ubiquitously exist in eukaryotic genomes. They replicate themselves in the genome by copy-paste mechanism with RNA as medium. In higher plants, manyactive LTR retrotransposons have been applied to analyze molecular marker technology, genetic tagging, insertion mutation and gene function. Here, we systematically review the characteristics of plant active LTR retrotransposons, including their structures, copy numbers and distributions. We further analyzed the gag (group-specific antigen) and pol (polymerase) sequence features of different plants active LTR retrotransposons and the distribution patterns of the cis-acting elements in LTR regions. The results show that autonomous active LTR retrotransposons must contain LTR regions and code Gag, Pr, Int, Rt, Rh proteins. Both LTR regions are highly homologous with each other and contain many cis-regulatory elements; RVT and RNase_H1_RT domain are essential for Rt and Rh protein respectively. These results provide the basis for subsequent identification of plant active LTR retrotransposons and their functional analysis.
Keywords:LTR retrotransposons, transposition, LTR, gag, pol
DOI:10.13345/j.cjb.150279
Corresponding author:Mingbing Zhou. Tel: +86-571-63731263; E-mail: zmbin@163.com
